牛源无乳链球菌对喹诺酮耐药的基因特征
2023-05-13孟肖潇吴建勇洪都孜波拉提李建军古努尔吐尔逊努尔拜合提努尔旦杨学云
孟肖潇, 吴建勇, 洪都孜·波拉提, 李建军, 古努尔·吐尔逊, 努尔拜合提·努尔旦, 杨学云
(新疆畜牧科学院兽医研究所,新疆 乌鲁木齐 830010)
乳腺炎是奶牛常见多发病,也是影响奶牛产奶性能的重要病因[1],无乳链球菌(Streptococcusagalactiae)是引起奶牛乳腺炎的重要机会性致病菌,在世界各地广泛流行[2].无乳链球菌传播易受卫生条件和环境因素影响,在20世纪20年代,90%的奶牛乳腺炎由无乳链球菌引起[3].随着饲养条件的改善和抗菌药物的使用,当前北美地区无乳链球菌性奶牛乳腺炎所占比例已不足5%[2];而在我国,乳腺炎奶样中无乳链球菌的检出率仍高达14%~20%[4-6],在奶牛场大罐奶中的检出率甚至超过90%[7].据统计,链球菌性奶牛乳腺炎可导致产奶量下降3.7%~6.6%[8],给我国奶牛场带来较为严重的经济损失.
目前国内没有无乳链球菌性奶牛乳腺炎疫苗,兽医临床上一般采用抗菌药物对其进行治疗,其中,喹诺酮类药物是兽医临床上常用的治疗奶牛乳腺炎的抗菌药物[9],这促使无乳链球菌在喹诺酮类药物选择压力下获得耐该类药物的能力.早在2003年日本就首次报道了喹诺酮耐药无乳链球菌[10],此后无乳链球菌对喹诺酮类药物的耐药率呈现逐年上升的趋势[11],一定程度影响了奶牛乳腺炎的临床治疗效果.因此对无乳链球菌进行药物敏感性检测是保障临床乳腺炎治疗效果的重要手段.
研究表明,喹诺酮耐药无乳链球菌主要通过DNA解旋酶和DNA拓扑异构酶Ⅳ基因喹诺酮耐药决定区(quinolone resistance-determining region, QRDR)突变、携带耐药基因质粒的横向转移以及非特异的多重药物外排泵等耐药机制实现[12].其中,QRDR基因突变所引起的喹诺酮耐药性较为常见.喹诺酮类抗菌药物通过结合DNA拓扑异构酶Ⅱ(DNA解旋酶、DNA拓扑异构酶Ⅳ),干扰细菌基因组DNA复制进而抑制细菌增殖,以达到控制细菌感染的目的.DNA解旋酶由2个GyrA和2个GyrB亚基组成,DNA拓扑异构酶Ⅳ由2个ParC和2个ParE亚基组成,4种亚基中的QRDR可发生基因突变,不同突变对喹诺酮耐药性的影响不同.目前发现喹诺酮耐药人源无乳链球菌中GyrA的第81位氨基酸和ParC的第79位氨基酸发生突变最为常见[13-14],而牛源无乳链球菌耐药机制的相关报道较少.据此,本研究对本实验室保存的110株奶牛隐性乳腺炎无乳链球菌进行喹诺酮类药物敏感性试验及QRDR基因检测,调查了解牛源无乳链球菌对喹诺酮耐药的基因特征,旨在为无乳链球菌性奶牛乳腺炎的临床治疗提供参考资料.
1 材料与方法
1.1 材料
1.1.1 菌株来源 110株无乳链球菌由本实验室保存,于2014—2017年分离自中国10省区21个规模化奶牛养殖场的隐性乳腺炎奶样[4],其中,新疆49株、内蒙古18株、四川11株、山东8株、辽宁8株、河南4株、浙江4株、陕西4株、山西3株、海南1株.质控菌株为无乳链球菌ATCC27956.
1.1.2 主要试剂 细菌基因组DNA提取试剂盒、DNA Marker、2×TaqPlus PCR MasterMix购自北京天根公司;左氧氟沙星、诺氟沙星、环丙沙星购自北京索莱宝科技有限公司;高分辨率琼脂糖-1000购自Life Technologies公司;CAMHB培养基购自青岛海博公司;常规试剂均为国产分析纯.
1.2 药敏试验
将试验菌株和质控菌株分别划线接种于改良的格拉纳达固体培养基上培养36~48 h[15],挑取单菌落接种于含50 mL·L-1裂解马血的CAMHB液体培养基中,采用美国临床和实验室标准协会(CLSI)推荐的微量肉汤稀释法[16]检测左氧氟沙星、诺氟沙星和环丙沙星对110株无乳链球菌的最小抑菌浓度(minimum inhibitory concentration, MIC).在该检测标准中,无乳链球菌对左氧氟沙星的MIC≤2 μg·mL-1为敏感菌株,2 μg·mL-1
1.3 QRDR基因的检测
1.3.1 基因组DNA 的提取 将无乳链球菌接种于改良的格拉纳达固体培养基上培养36~48 h,挑选单菌落接种至5 mL TSB液体培养基中,于37 ℃培养24 h后采用细菌基因组DNA提取试剂盒提取无乳链球菌基因组DNA.
1.3.2 QRDR基因的PCR扩增及测序 参考文献[13]的方法设计无乳链球菌gyrA、gyrB、parC和parE基因的特异性引物(表1),委托生工生物工程(上海)股份有限公司合成.PCR扩增产物经1.5%琼脂糖凝胶电泳分析并交由生工生物工程(上海)股份有限公司测序.测序后与GenBank数据库上的NC004116序列进行比对以确定其突变位点.
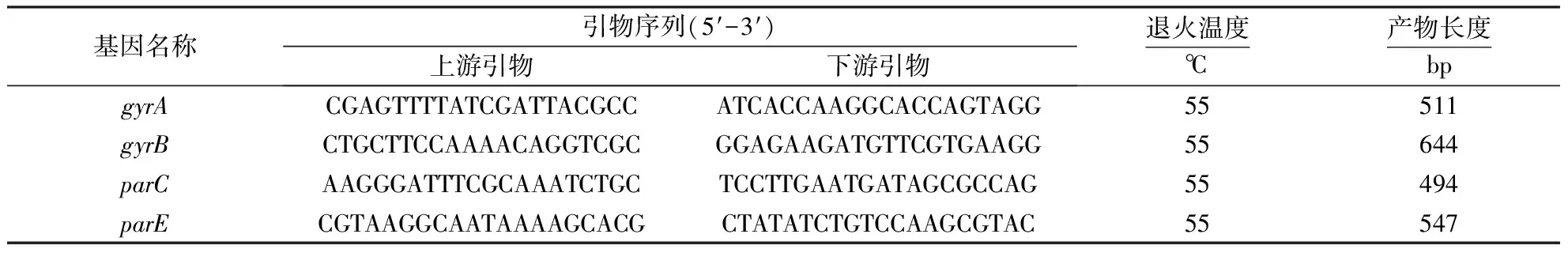
表1 QRDR基因PCR引物序列
2 结果与分析
2.1 无乳链球菌药敏试验结果
左氧氟沙星、诺氟沙星和环丙沙星对110株奶牛隐性乳腺炎无乳链球菌的MIC50分别为16、32、32 μg·mL-1,MIC90分别为16、32、64 μg·mL-1,耐药率分别为85.5%、98.2%、94.5%(表2).
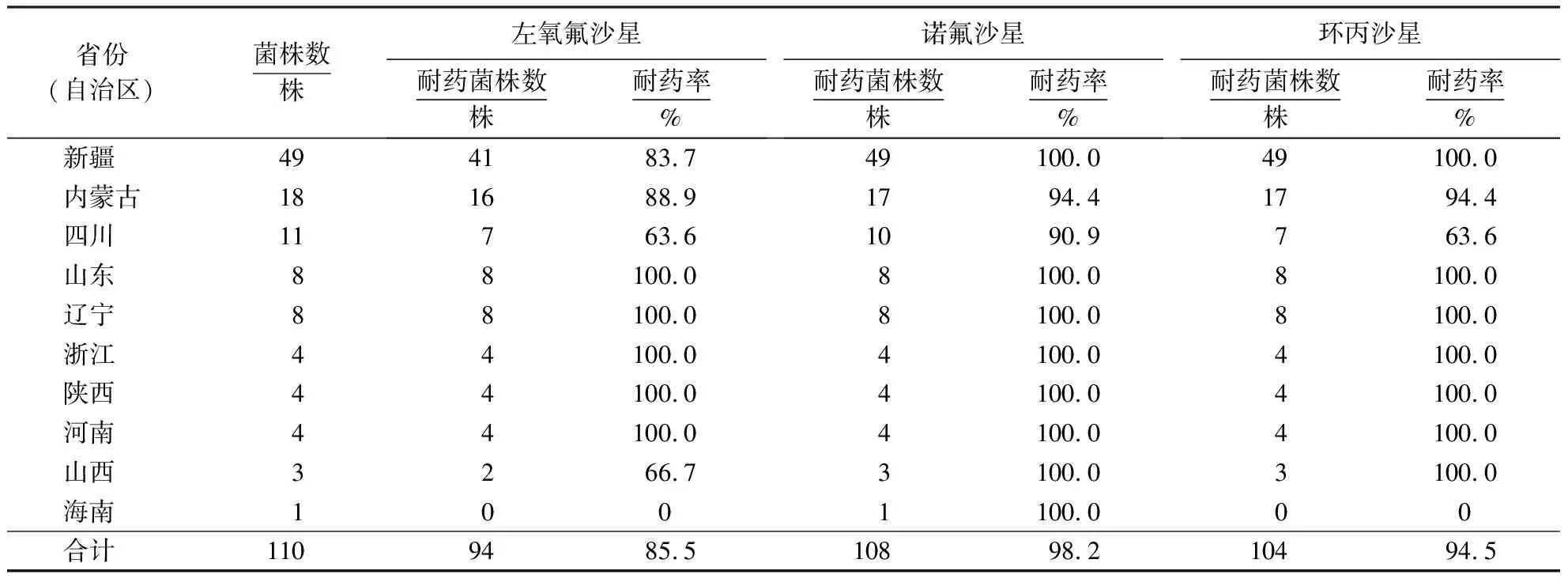
表2 不同省份(自治区)喹诺酮耐药性无乳链球菌的检出率
2.2 QRDR基因检测情况
110株奶牛隐性乳腺炎无乳链球菌均成功扩增出gyrA、gyrB、parC和parE基因,测序后与GenBank数据库上的NC004116序列进行氨基酸序列比对,存在GyrA(Ser81Leu)、GyrA(Glu85Lys)、GyrB(Val498Ala)、ParC(Asp83Tyr)、ParC(Ser79Phe)、ParE(Asp437Asn)、ParE(Ile503Val)7种突变.所有供试菌株和参考菌株2603V/R(GenBank序列号:NC004116)对比均存在GyrB(Val498Ala)和ParE(Ile503Val)突变,表明该突变与喹诺酮耐药性可能无关,在后续统计分析中不予考虑.通过统计GyrA、ParC和ParE的突变类型,在110株菌株中,89.1%(98/110)菌株发生QRDR基因突变,共存在6种突变类型(表3).其中,GyrA(Ser81Leu)+ParE(Asp437Asn)为最主要的突变类型,占比72.7%.ParC的Asp83Tyr和Ser79Phe突变类型对左氧氟沙星敏感,但对诺氟沙星和环丙沙星耐药.GyrA(Ser81Leu)、GyrA(Ser81Leu)+ParE(Asp437Asn)、GyrA(Ser81Leu)+ParC(Ser79Phe)、GyrA(Glu85Lys)+ParC(Ser79Phe)4种突变类型对3种喹诺酮类药物均耐药.
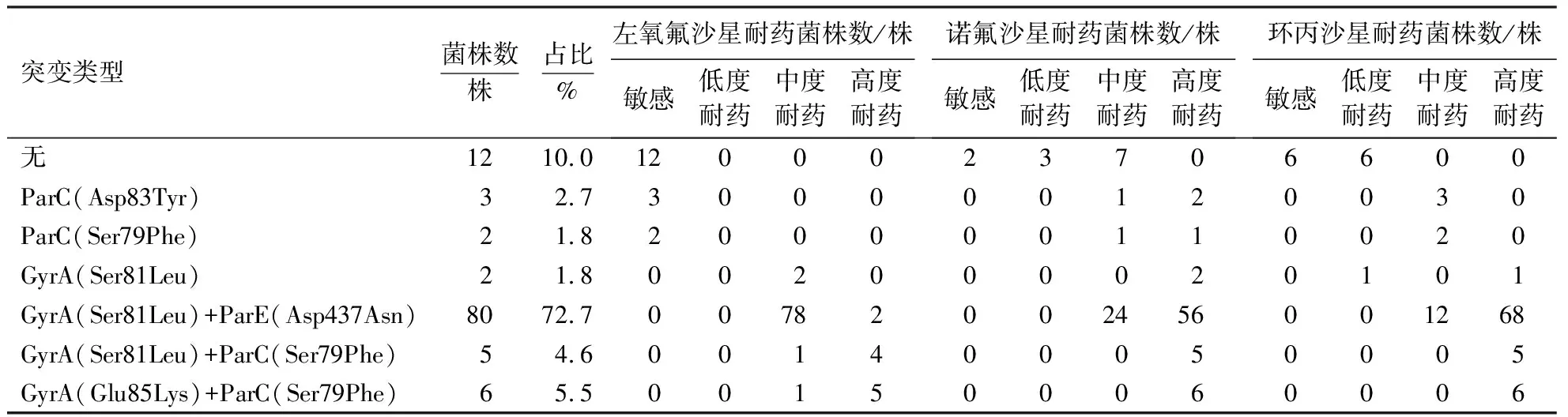
表3 菌株基因突变类型与喹诺酮类药物耐药性统计1)
2.3 菌株基因突变类型对3种喹诺酮类药物MIC的影响
分析菌株基因不同突变类型对左氧氟沙星、诺氟沙星和环丙沙星3种药物MIC的影响.结果(图1-3)显示,对左氧氟沙星耐药的菌株对诺氟沙星和环丙沙星也耐药,且诺氟沙星和环丙沙星的MIC约为左氧氟沙星的2倍.耐药菌株基因最主要的突变类型是GyrA(Ser81Leu)+ParE(Asp437Asn),该突变类型对喹诺酮类药物呈现中高度耐药.根据CLSI推荐的判定标准[16],菌株基因ParC的Asp83Tyr和Ser79Phe突变类型对左氧氟沙星敏感,该突变类型的菌株对左氧氟沙星的MIC略高于无突变菌株(图1),且ParC的这两种突变类型对诺氟沙星和环丙沙星耐药(图2-3).表明ParC的Asp83Tyr或Ser79Phe突变可能会导致无乳链球菌对左氧氟沙星的敏感性下降.当菌株基因存在ParC的Ser79Phe突变与GyrA的Ser81Leu或Glu85Lys突变联合时则表现出高度耐药.
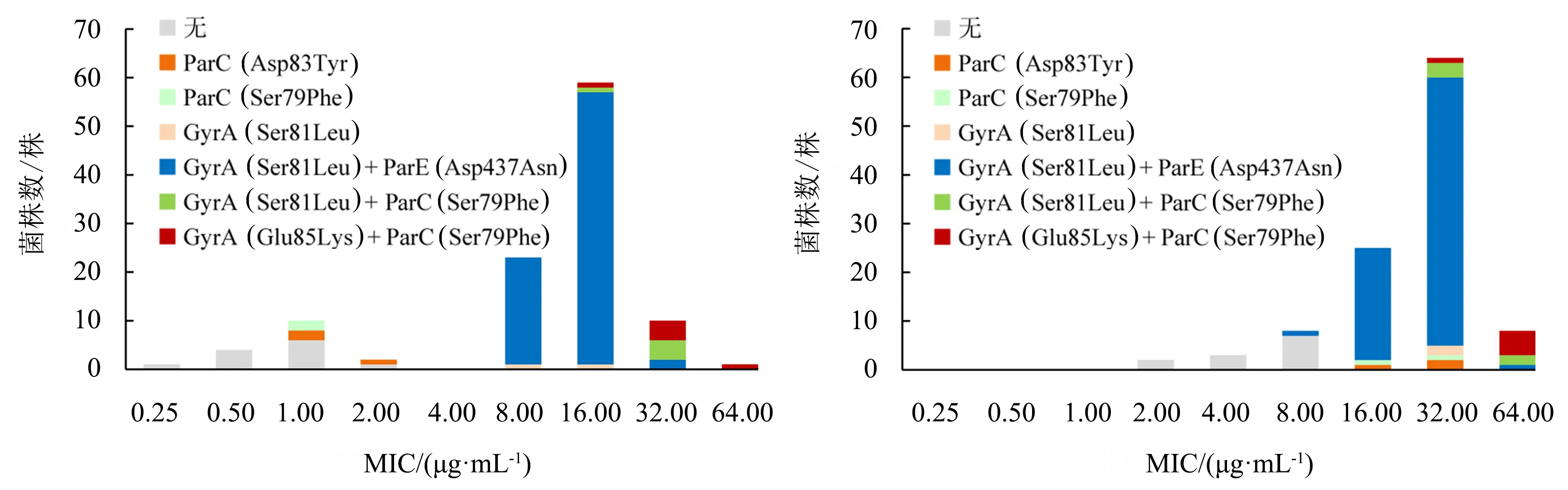
图1 菌株基因突变类型对左氧氟沙星MIC的影响

图3 菌株基因突变类型对环丙沙星MIC的影响
3 讨论与结论
我国奶牛养殖场隐性乳腺炎发病率高达50%以上[6,17],它不仅导致奶牛产奶量下降,还可由于治疗该病时抗菌药物残留、耐药菌产生等原因造成乳制品质量下降,甚至危及人类健康[18].近年来,无乳链球菌性奶牛乳腺炎的发生率呈现逐年上升的趋势[19],已成为部分规模化奶牛场乳腺炎防控的重点,但耐药菌的产生严重降低了对奶牛乳腺炎的治疗效果.
喹诺酮类药物是4-喹诺酮-3-羧酸结构化衍生物,自1962年由Lesher et al[20]初次合成至今,已有4代喹诺酮类药物问世,其中,氟喹诺酮类是喹诺酮类的第3代产物.喹诺酮类药物在治疗和预防动物疾病、促进动物生长、提高饲料转化率等方面应用广泛,临床上使用较多的是氧氟沙星、诺氟沙星和环丙沙星等药物.由于存在药物残留、产生耐药菌等影响动物产品质量安全和公共卫生安全的潜在风险,中华人民共和国农业部自2016年起禁止在食品动物中使用氧氟沙星、诺氟沙星、洛美沙星和培氟沙星4种喹诺酮类药物[21].尽管如此,喹诺酮类药物的品种和制剂数量多,仍然是兽医临床上应用最为广泛的抗菌药物之一.据统计,2018年我国养殖业使用的兽用抗菌药有69种,其中有10种为氟喹诺酮类药物,占总量的14.5%,排名第一[22].
人源无乳链球菌对左氧氟沙星的敏感率低,不敏感度高达50%~70%[23-24];牛源无乳链球菌对喹诺酮类药物的耐药率大多为0~50%,不同地区、养殖场的无乳链球菌对环丙沙星的耐药率差别较大[25].值得注意的是,近年来中介菌增多,在河北、内蒙古和宁夏分离的牛源无乳链球菌对环丙沙星的中介率分别为83%[26]、31%[27]、63%[28].中介菌的增多表明耐药率将呈现上升趋势.本研究对分离自我国10省区21个规模化奶牛养殖场的110株奶牛乳腺炎无乳链球菌进行了氟喹诺酮类药物的敏感性试验,发现耐药率超过80%,高于已往报道的数据[25],可能与养殖场用药种类和用药方法不同有关.比较分析本研究3种喹诺酮类药物对菌株的MIC发现,大多数耐药菌同时对3种喹诺酮类药物耐药.这与文献报道的“左氧氟沙星耐药菌对多种喹诺酮类药物均产生不同程度的耐药性”[29]一致.
QRDR基因突变是细菌对喹诺酮类药物耐药的主要机制之一.与参考菌株对比,本研究所有供试菌株基因均存在GyrB(Val498Ala)和ParE(Ile503Val)突变,而GyrB(Val498Ala)突变与菌株耐药性无明显关联[13,30],因此,本研究在进行基因突变统计分析时未考虑这两个位点.耐药菌基因最常见的突变位点为GyrA(Ser81Leu),占左氧氟沙星耐药菌的92.6%(87/94);其次为ParE(Asp437Asn),占左氧氟沙星耐药菌的85.1%(80/94).本研究中,菌株基因ParE(Asp437Asn)突变与GyrA(Ser81Leu)突变同时出现,且GyrA(Ser81Leu)+ParE(Asp437Asn)突变与GyrA(Ser81Leu)突变对喹诺酮类药物的敏感性并无明显差异,均呈现中高度耐药,因此,ParE(Asp437Asn)突变是否影响喹诺酮类药物敏感性尚需进一步验证.研究表明,基因GyrA和ParC的双突变会增强细菌的耐药程度[29],这与本研究结果一致.本研究中,菌株基因ParC第79和83位氨基酸突变虽然对左氧氟沙星敏感,但其对左氧氟沙星的MIC高于无突变菌株,且该菌株对环丙沙星和诺氟沙星耐药;菌株基因若同时存在ParC(Ser79Phe)突变和GyrA第81或85位氨基酸突变,则该菌株对喹诺酮类药物呈现高度耐药.本研究存在一些基因无突变的菌株,表现出对诺氟沙星或环丙沙星的低度耐药,这可能与其他耐药机制相关.除了QRDR基因突变以外,细菌携带含耐药基因的质粒、外排泵系统功能的增强和细胞膜通透性的降低也可导致其对喹诺酮类药物产生耐药性,但目前对于无乳链球菌的这些相关机制研究较少.
本研究结果表明,我国牛源无乳链球菌对喹诺酮类药物的耐药率较高,对左氧氟沙星、诺氟沙星和环丙沙星的耐药率分别为85.5%、98.2%、94.5%,其耐药性与菌株QRDR基因突变有关.本研究结果对兽医临床上合理使用喹诺酮类药物具有指导作用.
