南昌市食源性沙门菌多黏菌素耐药基因突变分析
2023-04-29邓灵芦晓萍翟平平李睿
邓灵 芦晓萍 翟平平 李睿

摘要:沙门菌耐药性对人类健康造成巨大威胁。多黏菌素号称人类对抗细菌感染的最后一道防线。本文对南昌市食品源沙门菌分离株进行药敏测定。并将4株多黏菌素耐药株进行三代全基因组测序。比较基因组分析结果表明,4株菌均不携带能导致多黏菌素耐药的基因mcr,但eptC、micA都存在基因突变,PhoP/PhoQ 和PmrA/PmrB系统存在的SNP位点可能是导致其耐多黏菌素的原因之一。本文结果为沙门菌耐多黏菌素的机制提供了新的数据参考。
关键词:沙门菌;全基因组测序;耐药;多黏菌素;基因突变
中图分类号:R978.1 文献标志码:A 文章编号:1001-8751(2023)03-0145-05
The Mutations of Polymyxin Resistance Genes of Foodborne Salmonella
Deng Ling1, Lu Xiao-ping1, Zhai Ping-ping2, Li Rui1
(1 College of Biological and Pharmaceutical Engineering,Wuhan Polytechnic Universitya, Wuhan 430023;2 Jiangxi General Institute of Testing and Certification Food Testing Institute, Nanchang 330000)
Abstract: The antimicrobial resistance of Salmonella poses a threat to human health. Polymyxin B is considered as last-line drug for bacterial infection. In this paper, four Polymyxin B-resistant Salmonella strains isolated from food sources were performed third generation whole-genome sequencing. The comparative genome analysis showed that the test strains did not carry mcr gene which was associated with polymyxin B resistance. However, the mutations on eptC and micA were found in the strains. The SNP sites present in PmrA/PmrB and PhoP/PhoQ system may be responsible for polymyxin B resistance of Salmonella in this paper. The results obtained in this paper provided new data for polymyxin B resistance research.
Key words: Salmonella; whole genome sequencing; antimicrobial resistance; polymyxin B; gene mutation
1 引言
沙门菌属(Salmonella spp.)是一种常见的食源性致病菌,革兰阴性、兼性、胞内细菌。1885年Salmon和Smith在霍乱流行时分离到猪霍乱沙门菌,故定名为沙门菌属[1]。在全球,尤其是发展中国家,每年大约有1600 万人感染沙门菌,导致大约60万人死亡[2]。在我国,由沙门菌引起的食源性中毒事件在食源性致病菌中毒事件中屡占首位(70%~80%)[3]。沙门菌感染最常见的原因是食用了被污染的肉、蛋或饮用被污染的牛奶[4]。
随着新一代基因组测序的发展,基于全基因组测序(Genome-wide sequencing, WGS)的分子分型技术在食源性疾病聚集性病例的识别和暴发溯源调查中显示出极大的应用价值和发展潜力[5]。基于全基因组测序有助于食源性微生物开展遗传与变异、致病与耐药机制及菌种进化等方面的基础研究。通过全基因组测序的结果,可以快速的完成对特异性基因之间比对,了解基因中存在的差异,从而解析耐药与致病机制。
沙门菌耐药性根据来源可以分为天然耐药性和获得性耐药性[6]。天然耐药性是沙门菌在自然界中赖以生存的一种特有性质;获得性耐药则可能是由于人类长时间在疾病治疗和预防等方面对抗生素的应用不当造成的。多黏菌素是20世纪50年代发现的一种环肽类抗生素,于1947年在多黏芽孢杆菌(Bacillus polymyxa)中发现,对绝大部分革兰阴性细菌具有抗菌活性[7]。临床上常用的有多黏菌素B(Polymyxin B)和多黏菌素E(Colistin)。目前,多黏菌素已经成为了治疗多重耐药病原菌引起严重感染的最后一道防线。但随着病原菌的耐药性进一步的加强,世界范围内不断有新的报道称检测出耐多黏菌素的致病菌[8]。沙门菌常见的多黏菌素耐药机制有脂多糖修饰、质粒介导的多黏菌素耐药、细菌在不利条件下分泌的多糖物质屏障、外排泵等[9]。目前国内研究者开展食源性沙门菌耐药性研究时很少测定多黏菌素抗性[10-11]。但沙门菌临床株多黏菌素耐药性较为常见。浙江大学分析了沙门菌临床株多黏菌素耐药性,其中肠炎沙门菌(S. enteritidis)和鼠伤寒沙门菌(S. typhimurium)多黏菌素耐药率分别高达83.9% (125/149) 和15.3% (9/59)[12]。因此很有必要开展食源性致病菌多黏菌素耐药性监测。
本文研究目的就是筛查44株沙门菌食源性菌株多黏菌素的耐药性,再选取代表性耐药菌株进行三代测序,采用比较基因组学技术分析多黏菌素耐药相关的耐药基因SNP突变,为多黏菌素耐药机制研究提供新数据。
2 材料和方法
2.1 实验材料
样本来源为2020年在江西南昌地区的多个菜市场的冷鲜、现杀或冰冻的鸡肉与猪肉中分离到的44株沙门菌。沙门菌SAL-007(NCBI登录号:CP071686.1)、SAL-020(CP071690.1)、SAL-045(CP071693.1)均分离自2020年食物中毒病人粪便样本。
2.2 主要试剂
多黏菌素B(批号Y26A11H122615,武汉飞扬生物有限公司)、胰蛋白胨(批号20220303,青岛海博生物公司)、氯化钠(批号20161109,国药集团化学试剂有限公司)、酵母提取物(批号4352785-02, OXOID)、MH肉汤(批号20220601,青岛海博生物公司)、96孔细胞培养板(购自武汉华顺生物有限公司)。
2.3 实验方法
2.3.1 多黏菌素耐药性测定
按照美国临床和实验室标准协会(CLSI)方案[13]配制多黏菌素原液,用二倍稀释法对多黏菌素进行梯度稀释,稀释范围为0.125、0.25、0.5、1、2、4、8、16、32 μg/mL。首先将44株沙门菌分批次进行37 ℃过夜培养。于超净工作台分别吸取4 mL实验菌株到对应的10 mL EP管中。离心机8000 r/min离心5 min,去上清液,加入4 mL PBS溶液,移液枪吹打至沉淀完全重悬,重复操作2次。用浊度仪调节细菌菌悬液浓度到0.5麦氏单位(1×108 CFU/mL)并用MH溶液稀释100倍待用。取96孔板按标记依次加入不同梯度的多黏菌素稀释液50 μL、稀释后菌液50 μL混合。阳性对照加入50 μL稀释后菌液、MH溶液50 μL混合。阴性对照加入100 μL MH溶液。将96孔板置于生化培养箱,37 ℃,避光静置18~24 h。以肉眼观察到能抑制细菌生长的最低药物浓度为最低抑菌浓度,记录各菌株MIC浓度。大肠埃希菌ATCC25922作为质量控制菌。
2.3.2 全基因组测序数据分析与挖掘
以三代测序数据为主,采用二代测序进行补测及纠错的策略进行全基因组测序。将多黏菌素耐药菌株培养到OD0.6左右收集菌体,放置于EP管中,然后用干冰保存快递到上海美吉生物公司进行Pacbio三代测序。利用SOAP denovo 2组装测序原始序列得到完整的基因组序列。
使用PlasmidFinder软件筛查质粒,通过在线工具PlasmidFinder 2.1、MobileElementFinder和PMLST预测质粒类型。使用快速注释网站(RAST, Version2.0)对沙门菌的染色体以及质粒基因进行注释。利用Resfinder(Version4.1)和综合抗生素研究数据库(Comprehensive antibiotic resistance database, CARD)对耐药基因进行预测注释。以上海美吉生物公司与RAST预测的耐药基因为准,用Resfinder和CARD进行校正。对于无法查证的基因则采用NCBI的BLAST进行比对确定。重点筛查mcr基因。然后将测序菌株的多黏菌素耐药基因序列进行Blast分析,查找SNP位点,重点筛查PhoPQ系统和Pmr A/Pmr B系统相关基因的SNP突变。
2.3.3 基于CVTree构建进化树
利用在线分析软件CVTree 3(http://tlife.fudan.edu.cn/cvtree/),将这四株菌全基因组序列导入系统一起做系统发育分析。
3 结果与分析
3.1 多黏菌素耐药性测定
44株沙门菌中有3株对多黏菌素B中介(MIC≤2 μg/mL),有4株对多黏菌素B耐药(MIC≥4 μg/mL),其余菌株为敏感型。菌株A7和A29-2的MIC为32 μg/mL,菌株A39的MIC值为8 μg/mL,菌株JXY0409-18的MIC值为4 μg/mL。
3.2 全基因组测序数据分析与挖掘
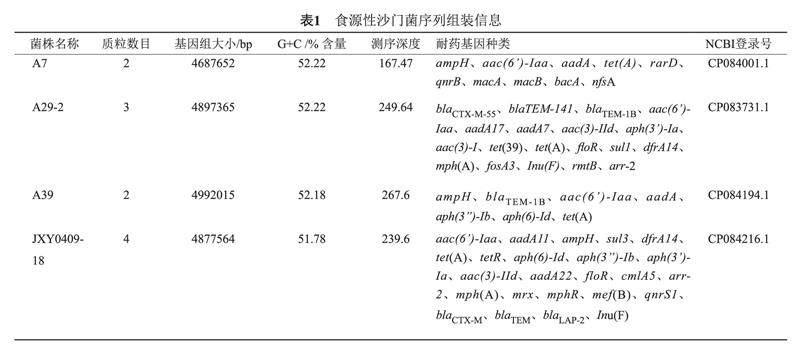
将4株多黏菌素耐药株送至上海美吉生物公司进行测序处理。全基因组测序基本信息如测序深度、GC含量、耐药基因种类等参见表1所示。由表1数据可知,4株菌都携带质粒和多种耐药基因。
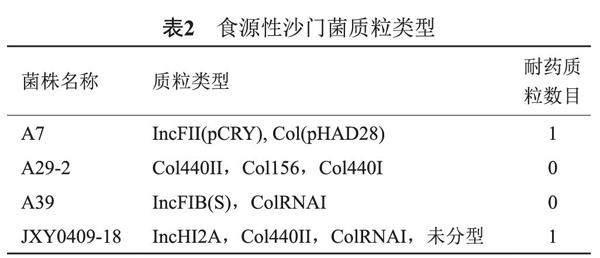
将4株沙门菌的质粒序列输入相关软件预测质粒类型,除了菌株JXY0409-18携带的一个小质粒无法进行分型外,其他质粒分类结果如表2所示。4株菌中仅有菌株A7和JXY0409_18携带耐药质粒。其中菌株A7耐药质粒只携带一种耐药基因,即喹诺酮类耐药基因qnrB。A7、A29-2、A39的耐药基因主要位于染色体上。菌株JXY0409_18与此相反,染色体仅携带3种耐药基因,如氨基糖苷类耐药基因aac(6')-Iaa、aadA11和β-内酰胺类耐药基因ampH,其余21种耐药基因都位于质粒上。
3.3 基于CVTree的进化树分析
将4株沙门菌测序株全基因组序列输入到CVTree3,软件将上传的序列和系统内置的细菌序列信息比较后生成亲缘关系树,如图1所示。4株菌分属于不同分支,根据与它们亲缘关系相近的菌株显示的血清型信息,可知A7属于S.give,A29-2属于S.kentucky,A39属于S.typhimurium,JXY0409-18属于 S.schwarzengrund。
3.4 多黏菌素耐药基因SNP突变分析
对多黏菌素相关耐药基因进行分析。经筛查4株菌均不携带导致多黏菌素耐药的mcr基因。文献报道PhoP/PhoQ和PmrA/PmrB系统存在SNP位点也可导致多黏菌素耐药[14]。沙门菌临床株SAL-007、SAL-020、SAL-045 均是肠炎沙门菌,且都对多黏菌素敏感,将4株多黏菌素耐药菌和这3株敏感菌一起对比,筛查PhoP/PhoQ和PmrA/PmrB系统的基因突变,包括eptA、eptB、eptC、pmrA、pmrB、mgrB、 micA、phoP、phoQ、lpxA、lpxC和lpxD。最后发现和敏感菌相比,耐药株在eptC、micA基因上均存在少量共同的SNP突变(表3),其他基因则没有发现规律性的SNP突变。敏感菌在这些基因上未发现SNP突变。
4 讨论与结论
本文发现江西省食品源沙门菌中有9%(4/44)耐多黏菌素,通过全基因组测序分析,可知4株沙门菌均不携带多黏菌素耐药基因mcr,而mcr是导致沙门菌耐多黏菌素最主要的耐药基因[15-16]。除此之外,PhoP/PhoQ和PmrA/PmrB系统对多黏菌素耐药有调控作用[17]。多黏菌素主要是通过破坏革兰阴性菌细菌脂多糖(Lipopolysaccharide,LPS)的结构来发挥杀菌作用的。通过与LPS带负电荷的脂质A成分相互作用,多黏菌素将带正电的残基(例如4-氨基-L-阿拉伯糖(L-Ara4N)、磷酸乙醇胺(PEtn)添加到LPS中,减少细菌表面上的负电荷,从而降低LPS的稳定性并破坏外膜的完整性,达到杀灭细菌的目的[18]。PhoP/PhoQ 和PmrA/PmrB系统可调控L-Ara4N、PEtn的合成,影响其添加到LPS中,因此PhoP/PhoQ和PmrA/PmrB调控系统及其调节器的突变会阻碍多黏菌素与细菌的结合,减弱多黏菌素的杀菌作用[6]。文献证实PhoP/PhoQ和PmrA/PmrB系统上的基因突变可导致多黏菌素耐药[19-20],通过比较多黏菌素耐药菌和敏感菌,发现4株多黏菌素耐药菌株虽然血清型不同,亲缘关系较远,但在eptC、micA基因上均存在少量共同的SNP突变,这可能是导致多黏菌素耐药的原因。本文在eptC、micA上发现的部分SNP突变位点目前未发现同类文献报道,这些SNP位点导致的基因表达变化和多黏菌素耐药效果还有待进一步实验证实。
质粒介导的耐药是获得性耐药的主要传播方式。多黏菌素耐药基因mcr主要位于耐药质粒上[21]。本文测序的4株菌耐药基因主要位于染色体上,仅仅一株菌JXY0409_18质粒上携带多种耐药基因。但4株菌都不携带mcr基因。因此南昌市食品源沙门菌多黏菌素耐药主要是固有耐药。
多黏菌素耐药机制较为复杂,近年来消毒剂和抗生素的过度频繁使用可能诱导PhoP/PhoQ和PmrA/PmrB系统产生更多的基因突变,导致更多多黏菌素耐药菌的产生,因此对耐药菌进行全基因组测序分析,有助于解析耐药机制,跟踪细菌耐药基因突变状况。
参 考 文 献
张庆贺, 张丹俊, 李槿年, 等. 沙门菌耐药性研究进展[J]. 安徽农业科学, 2018, 46(17): 27-29.
Parisi A, Crump J A, Glass K, et al. Health outcomes from multidrug-resistant Salmonella infections in high-income countries: a systematic review and meta-analysis[J]. Foodborne Pathog Dis, 2018, 15(7): 428-436.
罗荣, 任秀, 崔生辉. 食品中沙门菌快速检测技术研究进展[J]. 食品安全质量检测学报, 2016, 7(4): 1469-1472.
Chen Z, Bai J, Zhang X, et al. Highly prevalent multidrug resistance and QRDR mutations in Salmonella isolated from chicken, pork and duck meat in southern China, 2018-2019. Int J Food Microbiol[J], 2021, 340(2021): 1-7.
李晓然, 张若鸿, 杨洋, 等. 食源性致病菌全基因组测序及全球数据共享平台发展趋势[J]. 中国食品卫生杂志, 2020, 32(3): 339-344.
李立, 叶璟, 胡蕾, 等. 多黏菌素耐药性研究进展[J]. 医学综述, 2021, 27(17): 3376-3382.
王影, 李艳然, 韩镌竹, 等. 多黏菌素耐药性的进展[J]. 微生物学通报, 2017, 44(1): 200-206.
Wang W, Chen J, Shao X, et al. Occurrence and antimicrobial resistance of Salmonella isolated from retail meats in Anhui, China[J]. Food Sci Nutr, 2021, 9(9): 4701-4710.
Olaitan A O, Morand S, Rolain J M. Mechanisms of polymyxin resistance: acquired and intrinsic resistance in bacteria[J]. Front Microbiol, 2014, 5(643): 1-18
夏飞, 郑雪, 吴静, 等. 陕西省常见食源性致病菌耐药性研究进展[J]. 食品与生物技术学报, 2021, 40(7): 10-18.
张铭琰, 耿英芝, 于淼, 等. 辽宁省不同来源沙门菌耐药性分析[J]. 食品安全质量检测学报, 2020, 11(19): 7142-7146.
Luo Q, Wang Y, Fu H, et al. Serotype is associated with high rate of colistin resistance among clinical isolates of Salmonella[J]. Front Microbiol, 2020, 11: 592146.
Humphries R, Bobenchik A M, Hindler J A, et al. Overview of changes to the clinical and laboratory standards institute performance standards for antimicrobial susceptibility testing, M100, 31st Edition[J].J Clin Microbiol, 2021, 59(12):e0021321.
Sato T, Shiraishi T, Hiyama Y, et al. Contribution of novel amino acid alterations in PmrA or PmrB to Colistin resistance in mcr-negative Escherichia coli clinical isolates, including major multidrug-resistant lineages O25b: H4-ST131-H30Rx and Non-x[J]. Antimicrob Agents Chemother, 2018, 62(9): e00864-18.
Ling Z, Yin W, Shen Z, et al. Epidemiology of mobile colistin resistance genes mcr-1 to mcr-9[J]. J Antimicrob. Chemother, 2020, 75(11): 3087-3095.
王新兴, 翟真真, 常维山, 等. 多黏菌素耐药基因mcr-1的研究进展[J]. 中国动物传染病学报, 2020, 28(2): 110-114.
Binsker U, K?sbohrer A, Hammerl J A. Global colistin use: A review of the emergence of resistant Enterobacterales and the impact on their genetic basis[J].FEMS Microbiol. Rev, 2022, 46(1): fuab049.
陈涌泉, 王厚照. 多黏菌素耐药机制的研究进展[J]. 中国抗生素杂志, 2021, 46(8): 728-735.
Nirwan P K, Chatterjee N, Panwar R, et al. Mutations in two component system (PhoPQ and PmrAB) in colistin resistant Klebsiella pneumoniae from north Indian tertiary care hospital[J]. J Antibiot (Tokyo), 2021,74(7): 450-457.
Cannatelli A, Giani T, Aiezza N, et al. An allelic variant of the PmrB sensor kinase responsible for colistin resistance in an Escherichia coli strain of clinical origin[J]. Sci Rep, 2017, 7(1): 1-6.
Li Y, Dai X, Zeng J, et al. Characterization of the global distribution and diversified plasmid reservoirs of the colistin resistance gene mcr-9[J]. Sci Rep, 2020, 10(1): 1-10.
收稿日期:2022-10-29
基金项目:江西省市场监督管理局科技计划项目(GSJK202104)。
作者简介:邓灵,研究生,主要从事食品微生物的研究工作。
*通讯作者:李睿,教授,主要从事食品微生物与食品安全的研究工作。
