1株mphA和mcr-1阳性鼠伤寒沙门菌的鉴定及耐药特征分析
2023-03-14韩艺然李欣平朱坤鹏彭玉倩杨海燕邱少富向莹
韩艺然 李欣平 朱坤鹏 彭玉倩 杨海燕 邱少富 向莹
摘要:目的 1株mphA和mcr-1陽性鼠伤寒沙门菌的鉴定及耐药特征分析;方法 分离自4岁腹泻患者粪便样本中的1株鼠伤寒沙门菌,使用沙门菌血清凝集法进行菌株鉴定,质谱法复核鉴定结果;使用微量肉汤稀释法进行药敏试验;通过全基因组测序及耐药基因注释鉴定菌株所携带的耐药基因;通过质粒测序及生信分析进行质粒类型鉴定、质粒耐药基因、插入子等注释及质粒结构分析;通过质粒接合转移试验评估质粒的水平转移能力。结果 药敏试验结果显示分离株对包括阿奇霉素、多黏菌素、头孢曲松和环丙沙星等临床常用抗生素药物耐药。耐药基因鉴定结果显示:该分离株携带包括大环内酯类抗性基因mphA,多黏菌素抗性基因mcr-1在内的12类45个耐药基因及1个氟喹诺酮类耐药点突变gyrA S83F。质粒测序分析结果显示:该菌株携带一个260,008bp大小的IncHI2型质粒,该质粒携带包括mphA基因和mcr-1基因在内的19个耐药基因,15种插入子,4种转座子。质粒接合转移实验结果显示该质粒可以在肠杆菌目细菌之间水平转移。结论 研究结果为临床治疗鼠伤寒沙门菌感染中合理使用抗生素以及有效预防、控制耐阿奇霉素、多黏菌素鼠伤寒沙门菌的传播提供了科学依据。
关键词:鼠伤寒沙门菌;耐药性;mphA;mcr-1;质粒
中图分类号:R278;R378文献标志码:A
Identification and antimicrobial resistance analysis of a strain of Salmonella typhimurium positive for mphA and mcr-1
Han Yiran, Li Xinping, Zhu Kunpeng, Peng Yuqian, Yang Haiyan, Qiu Shaofu, and Xiang Ying
(1 College of Public Health, Zhengzhou University, Zhengzhou 450001;
2 The Centre of Disease Control and Prevention of Chinese People's Liberation Army, Beijing 100071)
Abstract Objective To identify a mphA and mcr-1-positive multidrug-resistant Salmonella typhimurium isolate and analyze its antimicrobial resistance. Methods The strain isolated from a fecal sample of a four years old kid and collected. The Salmonella strains were identified by serum agglutination method, and the results were verified by mass spectrometry . The antimicrobial sensitivity test was conducted by microbroth dilution method. The antimicrobial resistance(AMR) genes were identified by whole genome sequencing and AMR genes annotation. Plasmid sequencing and bioinformatics analysis were used to identify plasmid types, annotate plasmid AMR genes and insertion sequences, and analyze plasmid structure. The horizontal transfer ability of the plasmid was estimated by plasmid conjugative transfer assay. Results The results of antimicrobial sensitivity test showed that the isolates were resistant to commonly used antibiotics including azithromycin, colistin, ceftriaxone and ciprofloxacin. Identification of AMR genes showed that the isolated strain carried 45 AMR genes in 12 classes, including macrolide resistance gene mphA, colistin resistance gene mcr-1 and a point mutation in gyrA gene. Plasmid sequencing analysis showed that the strain carried a 260,008 bp size of IncHI2 plasmid, which carried 19 AMR genes, 15 classes of insertion sequences and 4 classes of transposons. The results of plasmid conjugative transfer assay showed that the plasmid could be transferred horizontally between intestinal strains. Conclusion This study provides a scientific basis for guiding the rational application of antibiotics and curbing the continuous spread of bacterial resistance.
Key words Salmonella typhimurium; Antimicrobial resistance; mphA; mcr-1; Plasmid
沙门菌感染是一个重大的全球性公共卫生问题,在许多国家引起了食源性疾病。食源性疾病主动监测网络(FoodNet)报告的数据显示,沙门菌已成为美国食源性细菌病原体的主要死亡原因[1]。目前,沙门菌已有2600多种血清型存在,鼠伤寒沙门菌是最常见的血清型之一[2]。在我国临床腹泻样本分离的沙门菌中,鼠伤寒沙门菌的分离比例多年来排名第一或第二,感染发病率也常年居首位[3-4]。近年来,随着抗生素的广泛及长期使用,鼠伤寒沙门菌已进化出多种耐药表型,呈现出耐药率高、耐药范围广的特点。由于常见用于治疗鼠伤寒沙门菌感染患者的抗生素,如环丙沙星、头孢曲松等的耐药率增加,阿奇霉素被FDA推荐用于鼠伤寒沙门菌感染临床治疗药物[5-6]。由于多黏菌素具有肾毒性和神经毒性的副作用,在20世纪80年代逐渐被其他抗菌类药物所替代[7]。然而,近年来,随着碳青霉烯类抗生素耐药性的日益流行,多黏菌素被认为是治疗多重耐药革兰阴性菌的最后手段[8]。随着阿奇霉素和多黏菌素在沙门菌感染治疗中的使用率增加,在病原监测过程中,发现了1株对阿奇霉素和多黏菌素共耐药的鼠伤寒沙门菌,这是极少见的,因此我们对其的耐药特征及其分子机制展开了研究。
1 材料和方法
1.1 材料
1.1.1 菌株來源
菌株分离于重庆市渝中区4岁急性腹泻患者粪便样本的鼠伤寒沙门菌。
1.1.2 主要试剂
沙门菌-志贺菌(SS)琼脂培养基(北京路桥技术股份有限公司)、营养琼脂培养基平板(广东环凯微生物科技有限公司)、沙门属诊断血清(泰国S&D公司)、CMV3AGNF药敏板(ThermoFisher Scientific)、Phoenixtm NMIC-413药敏板(碧迪医疗器械有限公司)、麦康凯琼脂粉(北京路桥股份技术有限公司)、CAMHB肉汤培养基(武汉金同浩博生物科技有限公司)、头孢曲松、环丙沙星Etest试纸条(意大利Liofilchem生物公司)、QIAamp DNA Minikit 试剂盒(德国 QIAGEN 公司)、质粒小提试剂盒(天根生化科技有限公司)。引物合成自生工(上海)生物工程有限公司。
1.1.3 主要仪器
Autof ms1000全自动微生物鉴定仪(郑州安图生物工程有限公司)、英国先德系列AutoReader自动快速微生物药敏及鉴定分析系统和SWIN微生物数据处理与报告系统、MiSeq platform 测序仪(Illumina公司)、美国Bio-Rad公司的PCR仪。
1.2 方法
1.2.1 菌株鉴定与复核实验
将实验室保存的菌株进行复苏,使用三区划线法在LB培养基上进行过夜培养,在培养好的营养琼脂平板上挑取单菌落使其在SS培养基上37 ℃培养16~18 h,然后根据典型菌落形态,(中心黑色边缘无色透明)挑选单菌落接种于LB培养基上进行过夜培养,37 ℃培养16~18 h。通过O抗原和H抗原的玻片凝集法进行血清型鉴定,血清型鉴定过程严格参考国家沙门菌检验标准(GB/T4789.4-2016),通过质谱试验对鉴定结果进行复核。
1.2.2 抗菌药物敏感性实验
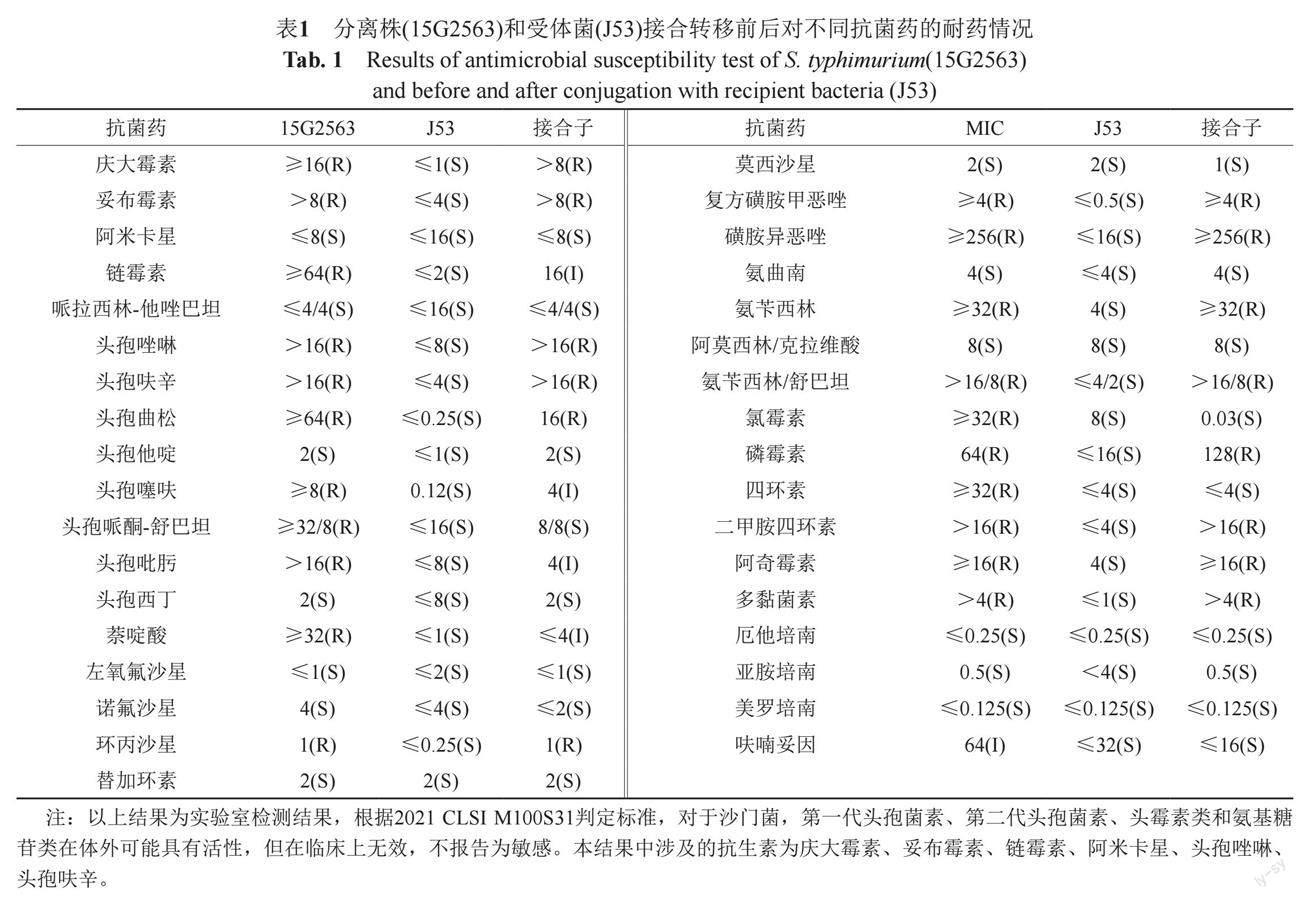
采用肉汤稀释法,选用CMV3AGNF和Phoenixtm NMIC-413商业化药敏板进行抗菌药物敏感性实验,以大肠埃希菌ATCC25922菌株作为质控菌株。测定共包含16类35种抗生素,包括氨基糖苷类:庆大霉素(GEN,0.25~16 μg/mL)、妥布霉素(NN, 2~8 μg/mL)、阿米卡星(AN,8~32 μg/mL)、链霉素(STR,2~64 μg/mL);抗菌青霉素+酶抑制剂:哌拉西林/他唑巴坦(TZP, 4/4~64/4 μg/mL));头孢类:头孢唑林(CZ, 2~16 μg/mL)、头孢呋辛(CXM, 4~16 μg/mL)、头孢曲松(CRO,0.25~64 μg/mL)、头孢他啶(CAZ, 1~32 μg/mL)、头孢噻呋(XNL,0.12~8μg/mL)、头孢哌酮-舒巴坦(SCP, 0.5/8~32/8 μg/mL)、头孢吡肟(FEP, 1~16 μg/mL)、头孢西丁(FOX,0.5~32μg/mL);喹诺酮类:萘啶酸(NAL,0.5~32μg/mL)、左氧氟沙星(LVX, 1~8 μg/mL)、诺氟沙星(NOR, 2~8 μg/mL)、环丙沙星(CIP,0.015~4 μg/mL)、莫西沙星(MXF, 0.5~2 μg/mL);磺胺类:复方磺胺甲恶唑(SXT,0.12/2.38~4/76 μg/mL)、磺胺异恶唑(FIS,16~256 μg/mL);
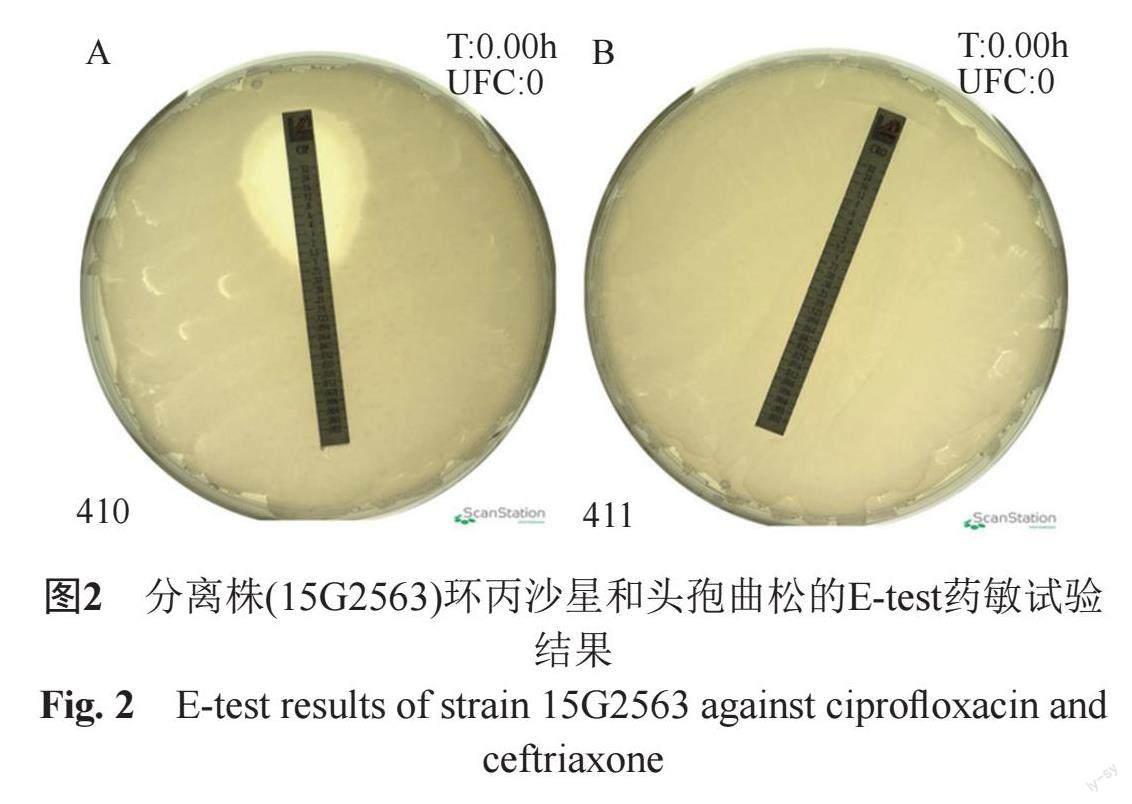
β-内酰胺类:氨曲南(ATM, 2~32 μg/mL);青霉素:氨苄西林(AMP,1~32 μg/mL);青霉素+酶抑制剂:阿莫西林/克拉维酸(AUG2,1/0.5~32/16 μg/mL)、氨苄西林/舒巴坦(SAM, 4/2~16/8 μg/mL);氯霉素类:氯霉素(CHL,2~32 μg/mL);磷酸类:磷霉素(FF,16~128 μg/mL);四环素类:四环素(TET,4~32μg/mL)、二甲胺四环素(MI, 1~16 μg/mL);大环内酯类:阿奇霉素(AZI,0.12~16μg/mL);多黏菌素:多黏菌素(CL, 1~4 μg/mL);碳青霉烯类:厄他培南(ETP, 0.25~2 μg/mL)、亚胺培南(IPM, 0.25~8 μg/mL)、美罗培南(MEM, 0.125~8 μg/mL);硝基呋喃类:呋喃妥因(FM, 16~24 μg/mL);甘氨酰环素:替加环素(TGC, 1~8 μg/mL)。使用E-test药敏法复核头孢曲松和环丙沙星药敏结果,挑取分离株单菌落于0.85% NaCl中,调整菌落形成单位为1.5×108 CFU/mL,用棉签蘸取适量菌液涂布于MH琼脂培养基上,将试纸条贴于MH琼脂培养基上,37 ℃培养16~18 h,观察抑菌情况并记录MIC值。结果按照2021年版临床和实验室标准协会指南(CLSI M100S31)进行解释[9]。
1.2.3 全基因组测序及耐药基因检测
使用QIAamp DNA试剂盒提取基因组DNA,使用NEBNext?Ultra?DNA library Prep kit for Illumina制备文库,并使用Illumina的高通量测序平台MiSeq生成配对端序列。质量控制和过滤通过Fastp (v0.20.1)[10]。使用SPAdes (v3.9.0)起草基因组组装,使用QUAST (v4.5)进行基因组组装质量评估,使用Prokka (v1.12)进行基因组注释[11-13]。
使用综合抗生素耐药性数据库(CARD)的耐药性基因标识符(RGI)预测抗微生物药物耐药性(AMR)基因的存在[14],使用BLAST(BLASTn)检测每个基因组序列中覆盖率超过70%的AMR基因组和80%的同源性,使用snippy软件进行耐药基因序列点突变比对分析。点突变基因进行PCR扩增,PCR产物进行一代测序,将测序结果与耐药基因序列比对以验证点突变。
1.2.4 质粒测序分析
使用Qiagen Plasmid Midi kit试剂盒来提取质粒DNA,质粒测序交由北京诺禾致源公司进行,并对质粒基因组进行拼接和组装;通过RAST在线网站对质粒进行初步注释;利用CGE网站中的PlasmidFinder功能鉴定质粒的复制子类型;使用Prokka (v1.12)进行基因组注释,利用card和ISFinder数据库对注释结果进行补充,完善序列结构;采用BLAST Ring Image Generator (BRIG)进行质粒序列比较分析。
1.2.5 质粒结合转移实验
采用质粒结合转移实验测定质粒的可转移性。以大肠埃希菌J53为受体,进行质粒偶联转移实验。取出菌株划线接种至LB培养基上,培养16~18 h,将单菌落接菌至5mL LB液体培养基的离心管中,放至37℃的摇床上增菌培养6 h;分别取20 μL的供体菌和60 μL的受体菌J53混于5 mL的LB液中,使供受体菌比例为1:3即可,放入 37 ℃恒温培养箱中,静置16~18 h;将上述菌液稀释1000倍后,吸取100 μL涂布在含有100 mg/mL叠氮化钠和25 mg/mL阿奇霉素及多黏菌素2 mg/mL的麦康凯培养基上,37℃静置培养36~72 h。挑选单菌落,通过PCR扩增供体和衍生的转偶联菌株中存在的质粒。
2 结果
2.1 菌株鉴定及药物敏感性
该分离株的0抗原为:1,4,[5],12;H抗原第一相为:i;H抗原第二相为:1,2,血清型鉴定为鼠伤寒沙门菌,质谱实验复核结果一致,SS培养基上菌落形态见图1。耐药性检测结果显示,该分离株对包括阿奇霉素、多黏菌素、环丙沙星、头孢曲松等多种临床常用抗生素耐药(表1)。E-test实验复核结果显示,该菌株对头孢曲松的MIC值为>32 μg/mL,对环丙沙星的MIC值为1 μg/mL,根据CLSI M100判定标准,复核结果均为耐药(图2)。
2.2 耐药基因检测
该分离株携带12类45个耐药基因,包括大环内酯类抗性基因mphA,黏菌素抗性基因mcr-1,氨基糖苷类抗性基因AAC(3)-Ⅳ、AAC(6)-Iaa、ANT(3)-IIa、APH(3)-Ib、APH(6)-Id、aadA2、acrD、kdpE,β-内酰胺类抗性基因blaCTX-M-14和blaTEM-1,磷霉素抗性基因fosA3,磺胺类抗性基因sul1、sul2,四环素类抗性基因tet(B),多肽类抗性基因bacA,二氨基嘧啶类抗性基因dfrA12,氯霉素抗性基因cmlA1、floR;外排泵相關基因CRP、acrA、ampH、H-NS、acrB、cpxA、golS、marA、mdsA、mdsB、mdsC、mdtB、mdtC、mdtK、msbA、oqxA、oqxB、qacH、sdiA、tolC和yojI,氟喹诺酮类抗性基因emrA、emrB和emrR(表2)。
该分离株携带1个氟喹诺酮耐药基因点突变gyrA S83F,测序结果显示,gyrA基因碱基序列的248位点由C突变为T,从而导致对应的氨基酸序列83位点由S(丝氨酸)突变为F(苯丙氨酸),该突变是产生氟喹诺酮耐药性的重要机制[15-16]。
2.3 质粒测序分析
提取质粒并进行测序分析,分析结果显示该质粒类型为IncHI2型。该质粒携带包括大环内酯类抗性基因mphA、多黏菌素抗性基因mcr-1在内的19个耐药基因(表2)。对该质粒结构进行比对分析,结果显示mphA基因位于IS26介导的转座单元中,mcr-1基因位于ISApl1介导的转座单元中,该质粒含有较多插入序列和转座子,如IS2、IS4321、Tn5393和TnAs3等。质粒比对发现,该质粒与分离于我国广州地区的圣保罗沙门菌中大小为237,710 bp的pS25质粒骨架相似,其覆盖度为81%,相似度为99.98%(图3)。
2.4 质粒结合转移
质粒接合转移成功之后的大肠埃希菌 J53 阳性接合子经 PCR 扩增验证mphA和mcr-1基因阳性(表3和图4),对结合菌进行药敏实验,实验结果阳性接合子对阿奇霉素(最低抑菌浓度为≥16 μg/mL)及多黏菌素耐药(最低抑菌浓度为>4 μg/mL)(表1)。
3 讨论
随着临床治疗和畜牧业长期使用抗生素,沙门氏菌的耐药率不断上升,多重耐药沙门菌的比例不断提升。鼠伤寒沙门菌是世界范围内沙门菌感染最常见的血清型之一,其耐药问题尤为突出。研究报道多重耐药鼠伤寒沙门菌blaCMY-2在墨西哥迅速广泛传播[17],对当地公共卫生造成极大威胁。多重耐药鼠伤寒沙门菌在我国鼠伤寒沙门菌中的占比也高达52.29%[18]。除了对常见抗生素的高耐药率外,近年来,阿奇霉素及多黏菌素因其治疗鼠伤寒沙门菌感染的有效性,这两类抗生素的临床应用相对增加,在抗生素选择压力下少数鼠伤寒沙门菌出现对阿奇霉素的耐药性、极少数菌株出现对多黏菌素的耐药性,这些耐药鼠伤寒沙门菌的出现和传播对临床治疗沙门菌感染提出了挑战。
肠杆菌目细菌对阿奇霉素的耐药机制主要包括①外排泵过表达,②多肽基tRNA水解酶过表达,③染色体突变,如核糖体蛋白改变和23S rRNA突变,④erm基因编码的甲基化酶(特别是ermA和ermB)介导的甲基化,⑤ereA和ereB基因编码的酯酶和/或mphA和mphB编码的磷酸转移酶介导的大环内酯失活。其中,mphA介导的阿奇霉素耐药性在肠杆菌目细菌中被多次报道[19-20]。
质粒介导的多黏菌素耐药基因mcr-1最初是2015年在中国的大肠埃希菌中发现的[21]。Hu等[22]通过已公开的细菌数据库分析发现mcr-1基因序列最早可在2011年中国人肠道菌群的研究中被检出。但mcr-1基因在肠杆菌目细菌中的检出水平依然很低,在我国大肠埃希菌中检出率为1.20%[23],沙门菌中检出率为1.08%[24]。由于mphA和mcr-1往往以质粒形式被携带,从而导致阿奇霉素、多黏菌素耐药性通过水平转移的方式在肠杆菌目细菌中快速传播,这些基因甚至可以与其他的耐药基因共同存在于单一质粒,表达后产生多种耐药机制[25]。
本研究报道了1株同时携带mphA和mcr-1的鼠伤寒沙门菌。该分离株对包括阿奇霉素、多黏菌素等多种临床重要抗生素药物耐药。值得关注的是该分离株对头孢曲松、环丙沙星这些沙门菌感染治疗的临床一线药物耐药,这引起我们对临床治疗的有效性的担忧。研究显示该菌株携带12类45个耐药基因及1个氟喹诺酮类耐药基因点突变gyrA S83F,耐药基因分析结果与耐药性结果相匹配,这些耐药基因的携带赋予了菌株广泛的耐药性。该分离株携带的大环内酯类抗性基因仅有mphA基因,多黏菌素抗性基因仅有mcr-1,说明mphA及mcr-1基因在该分离株中分别对阿奇霉素和多黏菌素的耐药中起到了关键作用,与已有的研究报道一致[26-27]。
质粒是能够在细胞之间自我传递的自主DNA分子,它们也能够通过称为高频重组(Hfr)的过程动员部分染色体[28]。质粒通过转座子或插入序列等可移动遗传元件获取新基因,以及它们在大范围宿主中复制的能力,使它们成为抗菌素耐药性(Anti-Microbial Resistance, AMR)传播的完美载体。肠杆菌目细菌中最常见的质粒为IncA/C、IncFIB、IncN和IncHI2,这些质粒在不同菌株间抗生素耐药性的传递中都起着重要作用[29-31]。在本研究中,mphA和mcr-1基因存在于IncHI2型质粒上,该类型质粒在鼠伤寒沙门菌中广泛分布,在其多重耐药的出现和快速传播中发挥了重要作用。鼠伤寒沙门菌中IncHI2质粒常对碳青霉烯类、头孢菌素类和氟喹诺酮类抗生素耐藥,而阿奇霉素耐药的报道较少[32-34]。通过质粒结构比较分析,结果显示p15G2563上存在15种插入序列和Tn3、Tn5393等4种转座子,这些插入元件均能独立转位,可介导耐药基因在质粒、整合子和染色体间转移。本研究发现mphA由转座子IS26转座单元介导,mcr-1基因位于ISApl1介导的转座单元中。IS26经常与编码抗生素抗性因子的基因相关,据报道质粒中IS26的存在大大增加了额外IS26插入该质粒的频率,从而形成包含多个抗生素抗性基因的区域,导致多重耐药的发生[35]。ISApl1属于IS30家族,是介导mcr-1基因最常见的插入元件,Laurent等人研究证实了复合转座子结构ISApl1-mcr-1-ISApl1的转位功能[36],但近年来发现许多mcr-1基因在只有一个拷贝ISApl1的情况下被介导转座[24]。质粒结合转移实验证明了p15G2563可以通过水平转移导致耐药性快速扩散。
本研究对该分离株进行了耐药性和耐药分子机制的研究,结果表明该分离株携带12类46个耐药基因,对阿奇霉素、多黏菌素、头孢曲松和环丙沙星等多种临床常用抗生素耐药。mphA和mcr-1基因的存在是该分离株分别对阿奇霉素和多黏菌素耐药的主要机制,且这两个基因均存在于p15G2563质粒上。p15G2563携带19个耐药基因和多个可转移元件,并且可以在肠杆菌目细菌之间水平转移。广泛耐药菌株及携带多个重要耐药基因的可转移质粒出现,给临床治疗带来了严峻挑战。因此,需进一步加强对鼠伤寒沙门菌的抗生素耐药特征和耐药谱变化的监测,并持续探索其耐药机制,特别是针对临床一线治疗药物的耐药性展开研究,这对于指导抗生素合理应用以及遏制细菌耐药性的不断蔓延具有重要意义。
参 考 文 献
Barton Behravesh C, Jones T F, Vugia D J, et al. Deaths associated with bacterial pathogens transmitted commonly through food: Foodborne diseases active surveillance network (FoodNet), 1996-2005[J]. J Infect Dis, 2011, 204(2): 263-267.
Simpson K M J, Hill-Cawthorne G A, Ward M P, et al. Diversity of Salmonella serotypes from humans, food, domestic animals and wildlife in New South Wales, Australia[J]. BMC Infect Dis, 2018, 18(1): 623.
余新华. 儿童腹泻患者沙门菌感染血清型分布及耐药性分析[J]. 中国现代药物应用, 2016, 10(1): 166-167.
Shen H, Chen H, Ou Y, et al. Prevalence, serotypes, and antimicrobial resistance of Salmonella isolates from patients with diarrhea in Shenzhen, China[J]. BMC Microbiol, 2020, 20(1): 197.
Kopper T J, Mcfarlane K E, Bailey W M, et al. Delayed azithromycin treatment improves recovery after mouse spinal cord injury[J]. Front Cell Neurosci, 2019, 13: 490.
Sj?lund-Karlsson M, Joyce K, Blickenstaff K, et al. Antimicrobial susceptibility to azithromycin among Salmonella enterica isolates from the United States[J]. Antimicrob Agents Chemother, 2011, 55(9): 3985-3989.
Biswas S, Brunel J M, Dubus J C, et al. Colistin: An update on the antibiotic of the 21st century[J]. Expert Rev Anti Infect Ther, 2012, 10(8): 917-934.
Landman D, Georgescu C, Martin D A, et al. Polymyxins revisited[J]. Clin Microbiol Rev, 2008, 21(3): 449-465.
Clinical and Laboratory Standards Institute (CLSI). CLSI M100S31 Performance standards for antimicrobial susceptibility testing; Thirty-one informational supplement[S]. 2021.
Wong M H, Yan M, Chan E W, et al. Emergence of clinical Salmonella enterica serovar Typhimurium isolates with concurrent resistance to ciprofloxacin, ceftriaxone, and azithromycin[J]. Antimicrob Agents Chemother, 2014, 58(7): 3752-3756.
Gurevich A, Saveliev V, Vyahhi N, et al. QUAST: quality assessment tool for genome assemblies[J]. Bioinformatics, 2013, 29(8): 1072-1075.
Seemann T. Prokka: rapid prokaryotic genome annotation[J]. Bioinformatics, 2014, 30(14): 2068-2069.
Larsen M V, Cosentino S, Rasmussen S, et al. Multilocus sequence typing of total-genome-sequenced bacteria[J]. J Clin Microbiol, 2012, 50(4): 1355-1361.
Jia B, Raphenya A R, Alcock B, et al. CARD 2017: Expansion and model-centric curation of the comprehensive antibiotic resistance database[J]. Nucleic Acids Res, 2017, 45(D1): D566-d573.
Campioni F, Souza R A, Martins V V, et al. Prevalence of gyrA mutations in nalidixic acid-resistant strains of Salmonella enteritidis isolated from humans, food, chickens, and the farm environment in Brazil[J]. Microb Drug Resist, 2017, 23(4): 421-428.
Ruiz J, Castro D, Go?i P, et al. Analysis of the mechanism of quinolone resistance in nalidixic acid-resistant clinical isolates of Salmonella serotype Typhimurium[J]. J Med Microbiol, 1997, 46(7): 623-628.
Zaidi M B, Leon V, Canche C, et al. Rapid and widespread dissemination of multidrug-resistant blaCMY-2 Salmonella typhimurium in Mexico[J]. J Antimicrob Chemother, 2007, 60(2): 398-401.
劉美玲, 李鑫鸽, 宁一桦, 等. 2008-2018年北京市人源鼠伤寒沙门菌的耐药与分子分型研究[J]. 疾病监测, 2022, 37(4): 523-530.
Darton T C, Tuyen H T, The H C, et al. Azithromycin resistance in Shigella spp. in Southeast Asia[J]. Antimicrob Agents Chemother, 2018, 62(4).
Nüesch-Inderbinen M, Heini N, Zurfluh K, et al. Shigella antimicrobial drug resistance mechanisms, 2004-2014[J]. Emerg Infect Dis, 2016, 22(6): 1083-1085.
Liu Y Y, Wang Y, Walsh T R, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: A microbiological and molecular biological study[J]. Lancet Infect Dis, 2016, 16(2): 161-168.
Mourand G, Jouy E, Chauvin C, et al. Dissemination of the mcr-1 colistin resistance gene among pigs: An experimental study[J]. Vet Microbiol, 2018, 221: 122-128.
Xie J, Liang B, Xu X, et al. Identification of mcr-1-positive multidrug-resistant Escherichia coli isolates from clinical samples in Shanghai, China[J]. J Glob Antimicrob Resist, 2022, 29: 88-96.
Liu G, Qian H, Lv J, et al. Emergence of mcr-1-harboring Salmonella enterica serovar sinstorf type ST155 isolated from patients with diarrhea in Jiangsu, China[J]. Front Microbiol, 2021, 12: 723697.
Falgenhauer L, Waezsada S E, Yao Y, et al. Colistin resistance gene mcr-1 in extended-spectrum β-lactamase-producing and carbapenemase-producing Gram-negative bacteria in Germany[J]. Lancet Infect Dis, 2016, 16(3): 282-283.
Gomes C, Martínez-Puchol S, Palma N, et al. Macrolide resistance mechanisms in Enterobacteriaceae: Focus on azithromycin[J]. Crit Rev Microbiol, 2017, 43(1): 1-30.
Gomes C, Ruiz-Roldán L, Mateu J, et al. Azithromycin resistance levels and mechanisms in Escherichia coli[J]. Sci Rep, 2019, 9(1): 6089.
Adelberg E A, Pittard J. Chromosome transfer in bacterial conjugation[J]. Bacteriol Rev, 1965, 29(2): 161-172.
Rychlik I, Gregorova D, Hradecka H. Distribution and function of plasmids in Salmonella enterica[J]. Vet Microbiol, 2006, 112(1): 1-10.
Hradecka H, Karasova D, Rychlik I. Characterization of Salmonella enterica serovar Typhimurium conjugative plasmids transferring resistance to antibiotics and their interaction with the virulence plasmid[J]. J Antimicrob Chemother, 2008, 62(5): 938-941.
Rozwandowicz M, Brouwer M S M, Fischer J, et al. Plasmids carrying antimicrobial resistance genes in Enterobacteriaceae[J]. J Antimicrob Chemother, 2018, 73(5): 1121-1137.
Zhang J F, Fang L X, Chang M X, et al. A Trade-off for maintenance of multidrug-resistant IncHI2 plasmids in Salmonella enterica Serovar Typhimurium through adaptive evolution[J]. mSystems, 2022, 7(5): e0024822.
Lian X, Wang X, Liu X, et al. oqxAB-positive IncHI2 plasmid pHXY0908 increase Salmonella enterica serotype Typhimurium strains tolerance to ciprofloxacin[J]. Front Cell Infect Microbiol, 2019, 9: 242.
Guo L, Zhao Y. Global spread and molecular characterization of CTX-M-producing Salmonella typhimurium isolates[J]. Antibiotics (Basel), 2021, 10(11).
Harmer C J, Moran R A, Hall R M. Movement of IS26-associated antibiotic resistance genes occurs via a translocatable unit that includes a single IS26 and preferentially inserts adjacent to another IS26[J]. mBio, 2014, 5(5): e01801-01814.
Poirel L, Kieffer N, Nordmann P. In vitro study of ISApl1-mediated mobilization of the colistin resistance gene mcr-1[J]. Antimicrob Agents Chemother, 2017, 61(7): e00127-17.
