‘凤丹’牡丹PoERF113基因的克隆及表达分析
2023-01-12魏祯祯宋程威郭丽丽郭琪侯小改
魏祯祯,宋程威,郭丽丽,郭琪,侯小改
‘凤丹’牡丹基因的克隆及表达分析
魏祯祯,宋程威,郭丽丽,郭琪,侯小改*
(河南科技大学农学院,河南 洛阳 471023)
为克隆‘凤丹’()花瓣衰老相关基因的序列,分析其编码蛋白性质,探索其在不同发育时期花瓣、不同组织和不同品种的表达模式及对乙烯的响应,采用RT–PCR技术,从‘凤丹’牡丹中克隆到花瓣衰老相关基因。该基因属于AP2/ERF家族,其开放阅读框(ORF)为636 bp,编码1个由211个氨基酸残基组成的蛋白,含有1个AP2保守结构域,不包含跨膜结构,蛋白相对分子质量约为53 000,理论等电点(pI)为5.13,为不稳定蛋白,具有亲水性;qRT–PCR分析结果表明,基因在露色期、初开期、半开期、盛开期、始衰期、衰败期均有表达,主要在‘凤丹’露色期的花瓣和叶片中表达;在早花‘凤丹’突变株系中表达量最高;基因在早花‘凤丹’突变株系与‘凤丹’中的序列相同,不存在碱基差异;基因对乙烯有响应,其表达量随处理时间和生育进程的延长呈逐渐升高的趋势,推测该基因可能与乙烯诱导后导致花衰老进程相关。
‘凤丹’牡丹;;基因克隆;表达分析
基因属于AP2/ERF转录因子家族ERF亚家族的B4类,编码的蛋白含有1个由58个基因组成的保守AP2结构域,在抵抗大豆疫霉、抵御非生物胁迫、调节花衰老、促进愈伤组织形成和根生长等植物发育过程中发挥重要的作用[1–3]。KHASKHELI等[4]在玫瑰中发现基因的表达受乙烯诱导,在萼片、花瓣、雄蕊和雌蕊等大多数花器官的衰老过程中表达量上调,采用VIGS技术使基因沉默,加速了玫瑰花的衰老,并且在花衰老过程中细胞分裂素含量呈降低的趋势,与细胞分裂素有关9个基因(),(),,(),,(),,()和的表达水平也随之降低,但这种衰老过程可以通过外源细胞分裂素处理得到恢复。
牡丹(Andr.)是芍药科(Paeoniaceae)芍药属()植物,原产于中国,是传统观赏植物,被广泛栽植,观赏价值高,市场需求量大[5–6]。目前关于牡丹花期调控的研究多注重于通过栽培、生长调节剂等手段延长花期,对其分子机制的研究不多[7–10]。本研究以‘凤丹’花瓣为试验材料,克隆获得‘凤丹’基因序列,采用生物信息学技术对PoERF113蛋白的理化性质、保守结构域、磷酸化位点、蛋白质高级结构等进行预测分析,运用实时荧光PCR技术研究基因的时空表达模式与不同花期、不同组织和不同品种的相关性及对乙烯的响应,并对基因在早花‘凤丹’突变株系与‘凤丹’中的基因序列进行比对,以期为后续基因的相关功能研究提供理论依据。
1 材料与方法
1.1 植物材料
九年生早花‘凤丹’种植于河南科技大学实验园区内;早花‘凤丹’突变株系种植于洛阳隋唐植物园;晚花‘莲鹤’种植于洛阳国际牡丹园。
1.2 试验方法
1.2.1总RNA提取及cDNA的合成
取‘凤丹’叶片、茎、花萼和不同时期(露色期、初开期、半开期、盛开期、始衰期、衰败期)的花瓣和用10 mg/L外源乙烯整株喷施圆桃期‘凤丹’后第1、2、4、8天的花瓣以及早花‘凤丹’突变株系、‘莲鹤’半开期的花瓣,采用天根RNAprep Pure多糖多酚植物总RNA提取试剂盒(离心柱型)完成总RNA的提取;采用PrimeScript™ II 1st Strand cDNA Synthesis Kit获取cDNA。
1.2.2‘凤丹’基因的克隆
从前期得到的‘凤丹’三代全长转录组数据库筛选出基因序列,利用Primer Premier 5.0设计基因克隆及实时荧光定量PCR特异性引物(表1)。引物由生工生物工程(上海)股份有限公司合成。50 μL PCR扩增体系包括2 μL cDNA模板、1 μL TransStart FastPfu DNA Polymerase、10 μL 5×Trans Start FastPfu Buffer、4 μL dNTP Mixture(2.5 mmol/L)、2 μL 引物–F和2 μL–R(10 μmol/L)、29 μL ddH2O。反应程序为95 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,33个循环;72 ℃延伸7 min。基因PCR产物经1%琼脂糖凝胶电泳检测,采用AxyPrep DNA凝胶回收试剂盒回收目的条带,与pMD18–T Vector载体连接后转至大肠埃希菌DH5α感受态细胞中,利用M13引物进行普通PCR检测,筛选出阳性单克隆送至生工生物工程(上海)股份有限公司测序。
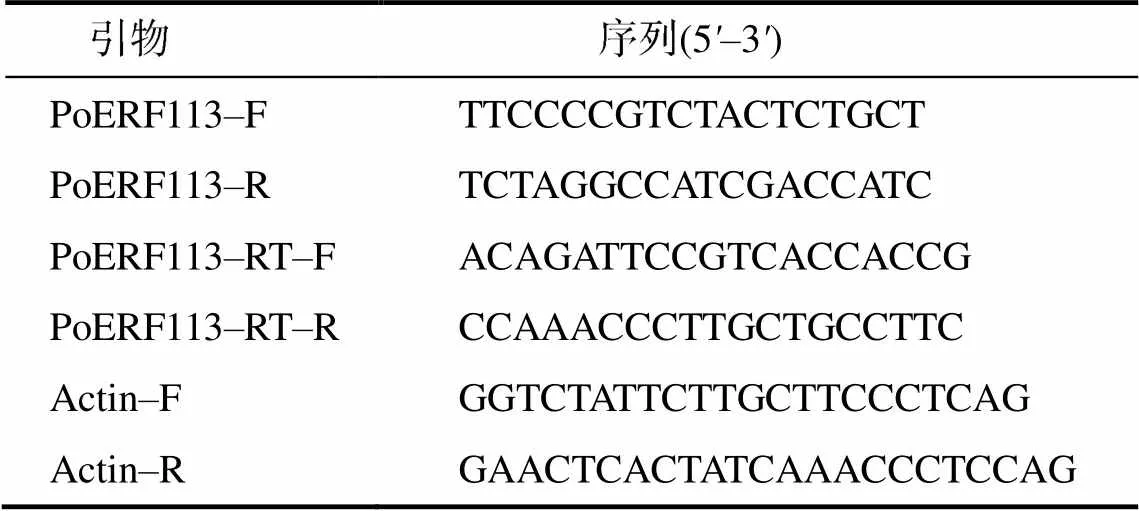
表1 PoERF113基因克隆及qRT–PCR分析引物
1.2.3‘凤丹’基因生物信息学分析
利用Translate(http://web.expasy.org/translate/)对基因推导的氨基酸序列进行分析;根据基因的开放阅读框(open reading frame,ORF),运用ProtParam(https://web.expasy.org/ protparam/)分析PoERF113蛋白特性理化性质;运用在线软件(http://www.ncbi.nlm.nih.gov/Structure/ cdd/wrpsb.cgi)分析PoERF113蛋白保守结构域;运用Protscale(https://web.expasy.org/protscale/)分析PoERF113蛋白亲疏水性;运用NetPhos(http://www. cbs.dtu.dk/services/NetPhos/)分析PoERF113蛋白磷酸化位点;运用TMHHMM(http://www.cbs.dtu. dk/services/TMHMM–2.0/)分析PoERF113蛋白跨膜结构;运用SPOMA(https://npsa–prabi.ibcp.fr/cgi– bin/secpred_sopma.pl)和SWISS–MODEL(https:// www.swissmodel.expasy.org/interactive)预测蛋白质的二级和三级结构。
1.2.4基因的表达模式分析
根据测序所得的基因的ORF序列,通过Primer Premier 5.0设计特异性引物,进行qRT–PCR试验。以基因为内参基因,使用宝生物工程(大连)有限公司的实时荧光定量试剂盒(TB GreenTM Premix Ex Taq II) 检测基因的表达量,每个样品设置3个生物学重复。
1.2.5数据分析
采用2–ΔΔCt法计算基因的相对表达量[11];运用Excel 2010进行数据整理与分析;运用SPSS 19.0进行统计分析;运用Origin 8.0绘图。
2 结果与分析
2.1 ‘凤丹’PoERF113基因的克隆及推导的氨基酸序列
提取‘凤丹’花瓣的总RNA,经凝胶电泳检测,结果显示条带明亮、清晰,28S带的亮度约为18S带亮度的1.5~2.0倍,RNA质量较好(图1)。

M DL2000 DNA Marker;1 总RNA。
以‘凤丹’花瓣为材料提取花瓣组织总RNA,反转录后获得cDNA,利用特异性引物PoERF113–F和PoERF113–R进行扩增,凝胶电泳检测结果(图2)显示:在约750 bp处发现1条清晰的特异性条带,与目的片段预测条带大小一致,该基因长706 bp。通过NCBI(National Center for Biotechnology Information)在线工具对其包含的开放阅读框进行分析,ORF为636 bp,编码211个氨基酸残基,ORF序列及推导的氨基酸序列如图3所示。

M DL2000 DNA Marker;1 PoERF113基因。

图3 ‘凤丹’PoERF113基因推导的氨基酸序列
2.2 ‘凤丹’PoERF113蛋白的预测分析
2.2.1‘凤丹’PoERF113蛋白特性分析及保守结构域预测
利用ProtParam对候选基因进行蛋白特性分析,结果表明该蛋白的相对分子质量为53 000,理论等电点(pI)为5.13,脂肪系数为28.93,不稳定系数为52.83,大于40,表明该蛋白为不稳定蛋白。
利用NCBI中CDD(cyclic delay diversity,循环延迟分集)保守结构域数据库对PoERF113蛋白进行保守结构域预测。结果(图4)表明,该基因编码的蛋白具有AP2超家族结构域和特殊的DNA结合位点,属于AP2家族一员。

图4 PoERF113基因编码蛋白保守结构域预测结果
2.2.2‘凤丹’PoERF113蛋白亲/疏水性预测和跨膜结构域分析
运用Protscale对PoERF113蛋白进行亲疏水性分析,结果(图5)显示,疏水氨基酸(正值)少于亲水氨基酸(负值),表明该蛋白为亲水性蛋白。

图5 PoERF113蛋白亲疏水性预测分析结果
通过TMHMM对基因进行跨膜结构域分析,结果(图6)表明,该基因编码蛋白的1~231位氨基酸定位于细胞膜表面,推测PoERF113蛋白可能不包含跨膜结构。
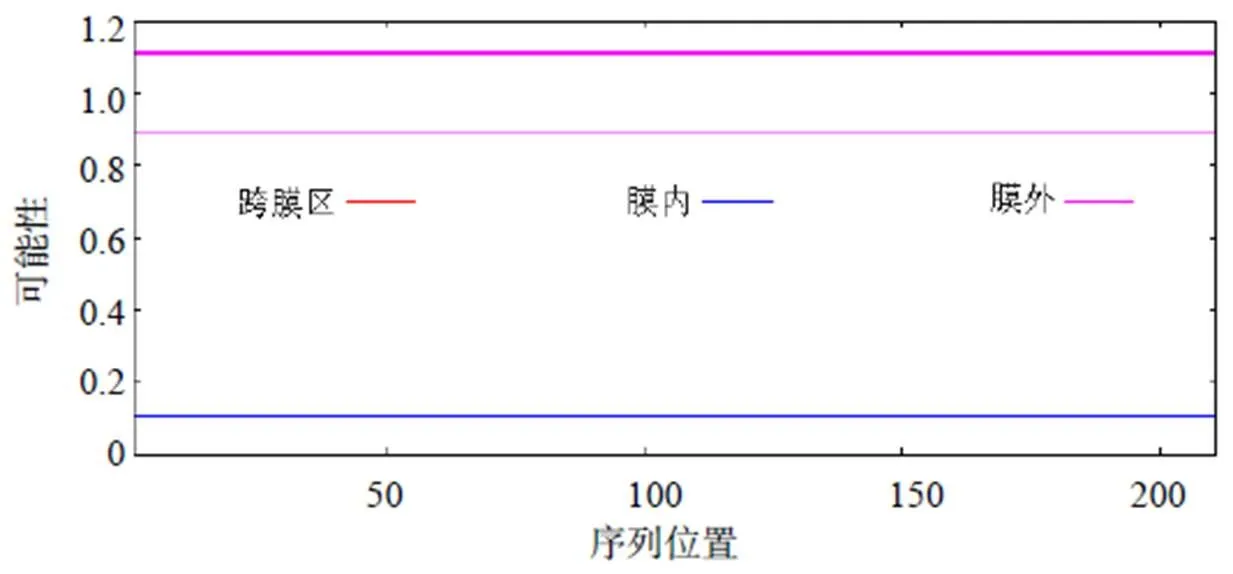
图6 PoERF113蛋白跨膜结构分析结果
2.2.3‘凤丹’PoERF113蛋白磷酸化位点分析
磷酸化主要集中在Tyr(酪氨酸)、Ser(丝氨酸)、Thr(苏氨酸)残基上,这些残基上具有游离的羟基,且本身不带电荷,磷酸化后的蛋白质具有电荷,引起结构和蛋白质活性的变化。NetPhos预测结果(图7)表明,基因编码的氨基酸序列中有19个Ser位点、10个Thr位点、4个Tyr位点。

图7 PoERF113蛋白磷酸化位点分析结果
2.2.4‘凤丹’PoERF113蛋白高级结构预测分析
运用SOPMA预测蛋白质二级结构,结果(图8)显示,PoERF113蛋白序列的二级结构中α–螺旋包含54个氨基酸,延伸链包含29个氨基酸,β–转角包含12个氨基酸,无规则卷曲包含116个氨基酸。如图9所示,PoERF113蛋白的三级结构与二级结构的预测结果保持一致。

图8 PoERF113蛋白二级结构预测结果
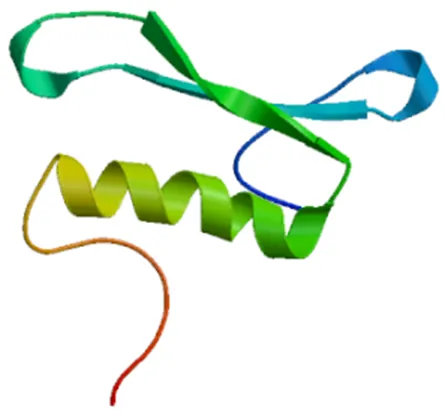
图9 PoERF113蛋白三级结构预测结果
2.3 ‘凤丹’PoERF113蛋白系统进化树分析
将基因的氨基酸序列上传至NCBI数据库中,采用bBLAST x算法,选择10条同源性较高的氨基酸序列进行多序列比对分析并构建系统进化树,这10条序列分别为美国山核桃(,KAG2678378.1),杨梅(,KAB1217539.1),大叶栎(,XP 030937739.1),苹果(,XP 017183622.1),非洲相思子(,XP 027360534.1),葡萄(,CBI15759.3),藤堤(,XP 034672620.1),南瓜(,XP 021622666.1),菠菜(,XP 021844726.1),藜麦(,XP 021731943.1),利用MEGA X软件分析‘凤丹’牡丹基因与其他物种的进化关系,比对结果如图10所示。从图10可以看出,‘凤丹’基因与南瓜、菠菜、葡萄的亲缘关系较近,与美国山核桃、杨梅等植物亲缘关系较远。

图10 ‘凤丹’PoERF113蛋白构建系统进化树分析结果
2.4 ‘凤丹’PoERF113基因表达模式分析
为确定基因在‘凤丹’牡丹花发育不同时期花瓣中的表达情况,利用荧光定量PCR技术,以基因为内参基因,测定其在露色期、初开期、半开期、盛开期、始衰期、衰败期花瓣的相对表达量。结果(图11)表明,基因在‘凤丹’牡丹各个花发育阶段中均有表达,且不同花期的相对表达量存在差异。在露色期花瓣的相对表达量最高,初开期花瓣中的相对表达量最低。

柱上不同字母示处理间的差异有统计学意义(P<0.05)。
为分析基因在组织中的表达模式,选取同时期叶片、茎、花萼和花瓣进行qRT–PCR,以‘凤丹’牡丹的花瓣为对照。结果(图12)表明,基因在‘凤丹’叶片、茎、花萼和花瓣中均有表达,在 ‘凤丹’叶片的基因的表达量极显著高于茎、花萼和花瓣中的表达量,说明基因主要在叶片中表达。

柱上不同字母示处理间的差异有统计学意义(P<0.05)。
为明确基因的表达特性,利用qRT–PCR技术对早花‘凤丹’突变株系、早花‘凤丹’和晚花‘莲鹤’中基因的表达进行分析。结果(图13)表明,基因在早花‘凤丹’、晚花‘莲鹤’的花瓣中均有表达。在早花‘凤丹’突变株系的相对表达量最高,约为‘凤丹’的相对表达量的4.88倍,约为‘莲鹤’的相对表达量的6.54倍。

柱上不同字母示处理间的差异有统计学意义(P<0.05)。
采用10 mg/L的外源乙烯处理‘凤丹’牡丹花瓣,发现基因的表达量随处理时间的延长逐渐上升(图14),且表达量存在显著差异性。基因在处理第8天时相对表达量最高,约为处理第1天的10倍,表明基因能被乙烯诱导表达,推测基因可能参与了乙烯信号传导途径。

柱上不同字母示处理间的差异有统计学意义(P<0.05)。
2.5 早花‘凤丹’突变株系及‘凤丹’PoERF113基因序列比对
为进一步探究基因在花期调控中的功能,以早花‘凤丹’突变体花瓣cDNA为模板,采用相同的引物与扩增条件进行RT–PCR扩增,利用DNAMAN 6.0对基因在自然状态下‘凤丹’和早花‘凤丹’突变株系中的序列进行比对。结果表明,早花‘凤丹’突变株系基因与‘凤丹’的序列不存在差异,2条序列的ORF框完全相同。
3 结论与讨论
APETALA2/ERF(Apetala2/Ethylene–responsive factor,AP2/ERF)转录因子家族作为最早被鉴定与乙烯响应基因相结合的转录因子,具有高度保守的AP2/ERF结构域,且在植物生长和发育中发挥着不同的功能[12–14]。研究人员在拟南芥、桃、苹果等多种植物进行了全基因组的鉴定及相应功能的研究,在拟南芥中鉴定出122个AP2/ERF家族成员[15],在桃中鉴定出131个AP2/ERF家族成员[16],在苹果中鉴定出259个AP2/ERF家族成员[17],在柑橘中鉴定出126个AP2/ERF家族成员[18]。
本研究通过克隆得到‘凤丹’牡丹基因,并对该基因编码的氨基酸序列进行了一系列生物信息学分析,发现该基因属于AP2/ERF家族,编码蛋白含有1个AP2保守结构域,不包含跨膜结构,为不稳定蛋白,与易萍等[19]鉴定出的芒果基因特征相符。基因在露色期、初开期、半开期、盛开期、始衰期、衰败期均有表达,但主要在露色期的花瓣中表达,与玫瑰基因在开花的第五个阶段(花朵完全绽放)表达量达到高峰的表达模式不同[4]。前人研究结果表明,同源基因在不同物种中的时空表达既有共性又有差异。蜻蜓凤梨基因在外叶、心叶、茎、根中均有表达,但表达量较低,成熟的根中的表达量高于幼根,在花器官中表达量维持在较低水平[20]。本试验结果显示,基因在‘凤丹’的叶片、花瓣、茎、萼片中均有表达,在叶片中的相对表达量最高,在茎中的相对表达量最低,这暗示基因可能主要调控叶片的生长发育。
花衰老是一个高度程序化的过程,由多方面因素共同作用,伴随着植物体内内源激素、自由基代谢、细胞内结构解体和膜系统的降解等一系列生理生化过程[21]。植物激素可以调节植物开花进程,乙烯、茉莉酸和脱落酸作为植物花衰老促进剂,生长素和细胞分裂素类激素发挥抑制花衰老的作用[22]。乙烯在植物生长和衰老中起重要作用,在花衰老期间,乙烯的积累成为促进植物衰老的关键步骤[23]。研究表明,外源乙烯处理‘洛阳红’切花可以促进其内源乙烯的生成[24];对于乙烯依赖型植物,内源乙烯的生成将导致植物花衰老[25];对于乙烯不依赖型植物,外源乙烯的应用不会引导其产生大量的内源乙烯,但也可以促进花衰老[26]。研究表明,拟南芥、蜻蜓凤梨等多个物种的同源基因均响应乙烯处理。外源乙烯处理6~24 h内,拟南芥基因表达量呈逐渐上升的趋势[2]。外源乙烯处理1 h后,蜻蜓凤梨基因在幼株和成株外叶、心叶、茎中表达量上升,响应乙烯诱导[20]。本研究中用乙烯喷施‘凤丹’植株,基因对乙烯有所响应,处理后,基因表达量升高,这与AGUSTÍ等[27]在柑橘()中的研究结果类似。推测该基因可能与乙烯诱导后导致花衰老进程相关。
[1] KONG X P,TIAN H Y,YU Q Q,et al.PHB3maintains root stem cell niche identity through ROS-responsive AP2/ERF transcription factors in[J].Cell Reports,2018,22(5):1350–1363.
[2] KRISHNASWAMY S,VERMA S,RAHMAN M H,et al.Functional characterization of four APETALA2- family genes(RAP2.6,RAP2.6L,DREB19 and DREB26) in[J].Plant Molecular Biology,2011,75(1/2):107–127.
[3] SUN F,LIU P Q,XU J,et al.Mutation in RAP2.6L,a transactivator of the ERF transcription factor family,enhancesresistance to[J]. Physiological and Molecular Plant Pathology,2010,74(5/6):295–302.
[4] KHASKHELI A J,AHMED W,MA C,et al.RhERF113 functions in ethylene-induced petal senescence by modulating cytokinin content in rose[J].Plant and Cell Physiology,2018,59(12):2442–2451.
[5] 李嘉珏.中国牡丹起源的研究[J].北京林业大学学报,1998,20(2):22–26.
[6] 洪德元,潘开玉.芍药属牡丹组的分类历史和分类处理[J].植物分类学报,1999,37(4):351–368.
[7] 李婷婷,薛璟祺,王顺利,等.秋发牡丹远缘杂交新材料筛选及系统优化[J].分子植物育种,2019,17(8):2761–2768.
[8] 岳高峰,韩志强,马俊强,等.不同遮阴处理对牡丹花期及花色品质的影响[J].湖北农业科学,2020,59(17):83–86.
[9] 黄金锋,陈学湘,刘菲.外源激素对牡丹花期延迟试验[J].山东林业科技,2016,46(6):22–25.
[10] 石丰瑞.牡丹长日照途径关键基因以及其上、下游基因、的克隆与表达分析[D].北京:中国农业科学院,2013.
[11] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCtmethod[J].Methods,2001,25(4):402–408.
[12] DUBOUZET J G,SAKUMA Y,ITO Y,et al.genes in rice,L.,encode transcription activators that function in drought-,high-salt- and cold- responsive gene expression[J].Plant & Cell Physiology,2003,33(4):751–763.
[13] MIZOI J,SHINOZAKI K,YAMAGUCHI-SHINOZAKI K. AP2/ERF family transcription factors in plant abiotic stress responses[J].Biochimica et Biophysica Acta(BBA) -Gene Regulatory Mechanisms,2012,1819(2):86–96.
[14] GU Y Q,YANG C,THARA V K,et al.is induced by ethylene and salicylic acid,and its product is phosphorylated by the Pto kinase[J].The Plant Cell,2000,12(5):771–786.
[15] NAKANO T,SUZUKI K,FUJIMURA T,et al. Genome-wide analysis of thegene family inand rice[J].Plant Physiology,2006,140(2):411–432.
[16] ZHANG C H,SHANGGUAN L F,MA R J,et al. Genome-wide analysis of the AP2/ERF superfamily in peach()[J].Genetics and Molecular Research,2012,11(4):4789–4809.
[17] GIRARDI C L,ROMBALDI C V,DAL CERO J,et al. Genome-wide analysis of thesuperfamily in apple and transcriptional evidence of ERF involvement in scab pathogenesis[J].Scientia Horticulturae,2013,151:112–121.
[18] XIE X L,SHEN S L,YIN X R,et al.Isolation,classification and transcription profiles of the AP2/ERF transcription factor superfamily in citrus[J].Molecular Biology Reports,2014,41(7):4261–4271.
[19] 易萍,黄方,李丽,等.芒果乙烯响应转录因子基因的克隆及表达分析[J].热带作物学报,2022,43(9):1751–1758.
[20] 雷明,李志英,王加宾,等.蜻蜓凤梨基因的克隆与表达特性[J].西北农业学报,2016,25(12):1851–1860.
[21] PARVEEN S,ALTAF F,FAROOQ S,et al.Is proline the quintessential sentinel of plants? A case study of postharvest flower senescence inL.[J].Physiology and Molecular Biology of Plants,2021,27(7):1597–1607.
[22] VAN DOORN W G,ÇELIKEL F G,PAK C,et al.Delay offlower senescence by cytokinins and jasmonates[J]. Physiologia Plantarum,2013,148(1):105–120.
[23] ZHANG H S,ZHOU C J.Signal transduction in leaf senescence[J].Plant Molecular Biology,2013,82(6):539–545.
[24] 周琳,贾培义,刘娟,等.乙烯对‘洛阳红’牡丹切花开放和衰老进程及内源乙烯生物合成的影响[J].园艺学报,2009,36(2):239–244.
[25] SHIBUYA K.Molecular mechanisms of petal senescence in ornamental plants[J].Journal of the Japanese Society for Horticultural Science,2012,81(2):140–149.
[26] SHIBUYA K.Molecular aspects of flower senescence and strategies to improve flower longevity[J].Breeding Science,2018,68(1):99–108.
[27] AGUSTÍ J,MERELO P,CERCÓS M,et al.Comparative transcriptional survey between laser-microdissected cells from laminar abscission zone and petiolar cortical tissue during ethylene-promoted abscission in citrus leaves[J]. BMC Plant Biology,2009,9:127.
Cloning and expression analysis ofgene in‘Fengdan’
WEI Zhenzhen,SONG Chengwei,GUO Lili,GUO Qi,HOU Xiaogai*
(College of Agriculture, Henan University of Science and Technology, Luoyang, Henan 471023, China)
To investigate the petal senescence-related geneof ‘Fengdan’ () expression patterns in petals, different tissues and different varieties at different flowering stages and their response to ethylene, we did cloning analyzed its coded protein properties in this study. First, we used RT-PCR technology to clone thepetal senescence related genegene from ‘Fengdan’. The gene belongs to AP2/ERF family. Its open reading frame(ORF) is 636 bp, encoding a protein consisting of 211 amino acid residues. It contains one AP2 conservative domain and does not contain transmembrane structure. The molecular weight of the protein is about 53 000, and the theoretical isoelectric point (pI) is 5.13. It is unstable protein and hydrophilic. The results of qRT-PCR analysis showed thatgene was expressed in the open color stage, the first open stage, the half open stage, the full bloom stage, the first decline stage, and the decline stage, mainly in the petals and leaves of ‘Fengdan’ in the open color stage; The highest expression was found in ‘Fengdan’ mutant strain of Zaohua; The sequence ofgene in the ‘Fengdan’ mutant line of Zaohua was the same as that in ‘Fengdan’ and there was no base difference;gene was responsive to ethylene, and its expression level gradually increased with the extension of treatment time and growth process. It was speculated that this gene might be related to the process of flower senescence induced by ethylene. This experiment laid a preliminary foundation for further study on the function ofgene and its molecular mechanism in peony petal senescence and florescence regulation.
‘Fengdan’;; gene cloning; expression analysis
Q786;S685.11
A
1007-1032(2022)06-0659-07
魏祯祯,宋程威,郭丽丽,郭琪,侯小改.‘凤丹’牡丹基因的克隆及表达分析[J].湖南农业大学学报(自然科学版),2022,48(6):659–665.
WEI Z Z,SONG C W,GUO L L,GUO Q,HOU X G.Cloning and expression analysis ofgene in‘Fengdan’[J].Journal of Hunan Agricultural University(Natural Sciences),2022,48(6):659–665.
http://xb.hunau.edu.cn

2022–10–27
2022–11–06
国家自然科学基金项目(U1804233);河南省创新型科技人才队伍建设工程(202101510003);河南省中药材产业技术体系(2021–24)
魏祯祯(1996—),女,河南郑州人,硕士研究生,主要从事牡丹抗旱栽培与分子生物学研究,z35089658@163.com;*通信作者,侯小改,博士,教授,主要从事牡丹栽培生理生态与分子生物学研究,hkdhxg@haust.edu.cn
10.13331/j.cnki.jhau.2022.06.005
责任编辑:毛友纯
英文编辑:柳正
