塔吉克斯坦地区自然发酵牛乳中乳酸菌的分离鉴定及多样性分析
2023-01-12刘琛丛琳刘文俊张和平
刘琛,丛琳,刘文俊,张和平*
(1 内蒙古农业大学 呼和浩特0100182 乳品生物技术与工程教育部重点实验室 呼和浩特010018 3 乳酸菌与发酵乳制品省部共建协同创新中心 呼和浩特 010018)
塔吉克斯坦坐落于中亚,地处北温带(北纬36°40' 至41°05'),其境内多山地高原,为典型的内陆高山国家[1]。其国内气候温和,雨水量充足,水草肥美,良好的气候环境造就了天然牧场,农业和畜牧业的发展占据了主导地位,而居民生活多为半定居、半游牧,沿袭着古老而传统的生产、生活方式。良好的气候环境和原生态生活方式使得酸牛乳、奶疙瘩、奶皮子等传统发酵乳制品在当地人一日三餐中占重要比例[2-3]。事实上,每个国家都有自己独特的饮食习俗以及特有的食品种类和制作方法[4-5]。不同地区的发酵乳制品都具有悠久的历史,像是一个个“天然的菌种宝库”,成为研究不同地域饮食中微生物多样性的良好材料。
乳酸菌分类鉴定的方法分为传统纯培养生理生化分类鉴定法和非培养分子生物学鉴定法。传统纯培养方法通过生理及表型鉴定,所得研究结果准确性较低,误差较难避免,而且费时、费力。而非传统的分子生物学方法可以提取样品中微生物宏基因组,利用菌株基因组序列之间的特异性对菌群结构、丰度、多样性进行研究。由于其可高效、快速、全面再现生境中微生物菌群等优势,因此基于宏基因测序的方法在乳酸菌分类鉴定中广泛应用[6-7]。通过纯培养的方法分离纯化得到样品中微生物并作鉴定,然后利用非培养的方法分析其多样性,两者相互补充,是分析微生物群落结构和多样性的科学方法。
塔吉克斯坦地理条件和气候条件独特,乳制品资源丰富。当地的游牧民族长期饮用的发酵牛乳是以新鲜牛乳为原料自然发酵而成的。其发酵过程大都不需添加商品发酵剂,而是用前1 d 的酸乳作为“引子”[8]。由于保持了相对原始的发酵过程,样品中乳酸菌的生物学特性和菌种的多样性得到很好地留存,这些丰富的乳酸菌资源有待于开发和利用。
本研究采用实验室纯培养方法结合16S rRNA 基因序列分析,对塔吉克斯坦地区自然发酵牛乳中的乳酸菌进行分离纯化及鉴定,为优良菌株的筛选提供资源。应用PacBio SMRT 测序技术揭示自然发酵牛乳中的细菌群落组成,研究该地区自然发酵牛乳中乳酸菌群落结构和多样性。
1 材料与方法
1.1 样品的采集
7 份自然发酵牛乳样品均采自塔吉克斯坦地区牧民家庭,编号为TJ1~TJ7。每个样品采集一式两份,均使用50 mL 无酶无菌管,每个样品采集20 mL,一份加入样品保护剂(无菌的碳酸钙0.1 g和淀粉5 g ),另一份加入15 mL DNA 保护剂(TaKaRa),样品混匀后封紧管口,立即置于低温冰箱(4 ℃)中,送至实验室进行后续分析。
1.2 主要试剂及仪器
MRS 和M17 培养基,英国Qxoid 公司;琼脂,北京康倍斯公司;TIANamp Bacteria DNA Kit(细菌基因组DNA 提取试剂盒),天根生化科技有限公司;Dneasy Power Food Kit Dneasy(能量食品试剂盒),德国QIAGEN 公司;KAPA HiFi Hot-StartReadyMix PCR Kit(卡帕高通量聚合酶链试反应试剂盒),美国KAPA 公司;PCR 所用试剂,上海派森诺生物科技有限公司。
微量紫外分光光度计,美国Nanodrop 公司;PCR 仪(PTC-200 型),美国Life Technologies 公司;显微镜(CX33),日本OLYMPUS 公司;电泳仪(DYY-12 型),北京六一仪器厂;凝胶成像分析仪(CDS8000 型),美国UVP公司;三代测序仪(PacBio RS II),美国Pacific Biosciences 公司。
1.3 乳酸菌的分离与鉴定
将样品充分混合均匀后采用十倍梯度稀释法[9]。吸取10-5,10-6,10-73 个稀释梯度的稀释液,7个样品3 个梯度各200 μL 均匀涂布于MRS 和M17 固体培养基(琼脂1.2 g/100 mL)上,将固体培养基平板倒置于37 ℃厌氧培养箱中培养24~48 h。菌落形成后,观察所有菌落的形态,如菌落颜色、大小、边缘状态等并详细记录,挑取形态特征不同的单个菌落于相应的MRS 和M17 固体培养基上,采用平板划线法依据实际情况进行2~3 次划线纯化[10]。当平板上出现单个菌落后,使用无菌接种环挑取菌落分别接种到MRS 和M17 的液体培养基中,培养条件如上文所述。对所有菌株进行过氧化氢酶活性检测,以及将菌株革兰氏染色后显微镜观察,对结果为过氧化氢阴性(无气泡产生)且革兰氏染色阳性(显微镜观察菌株呈现紫色)的菌株进行再培养,并冷冻干燥保藏,以备进一步鉴定。
采用试剂盒法提取菌株DNA,先加入溶菌酶(质量浓度50 mg/mL)菌株进行细胞壁破壁处理,后续的裂解、漂洗、裂解等DNA 提取步骤均参考细菌基因组DNA 提取试剂(离心柱型)盒说明书。获得菌株的基因组DNA 后,用0.8%琼脂糖凝胶电泳检测,浓度、纯度均符合要求后方可进行后续试验。
使用细菌通用扩增引物,正向引物和方向引物【正向:27F(5'-AGAGTTTGATCMTGGCTCAG-3'),反向:1492R(5'-ACCTTGTTACGACTT-3')】,将DNA 作为扩增模板进行扩增。扩增反应体系为50 μL 的PCR 反应,扩增条件是:预变性5 min(94 ℃);变性1 min(94 ℃,30 个循环),退火1 min(58 ℃,30 个循环),延伸2 min(72 ℃,30 个循环);末端延伸10 min(72 ℃)[11]。将电泳条带明亮,且无拖尾现象的PCR 产物在低温条件下送往上海派森诺生物公司进行纯化和双向测序,测序得到的序列在NCBI 网站(https://www.ncbi.nlm.nih.gov/)上和已公布的菌株序列进行同源性比对,运用MEGA7.0 软件,采用邻接法(Neighbor-Joining)构建菌株进化树。
1.4 酸牛乳中细菌宏基因组DNA 的提取
使用Dneasy 能量食品试剂盒,2 mL tubes(100)抽提样品宏基因组DNA,其步骤参照试剂盒说明书[12]。用ND-1000 紫外分光光度计和1.0%琼脂糖凝胶电泳检验提取DNA 的浓度、纯度与完整度,将符合条件的DNA 置低温冰箱(-20 ℃)保存备用。
1.5 细菌16S rRNA 基因序列全长扩增
PCR 扩增所需的试剂盒为KAPA HiFi Hot-Start Ready Mix PCR Kit,扩增反应体系(50 μL):10 μmol/L 16S rRNA 基因序列通用引物(同上述1.3 节)各1.5 μL,DNA 模板1.5 μL,KAPA Mix 4 种碱基混合液25.0 μL,ddH2O 20.5 μL。扩增条件:95 ℃预变性3 min;98 ℃变性20 s(30 个循环),60 ℃退火15 s(30 个循环),72 ℃延伸30 s(30 个循环);72 ℃末端延伸2 min[13]。
1.6 PacBio SMRT 测序及数据分析
对原始的测序数据进行质量控制,采用SMRT portal(v2.7)中RS ReadsOfinsert.1,测序时DNA 插入片段应在1 400~1 800 nt 之间。将满足要求的高质量序列根据每个样品的Barcode 信息做去除Barcode 处理,用QIIME 平台对符合要求的高质量序列进行微生物组分析。同时,吉尔吉斯斯坦和摩洛哥的自然发酵酸牛乳数据集也做相同的处理。利用PyNAST 软件[14]对齐序列,通过UCLUST 软件[15]以100%的相似度划分序列,建立可操作分类单元(Operational taxonomic units,OTU)。利用FastTree 软件[16]绘制基于OTU 代表序列的系统发育进化树,对数据进行计算分析后,用Mann-Whitny/Kruskal-Wallis 检验统计不同地区酸牛乳菌落结构的差异性,应用R 软件和Origin2017 等软件对QIIME 分析的微生物组数据进行可视化处理。
2 结果与分析
2.1 塔吉克斯坦酸牛乳中乳酸菌的分离及鉴定
从塔吉克斯坦采集的7 份酸牛乳样品中共分离得到93 株菌,根据过氧化氢酶阴性(观察到无氧气气泡产生)和革兰氏阳性(显微镜中菌株呈紫色)的表型结果,筛选得到疑似乳酸菌92 株。
通过16S rRNA 基因测序和BLAST 比对,从种水平上对分离株进行鉴定,确认92 株分离菌株为乳酸菌。使用MEGA7.0 软件将部分分离株与模式株的16S rRNA 基因序列绘制成乳酸菌系统发育进化树(图1)。经鉴定,93 株分离菌株;乳杆菌属有德氏乳杆菌(83 株),植物乳杆菌(Lactobacillus plantarum)(3 株),短乳杆菌(Lactobacillus brevis)(1 株),肠球菌属有粪肠球菌(Enterococcus faecium)(3 株),耐久肠球菌(Enterococcus duran)(1 株)和屎肠球菌(Enterococcus faecalis)(1 株),葡萄球属仅1 株人葡萄球菌(Staphylococcus hominis)(1 株)。
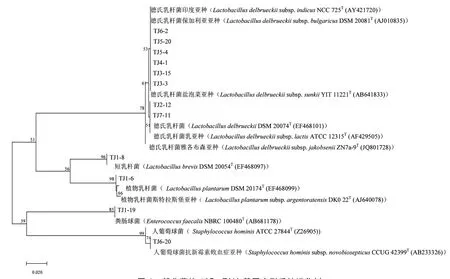
图1 部分菌株16S rRNA 基因序列系统进化树Fig.1 Phylogenetic relationship of 16S rRNA gene sequences of partial isolates
2.2 塔吉克斯坦酸牛乳的细菌多样性
应用PacBio SMRT 三代测序技术,对7 份塔吉克斯坦酸牛乳样品进行测序,最后共获得25 164 条高质量完整的基因序列,平均每个样品高质量序列3 595 条。经97%的相似度划分OTU,对获得的1 838 个OTU 进行分析,平均每个样品的OTU 数量为262 个,其中序列最多的样品为TJ3(5 008 个),序列最少的样品为TJ7(915个)。各样品具体序列数和OTU 数量见表1。

表1 自然发酵乳样品的测序信息Table 1 Sequences information of naturally fermented milk samples
图2a 中稀疏曲线表示未达到平衡仍呈上升趋势,测序深度会继续增加,同时样品中的物种数也会增加,说明随着测序深度的增加还会有新的物种被发现。图2b 中香农曲线达到平衡,香农多样性指数达饱和,表明此深度下测序,样品中的细菌的多样性已充分展现。本研究的测序深度已满足后续分析要求。

图2 香农多样性图(a)和稀疏曲线图(b)Fig.2 Shannon diversity index curves(a)and rarefaction curves(b)
2.3 塔吉克斯坦酸牛乳的细菌组成
利用PacBio SMRT 技术对塔吉克斯坦地区酸牛乳样品进行测序,采用RPD 和Greengenes 数据库进行序列比对,结果共鉴定到6 个细菌属和15 个细菌种。在属水平上鉴定到的6 个细菌属,分别是芽孢杆菌属(Bacillus)、拟杆菌属(Bacteroides)、肠球菌属(Enterococcus)、乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)、乳球菌属(Lactococcu)。不同样品细菌属水平群落组成如图3 所示。其中,酸牛乳样品中相对含量较高的为乳杆菌属,含量91.7%,其次为链球菌属,含量8.1%,其中乳球菌属极少,含量仅0.01%。7 份所测样品中,TJ4 样品菌种丰富度高,所有6 个菌属均检测到;乳杆菌属和链球菌属在所有样品中均可检出,其含量均在70%以上;链球菌属仅在TJ2 和TJ4 样品中检出,含量未达到1%。

图3 自然发酵乳在属水平上的相对丰富度和多样性Fig.3 Relative abundances and bacterial diversity of the microbiota of naturally fermented milk at the genus levels
在种水平上鉴定到15 个细菌种,包含7 个链球菌,4 个乳杆菌,耐久肠球菌等。不同样品细菌种水平群落组成如图4 所示。德氏乳杆菌、嗜热链球菌(Streptococcus thermophiles)和瑞士乳杆菌(Lactobacillus helveticus)在7 份样品中均有检出;德氏乳杆菌相对含量高达82%,并且在每份样品中占比均在47%以上,是所测样品的主要细菌群落,可视其为优势菌种;嗜热链球菌相对含量为7.36%,是含量第二高的菌种;瑞士乳杆菌的相对含量为6.62%;莱士曼氏乳杆菌(Lactobacillus leichmannii)占比为2.05%;其余11 种菌种含量极少,小于0.5%。不同酸牛乳样品的菌种分布及占比并不相同,TJ1 样品中德氏乳杆菌占比47.87%,瑞士乳杆菌占比44.87%,两者均为该样品的优势菌种。在TJ7 样品中嗜热链球菌占比26.88%,是该样品中含量第二高的菌种;TJ2 在7 份样品中菌种较为单一,并且只有德氏乳杆菌含量最高(97.75%)。

图4 自然发酵乳在种水平上的相对丰富度和多样性Fig.4 Relative abundances and bacterial diversity of the microbiota of naturally fermented milk at the species levels
2.4 不同地区酸牛乳细菌多样性比较
为了比较不同地域自然发酵牛乳微生物多样性,从公共平台上检索到摩洛哥和吉尔吉斯斯坦自然发酵牛乳的微生物多样性数据集进行比较分析。首先对3 个地区样品中微生物的多样性和丰富度进行分析,使用α 多样性的4 个指数来评估。如图5 所示,吉尔吉斯斯坦和塔吉克斯坦地区酸牛乳样品的chao1 指数和发现物种数比摩洛哥地区高,说明吉尔吉斯斯坦和塔吉克斯坦地区样品的细菌丰富度高于摩洛哥地区;另外,摩洛哥地区酸牛乳样品的香农指数指数和辛普森指数与其余两地区的样品相比较高,说明该地区酸牛乳样品的细菌多样性较高。各地区酸牛乳样品α 多样性指数详见表2。
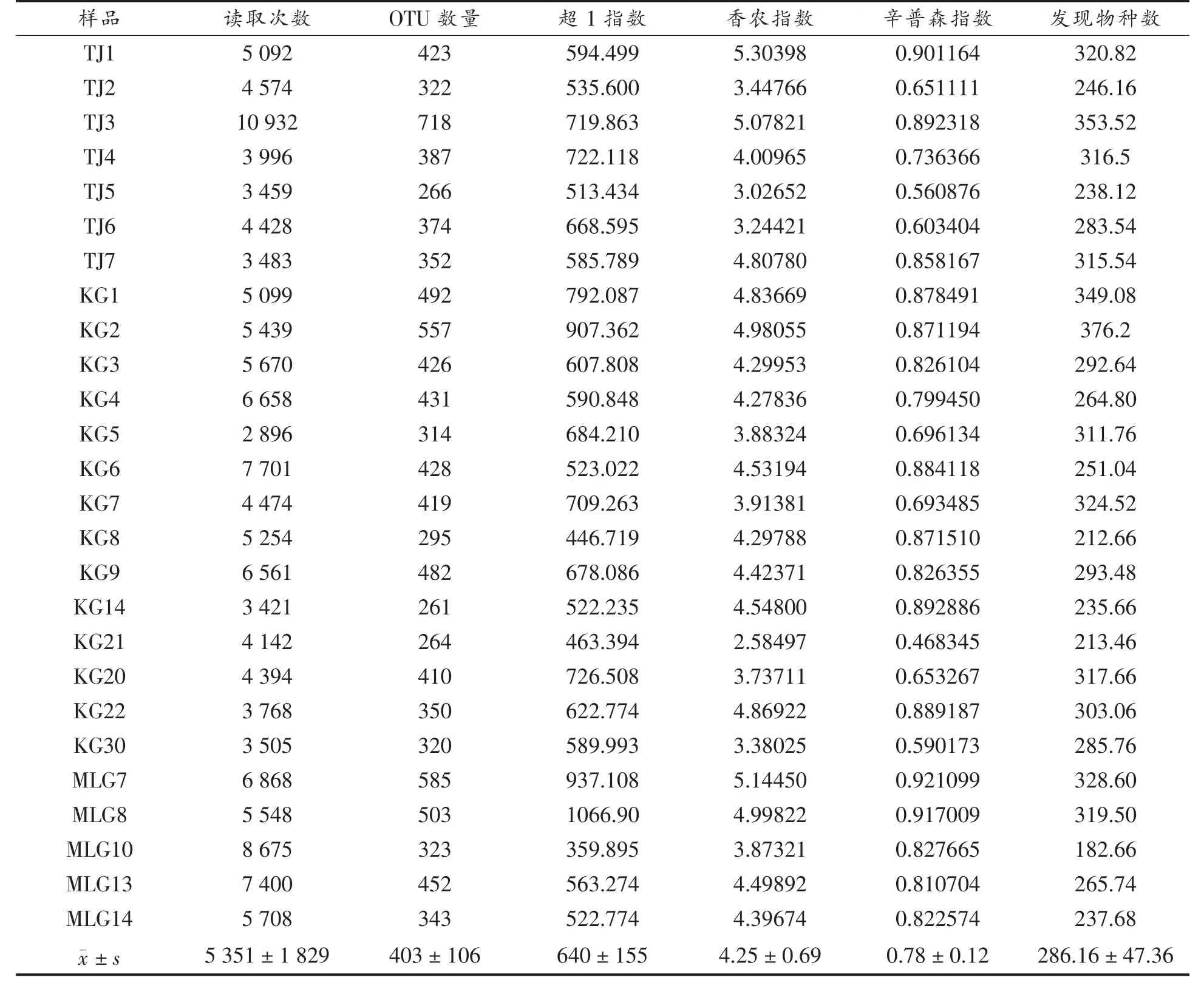
表2 塔吉克斯坦酸牛乳序列数和OTUs,微生物多样性和丰富度指数Table 2 Number of sequences and OTUs,microbial diversity and richness indexes of Tajikistan naturally fermented milk
利用β 多样性对3 个地区自然发酵牛乳样品的多样性进行分析。通过加权和非加权UniFrac距离的主成分分析,显示3 个地区微生物群落的差异。基于加权的UniFrac 距离来分析,可以显示物种丰度的不同,而非加权的UniFrac 距离考虑的是各样品序列间是否有显著的微生物群落差异[17]。以第1 主成分和第2 主成分来分析3 个地区样品微生物落群的多样性,结果见图5。图5a为UniFrac 加权距离,其第1 主坐标和第2 主坐标的贡献率分别为64.06%和19.30%,图5b 为UniFrac 非加权距离,第1 主坐标和第2 主坐标的贡献率分别为19.53%和8.75%。由图5 可知,摩洛哥地区样品与塔吉克斯坦和吉尔吉斯斯坦地区样品能够较好地区分,说明摩洛哥和两地区微生物组成结构存在一定差异;而塔吉克斯坦和吉尔吉斯斯坦样品呈现聚集状态,说明两地区的样品微生物菌群结构组成相似度高。图中摩洛哥和吉尔吉斯斯坦的样品存在部分交叠的现象,说明两个地区样品的微生物结构在一定程度上存在相似性。

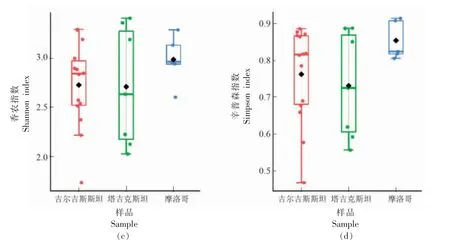
图5 自然发酵酸牛乳样品中α 多样性指数的箱型图Fig.5 Boxplots of α-diversity indexes of naturally fermented milk samples
进一步分析3 个地区酸牛乳样品微生物群落结构差异。一般来说,一个菌株有一条序列,序列数量庞大难以比对,因此以序列相似度来划分OTU,了解样品中的菌种和菌属情况[18]。统计3 个地区的OTU 数,共有10 497 个OTU,每个地区的样品都存在特有的OTU。从图6 来看,3 个地区酸牛乳样品的优势菌种组成存在差异,摩洛哥地区优势菌株为嗜热链球菌、乳酸乳球菌(Lactococcus lactis)和瑞士乳杆菌,其次马乳酒样乳杆菌(Lactobacillus kefiranofaciens)、副干酪乳杆菌(Lactobacillus paracasei)也较多;而塔吉克斯坦和吉尔吉斯斯坦地区优势菌种为德氏乳杆菌和嗜热链球菌,这两地区的菌群有细微的差别。吉尔吉斯斯坦样品中存在乳酸乳球菌、哈尔滨乳杆菌(Lactobacillus harbinensis)、食二酸乳杆菌(Lactobacillus diolivorans)、类谷糠乳杆菌(Lactobacillus parafarraginis)等菌株,然而丰度较小,在塔吉克斯坦样品中未检出。上述结果表明3 个地区的样品虽然微生物群落的多样性差异较小,在微生物群落结构上具有相似性,但是样品优势菌种的组成不同。

图6 基于主成分1 和2 的加权(a)和未加权(b)UniFrac 主成分分析(PCoA)得分图Fig.6 Weighted(a)and unweighted(b)UniFrac principal component analysis(PCoA)score plots based on principal components 1 and 2

图7 3 个地区自然发酵乳中菌种丰度气泡图Fig.7 Bubble chart of the abundance of bacteria in natural fermented milk in three regions
3 讨论
纯培养的方法是最直接、有效地获得乳酸菌菌种资源的方法,要深入全面获取样品中微生物多样性的信息,需结合分子生物学技术,克服纯培养的局限性[19]。本研究结合两种方法对塔吉克斯坦地区自然发酵乳中乳酸菌多样性进行分析。结果显示,德氏乳杆菌为该样品的优势菌种,两种方法检测结果一致,然而,测序结果发现存在嗜热链球菌和瑞士乳杆菌等菌株,而分离鉴定并未发现。以往研究者对酸牛乳中菌群结构的研究显示,新疆喀什地区传统酸乳中德氏乳杆菌是优势菌种,同时也可分离得到瑞士乳杆菌、嗜酸乳杆菌(Lactobacillus acidophilu)、屎肠球菌等菌种[20]。同样,俄罗斯地区采集的自然发酵乳中,乳杆菌属是最丰富的菌属[21],上述研究与本研究结果相近。通过测序分析,一些痕量微生物被检出,然而,受某些因素的限制,许多微生物较难分离培养。分子生物学技术虽可检测到更为丰富的微生物,但无法保留菌种资源。将二者结合来研究微生物多样性,具有开发乳酸菌资源的优势。
不同地区酸牛乳的微生物多样性存在差异。将吉尔吉斯斯坦和摩洛哥与塔吉克斯坦地区数据对比,结果显示,3 个地区的微生物菌群组成不同。吉尔吉斯斯坦和塔吉克斯坦地区群落组成差异较小,可能是两国地处中亚相邻而立,空间上地理位置接近,历史文化和饮食习惯相似。而摩洛哥位于非洲西北部地区,与两国相距较远,气候也相差甚远,因此微生物群落结构和其它两国存在差异。Sun 等[22]研究的蒙古国地区自然发酵酸牛乳中发酵乳杆菌(Lactobacillus fermentum)和瑞士乳杆菌是优势菌种,而Liu 等[23]研究的中国内蒙古地区发酵酸牛乳中乳酸乳球菌、瑞士乳杆菌是优势菌种。传统自然发酵乳制品具有不同的地域特色,因不同的制作工艺及地域环境,而使得其中蕴含的微生物资源差异较大。Zhong 等[24]研究表明:地理来源和样品类型塑造了天然发酵乳的微生物多样性,地理位置不仅代表着空间上的距离,也代表着温度、湿度、海拔等条件对微生物多样性的影响。综上,自然环境、气候等因素造就了丰富多样的乳酸菌资源。
4 结论
运用纯培养方法和PacBio SMRT 三代测序技术对塔吉克斯坦地区自然发酵酸牛乳中微生物多样性和群落结构,特别是乳酸菌菌群组成进行综合分析。分离鉴定出92 株乳酸菌,获得珍贵的乳酸菌资源。对不同地区发酵酸牛乳微生物群落组成分析发现,不同地区的自然发酵乳样品中微生物群落结构具有差异,地理位置是引起差异的一个因素。本研究结果可为具有区域特色的乳酸菌资源开发利用提供参考。
