利用生物信息学设计基于抗原表位的非洲猪瘟疫苗的总效果
2023-01-11何娜译自2020
何娜 译自,2020,9
范美红 校 李 红 审 张 涛 制图
非洲猪瘟(African swine fever,ASF)病毒是引发ASF 的病原体,是全球家猪和野猪最致命的病原体之一。20 世纪20 年代,在肯尼亚引进欧洲家猪后,该病首次被发现。在20 世纪上半叶,该病的暴发主要局限在撒哈拉以南的非洲东部和南部的猪群中。之后,ASF 病毒蔓延到全球各地,不仅在其他非洲国家,而且在欧洲、南美洲、大洋洲和亚洲的猪群中流行。最近一次从非洲传入欧洲的ASF 病毒引发了有史以来最大规模的ASF 疫情,成为当今全球养猪业的头号威胁。由于其潜在的破坏性影响,ASF 被列为必须向世界动物卫生组织强制通报的疾病。
在非洲,ASF 病毒的主要宿主是非洲野猪,如感染后无临床症的疣猪(Phacochoerus africanus)和布什猪(Potamochoerus larvatus)。在这些地区,ASF 病毒感染离不开钝缘蜱(Ornithodoros)属的蜱虫这类虫媒载体,它们负责向家猪传播ASF 病毒。如今,直接接触受感染的材料已经成为ASF 病毒在欧洲和亚洲的猪群中传播的主要风险因素,如病死猪的尸体或猪肉制品,以及接触感染猪的人。欧亚大陆的野猪也容易感染ASF 病毒,感染后表现出与家猪相似的临床症状和死亡率,影响了欧亚大陆防控ASF 的效果。ASF 病毒可引起感染猪出现急性出血热,这种病毒的毒力很强,死亡率几乎100%。目前,对ASF 的防控还没有疫苗或治疗药物,控制病毒传播的唯一有效方法是扑杀受影响地区的所有猪,即受感染的猪和可能与之接触过的猪。需要安全地处理病死猪的尸体和其他被污染的材料,这包括实施最大限度的控制措施和生物安全措施,以及限制猪及其衍生产品的流动。
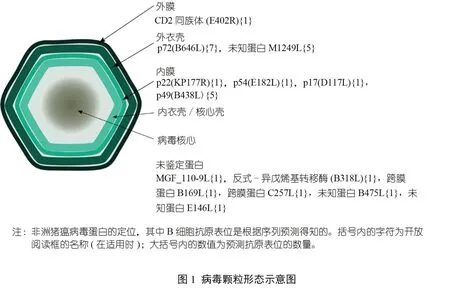
ASF病毒是非洲猪瘟病毒科(Asfarviridae) 的唯一成员,是属于核质大型DNA 病毒(nucleocytoplasmic large DNA viruses,NCLDVs)组的一种巨型病毒(约200 nm)。该病毒有一个复杂的多层结构,由多个脂质和蛋白层组成(图1),基因组被第一层蛋白层包裹着,该蛋白层由核心壳内衣壳组成,而核心壳内衣壳又被内脂双层膜覆盖,该膜包含在一个二十面体蛋白质衣壳中。细胞外颗粒通过质膜排出时获得脂质外膜。ASF 病毒的主要靶细胞是巨噬细胞,也感染树突状细胞和其他单核细胞。病毒基因组由一个长度为170~193 kb 的双链DNA 分子组成,可编码150 多个开放阅读框(open reading frames,ORFs),包括其自身的复制机制、基因调控、宿主防御逃避因子以及其整套结构蛋白。病毒在基因组长度和ORFs 数量上的差异主要归因于位于基因组两端可变区域的多基因家族(multigene family,MGF) 基因数量的不同,而在中央保守区域的基因中可以发现更细微的差异。值得注意的是,许多病毒蛋白的功能仍未知。
来自不同国家、不同发病地、具有不同毒力水平的多种ASF 病毒分离株已得到了确认。ASF 病毒的第一个完整的基因组序列来自20 世纪70 年代初在巴达霍斯(西班牙)获得的分离株,该分离株适合在Vero 细胞(Ba71V)中生长。此后,许多其他基因组也已完成,截至2020 年8 月,在通用蛋白质数据库(universal protein resource,UniProt)中获得了来自不同病毒突变体或分离株的46个蛋白质组。根据主要的衣壳蛋白p72 基因(B646L)3’端序列的分析,可将其分为22 种不同的基因型。从地理位置上看,在非洲,ASF 病毒的基因型变异最大,而在欧洲、美洲和亚洲,仅发现了基因Ⅰ型和基因Ⅱ型的分离株。到目前为止,还没有证据表明ASF 病毒的基因型与其毒力有关。
感染后存活的猪会产生免疫力,接种减毒病毒会诱发动物机体产生免疫反应和变异株特异性保护作用。因此,尽管安全问题限制了减毒活疫苗的田间应用,但仍可以推行疫苗接种策略来进行一定程度的预防。其他免疫方法,如蛋白质亚单位或DNA 构建体,虽然其中一些能诱发特异性免疫反应,但无法达到减毒活病毒所能达到的保护水平。尽管使用减毒活疫苗和亚单位疫苗获得的体内保护数据很少,但可以确定分子和免疫学机制参与了这种保护。一方面,尽管引发保护性反应的特异性T 细胞决定因素在很大程度上仍然未知,但已广泛证实特异性T 细胞起着关键作用。另一方面,已经发现了几种ASF 病毒血清学决定因素,但它们的保护作用尚不清楚。然而,据报道,ASF 病毒特异性抗体的被动转移可提供免疫保护,证明其在控制感染上能发挥一定的作用。此外,新的证据表明,微生物群可能在防御病毒方面起着重要的作用,因为将疣猪的粪便移植到家猪身上,可以实现对ASF 病毒的部分免疫保护。
另一种针对多种抗原的疫苗设计策略,能够在不影响安全性的情况下诱发动物机体产生保护性免疫反应,这可能是解决病毒生物学复杂性的一种方法。在这方面,反向疫苗学(reverse vaccinology,RV) 的应用,对筛选越来越多的ASF 病毒序列以寻找最佳靶抗原表位非常有帮助。为开发难以分离和培养的病原体的疫苗,反向疫苗学是基于对病原体基因组和/或蛋白质组数据的计算分析,以确定对假设的疫苗最感兴趣的蛋白质。使用计算机模拟方法(in-silico approach)还可以考虑病毒的变异性,特别是确定不变区域,有助于设计出具有广泛保护性疫苗(pan-protecting vaccines)。同样,它可以避免诱导保护性免疫反应所不需要的序列或可能会导致疾病恶化的序列。令人鼓舞的是,已经有一种利用反向疫苗学生产的针对脑膜炎奈瑟菌血清B 群的疫苗于2013 年获得许可。
1 结果
1.1 ASF 病毒蛋白空间分析
从UniProt 中46 个可用的病毒蛋白质组中下载了7 088 条ASF 病毒蛋白质序列。总的来说,由于蛋白质数量异常,有7 个蛋白质组被去掉:Kyev 2016(UP000321214)、波兰2016o23(UP000274966)、波兰2016o9(UP000268777)、波兰2017C220(UP000282187)、波兰2016o10(UP000278405)、撒丁岛 Sassari 2008(UP000266411)和南非1985/SPEC 57(UP000423628)。
用CD-HIT*CD-HIT是一个用于聚类和比较大量蛋白质或核苷酸序列集的快速程序,以减少序列冗余并提高其他序列分析的性能。进行蛋白质聚类产生了459 个聚类。为了考虑每个聚类中ASF 病毒的变异性,我们去掉了序列源自少于14 个不同蛋白质组的聚类。选择该阈值是因为这是来自ASF病毒基因Ⅱ型(格鲁吉Georgia 2007/1 分离株)的可用ASF 病毒蛋白质组的数量,目的是保留所有可能的ORFs 属于该基因型。因此,我们考虑进一步分析173 个包含14 个或更多不同ASF 病毒蛋白质组的蛋白质簇。值得注意的是,为量化位置的变异性而计算香农熵(Shannon entropy) 值显示,在集群的多序列排列(multiple sequence alignments,MSAs)中,82.5%的氨基酸位置是完全不变的,即熵值H=0。因此,为了尽可能的严格,所有后续分析都是用指定香农熵阈值为H=0 的不变的共识蛋白质组(consensus proteome)来完成的。
1.2 鉴定ASF 病毒假定暴露的蛋白质(putative-exposed proteins)
到目前为止,在ASF 病毒的表面只确定了一种蛋白质:CD2 同系物(ORF:E402R)。为了鉴定病毒蛋白,我们付出了更多的努力,这些蛋白可能存在于受感染细胞膜的外表面和/或细胞外病毒粒子,因此容易被抗体中和。为了确定假定的暴露蛋白质,我们用亚细胞定位预测工具预测ASF 病毒格鲁吉2007/1 分离株蛋白质组(UP000141072),根据注释规范(criteria of annotation)和预测的信号肽序列和/或至少一个跨膜结构域,共选择了49 个假定的暴露蛋白质。假定暴露的蛋白质包括那些被预测为暴露的蛋白质,以及那些以前被描述为形成一部分外衣壳的蛋白质,在无包膜的感染性颗粒的情况下是抗体的潜在靶位点。所有这些都被标记为“暴露”,并与ASF病毒蛋白质组的其他部分聚类,以找到所有ASF 病毒“暴露”蛋白质的同系物。在包括14个或更多ASF 病毒蛋白质组的173 个聚类中,只有43 个具有“暴露”蛋白质,并选中用于进一步探索B 细胞抗原表位的存在。
1.3 B 细胞抗原表位的选择
我们在免疫抗原表位数据库(immune epitope database,IEDB) 中发现了该病毒102 个经实验验证的、独特的抗原表位,其中76 个是B 细胞抗原表位。我们保留了那些来源于假定的暴露蛋白的序列,然后对照ASF 病毒不变的“暴露”蛋白质组解析了它们的全序列。结果发现了9 个保守的B 细胞抗原表位。为了进一步组织和管理这一选择,我们使用BLASTP 将这些序列与野猪蛋白质组和其微生物群的蛋白质组进行了比对。我们放弃了那些与野猪的蛋白质或其微生物群的蛋白质同源性大于70%的序列,将名单减少到来自3 个不同蛋白的4 个保守的B 细胞抗原表位:两个来自主要的衣壳蛋白p72(B646L),两个来自内衣壳蛋白p22(KP177R) 和p54(E183L)。结果发现,抗原表位VCKVDKDCGSGEHCV(p22 蛋白)和FPENSHNIQTAGKQD(p72 蛋白) 与基于序列预测的抗原表位具有相同的残基;后者也与结构化预测抗原表位共享残基。
1.4 重新(de novo)预测的B 细胞抗原表位
除了免疫抗原表位数据库(IEDB) 中传统的B 细胞抗原表位外,我们还采用了两种方法来预测新的抗原表位:基于序列的方法和基于结构的方法。按照基于序列的方法,我们检索到42 个在ASF 病毒中保守的潜在B 细胞抗原表位。对于那些我们预测的跨膜结构域的蛋白抗原表位,我们绘制了它们的图谱,以评估它们在“外部”的定位。任何映射到跨膜蛋白内测或跨膜位置内的表位都被丢弃。此外,我们用PyMOL 软件*PyMOL 软件一款功能强大的分子可视化软件,适用于创作高品质的小分子或生物大分子(特别是蛋白质)的三维结构图像;以Py+MOL 命名指,“Py”表示它是由一种计算机语言Python 所衍生出来的,“MOL”表示它是用于显示分子(英文为molecule)结构的软件。对主要的衣壳蛋白p72(PDB ID:6L2T)的潜在抗原表位进行了观察,并丢弃了那些没有暴露的抗原表位,即面向病毒粒子外衣壳抗原表位。通过这种方法,我们将列表中的抗原表位减少到29 个,并取消了另外两个抗原表位,因为它们与野猪或其粪便中微生物群的蛋白质序列的同源性大于70%。所选择的27 个预测的B 细胞抗原表位来自于13 种不同的蛋白。其中一些抗原表位与在IEDB 中验证的序列具有相同的残基。这是由于IEDB 中的抗原表位是根据共识蛋白质组进行整体解析的,这导致放弃了具有可变位置的长序列和/ 或与猪体内蛋白质的同源性大于70%的序列。
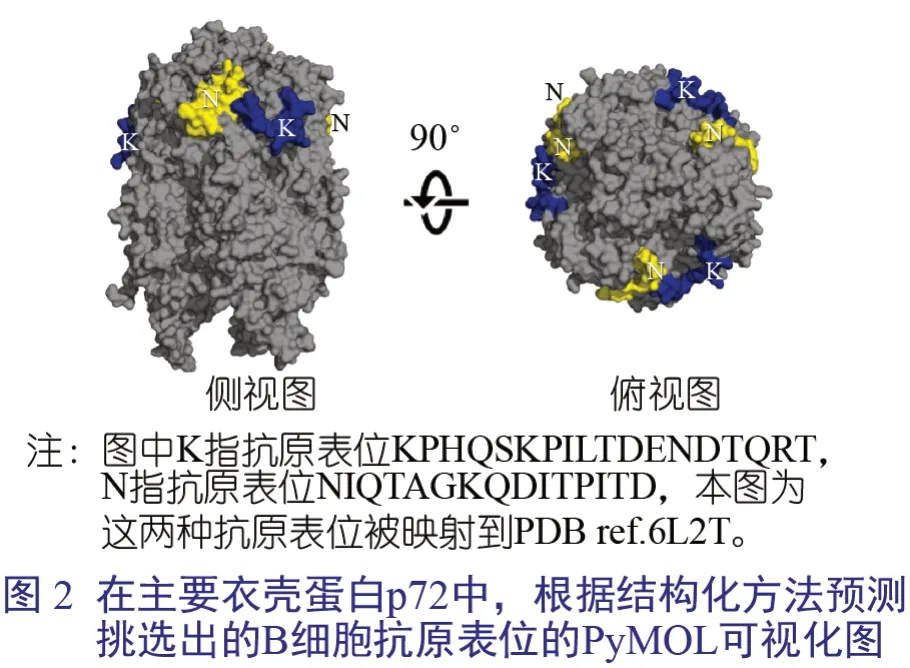
对于基于结构的方法,我们对蛋白质数据库(protein data bank,PDB)中提供的ASF病毒主要衣壳蛋白p72 两种结构上的残基的灵活性和残基相对可溶性(relative solvent accessibility,RSA) 施加了特定的限制:6KU9 和6L2T。我们确定了13 个潜在的抗原表位,这些抗原表位在“暴露”的不变蛋白质组中也是保守的。在丢弃非独特的肽后,由于p72 同源三聚体组装成壳体,并融合了重叠的抗原表位,我们最终得到了8 个独特结构预测的B 细胞抗原表位,其中仅有3 个与野猪及其微生物群的蛋白质的同源性不超过70%,均来自PDB 结构6L2T。在PyMOL 软件观察它们的易接近性时,我们去掉了位于该蛋白质底部的一个抗原表位,因此是非暴露的。其余两个潜在的抗原表位显示在图2 中。这两个抗原表位是基于序列的方法预测的。
1.5 CD4+T 细胞抗原表位的选择
在免疫抗原表位数据库的102 个经实验验证的抗原表位中,26 个是T 细胞抗原表位。这些抗原表位缺乏有关与主要组织相容性复合体(major histocompatibility complex,MHC)结合的信息,但考虑到它们的长度(≥15个氨基酸),我们认为它们是CD4+T 细胞抗原表位。只有三个是保守的,都来自同一个蛋白质CD2 同系物。然而,利用BLASTP 与猪及其微生物群的蛋白质进行比对时,这些蛋白质的同源性都不超过70%,所以去掉了它们。
1.6 预测新的CD4+T 细胞抗原表位
我们没有在IEDB 中找到符合选择标准的ASF 病毒CD4+T 细胞抗原表位,所以我们预测了新的CD4+T 细胞抗原表位,以确定它们是否可能会被纳入疫苗集合(ensemble)中。在不变的蛋白质组上应用 IEDB MHC Ⅱ类结合预测工具,共产生了13 342 个潜在的15-mer 抗原表位,我们继续研究前0.1%结合者中的273 个序列,其中大部分是较大肽的15-mer 片段。重叠序列的融合产生了74 个独特的肽序列。我们去掉了任何被预测为只与一个等位基因结合的抗原表位,因此,我们保留了16 个序列。利用BLASTP 对野猪及其微生物群的蛋白质进行对比后,我们从13 个不同的蛋白质中选择了14 个潜在的抗原表位,这些抗原表位与这些蛋白质的同源性不超过70%。
1.7 CD8+T 细胞抗原表位的选择
在免疫抗原表位数据库中没有经实验验证的CD8+T 细胞抗原表位,而文献检索也没有发现任何存在于不变蛋白质组中的抗原表位。因此,我们使用模仿这些抗原表位在细胞内自然处理途径的步骤序列来预测新的CD8+T 细胞抗原表位。
蛋白酶体加工不变的蛋白质组产生了277个最小长度为9 个氨基酸的保守肽,任何短于9 个氨基酸的片段都被去掉。我们从那些大于9 个氨基酸长度的蛋白酶体反应衍生的片段中创建了所有可能重叠9-mer 的多肽组合,产生了669 个潜在的抗原表位。我们采用log(IC50) 值为1 作为串联亲和纯化法(tandem affinity purification,TAP) 的阈值,将潜在的抗原表位列表限制在40 个肽的序列。IEDB MHC Ⅰ类结合工具预测了其中33 个在前1%内,其中22 个与野猪蛋白质的同源性不超过70%。然而,考虑到与微生物群的蛋白质的潜在交叉反应性,我们提高到了≤80%的同源性阈值。选定的潜在CD8+T细胞抗原表位的最终清单仅包含来自6 个不同蛋白质的6 条肽。
2 讨论
自2018 年以来,非洲、欧洲、亚洲和大洋洲的几十个国家都暴发了ASF,中国首次报道于2018 年,根据官方的消息,亚洲生猪总损失估计超过600 万头。
由于目前尚无有效的治疗方法,针对ASF病毒的防控显然迫切需要有效的方法,但还没有成功开发出安全且有效的疫苗。目前,大多数后期的免疫力都是利用自然发生或在实验室产生的减毒活病毒进行的。例如,用低毒力的NHV/1968 变异体进行免疫接种,可对密切相关但高毒力的ASF 病毒L60 分离株产生免疫力。因此,对异源性分离株的挑战只能达到部分免疫,并且对接种猪产生了有害的副作用。另一个有希望的疫苗接种策略是使用缺失CD2 同源蛋白的ASF 病毒Ba71 分离株。这种毒力减弱的病毒可提供对母体病毒的免疫力,也可提供对异源变体ASF 病毒E75 分离株和ASF 病毒格鲁吉2007/1 分离株的免疫,而不会产生明显的副作用。令人震惊的是,同样的原则在使用CD2 同源缺乏的Georgia 2007/1 分离株时未能产生免疫保护,突出了ASF 病毒的表型多样性。一个合理的解释可能是存在冗余的ORFs,可以覆盖CD2 同系物的基本功能。事实上,ASF 病毒格鲁吉2007/1 分离株与Ba71 分离株相比显示出更大的复杂性,与后者相比前者有更多的ORFs。这进一步加强了我们对ASF 病毒格鲁吉2007/1 分离株的关注,因为我们想尽可能地解决这种复杂性。
也有人尝试用转基因病毒进行免疫接种,但在诱发的免疫水平、获得对异源分离株的保护或出现有害的副作用方面结果各异。人们也在追求用ASF 病毒亚单位的特定病毒蛋白进行免疫。这类疫苗比上述方法更加安全,有各种基于亚单位免疫的许可产品的例子。然而,对于ASF 病毒来说,这种策略只能提供部分保护或根本不提供保护。
在本研究中,我们使用计算机辅助方法来确定一组ASF 病毒抗原表位,这可能对设计基于抗原表位的ASF 疫苗很有意义(图3)。为了激发广泛而强大的免疫反应,以抵御异源性分离株的挑战,我们优先选择了在不同ASF 病毒分离株中严格保守的潜在抗原表位序列。选择保守的抗原表位也有一个好处,就是对ASF病毒而言这些高度保守区域在生物学上可能是必需的,因此可用于制作疫苗的理想靶区。然而,任何选择的抗原表位必须能够诱导动物机体产生真正的保护性反应,因为即使存在中和抗体,涉及非中和(non-neutralized)ASF 病毒的慢性感染已被大量地报道。
在选择B 细胞、CD4+T 细胞和CD8+T 细胞的抗原表位时,我们采取了多种策略,包括查询免疫抗原表位数据库的传统实验方法,以及通过成熟的免疫信息学工具对抗原表位进行重新预测。对序列进行优先排序的依据是其与野猪及其微生物群的蛋白质的同源性不超过70%这一阈值。这样做的理由是:首先,任何具有与宿主中蛋白质序列不同的序列在传递时将有更大的机会获得免疫原性,这是未来疫苗的一个理想特征。其次,出于安全考虑,与宿主的蛋白质序列不同的序列应避免交叉反应。此外,由于微生物群最近被证实在动物抵抗ASF 上扮演一个有重大意义的角色,任何最佳的疫苗都应能够诱导机体产生全身性免疫和黏膜免疫,能够阻断病原体,同时避免针对不受宿主微生物群欢迎的疫苗诱导反应。在对CD8+T 细胞抗原表位进行优先排序时,利用BLASTP 比对微生物群的蛋白质序列时使用了≤80%的阈值,这一事实与这些抗原表位固有的短长度(9-mers)有关,否则在更严格的阈值下会被去掉。必须强调的是,由于在物种水平上缺乏猪的微生物群的数据,BLASTP是针对报告存在于猪体内的每个微生物属中所有微生物物种进行的,这可能包括未真正存在于猪体内的微生物物种。因此,我们决定将≤80%的阈值作为低分辨性和BLASTP 命中之间的良好权衡。

抗体在ASF 病毒免疫中具有重大的意义,因为从存活的猪上转移的抗ASF 病毒免疫球蛋白G(IgG)可以防止感染。我们遵循三种不同的方法来选择可能感兴趣的B 细胞抗原表位:一种是传统的实验方法,在IEDB 中搜索保守和经实验验证的抗原表位,还有两种重新预测的方法,一种是基于BepiPred-2.0 的序列方法,另一种是基于结构的方法,使用蛋白质数据库(PDB)的三维结构,只适用于p72 的情况。
ASF 病毒是一种包膜病毒,包膜隐藏在脂质双分子层之下(图1)。迄今为止,已知嵌入脂质外膜的唯一蛋白是CD2 同源蛋白(E402R),我们根据序列方法预测了它的抗原表位。该蛋白是不同ASF 病毒中差异最大的蛋白之一,因此在不同ASF 病毒分离株之间发现一个保守的抗原表位是有意义的。虽然这是一种包膜病毒,但据报道,细胞外和细胞内的“裸体”颗粒都具有感染性。ASF 病毒感染可导致细胞裂解,而释放的细胞内颗粒可能已经具有感染性。这些“裸”病毒的抗原表面主要由重要的衣壳蛋白p72 蛋白(B646L)的拷贝组成,它构成了外衣壳的大部分。此外,已经报道存在针对这种蛋白的中和抗体,证明了从p72 蛋白中优先选择B 细胞抗原表位的理由。我们从IEDB 中搜索并找到了两个保守的序列,此外还有2 个基于结构的方法预测的抗原表位和7个基于序列的方法预测的抗原表位。值得注意的是,从p72 三维结构分析中发现的两个抗原表位(图2) 都含有与基于序列方法预测的序列和IEDB 的实验验证序列相匹配的残基,其中一个还在保守序列FPENSHNIQTAGKQD 中发现。除了p72 蛋白,我们预测的另一个蛋白是M1249L 蛋白,Wang 等将其确定为帽状体骨架的一个重要部分,因此在“裸”病毒粒子的情况下可能被抗体锁定。
其他选定的潜在B 细胞抗原表位来自于该外衣壳下面的蛋白质,嵌入到内脂层中。p54 蛋 白(E183L) 和p22 蛋 白(KP177R) 的 抗原表位都是通过IEDB 传统实验搜索(legacy experimentation search)和基于序列方法发现的,在p22 蛋白的特殊情况下,根据序列预测的抗原表位与经实验验证的抗原表位共享一半以上的序列。此外,我们在p17 蛋白(D117L)中预测了一个抗原表位,在p49 蛋白(B438L)中预测了五个抗原表位,后者有一个抗原表位与在IEDB 验证的序列中发现的残基相同。如上所述,所有这些蛋白都位于病毒粒子的内层,有关它们的抗体的保护作用可能并不明确。尽管如此,有研究报道了由针对内层粒子蛋白p22 蛋白、p54 蛋白和p30 蛋白(CP204L) 的抗体介导的中和作用,表明存在一种机制,这些蛋白通过这种机制可以与抗体相接触。可以假设,病毒粒子层是动态结构,而不是静态结构,因此,存在于其中的蛋白质可能容易被中和。此外,还预测了来自B169L 蛋白、C257L蛋白、B475L 蛋白、E146L 蛋白、反式异戊烯基转移酶B318L 和MGF 110-9L 的抗原表位,但通过文献无法证实它们在病毒颗粒中的位置。我们选择这些蛋白质是为了鉴定可能暴露的病毒蛋白质。
综上所述,必须强调的是,以前的疫苗接种研究已经确定这些抗原中的一些会加重疾病,可能通过抗体依赖性增强感染,就像p30 蛋白、p54 蛋白、p17 蛋白、p72 蛋白甚至CD2 同系物的情况那样,在对它们进行实验验证时应格外谨慎。此外,预测方法的一个局限性是,用所遵循的方法我们无法预测B 细胞构象性抗原表位(conformational B-cell epitopes)。非蛋白质抗原表位如基于糖的抗原表位也没有被检索到。
以前用减毒活疫苗和DNA 疫苗进行的实验表明,CD8+T 细胞在产生对ASF 病毒的免疫力上发挥了主要作用,但我们没有发现任何经实验验证的CD8+T 细胞保守抗原表位。我们只能依靠计算方法来预测它们。最终选择的6 个抗原表位经预测可以与预测工具中45 个猪淋巴细胞抗原(swine lymphocyte antigens,SLA)I类等位基因中的38个结合。与该种类的SLA 等位基因的结合将确保能够覆盖广泛的种群。事实上,仅用抗原表位MAMQKLFTY 和KRHENIWML 就可以达到最大的覆盖率。值得注意的是,所有选定的CD8+T 细胞潜在抗原表位都来自于病毒的复制和转录机制。假定的DNA 定向RNA 聚合酶亚单位5 同源物(D205R)、非特征蛋白D339L(在别处称为RNA 聚合酶亚单位)、DNA 定向RNA 聚合酶亚单位β(EP1242L)、核糖核苷-二磷酸还原酶(F334L)、DNA 拓扑异构酶2(P1192R) 和非特征蛋白G1340L(在别处描述为可能的早期转录因子)。一系列的CD4+T 细胞抗原表位也被选中。初始T 辅助细胞(naive Th cells)通常由抗原呈递细胞(antigen-presenting cells,APCs) 激活,而这些细胞恰好是ASF病毒的主要靶点(巨噬细胞、单核细胞和树突状细胞)。这种现象可能会妨碍APCs 呈递抗原的能力,而这是形成有效的适应性免疫反应的关键步骤。CD4+T 细胞在平衡和维持细胞毒性和体液反应方面发挥着重要的作用,因此任何疫苗研究都应考虑这些细胞的作用。在本研究中,我们没有发现任何经实验验证的CD4+T 细胞在IEDB 中的保守抗原表位,所以我们被迫对其进行预测。我们的新方法发现了14 个潜在的CD4+T 细胞抗原表位。值得注意的是,CD2 同系物和p72 蛋白,即B 细胞介导反应的驱动因子,各提供一个CD4+T 细胞的潜在抗原表位。必须指出的是,大多数免疫信息学工具都是根据人类免疫学背景下的数据设计和校准的,这可能会对ASF 病毒CD4+T 细胞抗原表位的预测产生影响。
最终,我们确定了6 个CD8+T 细胞抗原表位、14 个CD4+T 细胞抗原表位和29 个B细胞抗原表位的优先顺序,这些抗原表位是独特且保守的。考虑到迄今为止所介绍的所有ASF 病毒分离株之间的变异性,这一清单为根据ASF 病毒抗原表位设计疫苗提供了广泛的可能性。下一步将包括通过预测方法对这些抗原表位进行实验验证,并全面评估它们的个体和/或群体诱发免疫反应的能力,以抵抗病毒攻击。在阐明这一点后,它们很可能有助于设计一个安全且有效的基于抗原表位的非洲猪瘟疫苗组合。
3 结论
我们确定了49 个抗原表位,包括B 细胞和T 细胞(CD4+和CD8+) 的抗原表位,它们有可能被用作基于抗原表位的非洲猪瘟疫苗的一部分。它们在ASF 病毒中都是严格保守的,并且与野猪及其微生物群的蛋白质显示出较低的一致性。然而,其中只有4 个抗原表位有实验性证据表明它们被猪的免疫系统合理地处理、递呈和识别。因此,本研究的下一步将对这些重新预测的抗原表位进行实验验证。如果可能的话,还应通过体外免疫学试验对它们进行功能鉴定,包括免疫原性和保护性的替代物。只有到那时,才有可能构思出一种包括这些抗原表位的疫苗,并将其应用于体内试验中。
