污泥与菌渣协同消化对微生物的影响分析
2022-11-25鲍王波
鲍王波
(中国联合工程有限公司,杭州 310052)
城镇人口的高速增长导致污水排放量激增,市政污水处理厂数量迅速增加,产泥量也同步增长。据报道,我国2010-2017年间市政污泥年均增长率高达4.7%[1]。市政污泥中含有大量经过驯化的微生物菌群,这为降解其他有毒有害物质提供了有利条件。同时,我国也是抗生素特别是青霉素生产和出口大国,据统计,2009年中国抗生素生产量已达14.7万吨,约占全世界生产量的70%,其中青霉素生产量占全球产量的75%以上[2],而这也同时产生大量生产废渣。众所周知,污水处理厂二沉池污泥大多属于微生物内源呼吸的产物,有机质含量低[3],营养物质匮乏,而青霉素菌渣中含大量未完全利用的糖类、蛋白质、脂类等[4]。因此,本研究探索将二沉污泥与青霉素菌渣进行协同消化处置,可达到以废治废的效果,并对其微生物活动进行了跟踪分析,以期为后续研究提供参考。
1 研究方法
1.1 青霉素残留检测
样品1g于15 mL离心管中,加入提取液(超纯水)5 mL,涡旋1~2 min充分混匀后,20℃超声40 min,4℃下4 000 r/min离心15 min,上清液转移到干净的离心管中待净化。
SPE净化:由于提取液中含有一定量的杂质,会影响色谱图峰型和仪器使用寿命,故进样前需净化处理。Oasis HLB小柱预先用5 mL甲醇活化、5 mL超纯水平衡,再将提取液以2~3 mL/min的流速通过小柱,5 mL 5%的甲醇水溶液淋洗小柱,2 mL乙腈+2 mL甲醇洗脱。洗脱液于40℃下N2吹干后,以超纯水定容至1 mL,0.22 μm滤膜过滤,采用高效液相色谱仪检测青霉素含量。
经试验探索优化,最终确定青霉素色谱检测条件:Thermo C18 1.7 μm 2.1×100 mm色谱柱,柱温30 ℃,流速0.15 mL/min,进样体积10 μL,PDA检测器,流动相A为0.1%的甲酸水溶液,B为乙腈,比例:A:B=60:40。
1.2 微生物基因组的PCR-DGGE指纹分析
1.2.1 样本总DNA提取及PCR扩增
样品总DNA采用美国MP公司土壤DNA提取试剂盒(MP Biomedicals)提取,DNA浓度采用核酸定量仪(Thermo NanoDrop 1000 spectrophotometer)测定。
PCR使用V6-V8区通用引物,序列:U968-GC(5’- CGCCCGCCGCGCCCCGCGCC CGGCCCGCCGCCCCCGCCCCAACGCGAAGAACCTTAC-3’);L1401(5’-CGGTGTGTACAAGACCC-3’),配置50μl PCR体系:5μl 10×PCR buffer (Mg2+free),4μl dNTP Mix,左右引物各1μl,3μl MgCl2,1.5μl taq酶,DNA模板4μl,ddH2O 34μl。
PCR程序:94℃预变性5min,94℃变性30s,65℃退火30s,72℃延伸30s,35个循环,最后72℃保持8min。PCR产物用1.5%的琼脂糖胶进行电泳。
1.2.2 PCR产物的DGGE指纹分析
PCR扩增产物进行变形梯度凝胶电泳(DGGE)分析,DGGE使用8%的聚丙烯酰胺(37.5:1丙烯酰胺:甲叉双丙烯酰胺)凝胶,变性范围:30%~50%(100%的变性剂中含有7 mol/L的尿素和40%的去离子甲酰胺),蠕动泵自动灌胶。每条泳道加反应体系样20μl,1×TAE电泳液,60℃,200V预跑10分钟,随后60V电泳17h,SYBR Green I(1:10000稀释)染色30分钟后于凝胶成像仪(Bio-Rad,美国)中扫描成像[5]。
1.2.3 DGGE条带克隆测序分析
(1)DNA回收
对DGGE 指纹图谱上的DNA条带进行割胶回收,溶于20 μl灭菌的去离子水中,4℃静置过夜。以上述胶水混合物为DNA模板再次进行PCR扩增,反应体系:10×PCR缓冲液5μl,dNTP(25 mmol/L)4μl,V3可变区通用引物(不带GC夹子)各0.5 μl,MgCl2(25 mmol/L) 3 μl,模板2 μl,TaqDNA聚合酶0.3 μl,dd H2O 36 μl,总体积50 μl。反应条件:94℃预变性5 min,94℃变性30s,65℃退火30s,72℃延伸30s,72℃保持7 min,共循环35次,4℃保温。
将50μl扩增到的PCR产物进行1.5% 的琼脂糖凝胶电泳,使用Axygen公司的凝胶回收试剂盒割胶回收,产物加入20μl去离子水-20℃保存用于后续的酶连与转化。
音乐,能够唤起人内心最真实的声音,也是构建情境教学法最有效的方法。在小学语文教学中,最注重的就是强化学生的读写能力。因为汉语是我国的母语,听说能力肯定都具备了,关键在于“读”和“写”。而用音乐来创设情境,就能够帮助学生很好地融入课本,在音乐的伴随下,学生与课本内容产生了共鸣。这样,学生在“读”的过程中才能调动自己的真实情感。如在进行《怒吼吧,黄河》一文的教学时,教师可以在阅读的同时播放《黄河大合唱》这首歌,让学生在感受音乐磅礴气势的同时,理解文章的真实情感,这大大提高了语文教学的课堂效率。
(2)酶接转化
4 ℃条件下,灭菌管中加入酶接体系:DNA片段4.5 μl,PMD-19载体0.5 μl,SolutionI 5 μl,总体积10 μl,16℃保存4h。
将酶接体系(10 μl)加入到感受态细胞中(感受态细胞要一直处于冰盒中),在冰盒中放置半小时,42℃水浴热激90s后,冰上放置2 min,加入800 μl LB液体培养基,于37℃恒温摇床中孵育30~40 min。然后将其涂布在含青霉素(100 μg/ml)的LB固体培养基上,于37℃过夜培养。
(3)克隆子的培养与验证
高温灭菌的96孔板中加入含有青霉素(100 μg/ml)的液体LB培养基600 μl,挑取平板上生长良好的单菌落接种到培养基中,37℃过夜培养。
取上述培养所得菌液进行PCR扩增(10×PCR缓冲液2.5 μl,dNTP(25 mmol/L)2 μl,M13左右引物各0.5 μl,MgCl2(25 mmol/l) 1.5 μl,菌液0.3 μl,TaqDNA聚合酶0.25 μl,dd H2O 17 μl)PCR条件:94℃预变性5 min,94℃变性30s,65℃退火30s,72℃延伸30s,35个循环,72℃保持7 min,4℃保温。PCR产物进行1.5%的琼脂糖电泳验证,根据电泳图谱条带位置选取酶接成功的克隆子进行DNA测序。
1.3 PCR-DGGE指纹图谱数据分析
DGGE图谱使用Quantity one软件进行数据提取分析导出至Excel工作表中。Quantity one软件自动提取条带丰度数据及条带数量S,由此计算多样性香农(Shannon)指数H及均匀度E,计算公式如下:
H=-ΣPi×lnPi[6]
E=Hmax/lnS[7]
其中,Pi=ni/N,ni为第i个条带的丰度值,N为第i个条带的泳道上所有条带丰度之和,Hmax为所有H值中最大的值。采用Quantity one软件对DGGE条带进行聚类分析。
1.4 实验设计
将青霉素菌渣(青霉素残留量1.09±0.58 mg/kg)与市政污水处理厂二沉池剩余污泥按填埋质量比0:1 (处理组1)、1:2(处理组2)、2:2(处理组3)的质量比混合消化,并于0、3、7、20、60天时采集填埋样品,检测样品中青霉素残留,每个样品3个平行;采用PCR-DGGE图谱分析样品微生物变化,每个样品3个平行(重复实验图谱未列出)。

图1 实验装置(A示意图;B实物图)
2 结果与讨论
2.1 青霉素残留检测
结果表明(图2),样品中残留青霉素降解迅速,一方面是污泥中可能存在抗生素降解菌及其分泌的酶加速青霉素降解,另一方面消化过程体系温度上升也促进青霉素的降解。
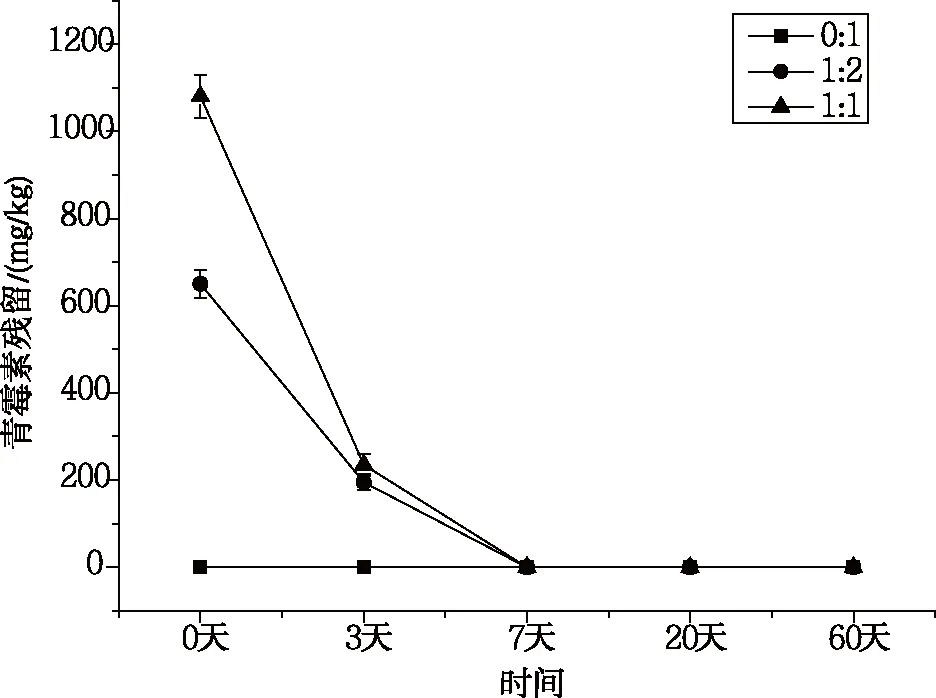
图2 样品中青霉素的残留量检测
2.2 样品微生物多样性分析
样品微生物DGGE指纹图谱见图3,图中共3个处理组,每组3个重复,1、2、3泳道代表第1处理组:m(菌渣)/m(污泥)=0:1;4、5、6泳道代表第2处理组:m(菌渣)/m(污泥)=1:2;7、8、9泳道代表第3处理组:m(菌渣)/m(污泥)=2:2。DGGE图谱中的条带编号分别代表切胶测序编号,结果未列出,下同。
DGGE指纹图谱(图3)显示,第0天三个处理组条带无明显差异,聚类分析提示三个处理组相似度达82%以上,说明微生物群落结构差异很小。第3天体系内的微生物结构与第0天相比发生明显变化,0:1组微生物丰富度明显降低,多数条带强度微弱,可能是缺乏营养微生物大量死亡或休眠,而1:2和2:2组添加了菌渣营养物质,都有各自的优势菌种,聚类分析表明,三组的相似度较低(仅46%),但三个处理组均各自聚成一簇,这说明三个组的微生物结构存在较大差异。第7天及第20天(与第7天基本相同,未列出)大体上持续第三天的情形,说明0:1组由于缺乏营养物质微生物持续休眠,而1:2和2:2组少数能够分解菌渣中营养物质的菌群持续繁殖,保持较高活性。第60天时,各组微生物多样性均得以恢复,说明微生物菌群经过调整后逐渐变得丰富,但组间差仍然很大,聚类分析(图4)表明三个处理组的组间相似度仅49%,明显低于0天的82%,这说明虽然60天后,三个处理组的微生物丰富度有一定程度恢复,但相似度却未增加。

图4 样品DGGE指纹图谱聚类分析
2.3 样品微生物多样性分析
利用Quantity one软件提取DGGE指纹图谱数据并分析多样性指数H及均匀度指数E,结果如图5。结果表明,各组微生物多样性及均匀度都呈现先下降后恢复趋势,但添加了营养物质菌渣的处理组微生物恢复较快,但60天后其2个指数均不能恢复至初期水平,这说明添加菌渣有利于体系中微生物快速恢复繁殖,但总体数量下降了。

图5 样品微生物多样性指数H(左)及均匀度E(右)
2.4 基因测序分析
第0天的样品条带2测序结果与沙门氏菌(Salmonellaenterica)相似度99%,为肠细菌科(Enterobacteriaceae.),分离自人或动物的肠道[8];条带3的测序结果与UnculturedChloroflexibacterium的相似度99%,Björnsson L等[9]通过FISH探针法研究表明该菌在生物活性污泥中含量丰富,是废水生物处理中重要微生物之一;条带4(河氏菌属,Hahellasp.,97%)、5(红环菌属,Rhodocyclaceaebacterium,99%)、8(红长命菌属,Rubrivivaxgelatinosusstrain,98%)则分别分离自海洋沉积物[10]、混合生物膜活性污泥[11]、湖泊沉积物[12]中。
第3天的样品条带3测序结果与厌氧绳菌属(Anaerolineaceaebacterium)98%相似,根据Yamadat等研究表明[13],其对青霉素敏感,故可推测其受到菌渣中残留青霉素的抑制作用而在2、3处理组未发现。条带5结果与埃氏巨型球菌(Megasphaeraelsdenii)99%相似,Marounek等[14]研究表明,埃氏巨型球菌以有机酸、氨基酸等有机物为底物,可耐受低浓度的青霉素,因此可以推测埃氏巨型球菌可能因缺乏易降解的营养物质而未成为第1处理组的优势菌属,又可能因高浓度青霉素抑制而未成为第3处理组的优势菌属。条带9测序结果与弯曲乳酸杆菌(Lactobacilluscurvatus)相似度99%,在第2、3处理组的强度高于第1处理组,Danielsen[15]、Mataragas等[16]研究发现Lactobacilluscurvatus可耐受较高浓度青霉素,说明菌渣中残留的青霉素对其无明显影响。另外,据报道,与条带5、9相近的菌属均为严格厌氧的化能有机异养型微生物,能够降解多糖、果胶、蛋白质等复杂的大分子聚合物,推测第3天时填埋处于厌氧反应的水解、酸化阶段。
第7天的样品条带4测序结果与梭菌属(Clostridiumjejuense)97%相似,研究表明Clostridiumjejuense为严格异养厌氧菌,能利用葡萄糖、氨基酸等发酵产生甲酸、乙酸、CO2等[17],说明该菌属能加速菌渣中有机物的降解。条带5与Sporanaerobacteracetigenes99%相似,研究表明Sporanaerobacteracetigenes[18]也是严格异养厌氧菌属,可利用葡萄糖、氨基酸等小分子有机物为底物,发酵产生乙酸、H2、CO2等,故可推测第2处理组此时可能进入有机物厌氧降解的第二阶段(产氢产乙酸)阶段。条带7(消化链球菌属Peptostreptococcusrussellii)、8(Peptostreptococcusanaerobius)在第3处理组强度最高,可利用葡萄糖等易降解有机物发酵产生醋酸、乳酸、甲酸等[19],因此推测第3处理组也已进入产氢产乙酸阶段。填埋20天的测序比对结果与第7天基本相同,故不再赘述。
由于数据库有限,第60天的样品测序结果未发现比对结果,说明第60天时微生物群落结构发生较大变化。
3 结论
(1)利用二沉池污泥与抗生素菌渣协同消化有利于加速抗生素降解进程,减轻残留物对环境的影响。
(2)PCR-DGGE指纹图谱的微生物多样性分析发现,在填埋不同时期,不同处理组的微生物菌落结构发生明显改变。不同处理组的微生物多样性演变都经历了从减少到恢复的过程,但群落结构发生了难以恢复的改变。
(3)测序分析表明,与不含菌渣的0:1处理组相比,菌渣与污泥混合填埋处理时,第3天发现与可将复杂大分子聚合物水解、酸化为单体或小分子聚合物的菌群Megasphaeraelsdenii、Lactobacilluscurvatus同源的DNA片段,第7天样品中发现与可利用小分子有机物或单体发酵产生甲酸、乙酸、H2、CO2等小分子化合物的菌群Clostridiumjejuense、Sporanaerobacteracetigenes、Peptostreptococcusrussellii、Peptostreptococcusanaerobius同源的DNA片段。
