基于非靶向代谢组学的促乳腺癌曲妥珠单抗耐药代谢产物分析*
2022-11-17董春涛魏棒棒曹怿玮张文钧许飞飞司鑫鑫
董春涛,魏棒棒,曹怿玮,张文钧,许飞飞,司鑫鑫**
1江苏海洋大学药学院 江苏省海洋药物活性分子筛选重点实验室,连云港 222005;2 南京医科大学 药学院,南京 211166
乳腺癌可依据分子分型分为管腔A 型、管腔B型、人类表皮生长因子受体2(HER2)阳性型和基底样型4 个亚型[1]。在乳腺癌患者中约有20%被诊断为HER2 阳性乳腺癌,因其预后差,复发率高,严重威胁到女性的生命安全。
目前,在临床上主要采用曲妥珠单抗(trastuzumab)联合化疗药物治疗HER2 阳性乳腺癌,但随着曲妥珠单抗的广泛应用,耐药问题日益突出。由于耐药诊断的滞后性,造成了曲妥珠单抗耐药患者不能得到及时的诊断,使患者错过最佳治疗窗,生存期降低。因此临床亟需耐药的预测指标以及逆转耐药的有效治疗方法。
非靶向代谢组学(untargeted metabolomics)是对整个代谢组进行系统地分析,从样本中获取物质分子及离子碎片信息,鉴定和识别大量代谢物,以发现用于疾病诊断的新型生物标志物[2]。目前,利用超高效液相色谱-串联质谱(UHPLC-MS/MS)非靶向代谢组学方法探究曲妥珠单抗耐药的报道很少。本研究选择SK-BR-3 和HCC1954 两株HER2 阳性乳腺癌细胞进行研究。
SK-BR-3 细胞是从女性乳腺癌患者胸腔积液中分离出来,HER2 蛋白过度表达,并对曲妥珠单抗敏感[3],HCC1954 细胞源自原发性ⅡA 期,3 级浸润性导管癌,HER2 蛋白过度表达,对曲妥珠单抗耐药[4]。上述两株细胞之间的差异筛选已被广为认可,已有研究从SK-BR-3 和HCC1954 细胞中筛选差异基因,用于曲妥珠单抗耐药诊断生物标志物的研究[5]。因此,本研究通过检测两株细胞之间代谢物含量变化,筛选差异代谢物,用于曲妥珠单抗耐药诊断的后续研究。
现采用了主成分分析(PCA)、正交偏最小二乘-判别分析(OPLS-DA)、层次聚类分析、相关性分析以及KEGG 通路富集分析,筛选出差异代谢物,为临床开发新的曲妥珠单抗耐药标志物和治疗靶点奠定基础。
1 材料与方法
1.1 仪器、材料与试剂
1.1.1 仪器 Dionex UltiMate 3000 液相色谱仪和Q ExactiveTM组合型四极杆OrbitrapTM质谱仪(美国赛默飞公司);CKX41 生物显微镜(日本奥林巴斯);真空离心浓缩仪(美国Labconco 公司);十万分之一AUW220D 电子天平(日本岛津公司);Allegra 64R台式高速冷冻离心机(美国贝尔曼库尔特公司)。
1.1.2 材料与试剂 McCoy’s 5A Medium 培养基粉末、对乙酰氨基酚、乙腈、甲醇和甲酸购自美国Sigma 公司;胎牛血清(FBS)和RPMI 1640 培养基粉末均购自美国Gibco 公司;HER2 阳性SK-BR-3和HCC1954 乳腺癌细胞,系购自美国模式培养物集存库(ATCC)。
1.2 方法
1.2.1 细胞培养 HER2 阳性乳腺癌曲妥珠单抗敏感细胞SK-BR-3,用含15%胎牛血清(FBS)和1%青霉素与链霉素的McCoy’s 5A Medium 培养基中培养。HER2 阳性乳腺癌曲妥珠单抗耐药细胞HCC1954 细胞,在含10% FBS 和1%青霉素与链霉素RPMI 1640 中的培养基中培养。SK-BR-3 和HCC1954 细胞均在37℃、5%CO2的环境中培养,细胞每两天换液一次,5~7 天需要传代一次,使用0.25%胰蛋白酶消化贴壁细胞使其悬浮,加入到含有新培养基的培养皿中,两株细胞系均培养5 皿,提取细胞代谢物。
1.2.2 细胞代谢物提取与样本制备 在细胞培养皿中加入1.5 mL 的超纯水,放置2 h 于-80℃冰箱内,以甲醇配制的1 μg·mL-1对乙酰氨基酚4.5 mL于培养皿中,用细胞刮刀轻轻刮下细胞,吸取细胞裂解液。再次加入预冷的75%甲醇水2 mL 于培养皿中,将两次的细胞裂解液进行合并,涡旋离心,取上清液,4℃旋转蒸发,用80%甲醇水复溶样品。在每个样本中各吸取10 μL 复溶后的溶液,制成质量控制(QC)样本,用来判断仪器稳定性与数据可靠性。
1.2.3 色谱条件 色谱柱:00D-4725-AN Kinetex EVO C18(100 mm×2.1 mm,2.6 μm);流动相A 为0.1%甲酸水溶液,流动相B 为0.1%甲酸乙腈溶液[线性梯度:B 10%(0 min)→30%(1 min)→95%(19 min)→95%(20 min)→10%(20.1 min)→10%(23 min)];柱温:40℃;流速:0.4 mL·min-1;进样量:5 μL。
1.2.4 质谱条件 Q ExactiveTM组合型四极杆OrbitrapTM质谱仪,分别采用电喷雾电离(ESI)正离子与负离子两种模式检测。ESI 源条件如下:毛细管温度:300℃,喷雾电压:±3.5 kV;质荷比(m/z)67~1000为一级质量扫描范围,二级碰撞能(30±15)eV,二级质量扫描范围m/z 为30~1000。
1.2.5 数据处理 采用MS-Covert 软件将原始数据格式转化为mzML 格式,利用MS-DIAL 软件对非靶向代谢组学数据进行程序化处理。将采集到的数据进行峰提取、解卷积、峰对齐和化合物的识别,得到化合物的峰值列表。列表中包含峰强度、峰面积、质荷比、保留时间与化合物离子碎片等重要信息[6]。
1.2.6 数据分析 数据分析包括统计学分析和生物信息学分析。主成分分析(PCA)和正交偏最小二乘判别分析(OPLS-DA)通过SIMCA-P(v14.0)软件完成。在分析过中需要判断模型的拟合程度。在回归分析中,通常用R2判断模型拟合性的效果,用Q2表征模型的预测能力,当R2和Q2越接近1 时,模型的稳定性和预测能力越好[7]。利用OPLS-DA 分析中获得变量重要性投影(VIP)值与差异倍数(FC)和P值进行差异代谢物的筛选,以VIP >1、P <0.05、FC >1.5 或FC <0.67 作为代谢物显著上调或下调的筛选标准[8]。代谢物鉴定是通过检索人类代谢组数据库(HMDB)和MassBank 数据库对分子离子峰和二级碎片进行预测,以及通过标准品进行验证。生物信息学分析主要包括层次聚类分析、相关性分析和KEGG 通路富集分析。通过层次聚类分析可以直观地体现代谢物间和样本间的关系;相关性研究可以观察代谢物之间的表达模式的异同;通路富集分析可对差异表达代谢物进行功能注释。
2 结果
2.1 耐药和敏感细胞代谢组分析
2.1.1 PCA 分析结果 通过PCA 分析,发现在正负离子两种模式下R2x均为0.707,表明了模型的拟合性较好。QC 样本聚集明显,表明仪器稳定性较好,实验数据结果稳定可靠。曲妥珠单抗耐药细胞和曲妥珠单抗敏感细胞之间分离趋势较为明显,说明组间具有显著性差异。PCA 得分图如图1 所示。

图1 曲妥珠单抗敏感细胞(SK-BR-3)和耐药细胞(HCC1954)在ESI 正离子(1A)和负离子(1B)PCA 得分图
2.1.2 OPLS-DA 分析结果 OPLS-DA 可以将与分类无关的信息过滤,从而能够准确分析两组样本之间的差异性。ESI 正离子模式下:R2x=0.765,R2y=0.993,Q2y=0.945,ESI 负离子模式下R2x=0.631,R2y=0.982,Q2y=0.933,表明模型拟合性较好,模型预测能力较强。两组样本之间的分离程度较为明显,两组细胞间代谢物有显著性差异,如图2 所示。
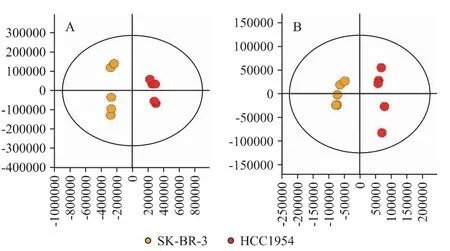
图2 曲妥珠单抗敏感细胞(SK-BR-3)和耐药细胞(HCC1954)在ESI 正离子(2A)和负离子(2B)OPLS-DA 图
2.2 差异代谢物鉴定与分析
2.2.1 差异代谢物鉴定 如图3 火山图所示,在正负离子模式下共得到3439 个代谢峰。以VIP>1、P<0.05、FC>1.5 或FC<0.67 作为代谢物显著上调或下调的筛选标准,得到383 个显著差异代谢峰,依据MS-DIAL 数据库分析对这些代谢峰的注释,筛选出15 个具有显著差异的内源性代谢物,为了进一步验证数据的准确性,利用HMDB 和MassBank数据库对上述代谢物进行鉴定,将实验所得分离离子峰和特征碎片离子峰与数据库进行匹配,结果发现与数据库一致。

图3 差异代谢峰火山图分布
另外对正亮氨酸、谷胱甘肽、谷氨酸、谷氨酰胺和色氨酸进行了标准品验证,分子离子峰和碎片离子峰均能与实验结果相一致,代谢物鉴定与筛选结果如表1 所示。然后对15 个差异代谢物进行分类,其中氨基酸及其衍生物5 种,脂肪酸类3 种、肉碱类3 种,葡萄糖类衍生物2 种,核苷酸类和磷脂类各1 种。

表1 差异代谢物信息
2.2.2 层次聚类分析 通过对代谢物进行层析聚类分析,更直观地观察各个样本之间的关联性。热图结果显示,谷氨酰胺、丙酰肉碱、正亮氨酸、谷胱甘肽、L-谷氨酸、色氨酸和L-肉碱在耐药细胞株中显著上调,其中曲妥珠单抗耐药组的丰度明显高于敏感组的代谢物,大部分为氨基酸及其衍生物。在耐药细胞中,6-磷酸葡萄糖酸、磷酰乙醇胺、D-葡萄糖酸、棕榈酸、硬脂酸、棕榈酰肉碱、次黄嘌呤和肉豆蔻酸下调,如图4 所示。
2.2.3 相关性分析 通过相关性分析可以帮助揭示代谢物之间的关联性。相关性结果显示,棕榈酸与肉豆蔻酸的变化趋势相同,呈正相关,相关性较强。丙酰肉碱与谷氨酰胺和L-肉碱的变化趋势差异较大,呈负相关;L-肉碱与肉豆蔻酸和硬脂酸呈负相关。相关性分析如图5 所示。
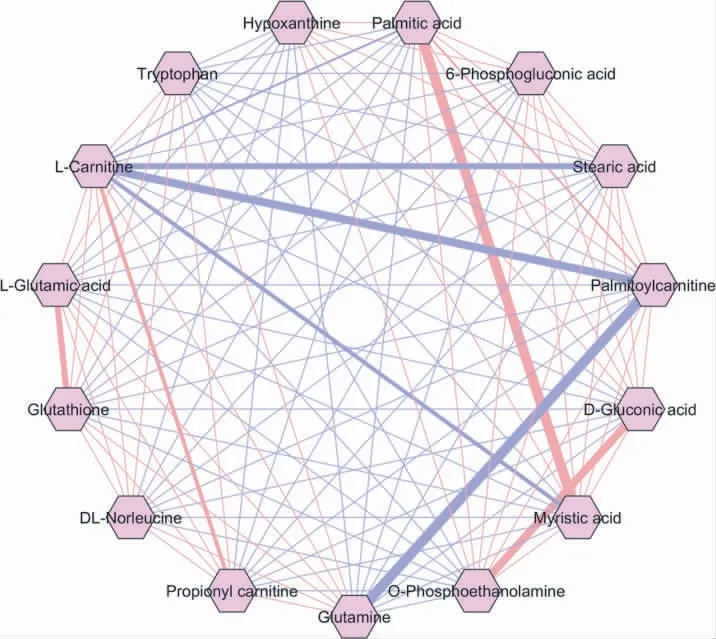
图5 相关性分析
2.2.4 差异代谢物的KEGG 富集通路分析 通路富集分析的气泡图如图6 所示,结果发现,有9 条代谢通路显著富集(P<0.05),其中氮代谢和谷氨酰胺与谷氨酸代谢通路最为显著,P 值为0.001 3。代谢通路信息如表2 所示。

表2 代谢通路信息统计
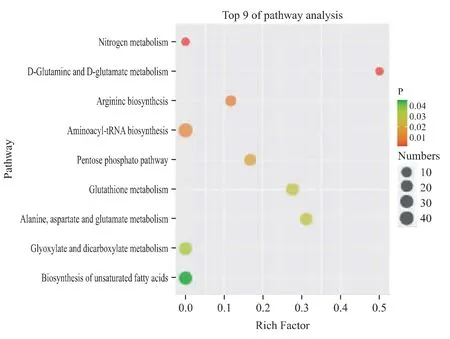
图6 通路富集分析
在9 条代谢通路中映射出的代谢物包括硬脂酸、色氨酸、谷氨酰胺、L-谷氨酸、谷胱甘肽、L-葡萄糖酸、棕榈酸和6-磷酸葡萄糖酸,其中谷氨酸和谷氨酰胺在多条代谢通路中被发现。
3 讨论
本研究基于非靶向代谢组学技术,以VIP>1、P<0.05、FC>1.5 或FC<0.67 作为代谢物上调或下调的筛选标准。在曲妥珠单抗敏感和耐药的乳腺癌细胞中筛选出15 个显著差异的代谢物,其中上调代谢物7 个,下调代谢物8 个。通过通路富集分析发现了氮代谢、谷氨酰胺与谷氨酸代谢和不饱和脂肪酸生物合成等9 条被显著富集的代谢通路,其中氮代谢、谷氨酰胺与谷氨酸代谢通路最为显著,并且在这两条代谢通路中都发现了谷氨酰胺和谷氨酸。因此,在代谢通路中,谷氨酰胺和谷氨酸的改变可能与肿瘤的发生、发展和耐药有关。
氮代谢在生物体内发挥着重要作用,氮是各种生物分子合成的必需物质,可以为细胞生长提供所需的核苷酸、己糖胺等物质[9]。肿瘤细胞增殖摄入大量核苷酸以维持细胞生长,研究发现,谷氨酰胺转运体SLC1A5 在肿瘤细胞中增加,加快了谷氨酰胺的转运,进而加速了氮代谢过程,为肿瘤细胞提供自身和合成所需的物质[10]。有研究对氮代谢通路中核糖焦磷酸氨基转移酶(PPAT)进行探索,发现磷酸核糖PPAT 的表达在肺腺癌中显著增加,对谷氨酰胺的氮转移做出了重大贡献,且与患者的疾病预后有关[11]。在氮代谢过程中,细胞内谷氨酸在谷氨酰胺合成酶作用下可以合成谷氨酰胺,有研究表明,通过谷氨酰胺合成酶作用产生的谷氨酰胺对于核苷酸生物合成在恶性胶质瘤等癌症中尤其重要[12]。
谷氨酰胺与谷氨酸代谢通路可以通过消耗谷氨酰胺和谷氨酸为三羧酸循环提供碳源,从而进一步支持肿瘤细胞的快速增殖[13]。细胞内的谷氨酰胺通过谷氨酰胺酶(GLS1)作用转化为谷氨酸,通过谷氨酸进一步生成α-酮戊二酸,为柠檬酸和脂肪酸的生物合成的三羧酸循环提供碳源[14]。有研究发现,肿瘤细胞对谷氨酰胺的分解具有依赖性,在谷氨酰胺代谢通路中,Wise DR 等[15]研究发现,谷氨酰胺被GLS1 分解得到的α-酮戊二酸参与三羧酸循环,为肿瘤细胞提供能量,维持肿瘤细胞的生长。有研究报道,在ER 阴性的乳腺癌中发现GLS1 过度表达,与肿瘤预后不良明显相关[16]。Zhao Y 等[17]研究了5-氟尿嘧啶可以增强GLS1 抑制,增加对直肠癌的抗肿瘤活性。
此外,既往研究也表明,谷胱甘肽代谢和磷酸戊糖代谢通路与多种恶性肿瘤的发生发展及耐药密切相关。在肿瘤细胞中需要谷胱甘肽代谢产生大量谷胱甘肽与活性氧维持平衡,减少活性氧对肿瘤细胞的损伤[18]。磷酸戊糖代谢通路不仅可以产生磷酸戊糖和核糖核苷,还可以为肿瘤细胞提供能量,并在细胞氧化还原稳态中起关键作用[19]。因此,在HER2 阳性乳腺癌曲妥珠单抗耐药细胞代谢通路中,蛋白或酶的改变可能与发生耐药有关。
在代谢通路中,代谢酶活性增强或减弱可能直接导致了下游代谢物发生改变。通过对曲妥珠单抗耐药细胞下游代谢物含量检测,可寻找用于曲妥珠单抗耐药诊断的标志物。本研究中发现谷氨酰胺和谷氨酸在多条代谢通路中发挥作用,尤其是在氮代谢和谷氨酰胺与谷氨酸代谢通路中,不仅为肿瘤细胞提供生长所需的核苷酸等必需物质,还为肿瘤细胞提供能量以支持其快速生长。因此,谷氨酰胺和谷氨酸的含量在代谢通路中发生改变可能与多种恶性肿瘤的发生发展以及耐药密切相关。既往研究表明,谷氨酰胺摄取量增加与肾细胞癌舒尼替尼的耐药有关[20]。在转移性去势抵抗性前列腺癌患者血浆中,较高的谷氨酰胺与不良预后有关,可以作为预后标志物对耐药患者进行诊断[21]。乳腺癌对阿霉素耐药的细胞中谷氨酸高度表达,并与乳腺癌的恶性程度有关[22]。在转移性食管鳞状细胞癌患者血清中,检测到谷氨酸水平较非转移性食管鳞状细胞癌明显上升,与恶性肿瘤预后不良有关,是诊断转移性食管鳞状细胞癌的重要血清标志物[23]。因此,谷氨酰胺和谷氨酸可能作为标志物用于HER2 阳性乳腺癌曲妥珠单抗耐药的诊断。
在HER2 阳性乳腺癌曲妥珠单抗耐药细胞中检测到的谷胱甘肽、色氨酸和次黄嘌呤还可能与肿瘤的发生发展及耐药有关。有研究表明,谷胱甘肽在对顺铂耐药的肺癌脑转移患者中高度表达,与标志物GPX4 和GSTM1 上调蛋白密切相关[24]。血清中犬尿氨酸和色氨酸的比值可用来评价卵巢癌对顺铂的耐药程度及预后[25]。次黄嘌呤可作为血清标志物用来预测卵巢癌铂类耐药[26]。这些代谢物在辅助诊断乳腺癌曲妥珠单抗耐药中可能具有潜在的价值。
通过对曲妥珠单抗耐药和敏感细胞中的差异代谢物以及代谢通路分析,初步发现了谷氨酰胺和谷氨酸等差异代谢物作为辅助诊断HER2 阳性乳腺癌曲妥珠单抗耐药的价值。另外在代谢通路中所涉及的代谢酶可能是治疗曲妥珠单抗耐药的潜在靶点。但是,本研究中基于细胞模型所筛选出的代谢物差异,尚未被证明与曲妥珠单抗耐药具有因果关系。因此,还需通过靶向代谢组学方法对细胞以及大量临床样本分析进行结果的验证。
4 小结
综上所述,本研究通过UHPLC-MS/MS 技术,采用非靶向代谢组学方法分析了HER2 阳性乳腺癌耐药和敏感细胞之间代谢物的差异,筛选出谷氨酰胺和谷氨酸等15 个差异代谢物,挖掘出9 条代谢通路具有统计学意义,从这些代谢通路中可能会寻找到一些与逆转曲妥珠单抗耐药有关的潜在靶点,为研究曲妥珠单抗耐药提供了一些思路;并且为进一步研究HER2 阳性乳腺癌细胞对曲妥珠单抗敏感,向曲妥珠单抗耐药转变过程中代谢物的差异变化,以及为生物标志物的探索提供了代谢组学的实验依据,为曲妥珠单抗耐药的研究提供相关理论参考。
