基于改进SegNet癌变组织图像语义分割算法
2022-11-15孟凡宏黄丹飞钟艾琦陈思阳赵成龙
孟凡宏,黄丹飞,钟艾琦,陈思阳,赵成龙
(长春理工大学 光电工程学院,长春 130022)
近年来,全球癌症的发病率和死亡率逐年上升,其中肺癌仍位居我国恶性肿瘤发病首位,高发癌症还有肝癌和甲状腺癌等,严重威胁着人类健康。以往的研究表明,偏振成像技术能够根据组织正常和恶性变化之间的微观结构差异,表现出不同的偏振特性[1]。背向散射光穆勒矩阵成像技术是一种有潜力的癌变组织表征手段,提供了反映组织微观结构信息的穆勒矩阵分解和变换参数,在检测组织微观结构方面有着独特优势。由于手术切除癌变组织干净与否很大程度上决定术后恢复的情况,得到准确完整的癌变组织位置是关键。然而目前作为癌症诊断“金标准”的病理切片显微镜检查需经过染色处理才能定性判断是否存在癌变,存在耗时长和易破坏组织微观结构的缺点,不利于大规模的研究和临床应用。临床上希望送检前给医生快速准确的边缘状况或癌变的具体位置,可以进行补切,避免二次手术和复发。所以研究一种准确的、快捷的针对穆勒矩阵参数图像癌变和正常组织分割算法是尤为重要的。
基于传统影像学分割肿瘤的方法有阈值、区域增长、基于统计学、基于模糊理论的分割方法[2]。近年来,深度学习被广泛应用于医学图像分割,Fukushim受生物学的启发,提出了卷积神经网络(CNN)的前身[3]。卷积网络通过卷积网络的端到端训练、像素到像素的训练等,可以达到语义分割的目的[4]。吕力兢[5]利用卷积神经网络及解码网络,实现了腺体的端到端的分割。国内外研究多采用染色处理后的病理切片图像,侧重于组织的光学强度信息,忽略了反映组织微观特征的偏振信息。本文对未染色、具有偏振信息的病理切片进行学习。为了给医生提供客观可靠的结果,定量分析癌变和正常组织至关重要。杨东等人[6]提出使用图像纹理特征参数定量比较乳腺导管组织不同阶段的差异。
为了减少术中癌残留,降低手术二次切缘的比例,同时量化组织信息,本文结合生物组织偏振特性和深度学习,对未染色病理切片的穆勒矩阵参数图像利用改进的语义分割网络叠加高级语义信息和低级语义信息,分割出癌变和正常组织,最后分别提取癌变和正常区域的图像纹理特征、中心矩参数,以期为癌症的病理诊断提供即时准确的组织定量分析。
1 基本原理
本文提出改进的语义分割网络模型,将生物组织偏振特性和深度学习结合,对正常与癌变组织的穆勒矩阵参数图像的差异进行学习,并采用图像纹理特征定量分析两区域的差异。首先使用背向散射光穆勒矩阵自动成像系统分别对肺癌和肝癌病理切片进行穆勒矩阵成像,获得相应的穆勒矩阵分解和变换参数图像。将选取的穆勒矩阵分解和变换参数图像进行加权平均融合后,采用数据增强方法以扩充数据集,将数据输入到语义分割网络中,进行三类分割,分割出背景、正常和癌变区域。最后分别提取正常和癌变区域的纹理特征,分析两区域的差异。如图1所示,为本文网络结构示意图。
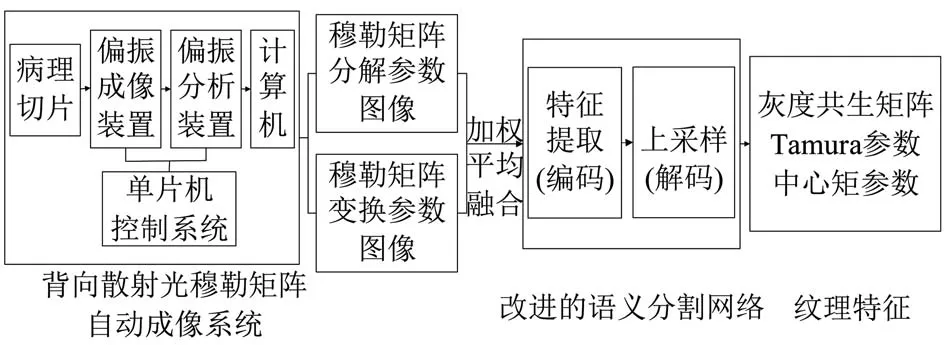
图1 本文网络结构示意图
1.1 穆勒矩阵参数
1.1.1 穆勒矩阵分解
穆勒矩阵极化分解(Mueller matrix polar decomposition,MMPD)是1996年Lu和Chipman提出的一种穆勒矩阵分解方法,将样本的穆勒矩阵分解为3个基本子矩阵的乘积,提取出二向色性D、散射退偏Δ和线性相位延迟δ三个与微观结构有关的参数。在一些癌变组织中,相位延迟参数可能会反映出病变区域原有的纤维结构被破坏引起的局部各向异性程度变化;另一些癌变组织中,未有明显的各向异性程度改变,散射退偏敏感与早期细胞代谢旺盛造成的细胞及细胞器密度增加和细胞核肿大等特征。偏振对于细胞中细胞核、细胞骨架、细胞器的形态、双折射特性以及纤维结构差异具有高敏感性[7-8]。
1.1.2 穆勒矩阵变换
基于穆勒矩阵提取反映微观结构信息的参数的计算方法,还有穆勒矩阵变换(Mueller matrix transformation,MMT)。它是从强度周期性变化的穆勒矩阵阵元中获得一组相互独立的参数,即A、b、t2、t3和 x。参数 A 反映样品各向异性程度;参数b与样品散射退偏、吸收有关;参数x体现各向异性结构在成像面内的方向;t2是散射介质的各向异性程度敏感;t3是组织双折射的指示器。b参数成像对比度主要来自于癌变和正常组织中代谢相关细胞器浓度的差异,A参数成像对比度来自癌变产生的纤维增生或破坏[9]。
1.2 加权平均融合
由于MMPD参数对纤维更敏感,MMT参数更容易快速地计算,因此本文采取加权平均融合方法来增强图像的细节,设F1和F2分别表示两参数图像,则加权平均融合后的图像可表示为:
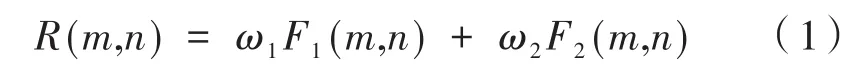
其中,m和n分别表示图像像素行和列号;ω1和ω2分别代表在图像F1和F2中的加权系数。当ω1+ω2=1且ω1= ω2=0.5时,表示加权平均融合,可以使用两幅图像的相关系数来确定公式中的权值,即:
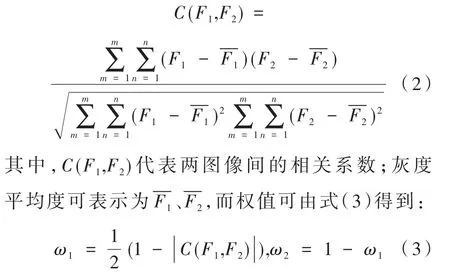
通过结合两参数图像中的像素灰度信息,可以有效减少图像中的噪声,提高信噪比[10]。对于肝癌,选择融合MMPD的D参数图像和MMT的t3参数图像,选择融合肺癌的MMPD的D参数图像和MMT的t2参数图像,通过两个图像的融合得到细节信息更丰富、更为准确的参数图像。
1.3 穆勒矩阵参数图像的语义分割网络
语义分割任务能够预测图像中的每个像素对应的类别。基于CNN的语义分割开始于全卷积神经网络FCN[11],随后的所有语义分割方法,都可以说是基于FCN的改进以及演化[12],如典型的编解码网络 U-Net、SegNet[13]、U-Net++等 。由于医学图像相比自然图像没有丰富的高级语义特征,而且病变区域相对于整张医学图像较小,过多的卷积层会导致小的病变在图像中消失。同时考虑到临床上对于获取癌变具体位置的时间需求,因此提出了一种改进的SegNet语义分割网络模型,通过学习图像所包含的穆勒矩阵偏振信息,将其转化成能够区分正常与癌变区域的特征,提升分割效果。其结构如图2所示。为降低癌变特征的丢失,编码部分将SegNet使用的VGG16的前13层网络替换为改进的VGG11网络作为主干网络,减少使用的卷积层数,去掉VGG11网络的全连接层和最后的分类层;为了统一训练样本使得网络学习速度加快和泛化能力提升,在每个卷积层输出后加上批量归一化层。解码部分为5个最大池化层和5个反卷积层,卷积层和反卷积层的卷积核大小均为3×3,最后得到分割结果图。
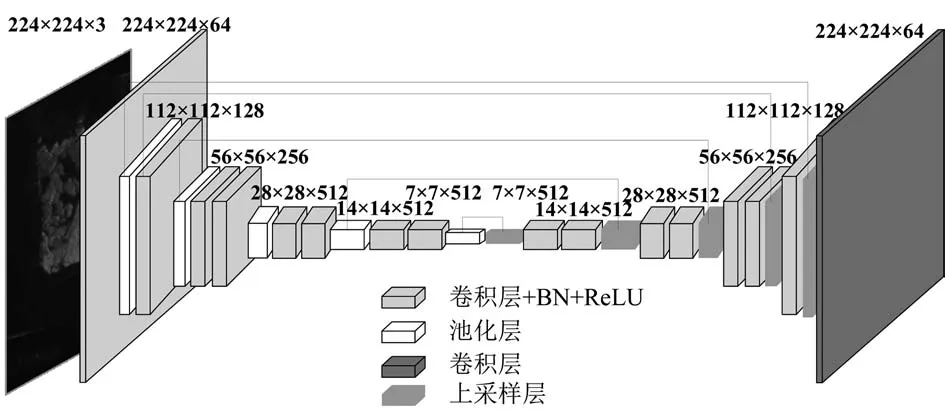
图2 语义分割网络结构
1.4 纹理特征分析
医学图像的纹理能呈现人体组织器官的结构等,因此本文采取纹理特征中灰度共生矩阵、Tamura和中心矩参数对分割出的癌变与正常组织进行定量分析。纹理特征定量分析了相邻像素之间分布的变化。
灰度共生矩阵(Gray level co-occurrence matrix,GLCM)灰度分析是一种考虑图像像素空间分布的图像纹理分析方法[14]。GLCM可导出能量(Energy,Er)、相关性(Correlation,Cr)、对比度(Contrast,Ct)和熵(Entropy)等多个二阶统计纹理特征。能量是对区域的均匀性的度量,相关性是用来表征与相邻区域像素的相关性程度,对比度计算的是与相邻区域像素间的强度对比度。图像纹理特征Tamura包括与视觉感知相对应的六个特征:对比度FCON、粗糙度FCRS、规则性FREG、粗略度FRGH、方向性FDIR和线性FLIN[15]。其中,对比度反映边缘的锐度和重复图案的周期,粗糙度和粗略度表征的是图像的粗糙度或平滑度。中心矩参数期望μ、方差σ2、偏度skewness和峰度kurtosis可由式(4)—式(7)得到:
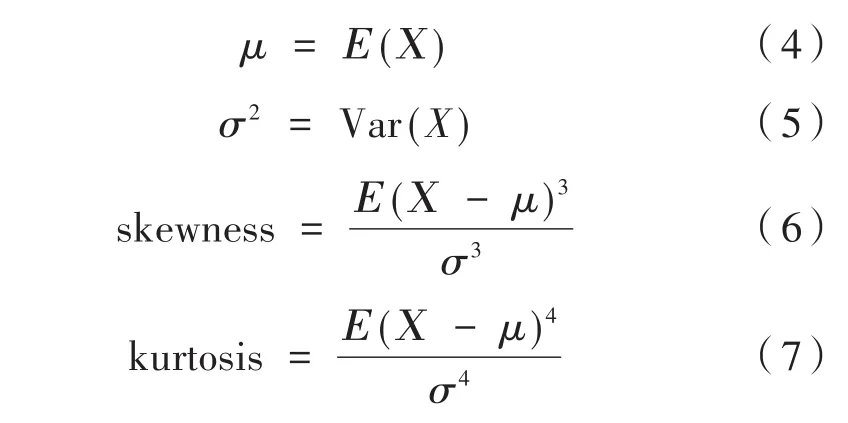
期望反应的是被测参数的中心趋势,方差是衡量数据分布广泛程度,即组织结构复杂程度的参数,偏度显示分布关于均值的不对称性,峰度是分布平坦或峰值程度的描述符。
将分割出的癌变与正常位置的像素分别映射到参数图像上,通过计算分别获得肝癌对应的癌变和正常组织的MMT参数t3的纹理特征:偏度skewness、对比度FCON和粗糙度FCRS等,肺癌对应的癌变与正常组织的MMT参数t2的纹理特征:对比度Ct和方向性FDIR等,来分析癌变与正常区域的差异。
2 实验结果与分析
2.1 数据集
肝癌病理切片采用的是未染色肝细胞肝癌组织,共60张。肺癌病理切片采用的是未染色肺腺癌组织病理切片,共60张。病理切片经穆勒矩阵成像获得的穆勒矩阵进行分解和变换得到参数b和D图像,最后得到加权平均融合图像。分别选择50张肝癌、50张肺癌病理切片作为训练集和验证集的原图进行预处理,剩余图片为测试图片不作处理。训练集中标签的区域标注由人工完成,根据病理医生标注结果,采用Labelme软件进行标注,获得训练集标签。类别癌变组织、正常组织和背景分别对应着标签cancer、normal和_background_,将生成的 json格式标签图转换成png格式标签图,对应的0~2像素值分别对应上面三种类别,如表1所示。由于癌症病理切片数量有限,经过随机裁剪、随机翻转和随机旋转-15°~15°任意角度的预处理,形成数据集。数据集中训练集为3 200个样本,验证集为800个样本,包含三类:癌变、正常组织和背景类。如图3、图4所示,其中图3(a)为肝癌的MMT参数t3图,上方圆形框内为肝癌组织,图3(b)为肝癌的 MMPD 参数 D图,图 3(c)、图3(d)分别为肝癌的一组数据集及对应标签图;图4(a)为肺癌的MMT参数t2图,左侧圆形框内为肺癌组织,图4(b)为肺癌的MMPD参数D图,图4(c)、图4(d)分别为肺癌的一组数据集及对应标签图,癌变、正常组织和背景分别用白色、灰色和黑色表示。

图3 肝癌数据集展示
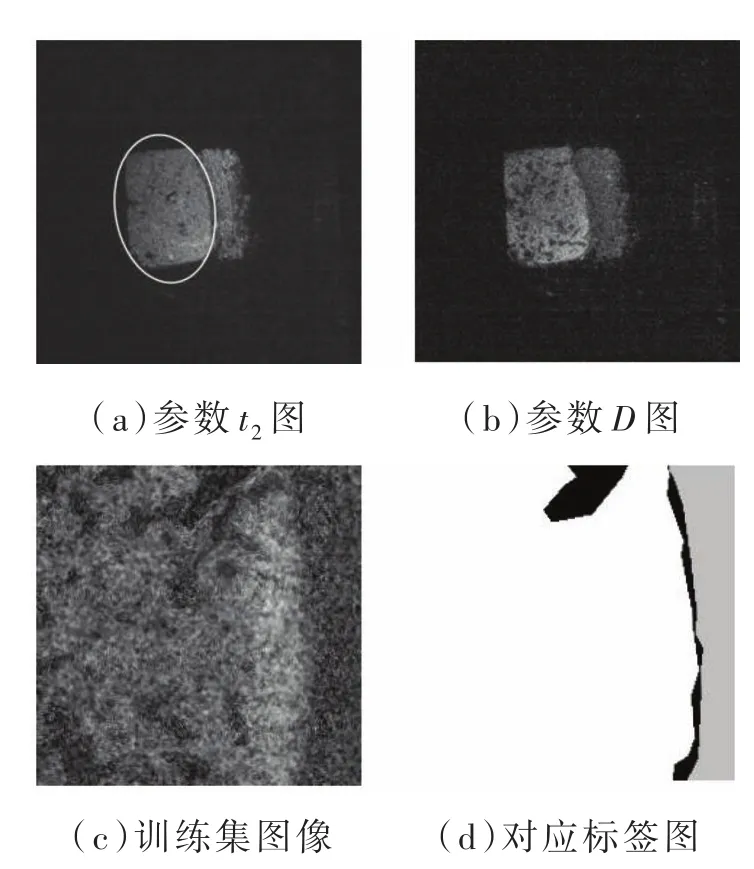
图4 肺癌数据集展示

表1 各算法评价指标比较
2.2 实验结果
本文采用Adam优化器优化损失函数,批量大小设置为8,训练迭代100次,图像大小为224×224。所使用的实验环境为Windows10系统下搭建的Pytorch深度学习框架,并使用英伟达3 090,中央处理器为i9十代处理器。本文实验分别与FCN、U-Net及SegNet算法进行比较,如图5、图6所示分别为FCN、U-Net、SegNet以及本文算法在肝癌和肺癌上的分割效果对比图。

图5 肝癌分割结果


图6 肺癌分割结果
2.3 评价分析
本文主要从主观评价和客观评价两方面来比较和分析实验结果。主观评价是根据视觉效果比较整体和边缘的分割效果。客观评价使用准确性(Accuracy)和平均交并比(Intersection over union,IoU)来评价分割结果,如表2所示为本文对比实验的评价结果。
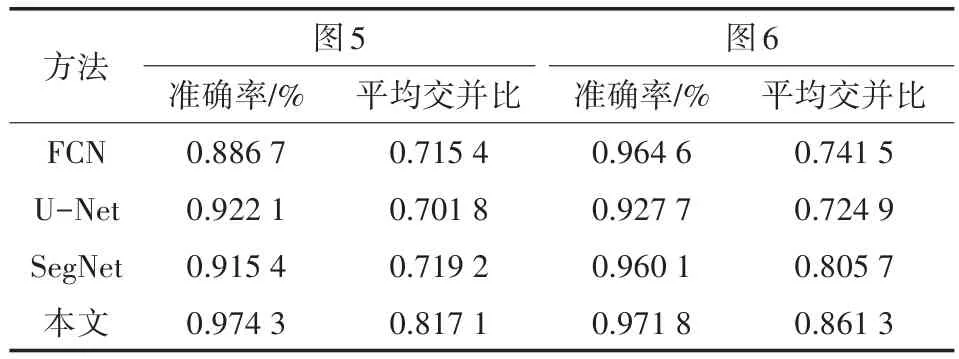
表2 各算法评价指标比较
本文提出的算法相较于U-Net和SegNet而言,肝癌和肺癌分割结果表现的主观效果更好,FCN分割效果良好,但对边缘细节不够敏感。本文方法的分割客观性能指标优于FCN、U-Net和SegNet,对于图5肝癌,总体测试准确性为0.974 3,平均交并比为0.817 1。对比SegNet的分割结果,准确性提升了5.89%,平均交并比提升了9.79%。对于图6肺癌,总体测试准确性为0.971 8,平均交并比为0.861 3,对比SegNet的分割结果,准确性提升了1.17%,平均交并比提升了5.56%,本文算法运行时间短,整体性能较优,在癌变和正常组织分割性能方面有很大的提升。
基于前述分割结果得到的不同区域进行纹理特征参数计算,肝癌和肺癌的相应的图像纹理特征如图7、图8所示,对于肝癌,图5(f)灰色区域MMT参数t3的偏度skewness、粗糙度FCRS和对比度FCON较大,白色区域参数t3的偏度skewness、粗糙度FCRS和对比度FCON较小。对于肺癌,图6(f)灰色区域MMT参数t2的标准差和对比度Ct较大,方向性FDIR较小,白色区域参数t2的标准差和对比度Ct较小,方向性FDIR较大。肝癌和肺癌的白色相对灰色区域更符合癌变特征。
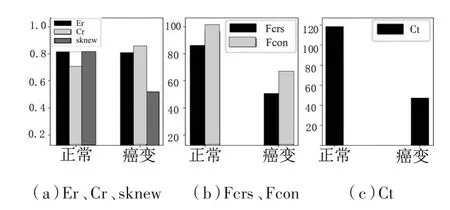
图7 肝癌参数t3图中正常、癌变组织的纹理特征分布
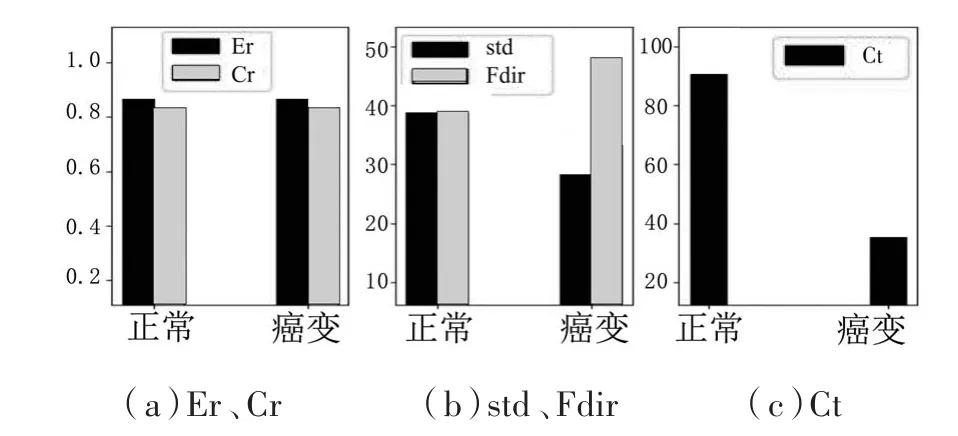
图8 肺癌参数t2图中正常、癌变组织的纹理特征分布
3 结论
提出了一种改进的SegNet语义分割算法,对癌变与正常组织的穆勒矩阵参数图像进行分割。首先,采用加权平均融合处理穆勒矩阵分解和变换参数图像,增强图像细节信息;然后在特征提取部分使用改进的VGG11网络作为主干网络,保留癌变组织的更多特征。实验结果表明,本文算法对肝、肺癌癌变组织穆勒矩阵图像具有较高的分割精度,与传统的SegNet算法相比较整体性能较优,在临床中可降低手术二次切缘的比例。
