探针ASPCR检测肺炎支原体微量耐药突变A2063G方法的建立
2022-11-07郭东星胡文娟李静宜吴赵永栗绍刚田秀君辛德莉
郭东星,胡文娟,李 丹,李静宜,吴赵永,栗绍刚,田秀君,辛德莉
肺炎支原体(Mycoplasma pneumoniae, MP)能够引起肺炎支原体肺炎(Mycoplasma pneumoniae pneumonia, MPP)和其他呼吸道感染性疾病[1-3]。大环内酯类抗生素、四环素类抗生素(多西环素及米诺环素等)和氟喹诺酮类药物是临床上常用于治疗MPP的药物。其中,氟喹诺酮类药物是治疗成人MPP的主要药物,由于氟喹诺酮类及四环素类药物可能对儿童的发育造成一定影响,大环内酯类抗生素成为治疗儿童MPP的首选药物。但伴随着抗生素的使用,相应的耐药MP也出现了较大规模的流行,尤其在亚洲地区,MP耐药率呈增高趋势[4-5],给治疗相关疾病带来了极大挑战。
23S rRNA基因可变区域肽基转移酶环的点突变可以干扰大环内酯类药物与rRNA的结合,是MP对大环内酯类抗生素耐药的主要机制[6-7]。其中,A2063G是最常见的突变,可导致MP对大环内酯类药物较高水平的耐药性[4,6,8-11]。前期研究结果表明,在MP感染者体内,耐药菌株与敏感野生菌株以混合菌群的形式存在[12],耐药菌群所占比例与临床用药种类、剂量及治疗效果密切相关。建立有效的检测MP菌群中耐药MP比例的方法,对于制定MPP临床治疗方案和研究MP耐药发生发展规律具有重要意义。本课题组前期研究发现,MP在感染者体内是以混合菌群的状态存在,另有病例报道称,MP感染者体内菌株的药物敏感性可能随着治疗的进程会发生变化[13]。因此,在MP感染者体内,支原体菌群可能随着治疗药物的使用和病程的进展发生耐药变异,并在一定的选择压力下,菌群中耐药菌株和敏感菌株的相对占比也在不断发生变化。目前临床中常用的检测方法仅能够检测是否MP感染或是否存在耐药MP,无法检测耐药MP的相应占比。
本课题组前期建立了检测咽拭子样本中MP耐药突变的ASPCR方法[12]。但在之前的研究中,ASPCR方法多为染料法[12,14-15],在检测临床样本时往往会出现非特异性的扩增,在结果分析时须要结合扩增曲线和熔解曲线进行判读,对临床检测人员的技术要求较高,且会耗费一定时间,一旦出现非特异性扩增就会直接影响耐药菌株比例计算结果的准确性,进而须要对样本进行重新检测。因此,为了克服以上缺陷,我们对前期的染料法ASPCR进行了改进,建立了检测MP A2063G耐药突变的探针ASPCR方法,并对该方法的灵敏度、特异度、准确度和检测临床样本的性能进行验证。
1 材料与方法
1.1 材料
1.1.1 咽试子样本 本研究中用于方法验证的58份临床咽拭子样本采集自2014年民航总医院儿科门诊及住院的临床诊断为疑似MMP的2~12岁患儿。
1.1.2 菌株 MP标准株M129(ATCC 29342)为本实验室长期保存菌株,大肠埃希菌、阴沟肠杆菌、人型支原体、金黄色葡萄球菌、肺炎克雷伯菌、黏液奈瑟菌、铜绿假单胞菌、肺炎链球菌、梨状支原体、发酵支原体、假单胞菌、解脲支原体等用于验证方法特异性的临床分离菌株,均由首都医科大学附属北京友谊医院检验科馈赠。
1.1.3 主要试剂和仪器 基因组提取试剂盒、PCR产物纯化及质粒提取试剂盒购自康为世纪生物科技有限公司。配制实时定量PCR反应体系用的TaqManTMUniversal Master Mix II和ABI Prism@7500型荧光定量PCR仪均为Thermo Fisher公司产品。
1.2 方法
1.2.1 方法建立 引物/探针组合设计:根据MP23S rRNA基因序列(GenBank, accession No.NR_077056)设计特异性引物/探针组(SP-2063-F,2063D-R和S2P-AS-18)和非特异性引物/探针组[NU-F(Np),2063D-R 和 N2P-AS-16](表1)。特异性引物/探针组合在一些特定的基因位点以次黄嘌呤核苷酸代替,以提高特异性引物/探针组合的特异性,最终能够选择性地扩增含有A2063G突变的DNA模板,用于对耐药菌株进行定量;而非特异性引物/探针组合能够扩增所有MP DNA模板,用于对总菌群进行定量。
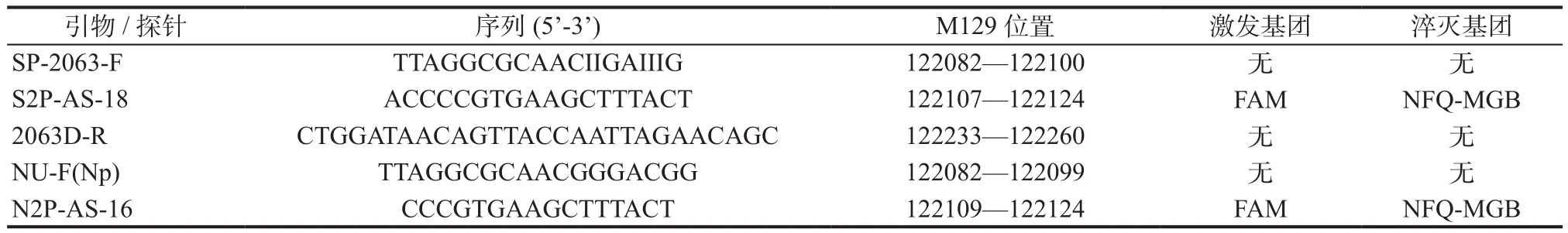
表1 引物及探针序列及其在M129基因组中的位置信息Table 1 Oligonucleotide sequences and locations on the M129 genome
标准品构建:以M129标准株为模板,扩增全长23S rRNA基因片段,连接至pMD18-T载体中,构建含2063位点的野生型质粒;在野生型质粒基础上对2063位点进行定点突变,构建突变型质粒,将2种基因型质粒梯度稀释为10~106拷贝/μl后作为标准品备用。
标准曲线绘制:用特异性引物/探针组和非特异性引物/探针组分别扩增含A2063G突变型标准品(3复孔,Ct=Ct平均值±标准差),横坐标为标准品拷贝数的对数值(lg),纵坐标为相应Ct值,分别绘制出定量突变型模板的特异性标准曲线和定量总MP的非特异性标准曲线。
反应体系:TaqManTMUniversal Master Mix II:10 μl,10 μmol/L 的 SP-2063-F/2063D-R 或 NU-F(Np)/2063D-R 引物各 1 μl,探针 S2P-AS-18或S2P-AS-16 0.5 μl,DNA 模板 1.0 μl,ddH2O 6.5 μl,总体积 20.0 μl。
扩增条件:60 ℃ 30 s;95 ℃ 5 min;95 ℃ 15 s,60 ℃ 1 min,40循环;60 ℃ 30 s,荧光检测信号激发基团和淬灭基团分别选为FAM和NFQ-MGB。
1.2.2 方法性能验证
1.2.2.1 检测MP特异性验证 采用非特异性引物/探针组扩增临床常见感染呼吸道且易与MP发生交叉反应的菌种(大肠埃希菌、阴沟肠杆菌、人型支原体、金黄色葡萄球菌、肺炎克雷伯菌、黏液奈瑟菌、铜绿假单胞菌、肺炎链球菌、梨状支原体、发酵支原体、假单胞菌、解脲支原体)和M129标准菌株扩增结果进行比较,以验证新建探针ASPCR方法检测MP的特异性。
1.2.2.2 检测MP A2063G耐药突变特异性验证 主要从以下2个方面进行验证:①用特异性引物/探针组分别扩增等量的突变型(2063G)与野生型(2063A)DNA模板,比较2者的Ct值的差值(△Ct),△Ct值的大小代表了特异性引物/探针组区分2种基因型能力的强弱,即△Ct值越大表明该方法检测A2063G突变的特异度越好;②用特异性引物/探针组测定野生型和突变型混合模板的Ct值,比较其Ct值与特异性引物/探针组扩增对应等量的突变型DNA模板的Ct值,2者一致性越好,说明该方法检测A2063G耐药突变的特异度越高。
1.2.2.3 检测MP灵敏度验证 用非特异性引物/探针组进行扩增反应, MP DNA模板为10~106拷贝/反应,验证该方法检测MP的灵敏度。
1.2.2.4 检测MP A2063G耐药突变灵敏度验证 使用特异性引物/探针组扩增105拷贝野生型模板,共重复3次,定义临界值=Ct平均值+标准差;使用特异性引物扩增突变比例为0.01%~100%的混合模板,得到相应的Ct值(Ct平均值±标准差)并与临界值进行比较,小于临界值说明混合物中存在突变模板,大于临界值说明突变无法检出,可检出最低突变比例即为该方法检测A2063G耐药突变的灵敏度。
1.2.2.5 准确度验证 用非特异性引物/探针组和特异性引物/探针组分别扩增突变模板含量为0.01%~100%的混合DNA,分别根据标准曲线进行定量,计算出混合模板的突变比例(检测突变比例=特异性扩增模板拷贝数/非特异性扩增模板拷贝数×100%)。比较该方法的检测突变比例与理论突变比例值,2者一致性越高,则该方法的准确度越高。
1.2.2.6 检测临床标本性能验证 采用新建立的探针ASPCR方法、巢式PCR联合DNA测序法和前期建立的染料ASPCR方法分别检测58份临床咽拭子标本,通过比较该方法与其他2种方法的检测结果,评价该方法检测MP感染的性能,通过比较该方法与染料ASPCR方法的检测结果,验证该方法检测野生型和耐药型MP比例的性能。
1.3 统计学处理 采用SPSS 19.0统计软件对实验结果进行统计分析,采用配对t检验对新建探针ASPCR与染料ASPCR方法的检测结果进行统计分析,对ASPCR方法与巢式PCR联合测序方法的检测结果进行阳性符合率、阴性符合率、总符合率分析。以P<0.05为差异有统计学意义。
2 结 果
2.1 标准曲线绘制 标准曲线特异性引物/探针组(SP-2063-F,2063D-R和S2P-AS-18)和非特异性引物/探针组[NU-F(Np),2063D-R和N2P-AS-16]分别扩增含A2063G突变型标准品(3复孔,Ct=Ct平均值±标准差),绘制的标准曲线,见图1。非特异性标准曲线:y=-3.15x+38.95,R²=1.00;特异性标准曲线:y=-3.2044x+38.961,R²=0.999;2条标准曲线几乎重合,表明新建探针ASPCR方法检测模板拷贝数与相应Ct值之间具有良好的相关性,能够应用该标准曲线进行定量。

图1 扩增A2063G的特异性和非特异性标准曲线◆所在直线为非特异性引物/探针组扩增A2063G突变型标准品得到的非特异性标准曲线;●所在直线为特异性引物/探针组扩增A2063G突变型标准品得到的特异性标准曲线Figure 1 Specific and non-specific standard curves of A2063G
2.2 方法性能验证
2.2.1 检测MP特异度验证 新建探针ASPCR方法检测用以验证特异性的菌种,均未出现扩增信号,结果见表2。由此可见,该方法检测MP具有良好的特异度。

表2 探针ASPCR方法的特异度Table 2 Specificity of the probe ASPCR
2.2.2 检测MP A2063G耐药突变特异度验证 ①用特异性引物/探针组分别扩增105拷贝的突变型(2063G)与野生型(2063A)DNA模板,△Ct值约为10.93(图2A);特异性引物/探针组能够正常扩增104和103拷贝突变型模板(见标准曲线),但扩增104和103拷贝野生型模板时,无扩增信号。②将107拷贝/μl的突变型模板视作突变型比例100%,用106拷贝/μl的野生型质粒梯度稀释含2063G的突变模板,得到突变比例为50%~0.01%的混合模板,对该混合模板进行检测,其Ct值仍与相应浓度的纯突变型模板具有良好的一致性(图2B)。说明特异性引物/探针组能够有效区分突变型和野生型,具有良好的检测A2063G耐药突变的特异度。

图2 探针ASPCR方法特异度验证A.分别为特异性引物/探针组扩增105拷贝/μl A2063G突变模板和野生型模板的扩增曲线,△Ctab(Ctb- Cta)=10.93;B.◆所在直线为特异性引物/探针组扩增A2063G突变型标准品得到的特异性标准曲线,●所在直线为特异性引物/探针组扩增梯度稀释的A2063G突变型模板和5×106拷贝/μl野生型模板的混合物DNAFigure 2 Allelic discrimination of the probe ASPCR assay
2.2.3 灵敏度与准确度验证 用特异性和非特异性引物/探针组分别扩增相应的标准品,所得扩增曲线梯度均匀,灵敏度均达10拷贝(图3~4)。

图3 特异性引物/探针组扩增A2063G突变基因的灵敏度特异性引物/探针组扩增A2063G突变型标准品的扩增曲线,从左到右依次为108~101拷贝Figure 3 Sensitivity of the specific primers and probe for testing A2063G
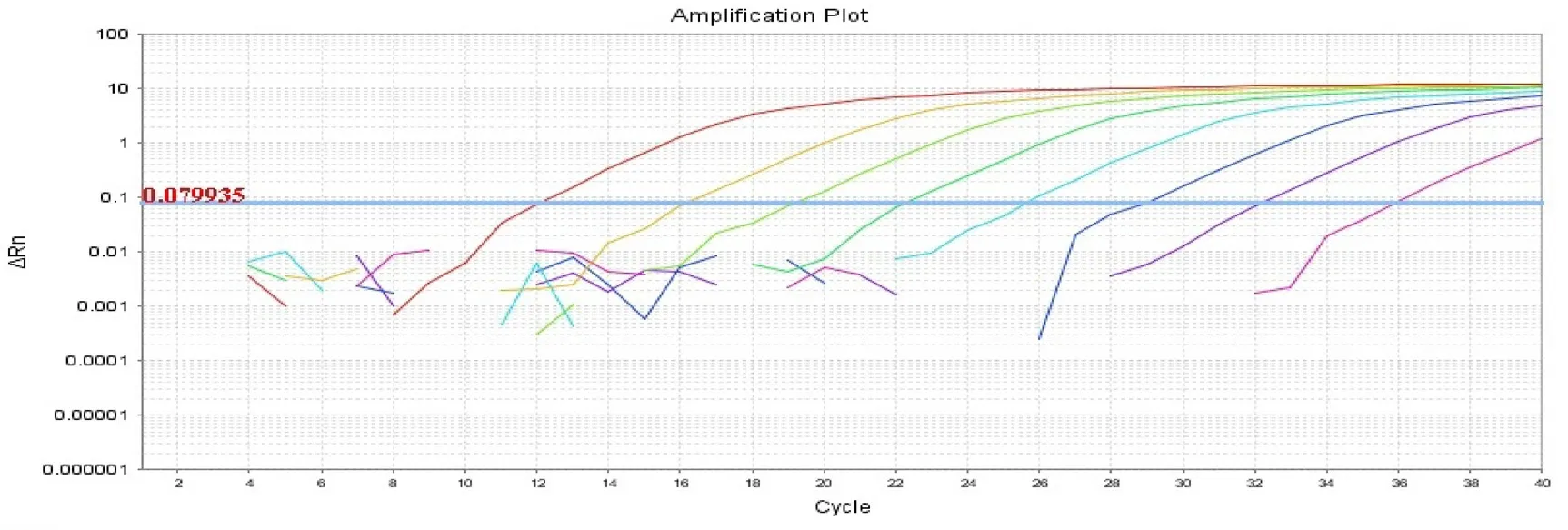
图4 非特异性引物/探针组扩增目的基因的灵敏度非特异性引物/探针组扩增A2063G突变型标准品的扩增曲线,从左到右依次为108~101拷贝Figure 4 Sensitivity of the non-specific primers and probe
根据计算所得临界值为33.96;使用特异性引物扩增突变比例为100%~0.01%的混合模板,得到相应的Ct值均<临界值(图5)。用非特异性和特异性引物/探针组同时扩增混合模板,实际检测突变比例与理论突变比例值具有良好的一致性(图6),说明探针ASPCR方法检测MP A2063G耐药突变具有良好的准确度。另外,当理论突变比例为0.01%时其相应的Ct值仍显著小于临界值;当理论突变模板比例为0.1%时,与检测到的比例具有良好一致性,低于0.1%时,测得值将显著偏离理论值,因此结合灵敏度和准确度的验证结果,最终确定探针ASPCR方法检测MP耐药位点的灵敏度可达0.1%。

图5 探针ASPCR方法检测A2063G灵敏度图中直线为特异性引物/探针组扩增105拷贝/μl野生型模板得到的临界值;●为特异性引物/探针组扩增A2063G突变模板比例为100%~0.01%的混合物所得Ct值Figure 5 Sensitivity of the probe ASPCR assay
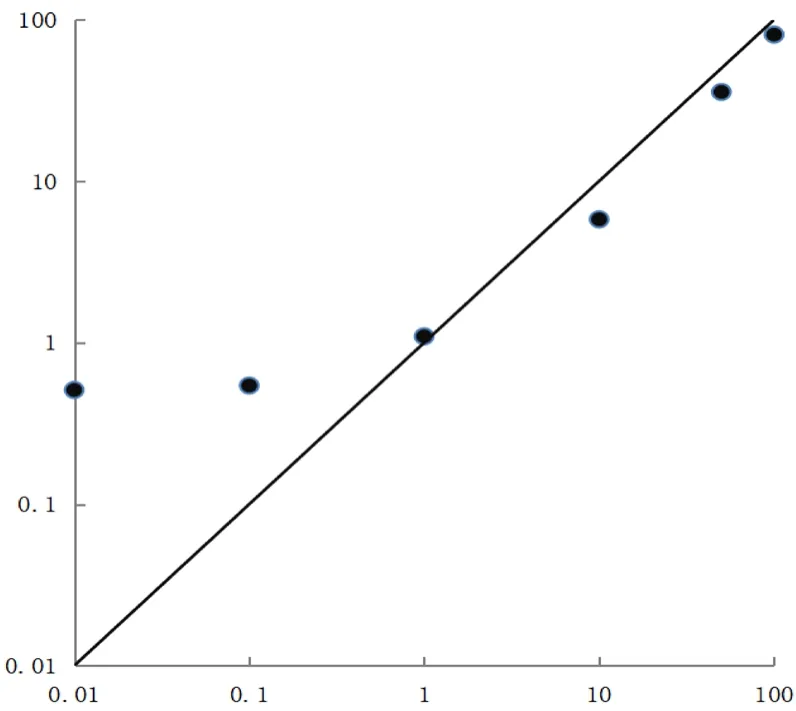
图6 探针ASPCR方法检测A2063G准确度横坐标轴为A2063G突变模板理论比例,纵坐标轴为0.01%~100%的混合模板的检测比例Figure 6 Accuracy of of the probe ASPCR assay
2.2.4 临床标本检测应用 使用新建探针ASPCR方法、染料ASPCR方法和巢式PCR联合DNA测序法分别检测58份临床咽拭子标本。探针ASPCR方法和染料ASPCR方法检测MP感染阴、阳性结果一致,总阳性率相同,均为94.83%(55/58);巢式PCR联合DNA测序法检测MP阳性率为75.86%(44/58),ASPCR和巣式PCR方法同时检测为阳性样本44份,同为阴性3份,阳性符合率是80.00%,阴性符合率是100%、总符合率是81.00%。其中探针法和染料法2种ASPCR方法检测结果中野生型、A2063G突变型和混合菌群的样本数量分别为20/16(<S%)、10/14(100%)、20/21(≥20%,<100%)(见表3)。配对数据t检验结果显示,t=2.057 ,P=0.044,说明染料ASPCR方法检测的A2063G耐药突变比例显著高于探针ASPCR方法检测的结果。探针ASPCR方法检测临床样本的扩增结果见图7~9。

表3 探针ASPCR方法和染料ASPCR方法检测A2063G结果比较(例)Table 3 Comparison of the results of dye-ASPCR and probe-ASPCR for testing A2063G (cases)
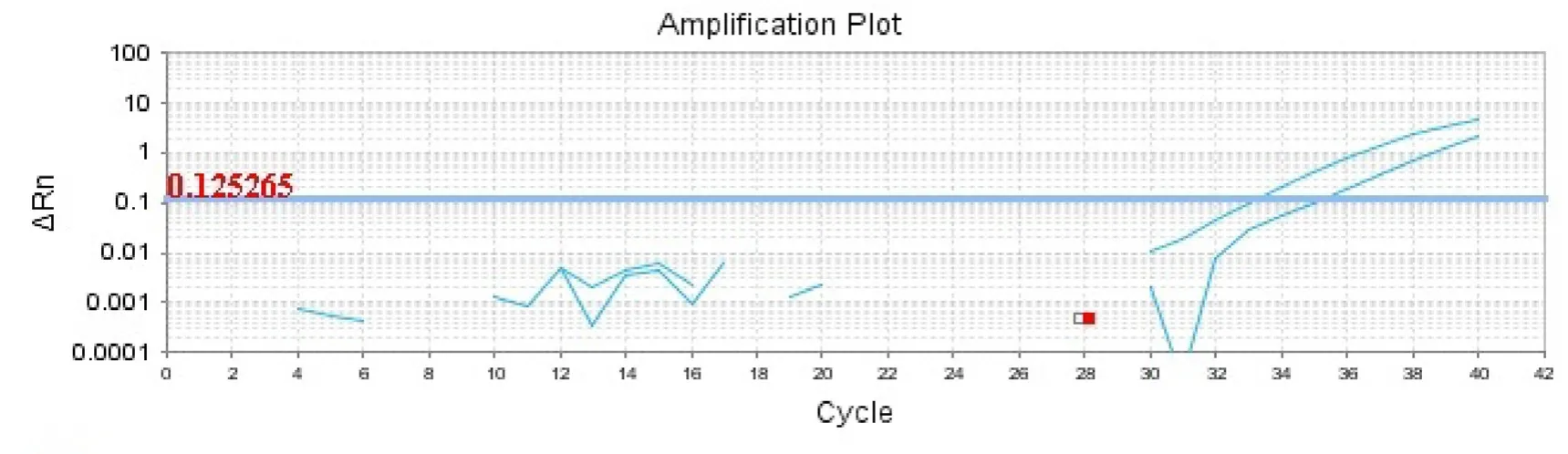
图7 混合基因型临床样本检测结果探针ASPCR方法检测混合(野生型+A2063G)临床样本的扩增曲线:a.是用非特异性引物扩增的扩增曲线;b.是A2063G特异性引物扩增的扩增曲线图,说明样本中肺炎支原体是混合菌群,同时含有野生型和耐药突变型Figure 7 Results of probe ASPCR testing clinical sample with mixed genotype

图8 野生型基因型临床样本检测结果探针ASPCR方法检测野生型临床样本的扩增曲线:a.是用非特异性引物扩增的扩增曲线;b.是A2063G特异性引物扩增反应结果(未发生扩增反应),说明样本中肺炎支原体均为敏感型,无耐药突变Figure 8 Results of probe ASPCR testing clinical sample with wild genotype
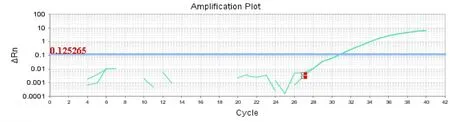
图9 突变基因型临床样本检测结果探针ASPCR方法检测突变型临床样本的扩增曲线,特异性引物/探针组和非特异性引物/探针组扩增样本的曲线完全重合,说明样本中肺炎支原体耐药比例为100%Figure 9 Results of probe ASPCR testing clinical sample with mutant genotype
3 讨 论
位于23S rRNA V区域的A2063G和A2064G点突变能够干扰大环内酯类药物与MP rRNA的结合,是MP耐药发生的主要机制[15-16]。其中,A2063G突变是耐药MP的最主要突变类型[17-18]。本研究建立了具有高灵敏度的探针ASPCR方法,能够准确检测微量A2063G耐药突变。采用该方法能够研究MP感染者体内的不同时期的23S rRNA变异情况,动态监测MP药物敏感性的变化情况,是进一步研究大环内酯类药物耐药发生发展机制的有利工具,并能够为临床治疗方案的制定提供有效依据。
探针ASPCR方法建立的难点在于特异性引物和相应探针组合的设计,要求该引物探针组合能够特异性地扩增突变样本,就需要尽量降低其扩增野生型模板的效率但其扩增突变型模板的效率不能有明显影响。理论上,当引物3’末端碱基与模板碱基不完全匹配时,其扩增反应效率则会大幅降低,甚至无法扩增,但在研究过程中我们发现,特异性引物序列当中仅末端碱基不匹配时,在模板足量的情况下,野生型模板仍能够有较高程度的扩增。为此,除了末端碱基外,我们在特异性引物的其他位置也设计了数个不完全匹配碱基(次黄嘌呤),以提高特异性引物对突变型和野生型模板的区分能力。次黄嘌呤的位置及数量对引物的特异性影响很大,须反复进行实验验证。另外,研究过程中我们发现,在特异性引物/探针组合中,除了特异性引物外,探针的序列对组合的特异性也有一定影响。在本研究中,对18组引物/探针组合进行了实验筛选,最终选定表1中所列出的特异性引物/探针组合。
本研究中的探针ASPCR方法是在前期建立的染料ASPCR方法的基础上重新设计引物/探针组合建立的一种新的ASPCR方法。对2种方法进行比较发现:①2者判读结果的方法存在差异。探针ASPCR方法在能够根据扩增曲线直接读取结果,而染料ASPCR方法对多种上呼吸道感染常见病原体均出现扩增反应,需要同时结合扩增曲线(Ct值)和熔解曲线(Tm值)对检测结果进行判读。②2种ASPCR方法检测临床样本的能力不同。临床样本检测结果中常有耐药菌和敏感菌混合存在的情况,采用染料ASPCR方法常会发生非特异性扩增的情况,此时其Ct值会小于其真实值,最终导致计算出的MP耐药比例存在一定误差,这也是导致2种ASPCR方法检测临床样本时,染料ASPCR方法检测临床样本的耐药比例高于探针ASPCR方法检测结果(表3)的主要原因,甚至当熔解曲线出现双峰时,还需要对样本进行复查。因此,该方法检测临床样本的特异性相对探针ASPCR方法较低,对实验者的要求也较高。而本研究中新建立的探针ASPCR方法,因其加入了探针,提高了扩增的特异性,可以根据Ct值即可直接判读结果,符合临床检验工作人员的判读习惯。
本研究将2种ASPCR方法检测临床样本的结果与巢式PCR联合DNA测序法的结果进行了比较:ASPCR方法法检测MP的阳性率高于传统巢式PCR方法。巢式PCR联合DNA测序法一般只能检测出主要基因型,不能检测出混合株中的其他微量基因型,在这方面,ASPCR方法检测临床标本检测能力明显优于传统巢式PCR联合DNA测序法。另外,本研究中,探针ASPCR方法检测临床样本结果显示,在近一半[45.45%(25/55)]的感染者体内,MP以敏感菌和耐药菌混合菌群的形式存在,且在MP阳性样本中,耐药菌和敏感菌都可能以优势菌种的形式存在。这说明,在临床用药前,很有必要采用ASPCR方法对临床样本进行检测,分析其中耐药菌和敏感菌的组成比例及其优势种群,为临床用药选择提供实验室依据。
