结直肠癌组织T 细胞受体组库多样性与患者临床特征和具核梭形杆菌丰度的关联分析
2022-10-21胡慕妮季林华张昕雨沈超琴陈豪燕
胡慕妮,季林华,张昕雨,沈超琴,洪 洁,陈豪燕
1.上海交通大学医学院附属仁济医院消化科,上海 200001;2.上海交通大学医学院附属仁济医院胃肠外科,上海 200127
结直肠癌(colorectal cancer,CRC)是全球第五大确诊的恶性肿瘤,为癌症相关死亡的第三大常见原因;据估计全球每年新增CRC病例超过10万例[1]。CRC的发生和发展与遗传、免疫、环境及肠道微生物等多种因素密切相关。研究[2]表明具核梭形杆菌(Fusobacterium nucleatum,Fn)在CRC患者粪便及肿瘤组织中的丰度较正常人群明显升高。同时Fn能够导致肠上皮细胞DNA损伤,诱发慢性炎症反应,促进CRC的发生和发展[3-4]。CRC患者肿瘤组织中高丰度的Fn与患者不良的预后也密切相关[4]。研究CRC发生和发展过程中的分子变化,寻找与之相关的新型生物学标志物对CRC相关研究的开展具有重要意义。
随着高通量技术的快速发展,多组学相关研究促进了肿瘤相关生物学标志物的发掘。既往研究[5]发现T 细胞受体(T cell receptor,TCR)组库可以反映特定时间段内机体内T细胞的多样性以及机体免疫系统应对外界刺激应答的能力。当患者受到某一抗原的刺激时,其相对应的T细胞会呈现单克隆或寡克隆反应性增生,导致TCR 组库多样性有所下降[6],这提示TCR 组库多样性可作为潜在的新型生物学标志物来评价机体的疾病状态。
T 细胞受体测序(T cell receptor sequencing,TCR-seq)以T 细胞为研究对象,应用多重聚合酶链式反应或5'cDNA 末端快速扩增(rapid amplification of 5'cDNA end,5'RACE)技术扩增决定TCR 多样性的互补决定区(complementary determining regions,CDR);该技术结合高通量测序技术常应用于TCR 组库多样性的评估中[7]。2021 年,来自哈佛大学的X Shirley Liu 团队开发的一种新的算法TRUST4[8],可以针对组织或者外周血的RNA 测序(RNA sequencing,RNA-seq)数据挖掘免疫组库信息;其能 与SMART-seq (switching mechanism at 5'end of the RNA transcript sequencing)和10× Genomics 平台兼容,可用于后续进一步的数据分析,有助于快速准确地评估TCR 序列信息,并深入研究免疫组库与疾病间的联系。TCR 组库多样性的分析主要在TCRα、β 链的基因水平(基因种类、频率、克隆数量及基因组合的差异表达等)与氨基酸水平(TCR β 链CDR3氨基酸的种类、频率、长度分布等)上开展[9],通常采用的指标包括香农熵(Shannon entropy)、Chao1指数、Simpson 指数、Hill 值等[10-11]。香农熵用于评估T细胞克隆的多样性,通过估计克隆型的相对数目和每种克隆型的丰度或分布反映TCR 的变异程度;香农熵值越高,提示样本中T 细胞克隆的多样性越高。较高的Chao1 指数和Hill 值以及更小的Simpson指数也可反映更高的TCR 多样性。通过分析TCR 组库多样性的特点,能够进一步了解机体免疫应答状态,从而有助于评估疾病进展和治疗效果[12]。
近年来,不少学者围绕TCR 组库多样性、肿瘤状态以及肿瘤免疫治疗等相关问题展开研究,并揭示了其中的密切联系,尤其在恶性黑色素细胞瘤、非小细胞肺癌等肿瘤中取得较多研究成果[13-14]。然而,TCR 组库分析技术在CRC 中的相关研究和应用较少。本研究利用TRUST4 算法分析53 例CRC 患者肿瘤组织及其匹配的癌周正常组织的RNA-seq 数据,探究CRC 组织TCR 组库多样性与临床特征以及肿瘤组织中Fn 丰度的相关性,以期为CRC 发生和发展机制研究提供参考。
1 对象与方法
1.1 研究对象
选择2016 年7 月—2016 年12 月上海交通大学医学院附属仁济医院胃肠外科收治的63 例行CRC 根治术患者作为研究对象。所有病例均经病理活检确诊。收集患者性别、年龄、肿瘤位置、肿瘤TNM 分期等临床资料,获取术中切除的肿瘤组织及其癌周正常黏膜组织样本(距离肿瘤组织边缘>5 cm)。
1.2 研究方法
1.2.1 组织处理 在组织离体0.5 h 内,使用生理盐水对其进行冲洗,随后放入装有RNAlater 组织保存液的10 mL 离心管中。4 ℃冰箱过夜之后使用DEPC水洗去组织表面的RNAlater,然后转入冻存管中,保存于-80 ℃冰箱内。
1.2.2 RNA-seq以及16S rDNA测序 使用TRizol法提取63对新鲜CRC肿瘤组织及配对癌旁组织的总RNA,通过RNeasy Mini Column(Qiagen,美国)以及DNA酶进行纯化,并通过Agilent 2100 Bioanalyzer进行质量检测。其中10对样本的RNA完整性指标(RNA integrity number,RIN)低于6,未达到测序平台的最低标准,予以排除。最终对质量合格的53 对样本进行深度RNA-seq,测序方案为双端测序(pair-end 150 bp,PE150),每例样品测序数据量为10 Gb。选取16S rDNA的特定序列进行高通量测序分析以估计细菌丰度。以纯化的DNA作为模板,利用PCR扩增特定目的片段,对回收后的PCR 纯化产物的样品使用Qubit dsDNA HS Assay Kit试剂盒(Life Technologies公司,美国)进行定量检测,最后在MiSeq高通量测序仪(Illumina公司,美国)PE250平台进行测序。
1.2.3 测序数据处理 使用FastQC软件对原始RNAseq数据进行质控处理,质控处理后的序列通过HISAT2.45软件与人类基因组序列GRCh38进行比对,利用基因组注释软件GENCODE v22对转录组进行定量分析。本研究RNA-seq 结果已上传至基因表达数据库(Gene Expression Omnibus,GEO)(GSE158559)。原始RNAseq数据利用TRUST4算法(https://github.com/liulabdfci/TRUST4)进行处理,获得每个样本的免疫组库信息。TRUST4首先将所测测序片段(reads)比对到参考基因组上,将比对上的reads组装成重叠群(Contig),然后根据国际免疫遗传学信息系统(International Immunogenetics Information System,IMGT)进行注释。使用FastQC软件对16S rDNA测序样本数据进行质量控制处理,再通过UCHIME算法去除嵌合体。利用USEARCH序列分析软件将拼接好的标签(Tag)在97%的相似度水平下进行聚类,获得OTU信息,并基于rdp_16s_v16.fa分类学数据库对OTU进行分类学注释。
1.2.4 TCR 组库多样性评估 基于TRUST4 算法处理后的免疫组库信息数据,使用immunarch R 进一步对免疫组库数据进行可视化分析,采用repClonality函数比较肿瘤组织及其正常组织中克隆型的数目、不同扩增程度克隆型的占比、不同克隆型的丰度;使用repExplore 函数比较不同长度CDR3 氨基酸序列分布上克隆型数目,并利用repDiversity 函数计算多样性指数(Chao1 指数、逆Simpson 指数、Hill 数值)。Chao1 指数、逆Simpson 指数、Hill 数值越高,TCR多样性越高。
1.2.5 临床特征及Fn 丰度评估 临床特征评估纳入性别、年龄、肿瘤位置、肿瘤TNM 分期等因素。年龄以65 岁作为临界值。根据肿瘤所在部位将肿瘤位置定义为直肠和非直肠。TNM 分期参照美国癌症联合 委 员 会(American Joint Committee on Cancer,AJCC)结直肠癌TNM 分期第八版[15],分为Ⅰ/Ⅱ期和Ⅳ/Ⅲ期。将性别、肿瘤位置、肿瘤TNM 分期转化为二分类变量进行分组统计。Fn 丰度评估使用OTU的reads数目的中位数作为临界值,>临界值被定义为Fn高负荷,≤临界值被定义为Fn低负荷。
1.3 统计学分析
使用R studio(4.1.1 版)软件进行统计学分析。符合正态分布的定量资料采用表示,2 组间差异比较使用t检验。对于非正态分布的定量资料,采用M表示,2 组间差异比较使用Wilcoxon 检验。P<0.05表示差异有统计学意义。
2 结果
2.1 肿瘤组织及癌周正常黏膜组织TCR克隆型分析
2.1.1 克隆型数目 对53 对样本的TCR 克隆型数目进行分析,总体上来看,肿瘤组织中克隆型数目较癌周正常黏膜组织下降(图1A)。统计分析结果显示,肿瘤组织TCR 克隆型数目明显少于癌周正常黏膜组织,差异具有统计学意义(P=0.000,图1B),且肿瘤组织中占据免疫组库10%的克隆型数目也低于癌周正常黏膜组织(P=0.000,图1C)。此外,对肿瘤组织和癌周正常黏膜组织TCR β 链CDR3氨基酸序列不同长度分布上的克隆型数目进行分析,结果显示,在不同长度的CDR3序列上,肿瘤组织的克隆型数目均低于癌周正常黏膜组织(图1D)。以上结果提示癌周正常组织TCR组库多样性更高。

图1 肿瘤组织和癌周正常组织的TCR克隆型数目差异分析Fig 1 Comparison of the number of TCR clonotypes between tumor and adjacent normal tissues
2.1.2 不同扩增水平的克隆型 将TCR 克隆型按照扩增水平从高到低排序,分析排在前10、11~100、101~1 000、1 001~3 000、3 001~10 000、10 001~30 000、30 001~50 000位的克隆型所占的比例。肿瘤组织中扩增水平排在前10 位的TCR 克隆型所占比例明显高于癌周黏膜正常组织,差异有统计学意义(P=0.000,图2),提示肿瘤组织的TCR 的扩增程度高于癌周正常黏膜组织。

图2 肿瘤和癌周正常组织不同扩增水平的TCR克隆型差异分析Fig 2 Comparison of proportion of TCR clonotypes with different amplification levels between tumor and adjacent normal tissues
2.1.3 不同频率克隆型的相对丰度 进一步分析肿瘤组织和癌周正常黏膜组织不同频率TCR克隆型在整个克隆空间内的相对丰度。克隆型的频率包括:稀有频率(>0且≤10-5)、小频率(>10-5且≤10-4)、中频率(>10-4且≤0.001)、高频率(>0.001且≤0.01)、超高频率(>0.01且≤1)。肿瘤组织和癌周正常黏膜组织中,TCR克隆型均以小频率(>10-5且≤10-4)和中频率(>10-4且≤0.001)为主。肿瘤组织中高频率(>0.001且≤0.01)和超高频率(>0.01且≤1)克隆型的相对丰度高于癌周正常黏膜组织,稀有频率(>0且≤10-5)和小频率(>10-5且≤10-4)克隆型的相对丰度低于癌周正常黏膜组织,差异具有统计学意义(均P=0.000,图3)。

图3 肿瘤和癌周正常组织不同频率TCR克隆型的丰度差异Fig 3 Comparison of relative abundances in different TCR clonotypes with specific frequencies between tumor and adjacent normal tissues
2.2 肿瘤组织及癌周正常黏膜组织TCR 组库多样性指数评估
利用Chao1 指数、逆Simpson 指数以及Hill 数值曲线对肿瘤组织及癌周正常黏膜组织的TCR 组库多样性进行分析比较。肿瘤组织的Chao1 指数和逆Simpson 指数均小于癌周正常黏膜组织,差异具有统计学意义(均P=0.000,图4A、B)。Hill 数值曲线也显示,肿瘤组织TCR 组库多样性较癌周正常黏膜组织明显下降(P=0.000,图4C)。
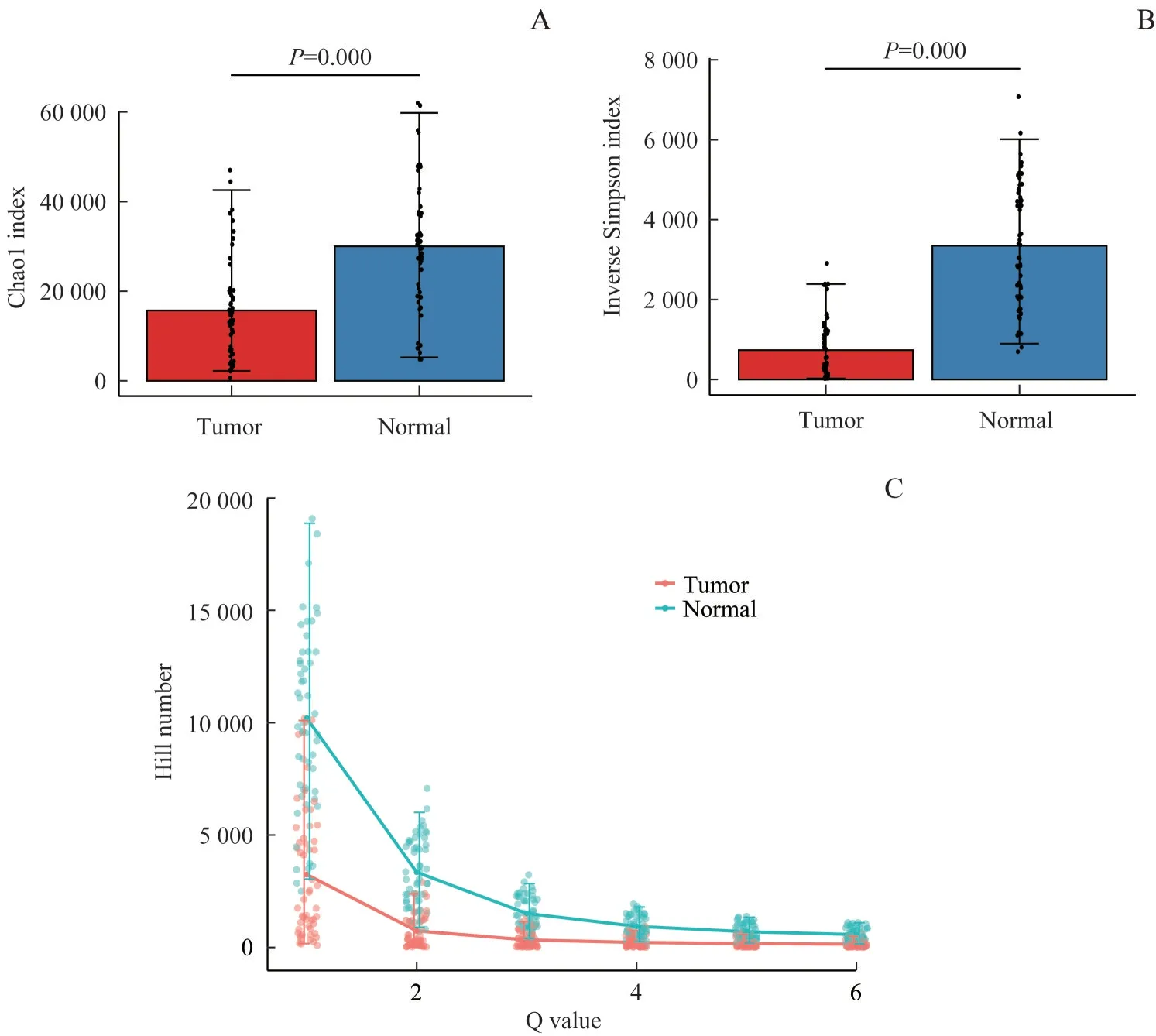
图4 肿瘤和癌周正常组织间TCR组库多样性指数比较Fig 4 Comparison of TCR repertoire diversity indexes between tumor and adjacent normal tissues
2.3 不同临床特征肿瘤患者的TCR组库多样性分析
发生于直肠的CRC 患者TCR 克隆型的数目高于非直肠位部(P=0.040,图5A),提示直肠部位肿瘤TCR 组库多样性更高。Fn 高负荷CRC 肿瘤组织的克隆型的数目(P=0.040,图5B)和Chao1 指数(P=0.030,表1)低于Fn 低负荷CRC 肿瘤组织,差异具有统计学意义;提示不同Fn 丰度下CRC 肿瘤组织的TCR 组库多样性具有差异,Fn 丰度越高的肿瘤组织TCR 组库多样性越低。而不同年龄、性别(≥65 岁vs<65 岁)、TNM 分期(Ⅰ/Ⅱ期vsⅢ/Ⅳ期)的CRC患者,肿瘤组织Chao1指数的差异无统计学意义(均P>0.05,表1)。
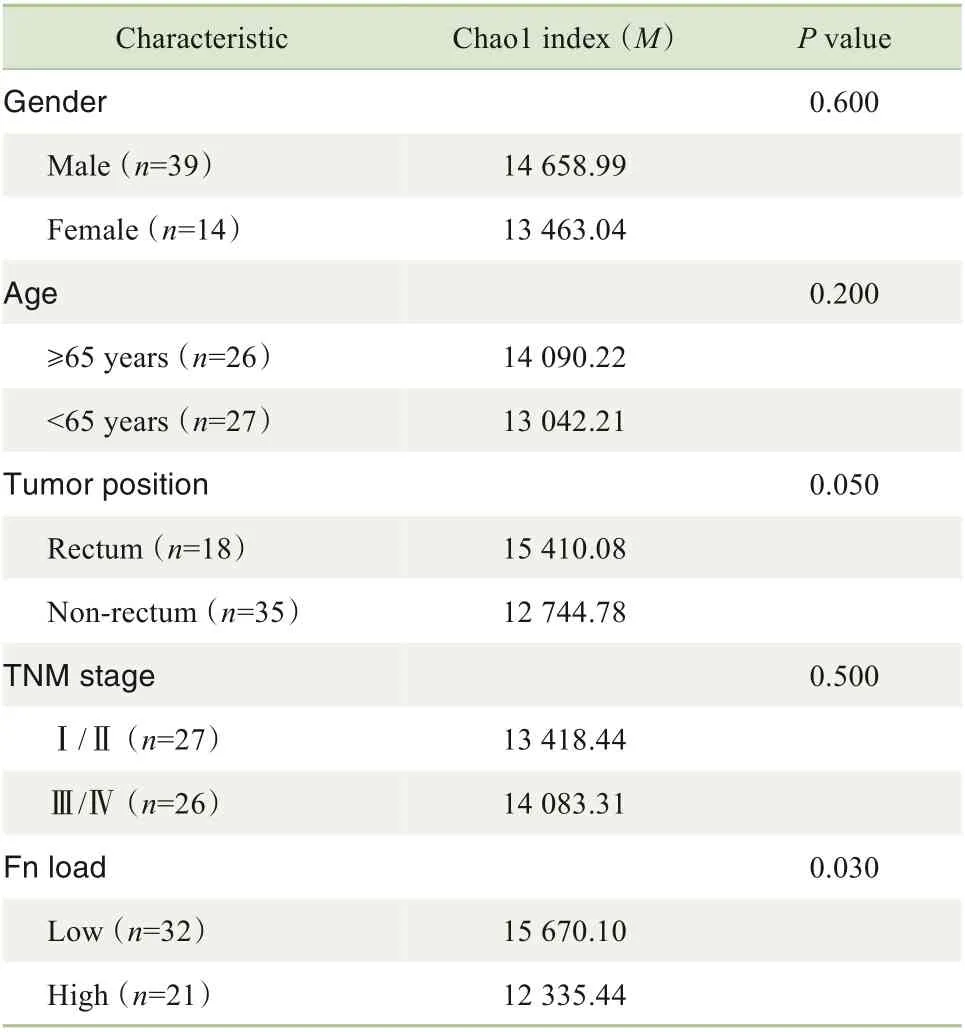
表1 不同临床特征和不同Fn丰度下Chao1指数比较Tab 1 Comparison of Chao1 indexes in different clinical features and different Fn loads

图5 不同肿瘤部位以及不同Fn丰度下TCR克隆型比较Fig 5 Comparison of TCR clonotypes in different tumor locations and different Fn loads
3 讨论
近年来,不少实体肿瘤的研究试图深入挖掘免疫组库信息,从而为疾病的诊断、免疫状态监测和患者预后评估提供新型生物学标志物[16-18]。然而关于TCR 组库在CRC 中的相关研究开展得较少。SHERWOOD 等[19]通过对比分析15 例CRC 患者的TCR β 链测序的结果,发现肿瘤浸润性T 细胞的多样性变异远低于癌周正常黏膜组织,同时肿瘤组织中TCR 组库的寡克隆性较高,提示肿瘤组织和周围正常黏膜组织内的免疫应答反应不同。既往关于TCR组库多样性的研究大多依靠TCR-seq技术获得样本的TCR 序列。尽管测序技术能够较为准确地呈现TCR序列特征,但测序费用高昂,且既往研究纳入的样本量都比较少。本研究使用TRUST 算法挖掘CRC 肿瘤组织和其匹配的癌周正常黏膜组织RNA-seq数据中的免疫组库信息,相比于单独针对样本进行TCR-seq处理,经济成本有所降低,数据处理更加高效,并能获取机体更多的生物学信息。此外,基于既往研究[3]发现的Fn 在CRC 的发生和发展过程的重要作用,我们试图寻找CRC 组织中的TCR 组库多样性与Fn 丰度之间的关系。
本研究对比分析53 例CRC 患者的肿瘤组织及其匹配的癌周正常黏膜组织的TCR 组库特点,发现在肿瘤组织中扩增水平前10 位克隆型的占比显著高于癌周正常黏膜组织,这提示肿瘤状态下T细胞受到外界抗原刺激,可能存在单克隆或寡克隆反应性增生,导致TCR 某一序列克隆比例显著上升,而多样性却呈现总体下降趋势。肿瘤组织和癌周正常组织间TCR 组库多样性的差异,对于研究肿瘤发生和发展的机制具有重要的参考价值。CRC 中发生于直肠部位的肿瘤通常表现出对治疗的高敏感性以及更好的预后[1]。本研究中,发生于直肠部位的肿瘤的TCR 组库多样性较非直肠部位肿瘤更高,这提示TCR 组库可能有助于CRC 的临床治疗和预后评估。此外,本研究发现CRC 肿瘤组织内的Fn 丰度与TCR 组库多样性有关,在较低的Fn 负荷下,肿瘤的TCR 组库多样性更高。Fn 与CRC 的发生和发展密切相关,肿瘤组织内Fn 的高负荷与患者的不良预后密切相关[2]。TCR 组库多样性特点与CRC 患者的Fn 丰度相关,进一步提示TCR 组库多样性能够作为潜在的生物学标志物应用于CRC发生和发展机制的研究中。
本研究尚存在一定的局限性。TCR β 链的CDR3序列信息是通过TRUST 算法计算得出的,尽管该算法十分高效,但难以避免存在一定的误差。这需要未来开发更多更高效的检测手段或者计算方法来尽可能地还原生物原始的CDR3序列信息。同时,TCR 组库多样性在CRC 的临床诊治及预后评估过程中的相关价值也有待于扩大样本量进一步研究。在CRC 的发生和发展过程中,TCR 组库多样性发生改变机制如何,是否存在疾病特征性的TCR 单克隆序列,TCR组库多样性特点是否可以用于直接评估CRC 患者的预后和免疫治疗效果,都有待于进一步探讨。
综上所述,本研究利用TRUST 算法从53 例CRC患者的RNA-seq数据中挖掘患者的免疫组库信息,发现TCR 组库多样性与患者多项临床特征以及肿瘤组织内的Fn 丰度有关,为CRC 发生和发展的机制研究提供了重要信息。
利益冲突声明/Conflict of Interests
所有作者声明不存在利益冲突。
All authors disclose no relevant conflict of interests.
伦理批准和知情同意/Ethics Approval and Patient Consent
本研究涉及的所有实验均已通过上海交通大学医学院附属仁济医院科学伦理委员会的审核批准(2016-01-15)。受试对象或其亲属已经签署知情同意书。All experimental protocols in this study were reviewed and approved by Renji Hospital,Shanghai Jiao Tong University School of Medicine(2016-01-15). Consent letters have been signed by the research participants or their relatives.
作者贡献/Authors'Contributions
陈豪燕、洪洁参与了实验设计;胡慕妮完成主要统计学分析和绘图;季林华、张昕雨和沈超琴主要负责标本收集和实验部分;胡慕妮、陈豪燕参与了论文的写作和修改。所有作者均阅读并同意了最终稿件的提交。
CHEN Haoyan and HONG Jie were responsible for the experimental design. HU Muni completed the main statistical analysis and mapping.JI Linhua,ZHANG Xinyu and SHEN Chaoqin mainly prepared for specimen collection and experiments.CHEN Haoyan and HU Muni participated in the writing and revision of the paper. All authors have read and approved the submission of the final manuscript.
·Received:2022-01-24
·Accepted:2022-05-27
·Published online:2022-07-25
参·考·文·献
[1] SIEGEL R L, MILLER K D, GODING SAUER A, et al. Colorectal cancer statistics,2020[J]. CA Cancer J Clin,2020,70(3):145-164.
[2] HARUKI K, KOSUMI K, HAMADA T, et al. Association of autophagy status with amount ofFusobacterium nucleatumin colorectal cancer[J]. J Pathol,2020,250(4):397-408.
[3] YU T, GUO F F,YU Y N, et al.Fusobacterium nucleatumpromotes chemoresistance to colorectal cancer by modulating autophagy[J].Cell,2017,170(3):548-563.e16.
[4] MIMA K, NISHIHARA R, QIAN Z R, et al.Fusobacterium nucleatumin colorectal carcinoma tissue and patient prognosis[J].Gut,2016,65(12):1973-1980.
[5] VUJOVIC M, DEGN K F, MARIN F I, et al. T cell receptor sequence clustering and antigen specificity[J]. Comput Struct Biotechnol J,2020,18:2166-2173.
[6] CHEN M, CHEN R Z, JIN Y, et al. Cold and heterogeneous T cell repertoire is associated with copy number aberrations and loss of immune genes in small-cell lung cancer[J]. Nat Commun,2021,12(1):6655.
[7] SIMNICA D,AKYÜZ N,SCHLIFFKE S,et al. T cell receptor nextgeneration sequencing reveals cancer-associated repertoire metrics and reconstitution after chemotherapy in patients with hematological and solid tumors[J]. Oncoimmunology,2019,8(11):e1644110.
[8] SONG L, COHEN D, OUYANG Z Y, et al. TRUST4: immune repertoire reconstruction from bulk and single-cell RNA-seq data[J].Nat Methods,2021,18(6):627-630.
[9] SHAO H W, LIN Y M, WANG T, et al. Identification of peptidespecific TCR genes byin vitropeptide stimulation and CDR3 length polymorphism analysis[J]. Cancer Lett,2015,363(1):83-91.
[10] LOONEY T J, TOPACIO-HALL D, LOWMAN G, et al. TCR convergence in individuals treated with immune checkpoint inhibition for cancer[J]. Front Immunol,2020,10:2985.
[11] STEWART J J, LEE C Y, IBRAHIM S, et al. A Shannon entropy analysis of immunoglobulin and T cell receptor[J]. Mol Immunol,1997,34(15):1067-1082.
[12] HOPKINS A C, YARCHOAN M, DURHAM J N, et al. T cell receptor repertoire features associated with survival in immunotherapy-treated pancreatic ductal adenocarcinoma[J]. JCI Insight,2018,3(13):e122092.
[13] HAN J F, DUAN J C, BAI H, et al. TCR repertoire diversity of peripheral PD-1+CD8+T cells predicts clinical outcomes after immunotherapy in patients with non-small cell lung cancer[J].Cancer Immunol Res,2020,8(1):146-154.
[14] JOSHI K, MILIGHETTI M, CHAIN B M. Application of T cell receptor (TCR) repertoire analysis for the advancement of cancer immunotherapy[J]. Curr Opin Immunol,2022,74:1-8.
[15] AMIN M B, GREENE F L, EDGE S B, et al. The Eighth EditionAJCC Cancer Staging Manual: continuing to build a bridge from a population-based to a more“personalized”approach to cancer staging[J]. CA Cancer J Clin,2017,67(2):93-99.
[16] BUHLER S,BETTENS F,DANTIN C,et al. Genetic T-cell receptor diversity at 1 year following allogeneic hematopoietic stem cell transplantation[J]. Leukemia,2020,34(5):1422-1432.
[17] FARMANBAR A, KNELLER R, FIROUZI S. RNA sequencing identifies clonal structure of T-cell repertoires in patients with adult Tcell leukemia/lymphoma[J]. NPJ Genom Med,2019,4:10.
[18] CHARLES J, MOURET S, CHALLENDE I, et al. T-cell receptor diversity as a prognostic biomarker in melanoma patients[J].Pigment Cell Melanoma Res,2020,33(4):612-624.
[19] SHERWOOD A M, EMERSON R O, SCHERER D, et al. Tumorinfiltrating lymphocytes in colorectal tumors display a diversity of T cell receptor sequences that differ from the T cells in adjacent mucosal tissue[J]. Cancer Immunol Immunother,2013,62(9):1453-1461.
