KRAS4AG12C和KRAS4BG12C对人肺上皮细胞生长和运动的作用差异及机制研究
2022-10-21邹菁华宫淼淼
邹菁华,宫淼淼,沈 瑛
1.上海交通大学基础医学院药理学与化学生物学系,上海200025;2.上海市转化医学协同创新中心,上海200025
肺癌的发病率和病死率都高居恶性肿瘤前列,预计5 年生存率仅为18%,严重威胁着人类生命健康[1]。在疾病进展过程中,肺癌原发部位的肿瘤细胞会通过局部浸润以及血管和淋巴管的远端转移而定植到其他部位生长,称为肿瘤转移[2]。转移不仅是肿瘤耐药复发的主要原因,更是患者常见的死因[3-4]。
大鼠肉瘤病毒(rat sarcoma,Ras)家族是肿瘤中突变频率最高的基因家族[5],其中鼠类肉瘤病毒癌基因(kirsten rat sarcoma viral oncogene,Kras)的恶性程度最高,是肺癌(30%)、结肠癌(40%)、胰腺癌(80%) 三大致命癌症的主要致癌驱动基因[6]。人类约13%的恶性肿瘤伴随KRAS基因突变[7],肺癌中KRAS主要突变亚型包括KRASG12C、KRASG12D、KRASG12V、KRASQ61H等,其 中KRASG12C占比高达46%[8]。然而,由于KRAS 与鸟苷三磷酸(guanosine triphosphate,GTP)的高亲和力以及表面缺少可以与小分子紧密结合的疏水口袋,KRAS 一度被认为是不可成药的靶点[8-10]。 近年来,KRASG12C共价抑制剂AMG510 和MRTX849 成功研发,临床试验疗效显著,给KRASG12C突变患者带来新的希望,然而长期使用均不可避免发生适应性或获得性耐药[11]。
KRAS基因转录表达的过程中,其第四外显子发生选择性剪切,得到KRAS4A 和KRAS4B 这2 种剪接形式的蛋白,这2 种蛋白在保守结构域(1-165残基) 具有高度同源性,其中包括鸟苷二磷酸(guanosine diphosphate,GDP)/GTP 结合区和效应因 子 结 合 区[12-13], 在 C 端 高 度 可 变 区(hypervariable region,HVR,166-188 或189 残基)存在差异,这一区域在膜靶向性、蛋白-蛋白相互作用和信号转导方面起着重要作用[14]。KRAS4A和KRAS4B在人类胚胎发育早期表达水平相当,而在成体组织中差异表达[15],且均能编码致癌蛋白[16]。在肿瘤细胞中,KRAS通常在第一或第二外显子的12、13、61 密码子发生点突变,蛋白构象发生改变,从而处于异常激活状态,促进癌症的发生发展[17-19]。有研究报道,KRAS4A 具有独特的双膜靶向结构,能够特异性结合并激活己糖激酶1(hexokinase 1,HK1)[20],而KRAS4B 通过C 端高度可变区与钙调蛋白(calmodulin,CaM) 结合,有利于保持肿瘤细胞的干性状态[21]。在肺癌细胞系中,KRAS4BmRNA 水平显著高于KRAS4AmRNA[16]。然而,关于KRAS4A 和KRAS4B 在肺癌转化过程中的生物学差异尚未研究清楚。本研究将围绕KRAS4A 和KRAS4B 促进人肺支气管上皮细胞生长和运动的差异与分子机制进行深入探究,构建人正常肺支气管上皮细胞BEAS-2B KRAS4AG12C和KRAS4BG12C过表达细胞株,通过细胞增殖、细胞迁移、细胞黏附实验观察细胞表型差异,全转录组RNA-seq 方法检测差异基因及信号通路变化并用实时荧光定量PCR (quantitative real-time PCR,qPCR)进一步验证,通过探究不同剪接体致癌机制的差异,为KRAS 特异性靶向抑制剂的研究提供一定的实验依据和理论基础。
1 材料与方法
1.1 材料、试剂及仪器
1.1.1 细胞系 人正常肺支气管上皮细胞系BEAS-2B,人胚肾上皮永生化细胞系293FT 均在上海交通大学基础医学院药理学与化学生物学系保存并使用。
1.1.2 主要试剂 0.25%胰蛋白酶、磷酸缓冲盐溶液(phosphate buffered saline,PBS)、青霉素-链霉素(双抗)溶液及谷氨酰胺溶液购自上海源培生物科技股份有限公司,Takara RNA 提取试剂盒、PrimeScriptTMRT Master Mix (Perfect Real Time)试剂盒购自日本Takara 公司,转染专用减血清培养基、DMEM 高糖培养基、BCA 蛋白定量试剂盒、预染蛋白分子量marker、Lipofectamine 3000 试剂、P3000试剂及ECL 显色液购自美国Thermo Fisher Scientific公司,胎牛血清购自美国Gemini 公司,Ku86 抗体购自美国Santa Cruz Biotechnology 公司,Tubulin 抗体购自美国Proteintech 公司,RAS 抗体、辣根过氧化物酶标记山羊抗兔IgG 及抗小鼠IgG 购自美国Cell Signaling Technology 公司,RIPA 裂解液、Western blotting 一抗稀释液及蛋白酶抑制剂购自上海碧云天公司。
1.1.3 主要仪器 生物安全柜、CO2细胞培养箱购自美国Thermo Fisher Scientific 公司,制冰机购自美国Scotsman 公司,蛋白电泳及转印系统、预制胶、多功能酶标仪购自美国Bio-Rad 公司,实验用超纯水系统购自美国Millipore 公司,Incucyte 活细胞动态实时成像仪购自美国Essen 公司,Odyssey 双色红外成像系统购自美国LI-COR 公司,倒置相差显微镜购自日本Nikon 公司,磁力搅拌器购自德国IKA 公司,移液器、低温高速离心机购自德国Eppendorf 公司。
1.2 研究方法
1.2.1 细胞培养及传代 配制DMEM 高糖完全培养基(含1%谷氨酰胺、1%双抗溶液、10%胎牛血清)。PBS 清洗细胞,37 ℃预热的0.25%胰蛋白酶(含0.038% EDTA)消化1~2 min,新鲜完全培养基终止消化。1∶3 传代,持续观察细胞状态。
1.2.2 构建过表达KRAS4AG12C和KRAS4BG12C细胞系 按比例配制转染专用减血清培养基、Lipo 3000试剂、P3000 Enhancer Reagent、包装质粒、目的基因质粒混合物转染293FT 细胞。收集24 h 和48 h 病毒。使用含病毒培养液和Polybrene 感染BEAS-2B 细胞,1 μg/mL 嘌呤霉素筛选,扩增、保种及后续实验。
1.2.3 蛋白质印迹法检测 蛋白质印迹法(Western blotting)检测模型的构建。配制10% SDS-PAGE 胶,电泳2~3 h;使用甲醇活化PVDF 膜,4 ℃恒流转膜2~3 h;5%脱脂奶粉室温封闭1~2 h;一抗冷室孵育过夜;二抗室温孵育1 h;ECL 显色液显影。
1.2.4 划痕实验分析 取对数生长期细胞(20 000个/孔)接种,待细胞汇合度达到95%时,无菌划痕仪划痕,PBS 清洗2 次,换用新鲜培养基。使用Incycute 活细胞动态实时成像仪拍照并分析数据。
1.2.5 Transwell 实验分析 于上层小室中接种细胞,50 000 个/孔,上层小室和下层小室分别为基础培养基和完全培养基,孵育20 h。PBS 清洗2 次,4%多聚甲醛固定25 min,结晶紫染色过夜。用棉签轻柔擦掉上室细胞,PBS 清洗2 次,于显微镜下观察及拍照。待液体蒸发后用10% 冰醋酸萃取,595 nm 波长下检测吸光度值进行定量分析。
1.2.6 RNA-seq 检测 提取RNA 样品后送至武汉华大基因科技有限公司检测。
1.2.7 基因集富集分析 采用基因集富集分析(Gene Set Enrichment Analysis,GSEA)对RNA-seq数据进行分析做图,具体操作步骤见文献[22]。其中标准化富集得分(normalized enrichment score,NES)绝对值大于1,normalP<0.05 的通路下的基因集合是有意义的。
1.2.8 RNA 提取及qPCR 检测 参照Takara RNA 提取试剂盒说明书提取RNA,经细胞裂解、DNA 清除、RNA 纯化、洗脱、DNA 酶消化、洗脱等步骤得到50 μL RNA。使用Nanodrop 检测RNA 样品的R 值及浓度。配制反转录体系:4 μL 1.5×PrimeScript RT Master Mix+RNA 1 000 ng+无RNA 酶水。参照仪器内反转录程序,关键步骤为37 ℃15 min,85 ℃5 s。qPCR 反 应 体 系 配 制:6.4 μL 无RNA 酶 水+10 μL SYBR Premix Ex Taq Ⅱ+0.8 μL PCR 上 游 引 物(10 μmol/L)+0.8 μL PCR 下游引物(10 μmol/L)+2 μL cDNA。预变性条件为95 ℃反应30 s;扩增条件为95 ℃反应5 s,60 ℃反应34 s,循环40 次。引物序列见表1。
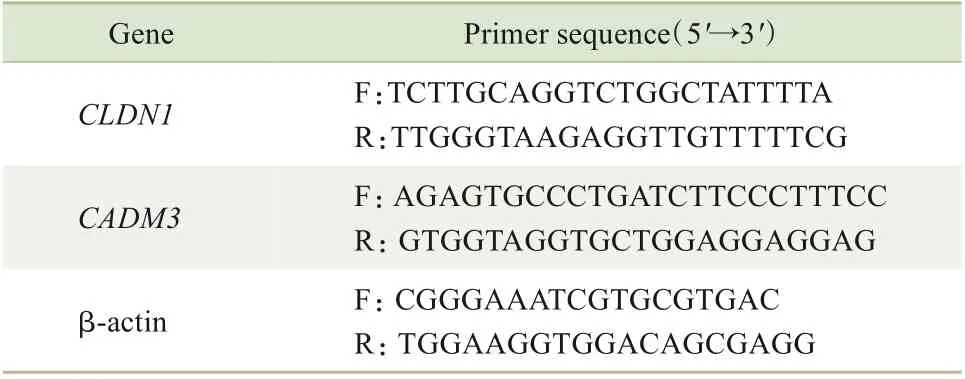
表1 qPCR所用的引物序列Tab 1 Sequences of primers for qPCR
1.3 统计学方法
使用GraphPad Prism 8 软件对数据进行统计分析及作图。定量结果采用xˉ±s表示,使用student'st检验进行组间比较。P<0.05 表示差异具有统计学意义。
2 结果
2.1 KRAS4AG12C和KRAS4BG12C过表达引起人正常肺支气管上皮细胞形态变化并增强其增殖能力
Western blotting 结果显示BEAS-2B KRAS4AG12C和BEAS-2B KRAS4BG12C稳定过表达细胞株构建成功(图1A、1B)。Incucyte 实时观测细胞增殖变化,结果显示与亲本相比,BEAS-2B KRAS4AG12C和BEAS-2B KRAS4BG12C细胞增殖能力增强(图1C)。为了探究KRAS4AG12C和KRAS4BG12C过 表 达 对BEAS-2B 细胞形态的影响,于显微镜下观察细胞形态(图1D):BEAS-2B 对照组细胞整体形态偏圆,细胞较薄,立体结构不明显。BEAS-2B KRAS4AG12C细胞趋向不规则形状生长,有伪足状结构生成;BEAS-2B KRAS4BG12C细胞立体结构明显,形状变为长梭形,细胞间连接增多,有伪足状结构生成。这些结果提示KRAS4AG12C和KRAS4BG12C过表达促进BEAS-2B细胞增殖并引起形态改变。
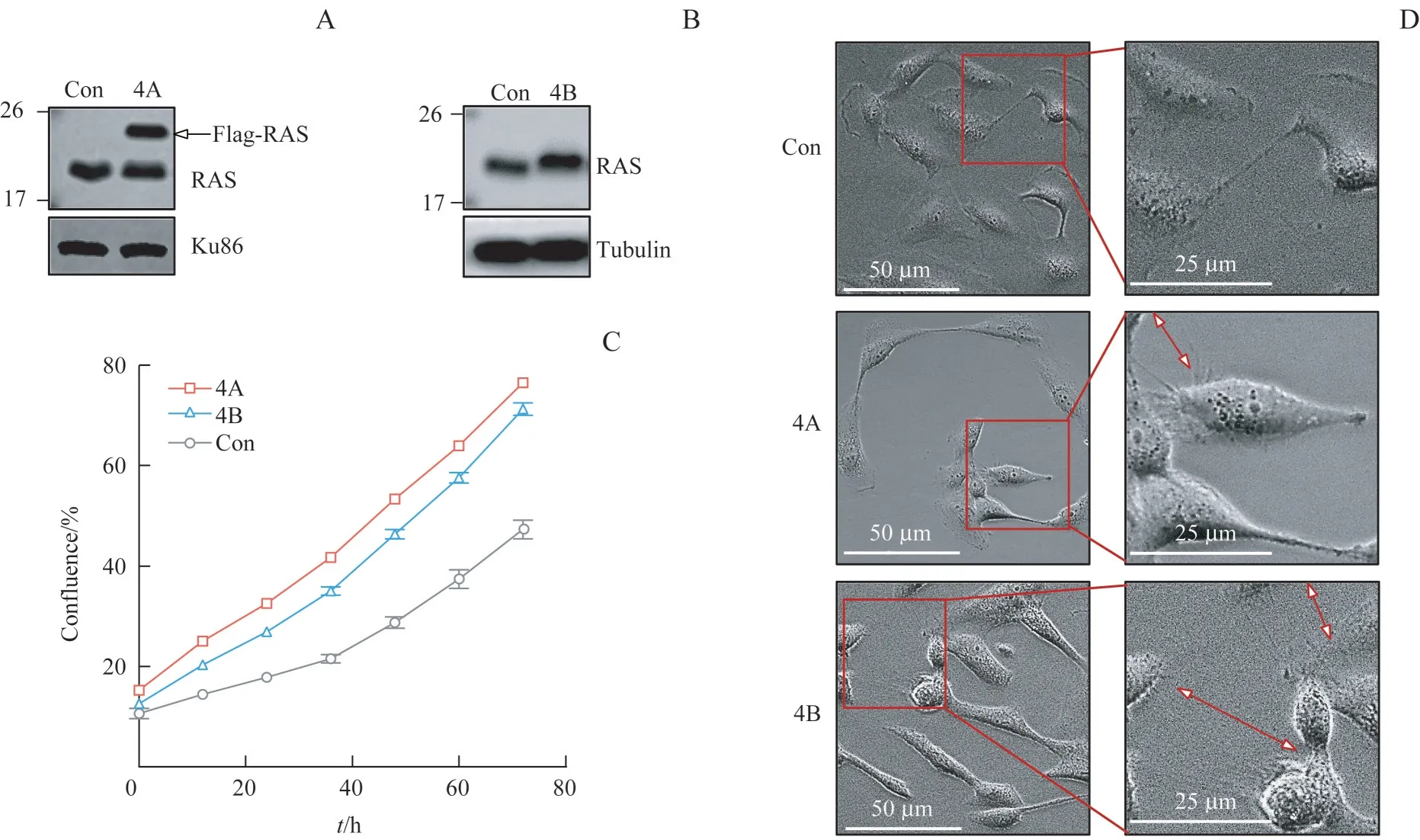
图1 KRAS4AG12C和KRAS4BG12C过表达对肺上皮细胞形态及增殖的影响Fig 1 Effects of KRAS4AG12C or KRAS4BG12C overexpression on pulmonary epithelial cells morphology and proliferation
2.2 KRAS4BG12C引起细胞运动能力增强效果强于KRAS4AG12C
进一步探究KRASG12C不同剪接体对人肺上皮细胞运动能力的影响。划痕实验结果显示12 h后,BEAS-2B KRAS4BG12C细胞划痕区完全愈合,而BEAS-2B KRAS4AG12C细胞与亲本细胞划痕区尚未愈合,表明BEAS-2B KRAS4BG12C细胞运动能力显著强于BEAS-2B KRAS4AG12C细胞(P=0.006)及BEAS-2B 亲本细胞(P=0.000,图2A、2B)。同样,Transwell 实验结果也证实BEAS-2B KRAS4BG12C细胞迁移能力显著强于BEAS-2B KRAS4AG12C细胞(P=0.048),且显著强于BEAS-2B 亲本细胞(P=0.033,图2C、2D)。这些结果说明过表达KRAS4BG12C引起人肺支气管上皮细胞运动能力增强效果强于KRAS4AG12C。

图2 KRAS4AG12C和KRAS4BG12C过表达对肺上皮细胞运动的影响Fig 2 Effects of KRAS4AG12C or KRAS4BG12C overexpression on pulmonary epithelial cells migration
2.3 KRAS4BG12C过表达促进BEAS-2B细胞黏附相关基因CLDN1和CADM3表达上调
为了进一步探究过表达KRAS4BG12C促进人肺上皮细胞运动的分子机制, 我们对BEAS-2B KRAS4AG12C过表达细胞和BEAS-2B KRAS4BG12C过表达细胞进行RNA-seq 测序,后对测序结果进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG) 通 路 富 集 分 析 及GSEA,寻找差异基因及信号通路变化。KEGG 分析的结果显示,细胞黏附分子信号通路排名第二(P=0.000,图3A)。GSEA 分析的结果显示,与BEAS-2B KRAS4AG12C过表达细胞相比,BEAS-2B KRAS4BG12C过表达细胞中的细胞黏附分子相关信号通路发生显著上调(P=0.002,图3B)。使用qPCR对细胞黏附分子通路中关键基因CLDN1和CADM3的mRNA 水平进行检测, 发现与BEAS-2B KRAS4AG12C过表达细胞相比,BEAS-2B KRAS4BG12C过表达细胞中黏附相关基因CLDN1和CADM3的mRNA 水平显著升高(P=0.000,图3C、3D)。这些结果提示KRAS4BG12C促进黏附相关基因CLDN1和CADM3上调,促进细胞运动。

图3 KRAS4AG12C和KRAS4BG12C过表达对黏附相关基因CLDN1和CADM3表达的影响Fig 3 Effects of KRAS4AG12C or KRAS4BG12C overexpression on the mRNA levels of CLDN1 and CADM3
3 讨论
KRAS作为多种癌症中的主要致癌驱动基因,30多年来一直被认为是不可成药的靶点。近年来,KRASG12C特异性共价抑制剂AMG510 的上市给KRAS突变癌症患者带来了希望[23]。但KRAS基因不同剪接变体在驱动癌症进展方面的分子机制以及KRAS不同基因突变类型致癌分子机制均尚未研究清楚,也增加了KRAS 抑制剂研发的难度。最近有研究报道KRAS4A 和KRAS4B 在各种癌症细胞株中均广泛表达,肺癌和胰腺癌细胞中以KRAS4B 表达为主,而结肠癌和黑色素瘤细胞中KRAS4A 和KRAS4B 表达水平相当[16]。为了研究KRASG12C不同剪接变体促进肺上皮细胞生长和运动的差异及其机制,我们通过慢病毒感染在人正常肺支气管上皮细胞BEAS-2B 中成功构建KRAS4AG12C和KRAS4BG12C(2 种剪接体)稳定过表达细胞株,镜下观察发现KRAS4AG12C和KRAS4BG12C过表达均引起肺上皮细胞形态变化,并且均可以促进细胞增殖。于是我们提出科学假设:携带活化突变的KRAS4AG12C和KRAS4BG12C2 种剪接体对肺上皮细胞的运动能力有何影响?哪种恶性程度更高?引起肺上皮细胞运动能力变化的分子机制如何?
为了解决这些问题,我们使用划痕实验和Transwell 实验模拟体内细胞迁移的过程,结果显示过表达KRAS4BG12C引起肺上皮细胞运动能力增强效果强于过表达KRAS4AG12C。 为了进一步探究KRAS4BG12C促进肺上皮细胞运动的分子机制,我们使用RNA-seq 方法对细胞全基因转录组进行测序并用GSEA 进行基因集富集分析,寻找差异基因及信号通路变化。结果显示与BEAS-2B KRAS4AG12C细胞相比,BEAS-2B KRAS4BG12C细胞中细胞黏附分子相关信号通路发生显著上调。使用qPCR 对细胞中黏附相关基因CLDN1和CADM3的表达水平进一步验证发现,与过表达KRAS4AG12C相比,过表达KRAS4BG12C促进黏附相关基因CLDN1和CADM3表达上调。有文献报道,结肠癌中CLDN1高表达,诱导肿瘤细胞发生上皮间充质转化,促进肿瘤转移[24];RAS突 变 合 并CLDN1表 达 上 调 与 肺 腺 癌 患者预后不良密切相关[25];结肠癌中CADM3 高表达与患者预后不良相关[26]。综上,KRAS4BG12C促进细胞运动能力更强,与CLDN1 表达水平升高相关,这也与肺癌细胞系中KRAS4B 更高的表达水平相匹配。虽然KRAS4AG12C促进肺上皮细胞运动较弱,但KRAS4AG12C同样可以促进肺上皮细胞增殖,具有一定的致癌活性。因此我们认为,针对KRAS 突变肿瘤开发的治疗策略仍然需要考虑对KRAS 2 种蛋白剪接体的抑制作用。
本研究在细胞实验中探究了KRAS4AG12C和KRAS4BG12C促进人肺上皮细胞BEAS-2B 生长和运动的差异,即初步证明人肺上皮细胞BEAS-2B 有发生恶性转化的趋势,关于是否发生恶性转化还需体内成瘤实验进一步深入探究。 而KRAS4AG12C和KRAS4BG12C2 种剪接体之间的这种差异与其哪个功能区相关,还需构建蛋白截断体及失活突变过表达细胞模型进一步深入探究。
综上,本研究表明KRAS4AG12C和KRAS4BG12C均可促进人肺上皮细胞增殖,KRAS4BG12C促进肺上皮细胞运动能力更强,并初步探究了相关分子机制。本研究进一步揭示了KRASG12C不同剪接体在肺癌中生物学差异的分子机制,为KRAS特异性靶向抑制剂的研究提供了一定的实验依据和理论基础。
利益冲突声明/Conflict of Interests
所有作者声明不存在利益冲突。
All authors disclose no relevant conflict of interests.
作者贡献/Authors'Contributions
沈瑛和邹菁华参与实验设计;沈瑛、邹菁华、宫淼淼参与论文的写作和修改。所有作者均阅读并同意最终稿件的提交。
The study was designed by SHEN Ying and ZOU Jinghua. The manuscript was drafted and revised by SHEN Ying,ZOU Jinghua and GONG Miaomiao. All the authors have read the last version of paper and consented for submission.
·Received:2022-01-10
·Accepted:2022-03-08
·Published online:2022-04-26
参·考·文·献
[1] TURNER M C,ANDERSEN Z J, BACCARELLI A, et al. Outdoor air pollution and cancer: an overview of the current evidence and public health recommendations[J]. CA A Cancer J Clin, 2020, 70(6):460-479.
[2] TSAY J C J,WU B G,SULAIMAN I,et al. Lower airway dysbiosis affects lung cancer progression[J]. Cancer Discov,2021,11(2):293-307.
[3] BIRKBAK N J, MCGRANAHAN N. Cancer genome evolutionary trajectories in metastasis[J]. Cancer Cell,2020,37(1):8-19.
[4] BERGERS G, FENDT S M. The metabolism of cancer cells during metastasis[J]. Nat Rev Cancer,2021,21(3):162-180.
[5] PRIOR I A, LEWIS P D, MATTOS C. A comprehensive survey ofRasmutations in cancer[J]. Cancer Res,2012,72(10):2457-2467.
[6] FERNÁNDEZ-MEDARDE A, SANTOS E. Ras in cancer and developmental diseases[J]. Genes Cancer,2011,2(3):344-358.
[7] AACR Project GENIE Consortium. AACR project GENIE:powering precision medicine through an international consortium[J]. Cancer Discov,2017,7(8):818-831.
[8] MOORE A R, ROSENBERG S C, MCCORMICK F, et al. RAStargeted therapies: is the undruggable drugged? [J]. Nat Rev Drug Discov,2020,19(8):533-552.
[9] KESSLER D, GMACHL M, MANTOULIDIS A, et al. Drugging an undruggable pocket on KRAS[J]. Proc Natl Acad Sci USA, 2019,116(32):15823-15829.
[10] LI S Q,BALMAIN A,COUNTER C M. A model for RAS mutation patterns in cancers:finding the sweet spot[J]. Nat Rev Cancer,2018,18(12):767-777.
[11] ZHANG B Y, ZHANG Y, ZHANG J W, et al. Focal adhesion kinase(FAK) inhibition synergizes with KRAS G12C inhibitors in treating cancer through the regulation of the FAK-YAP signaling[J]. Adv Sci(Weinh),2021,8(16):e2100250.
[12] VIGIL D,CHERFILS J,ROSSMAN K L,et al. Ras superfamily GEFs and GAPs:validated and tractable targets for cancer therapy?[J]. Nat Rev Cancer,2010,10(12):842-857.
[13] BOS J L, REHMANN H, WITTINGHOFER A. GEFs and GAPs:critical elements in the control of small G proteins[J]. Cell, 2007,129(5):865-877.
[14] AHEARN I M, HAIGIS K, BAR-SAGI D, et al. Regulating the regulator: post-translational modification of RAS[J]. Nat Rev Mol Cell Biol,2011,13(1):39-51.
[15] PELLS S, DIVJAK M, ROMANOWSKI P, et al. Developmentallyregulated expression of murine K-ras isoforms[J]. Oncogene, 1997,15(15):1781-1786.
[16] TSAI F D, LOPES M S, ZHOU M, et al. K-Ras4A splice variant is widely expressed in cancer and uses a hybrid membrane-targeting motif[J]. Proc Natl Acad Sci U S A,2015,112(3):779-784.
[17] SIMANSHU D K, NISSLEY D V, MCCORMICK F. RAS proteins and their regulators in human disease[J]. Cell,2017,170(1):17-33.
[18] SMITH M J, NEEL B G, IKURA M. NMR-based functional profiling of RASopathies and oncogenic RAS mutations[J]. Proc Natl Acad Sci USA,2013,110(12):4574-4579.
[19] HANCOCK J F. Ras proteins: different signals from different locations[J]. Nat Rev Mol Cell Biol,2003,4(5):373-384.
[20] AMENDOLA C R, MAHAFFEY J P, PARKER S J, et al. KRAS4A directly regulates hexokinase 1[J]. Nature,2019,576(7787):482-486.
[21] WANG M T, HOLDERFIELD M, GALEAS J, et al. K-ras promotes tumorigenicity through suppression of non-canonical Wnt signaling[J].Cell,2015,163(5):1237-1251.
[22] SUBRAMANIAN A, TAMAYO P, MOOTHA V K, et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles[J]. Proc Natl Acad Sci USA, 2005,102(43):15545-15550.
[23] LANMAN B A, ALLEN J R, ALLEN J G, et al. Discovery of a covalent inhibitor of KRASG12C (AMG 510) for the treatment of solid tumors[J]. J Med Chem,2020,63(1):52-65.
[24] DHAWAN P, SINGH A B, DEANE N G, et al. Claudin-1 regulates cellular transformation and metastatic behavior in colon cancer[J].J Clin Investig,2005,115(7):1765-1776.
[25] SUN B S,YAO Y Q, PEI B X, et al. Claudin-1 correlates with poor prognosis in lung adenocarcinoma[J]. Thorac Cancer, 2016, 7(5):556-563.
[26] CHEN W Z, HUANG JF, XIONG J B, et al. Identification of a tumor microenvironment-related gene signature indicative of disease prognosis and treatment response in colon cancer[J]. Oxid Med Cell Longev,2021,2021:6290261.
