Prrx1+牙周膜干细胞在牙移动过程中的谱系示踪
2022-10-21汪席均金安婷黄湘如徐弘远杨屹羚代庆刚江凌勇
汪席均,金安婷,黄湘如,徐弘远,高 昕,杨屹羚,代庆刚,江凌勇
1.上海交通大学医学院附属第九人民医院口腔颅颌面外科,上海交通大学口腔医学院,国家口腔医学中心,国家口腔疾病临床医学研究中心,上海市口腔医学重点实验室,上海 200011;2.上海交通大学医学院附属第九人民医院口腔第二门诊部,国家口腔医学中心,国家口腔疾病临床医学研究中心,上海市口腔医学重点实验室,上海 200011
正 畸 牙 移 动 (orthodontic tooth movement,OTM)的生理学基础是正畸应力作用下的牙周改建,包括压力区牙周膜的压缩及旧牙槽骨基质的吸收、张力区牙周膜的拉伸及新骨基质的形成[1]。牙周膜干细胞(periodontal ligament stem cells,PDLSCs)是存在于牙周膜中重要的成体牙源性干细胞,具有间充质干细胞的生物学特性[2],其能够响应正畸应力,并在该应力刺激下的牙周改建中发挥关键作用[3]。长久以来,在OTM 领域中,应力调控PDLSCs 重塑牙周组织的机制研究是诸多学者关注的热点。既往,国内外学者多采用传统方法如体外细胞加力、体内简单动物模型构建等开展研究,但由于体内外环境的差异、体内微环境的复杂性、生长发育的影响等,PDLSCs 在应力刺激下的真实转归与其重塑牙周组织的机制难以被深入解析。
细胞谱系示踪技术是一项利用各种方式对细胞进行标记,并对包括该细胞的后代所有细胞的增殖、分化及迁移等活动进行追踪观察的技术。目前谱系示踪技术包括以下几种:直接显微镜观察法示踪技术、细胞移植示踪技术、Y 染色体示踪标记、染料和放射性示踪剂标记、基因转染技术、MRI 磁性标记成像技术、基因打靶和转基因技术等[4-5]。近年来,基因打靶和转基因技术被广泛应用于对外源标志物基因导入靶细胞的示踪,其克服了传统物理标记中的细胞损伤及标记稀释等问题,但需利用基因组中的位点特异性重组酶序列[6]。目前在位点特异性重组酶系统中,Cre/loxP 和FLP/FRT 重组酶系统使用最为广泛;其中,Cre/loxP 重组酶系统效率更高,且根据时间是否可控分为一般型和诱导型。与一般型相比,诱导型Cre/loxP 重组酶系统可在特定时间点诱导激活重组酶,实现对该时间点被标记细胞的增殖、分化及迁移活动的观察。当前,已有众多学者利用细胞谱系示踪技术研究体内某一特定分子的作用,如利用Gli1-CreERT2小鼠的研究发现神经胶质瘤相关癌基因同源物1 (glioma-associated oncogene homolog 1,Gli1) 可标记骨形成和骨折修复的成骨祖细胞[7],通过Gli1-CreERT2小鼠的研究发现围绕牙槽骨脉管系统的Gli1+细胞是支持种植体骨整合的干细胞[8],利用Osx-CreERT2小鼠的研究发现成骨细胞特异性转录因子(Osterix,Osx)可标记骨发育中的祖细胞[9]。
配对相关同源基因1(paired related homeobox 1,Prrx1)是编码转录因子的同源盒基因,在小鼠间充质中广泛表达。有研究[10]显示,Prrx1+细胞在颅颌面及四肢骨骼的发育中均发挥了重要作用。且有学者发现,Prrx1存在于小鼠切牙及磨牙的PDLSCs 中,并参与了小鼠切牙及磨牙的发育、牙周膜的组织修复再生和持续改建的过程[11-12]。因此,本研究基于Cre/loxP 重组酶系统,构建诱导型条件性荧光表达小鼠,并创新性地将细胞谱系示踪技术与体内OTM 模型相结合,通过免疫荧光染色观察Prrx1+细胞谱系在正畸应力作用下的动态分布,为今后深入研究Prrx1在牙移动过程中的功能提供新的模型与方法。
1 对象和方法
1.1 实验动物
6 周龄、雄性诱导型Prrx1-CreERT2小鼠由上海交通大学生命科学技术学院李保界教授惠赠,体质量约20 g。6 周龄、雌性R26tdTomato荧光标记小鼠购买于江苏集萃药康生物科技股份有限公司,体质量为20~25 g,生产许可证:SCXK(苏)2018-0008。将上述2 种小鼠作为种鼠,以雄性∶雌性=1∶2 进行交配繁殖,获得的子代小鼠用于后续实验。
子代小鼠饲养于上海交通大学医学院附属第九人民医院SPF级动物房,使用许可证:SYXK(沪)2020-0025。饲养条件:温度(24±2)℃、湿度(60±5)%、12 h昼夜循环、自由饮水,并行常规饲料喂养。
1.2 主要仪器与试剂
PCR 仪(BioRad,美国),凝胶成像系统(GE Healthcare,美国),冰冻切片机(Leica,美国),正置荧光显微镜(Olympus,日本)。他莫昔芬(上海昂一生物科技有限公司),DAPI染色液(上海碧云天生物技术有限公司),Alexa Fluor 555 标记的驴抗兔多克隆抗体IgG (Invitrogen,美国),RFP 抗体(Rockland,美国)。
1.3 研究方法
1.3.1 子代小鼠基因型的鉴定 出生2周后,取子代小鼠尾部末端1~2 mm 组织,加入裂解液及蛋白酶K后置于55 ℃恒温水浴锅过夜。而后,向其中加入等量异丙醇,出现絮凝的DNA 沉淀后于10 000×g离心10 min,弃上清液,用ddH2O 溶解沉淀。采用常规PCR对小鼠的基因型进行鉴定,引物序列见表1。

表1 PCR引物序列Tab 1 Primer sequences for PCR
1.3.2 子代小鼠牙周膜中Prrx1+细胞谱系的标记、OTM 模型的构建及分组 取8 只、2 周龄的子代小鼠,于腹腔注射他莫昔芬(10 mg/mL),剂量为200 mg/kg[13],每间隔2 d 注射1 次,连续注射3 次。如小鼠牙周膜中的Prrx1+细胞谱系均表达tdTomato红色荧光蛋白,则细胞标记完成(即为tdTomato+细胞)。
当Prrx1+细胞谱系被标记1周后,构建小鼠OTM模型,具体方法参考本课题组的既往报道[14]。即将自制加力装置的一端结扎固定于左侧上颌第一磨牙,另一端结扎后用树脂固定于上颌切牙;放置加力装置的一侧为OTM 侧,未放置的一侧为对照侧。每日对小鼠口内牙移动装置是否完整、无脱落检查1次。
分别于牙移动第3 日(OTM 3 d)、第7 日(OTM 7 d)时处死小鼠(即OTM 3 d 组、OTM 7 d组),收集其双侧上颌第一磨牙及周围牙周组织,通过体视镜观察并测量上颌第一磨牙(M1)及第二磨牙(M2)间距离。
1.3.3 上颌第一磨牙及牙周组织的冰冻切片的获取 将收集的小鼠双侧上颌第一磨牙及周围牙周组织浸入4%多聚甲醛中固定,并于4 ℃过夜。次日,将其置于10% EDTA(pH7.4)脱钙液中进行脱钙。当上颌第一磨牙可被针尖轻易穿透,视为脱钙完成。随后,将其转入含有20%的蔗糖与2%聚乙烯吡咯烷酮(polyvinyl pyrrolidone,PVP)的混合溶液中进行脱水,再用8%明胶、20%蔗糖和2%PVP的混合冰冻包埋剂进行包埋[15]。使用冰冻切片机在平行于上颌第一磨牙近远中方向进行连续切片,切片厚度为10~12 μm,用于后续观察。
1.3.4 冰冻切片的染色分析 采用苏木精-伊红染色(hematoxylin-eosin staining,H-E 染色)对OTM 3 d及OTM 7 d小鼠双侧上颌第一磨牙近中(压力区)和远中(张力区)的牙周膜进行观察。取“1.3.3”制备的冰冻切片进行固定,于苏木精染液中染色30~60 s,再用1%盐酸乙醇处理1 s后水洗终止反应。待切片返蓝后,加入伊红染液染色20 s,再依次行乙醇梯度脱水、二甲苯中透明、中性树脂封片。而后,于光镜下观察OTM 3 d、OTM 7 d 小鼠压力区及张力区中牙周膜的改变。
1.3.5 冰冻切片的免疫荧光染色分析 采用免疫荧光染色对OTM 3 d及OTM 7 d小鼠双侧上颌第一磨牙近中(压力区)和远中(张力区)的牙周膜进行观察。取“1.3.3”制备的冰冻切片,行抗原修复,经磷酸盐缓冲液(phosphate-buffered saline,PBS)清洗后滴加封闭液,于室温封闭1 h,而后滴加RFP 抗体(1∶1 000) 于4 ℃孵育过夜。次日,加入Alexa Fluor 555 标记的IgG 抗体(1∶1 000)于室温孵育2 h。采用DAPI染色后封片,于共聚焦显微镜下观察Prrx1+细胞谱系在压力区及张力区的动态分布。
1.4 统计学方法
所有数据使用SPSS 16.0 软件进行统计分析。定量资料以表示,采用独立样本t检验进行2组间比较。P<0.05表示差异具有统计学意义。
2 结果
2.1 子代小鼠基因型的鉴定及OTM模型的构建
采用PCR对子代小鼠的基因型进行检测,结果(图1)显示,小鼠基因型为Prrx1-CreERT2;R26tdTomato。

图1 PCR检测子代小鼠基因型Fig 1 Detection of genotype of offspring mice by PCR
通过向子代小鼠的腹腔注射他莫昔芬,获得Prrx1+细胞谱系小鼠。随后,依据图2A 所示,建立OTM 模型。所有构建OTM 模型的Prrx1-CreERT2;R26tdTomato小鼠均全程耐受,可自由饮水、进食软食。于体视镜下对小鼠OTM 侧M1 与M2 间距离进行观察并测量,结果(图2B)显示,OTM 侧M1 发生了明显的近中移动,M1 与M2 间距离随正畸应力作用时间而增加,即OTM 7 d 时的间隙[(87.44±4.02)μm]大于OTM 3 d 时[(42.81±5.04)μm]。因此,本研究的牙移动规律符合先前的研究报道[16],表明OTM模型构建成功。

图2 子代小鼠OTM模型的构建及验证Fig 2 Construction and validation of OTM model in offspring mice
2.2 压力区牙周膜的改变及Prrx1+细胞谱系的表达
正畸应力作用下,采用H-E染色对切片的压力区进行观察,观察区域如图3A 所示。结果(图3B)显示:OTM 3 d 时,小鼠OTM 侧压力区的牙周膜间隙变窄、纤维及管腔被压缩;OTM 7 d 时,OTM 侧的牙周膜发生改建,宽度逐渐被恢复。采用免疫荧光染色对OTM 3 d 和OTM 7 d 小鼠的OTM 侧及对照侧的压力区tdTomato+细胞数量进行分析,结果(图3C、3D)显示:OTM 3 d 小鼠的OTM 侧压力区的牙周膜中tdTomato+细胞数量[(6.35±2.15)%]较对照侧[(13.26±3.02)%]有所减少(P=0.007),OTM 7 d小 鼠 的OTM 侧tdTomato+细 胞 数 量[ (60.41±4.59)%]较对照侧[(20.74±2.62)%]有所增加(P=0.000);且随着正畸应力作用时间的增加,OTM侧tdTomato+细胞数量亦逐渐增加(P=0.000),在OTM 7 d 时tdTomato+细胞已广泛分布于压力区牙周膜内。
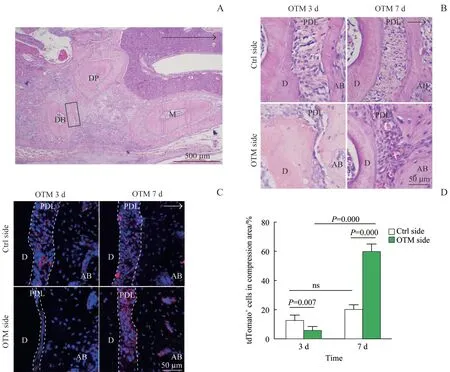
图3 压力区牙周膜的改变及Prrx1+细胞谱系的表达Fig 3 Changes of periodontal ligament and expression of Prrx1+cell lineage in the compression area
2.3 张力区牙周膜的改变及Prrx1+细胞谱系的表达
正畸应力作用下,采用H-E染色对切片的张力区进行观察,观察区域如图4A 所示。结果(图4B)显示:OTM 3 d 时,小鼠OTM 侧张力区的牙周膜被拉伸,牙周膜间隙增宽;OTM 7 d 时OTM 侧的牙周膜排列较OTM 3 d 时更加规则。采用免疫荧光染色对OTM 3 d 和OTM 7 d 小鼠的OTM 侧及对照侧的张力区tdTomato+细胞数量进行分析,结果(图4C、4D)显示:OTM 3 d 小鼠的OTM 侧张力区的牙周膜中tdTomato+细胞数量[(44.48±3.87)%] 较对照侧[(13.64±2.07)%] 有所增加,OTM 7 d 小鼠的OTM 侧tdTomato+细胞数量[(66.88±6.42)%]较对照 侧[(20.90±5.78)%] 亦 有 所 增 加(均P=0.000);且随着正畸应力作用时间的增加,OTM 侧tdTomato+细胞数量逐渐增多(P=0.001),在OTM 7 d时tdTomato+细胞已广泛分布于张力区牙周膜内。

图4 张力区牙周膜的改变及Prrx1+细胞谱系的表达Fig 4 Changes of periodontal ligament and expression of Prrx1+cell lineage in the tension area
3 讨论
随着医疗技术的发展及人民健康意识的增加,牙颌面畸形的诊疗需求与日俱增。如何安全有效地缩短正畸治疗周期、减少正畸复发与并发症的发生,一直是正畸学者密切关注的热点。OTM 是基于牙槽骨重塑的过程,骨髓间充质干细胞(bone marrow mesenchymal stem cells,BMSCs)及PDLSCs 在其过程中起到了重要作用。BMSCs 是体内骨细胞的主要来源[17],在应力刺激下可直接诱导成骨向分化,且分化后的成骨细胞会分泌相关因子间接调控破骨细胞;而PDLSCs 是存在于牙周膜中的力学敏感的牙源性间充质干细胞,可直接感知正畸应力并调节破骨细胞的活动,同时其还可在正畸应力下诱导牙周膜的增殖和分化、调节牙周膜稳态[18]。在体内PDLSCs 所处的微环境较为复杂,体外研究尚无法反映该细胞的真实生物学行为,因此在体内对PDLSCs 进行标记与追踪是研究该细胞在正畸应力下调控牙周改建的前提。
谱系示踪是一项针对单个细胞及其全部后代细胞进行标记及鉴定,以探究该细胞的谱系发生,并对其后代细胞的数量、位置、分化状态等进行追踪观察的技术。作为谱系示踪技术中使用较为广泛的重组酶系统,Cre/loxP 系统可在报告基因前插入loxP-STOPloxP 序列,当Cre 重组酶被激活时,其可切除插入序列中的STOP 序列,使报告基因在所有表达Cre 重组酶的细胞及其后代细胞中表达[19];同时,Cre重组酶还可与突变的ER(Cre-ER)相结合,当给予他莫昔芬后,Cre-ER 能够进入细胞核并发挥重组效应,实现对诱导型特异性荧光蛋白的标记[20]。近年来,诱导型细胞谱系示踪技术已成为研究干细胞谱系发生与转归的关键技术,这也为本课题组突破现有的牙移动机制研究提供了有力工具。然而在OTM 领域中,鲜少有利用谱系示踪技术进行研究的报道。
目前,尚未发现PDLSCs 的特异性标志物。既往研究中,部分学者使用轴向抑制蛋白2 (axis inhibition protein 2,Axin2)、Gli1及Prrx1对PDLSCs进行标记。在成牙本质细胞、成牙骨质细胞和成骨细胞中Axin2表达广泛[21],使得其对PDLSCs 的特异性不足。而Gli1+及Prrx1+细胞在牙周膜改建、小鼠磨牙生长中发挥了重要作用。其中,Prrx1是编码转录因子的同源盒基因,在颅面部骨骼发育和胚胎肢芽形成中高度表达[22],敲除Prrx1基因则会导致小鼠牙颌面骨发育障碍[23-26];同时,Prrx1还参与维持神经干细胞的自我更新能力,高表达于小鼠间充质细胞[27]。这些研究均提示,Prrx1具备干细胞标志物的条件,或参与了牙周组织的发育、稳态维持及修复再生。基于此,本研究选用Prrx1基因作为小鼠PDLSCs 的研究标志物,构建Prrx1+细胞谱系示踪小鼠OTM 体内力学模型,追踪Prrx1+细胞在应力作用下的改变;通过H-E 染色、免疫荧光染色观察后发现,tdTomato+细胞能够标记PDLSCs,且OTM 3 d 和OTM 7 d 小鼠的OTM 侧张力区tdTomato+细胞数量均较对照侧有显著增加。
本研究成功构建了诱导型Prrx1+细胞谱系示踪小鼠的牙移动模型,初步证实了Prrx1+细胞谱系参与OTM 的牙周改建。后续,我们将对成骨、破骨及成纤维细胞的标志物进行检测,对Prrx1+细胞的转归行进一步探究。值得注意的是,作为牙源性干细胞,PDLSCs 具有一定的异质性,存在于多个不同的功能亚群。未来,我们还将利用单细胞测序技术,通过转录组学对PDLSCs 进行精确分群,并对其组织特异性标志物进行筛选及鉴定,为牙周组织的再生提供新思路。此外,本研究构建的Prrx1-CreERT2;R26tdTomato小鼠还可用于牙周损伤、牙周炎等模型的分析,以标记并追踪PDLSCs 参与的其他重要生物学过程及谱系转归。
综上,本研究实现了在正畸应力作用下,时空特异性标记、追踪牙源性干细胞PDLSCs,是国内利用谱系示踪技术结合OTM 模型的较早探索,或将为牙移动机制研究提供全新的动物模型与较理想的研究策略。
利益冲突声明/Conflict of Interests
所有作者声明不存在利益冲突。
All authors disclose no relevant conflict of interests.
伦理批准和知情同意/Ethics Approval and Patient Consent
本研究涉及的所有动物实验均已通过上海交通大学医学院附属第九人民医院科学伦理委员会的审核批准(SH9H-2020-A235-1)。所有实验过程均遵守《关于善待实验动物的指导性意见》。
All experimental animal protocols in this study were reviewed and approved by Shanghai Ninth People's Hospital,Shanghai Jiao Tong University School of Medicine (Approval Letter No. SH9H-2020-A235-1,dated 03/03/2020),and all experimental animal protocols were carried out by following the guidelines ofInstructive Notions with Respect to Caring for Laboratory Animals.
作者贡献/Authors'Contributions
汪席均负责实验、数据整理分析、论文撰写;金安婷参与论文的写作和修改;黄湘如负责动物培育及基因型鉴定;徐宏远、高昕负责资料收集与分析;杨屹羚负责购买试剂、参与实验分析;代庆刚、江凌勇参与了实验设计及指导。所有作者均阅读并同意了最终稿件的提交。
The experiment,data compilation and analysis and paper writing were completed by WANG Xijun. The participation in writing and revising was done by JIN Anting. The animal breeding and genotype identification was done by HUANG Xiangru. The data collection and analysis were performed by XU Hongyuan and GAO Xin. The purchase of reagents and the participation in the experimental analysis were done by YANG Yiling. The study was designed by JIANG Lingyong and DAI Qinggang.All the authors have read the last version of paper and consented for submission.
·Received:2022-04-14
·Accepted:2022-08-13
·Published online:2022-08-28
参·考·文·献
[1] LI Y N, JACOX L A, LITTLE S H, et al. Orthodontic tooth movement: the biology and clinical implications[J]. Kaohsiung J Med Sci,2018,34(4):207-214.
[2] SEO B M, MIURA M, GRONTHOS S, et al. Investigation of multipotent postnatal stem cells from human periodontal ligament[J].Lancet,2004,364(9429):149-155.
[3] WISE G E, KING G J. Mechanisms of tooth eruption and orthodontic tooth movement[J]. J Dent Res,2008,87(5):414-434.
[4] 赵闫,欧阳宁鹃,代杰文,等. 细胞谱系示踪技术及其在口腔医学研究中的应用进展[J]. 口腔生物医学,2017,8(4):205-210.ZHAO Y,OUYANG N J,DAI J W,et al. Development of cell lineage tracing technology and its application in stomatology research[J]. Oral Biomed,2017,8(4):205-210.
[5] BULTE J W M,DOUGLAS T,WITWER B,et al. Monitoring stem cell therapyin vivousing magnetodendrimers as a new class of cellular MR contrast agents[J]. Acad Radiol,2002,9(Suppl 2):S332-S335.
[6] NAGY A. Cre recombinase: the universal reagent for genome tailoring[J]. Genesis,2000,26(2):99-109.
[7] SHI Y, HE G X, LEE W C, et al. Gli1 identifies osteogenic progenitors for bone formation and fracture repair[J]. Nat Commun,2017,8(1):2043.
[8] YI Y,STENBERG W,LUO W,et al. Alveolar bone marrow Gli1+stem cells support implant osseointegration[J]. J Dent Res, 2022, 101(1):73-82.
[9] MIZOGUCHI T, PINHO S,AHMED J, et al. Osterix marks distinct waves of primitive and definitive stromal progenitors during bone marrow development[J]. Dev Cell,2014,29(3):340-349.
[10] KAWANAMI A, MATSUSHITA T, CHAN Y Y, et al. Mice expressing GFP and CreER in osteochondro progenitor cells in the periosteum[J]. Biochem Biophys Res Commun, 2009, 386(3):477-482.
[11] BASSIR S H, GARAKANI S, WILK K, et al. Prx1 expressing cells are required for periodontal regeneration of the mouse incisor[J].Front Physiol,2019,10:591.
[12] GONG X Y, ZHANG H, XU X Q, et al. Tracing PRX1+cells during molar formation and periodontal ligament reconstruction[J]. Int J Oral Sci,2022,14(1):5.
[13] ZHONG Z A, SUN W H, CHEN H Y, et al. Optimizing tamoxifeninducible Cre/Loxp system to reduce tamoxifen effect on bone turnover in long bones of young mice[J]. Bone,2015,81:614-619.
[14] DAI Q G, ZHOU S R, ZHANG P, et al. Force-induced increased osteogenesis enables accelerated orthodontic tooth movement in ovariectomized rats[J]. Sci Rep,2017,7(1):3906.
[15] KUSUMBE A P, RAMASAMY S K, STARSICHOVA A, et al.Sample preparation for high-resolution 3D confocal imaging of mouse skeletal tissue[J]. Nat Protoc,2015,10(12):1904-1914.
[16] 孙聪,刘文佳,金岩,等. 小鼠正畸牙齿移动模型的制作[J]. 牙体牙髓牙周病学杂志,2017,27(5):280-283.SUN C, LIU W J, JIN Y, et al. Establishment of mouse model of orthodontic tooth movement[J]. Chin J Conserv Dent, 2017, 27(5):280-283.
[17] ABDALLAH B M, KASSEM M. Human mesenchymal stem cells:from basic biology to clinical applications[J]. Gene Ther, 2008,15(2):109-116.
[18] WANG K, XU C, XIE X, et al. Axin2+PDL cells directly contribute to new alveolar bone formation in response to orthodontic tension force[J]. J Dent Res,2022,101(6):695-703.
[19] KIM H, KIM M, IM S K, et al. Mouse Cre-LoxP system: general principles to determine tissue-specific roles of target genes[J]. Lab Anim Res,2018,34(4):147-159.
[20] FEIL R, BROCARD J, MASCREZ B, et al. Ligand-activated sitespecific recombination in mice[J]. Proc Natl Acad Sci U S A, 1996,93(20):10887-10890.
[21] XIE X, WANG J, WANG K, et al. Axin2+-mesenchymal PDL cells,instead of K14+epithelial cells, play a key role in rapid cementum growth[J]. J Dent Res,2019,98(11):1262-1270.
[22] LI B, WANG Y G, FAN Y, et al. Cranial suture mesenchymal stem cells:insights and advances[J]. Biomolecules,2021,11(8):1129.
[23] MARTIN J F, BRADLEY A, OLSON E N. The paired-like homeo box gene MHox is required for early events of skeletogenesis in multiple lineages[J]. Genes Dev,1995,9(10):1237-1249.
[24] LU M F, CHENG H T, KERN M J, et al.Prx-1functions cooperatively with another paired-related homeobox gene,prx-2, to maintain cell fates within the craniofacial mesenchyme[J].Development,1999,126(3):495-504.
[25] MITCHELL J M, HICKLIN D M, DOUGHTY P M, et al. ThePrx1homeobox gene is critical for molar tooth morphogenesis[J]. J Dent Res,2006,85(10):888-893.
[26] BALIC A, ADAMS D, MINA M N. Prx1 and Prx2 cooperatively regulate the morphogenesis of the medial region of the mandibular process[J]. Dev Dyn,2009,238(10):2599-2613.
[27] WANG J, SARASWAT D, SINHA A K, et al. Paired related homeobox protein 1 regulates quiescence in human oligodendrocyte progenitors[J]. Cell Rep,2018,25(12):3435-3450.e6.
