一种lncRNA与疾病关联的多层线性投影预测算法
2022-10-15谢国波韩玉琼林志毅
谢国波,韩玉琼,林志毅
(广东工业大学 计算机学院,广州 510006)
E-mail:lzy291@gdut.edu.cn
1 引 言
在ncRNA中,长度大于200个核苷酸的被称为长非编码RNA(lncRNA)[1,2],占所有ncRNA的80%~90%.越来越多基于生物学实验的证据表明,大量的lncRNA在各种基本和重要的生物学过程中发挥着至关重要的作用[3],例如分子诱饵,分子向导,分子支架和信号通路调节剂[4].与lncRNA相关的各种生物学数据集存储在公共数据库中,例如NRED[5],lncRNAdb[6],NONCODE[7]等,其中包括lncRNA的序列,表达和功能等信息.
近年来,研究人员开始更加关注和分析已知的lncRNA-疾病关联,并探索其关联机制[8].考虑到生物学实验需要昂贵的成本并消耗大量时间,实验证实的lncRNA-疾病关联仍然有限.因此,研究人员已经开发出了许多计算模型预测潜在的lncRNA-疾病关联[9].目前,许多有效预测潜在的lncRNA-疾病关联的计算方法已经被提出,其可以分为以下两类.
1)基于生物网络的计算方法,通过构建lncRNA与疾病的生物信息网络预测未知的关联[10,11].例如,Sun等人[12]提出了一种名为RWRlncD的计算方法,通过在lncRNA的功能相似性网络上实施重启随机游动预测潜在的lncRNA-疾病关联,但是这个方法数据单一,未融合丰富的生物数据信息.Xie[13]等人使用标签传播将初始关联矩阵与lncRNA和疾病的特征混合,然后利用特征编码计算出预测关联矩阵,但是该方法存在参数过多,尚无法解决最优参数的选择问题.由于相似性网络数据的准确性难以保证,因此基于生物网络的计算方法存在着难以构建信息全面的网络相似性模型的问题.
2)基于机器学习的计算方法,根据已知的lncRNA-疾病关联对未知的关联关系进行训练分类,通过预测得分对候选样本进行分类[14-16].Chen等人[17]在相似的疾病通常与功能性相似的lncRNA相关的假设下提出一种半监督学习方法,该方法使用拉普拉斯正则最小二乘法(LRLSLDA)预测lncRNA-疾病关联,通过整合已知的lncRNA-疾病关联信息和lncRNA表达谱,可以有效地识别潜在的疾病-lncRNA关联,但是这个方法仍然存在负样本难以获得的问题.Tan等人[18]从多视图学习共识图,同时基于多标签学习和优化来预测lncRNA-疾病关联概率,但是这个方法没有考虑数据的全局信息,使得预测精度低.Lu[19]等人开发了一种基于矩阵补全的算法(SIMCLDA)预测lncRNA与疾病之间的潜在关联.目前,基于机器学习的大部分计算方法存在着负样本缺少的问题,其预测准确性难以进一步提高.
基于机器学习的计算方法会考虑到所有的lncRNA和疾病节点,可以有效避免基于生物网络的计算方法的缺陷,但是该类方法仍然存在着没有充分利用lncRNA-疾病关联数据的局部结构和全局结构信息的问题.因此,本文提出一种新的lncRNA与疾病关联的多层线性投影预测计算方法(MLPLDA).首先,MLPLDA通过组合加权方法对lncRNA的两个相似性和疾病的两个相似性进行整合,以获得更加完善的lncRNA-lncRNA相似性矩阵和疾病-疾病相似性矩阵.然后,使用加权的K最近邻已知邻居算法(WKNKN)对原始的lncRNA-疾病关联矩阵进行重构,解决了原始lncRNA-疾病关联矩阵中数据稀疏性问题,从而有效提高MLPLDA模型的预测性能.最后,MLPLDA通过多层线性投影方法来预测lncRNA与疾病潜在的关系,多层线性投影方法引入了层堆叠策略,将上一层的输出结果作为下一层的输入,从而充分利用了lncRNA-疾病关联数据的局部结构和全局结构信息,显著地提高了模型的预测性能.实验结果表明,MLPLDA在留一交叉验证[20](LOOCV)和五折交叉验证(5-fold-CV)[21]下的AUC值分别为0.8807和0.8563±0.0045与其他方法(RWRlncD,LRLSLDA,SIMCLDA和LLCPLDA)相比具有优越性能.此外,我们还对肺癌、乳腺癌和骨肉瘤3种疾病进行案例分析,以进一步证明MLPLDA模型的预测能力.
2 数 据
2.1 lncRNA-疾病关联数据
本文从LncRNADisease[22]数据库下载了已知的lncRNA-疾病关联数据.该数据集包括了115个lncRNA与178种疾病以及540个经过实验验证的lncRNA-疾病关联.本文用A∈Rnl×nd表示邻接矩阵,其中nl和nd分别表示lncRNA和疾病的数量.如果lncRNAli与疾病dj相关,则A(li,dj) = 1,否则为0.
2.2 LncRNA表达相似性
本文从ArrayExpression[23]下载了表达谱,并通过高通量测序收集了超过150万个表达谱信息,然后根据Spearman相关性计算不同lncRNA之间的表达相似性[11].我们用矩阵SL∈Rnl×nl来表示lncRNA的表达相似性,其中SL(li,lj)代表lncRNAli和lncRNAlj之间的相似度.
2.3 疾病语义相似性
使用有向无环图(DAG)来描述疾病语义相似性是一种常见的方法,本文在美国国家医学图书馆下载了MeSH[24]疾病描述符,以获取原始的语义相似性数据,并在下载的描述符上重构了每种疾病的DAG.对于疾病di的DAG,首先计算疾病的语义值.
计算出疾病t对疾病di的语义值,如式(1)所示:
(1)
疾病di的语义值是其祖先疾病和di贡献值的总和,如公式(2)所示:
D(di)=∑t∈DAG(di)Ddi(t)
(2)
两种疾病之间的语义相似度定义为与疾病di和dj的DAG共享的节点对其语义相似度的贡献之和,如公式(3)所示:
(3)
SD代表疾病语义相似度矩阵;D(di)和D(dj)分别是疾病di和dj的语义值.
2.4 lncRNA疾病高斯核相似性
本文利用已知的lncRNA-疾病关联关系网络分别构建了lncRNA和疾病的高斯核相似性矩阵[25],将lncRNAli和lncRNAlj之间的高斯核相似性定义为GL,将疾病di和dj的高斯核相似性定义为GD.GD和GL计算如下:
(4)
(5)
其中,IP(li)和IP(di)代表第i行向量,分别可以称为lncRNAli和疾病di的相互作用谱,作为高斯核的特征向量.
这里,核带宽参数γl和γd的定义如下:
(6)
(7)
根据相关文献[26],本文将原始核带宽参数γ′l和γ′d的值设置为1.
3 lncRNA-疾病关联的多层线性投影算法(MLPLDA)
本文提出了一种lncRNA与疾病关联的多层线性投影预测算法(如图1所示):1)采用组合加权的方式整合lncRNA和疾病的两种相似性;2)使用WKNKN重构初始的lncRNA-疾病的关联矩阵,以减少原始关联矩阵的稀疏性;3)通过使用堆叠层策略的多层线性投影方法来进一步捕获潜在的交互作用,最终得到lncRNA-疾病关联预测评分矩阵.

图1 MLPLDA方法流程图Fig.1 Flowchart of MLPLDA method
3.1 组合加权法构建lncRNA与疾病的整合相似性
为了获得更加丰富的生物学信息并增强相似性内核,我们分别将高斯核相似性与lncRNA表达相似性和疾病语义相似性通过线性组合加权的方法来构建lncRNA的整合相似性矩阵与疾病的整合相似性矩阵,如公式(8)和公式(9)所示:
NLS=α×SL+(1-α)×GL
(8)
NDS=β×SD+(1-β)×GD
(9)
3.2 加权的K最近邻已知邻居(WKNKN)算法
原始lncRNA-疾病关联矩阵A是一个稀疏矩阵,其中大多数值为零,这种情况可能会导致在预测潜在的lncRNA-疾病关联中表现不理想.
为此,本文使用加权的K最近邻已知邻居(WKNKN)算法[27]来估计未知的lncRNA-疾病关联.首先,通过使用疾病的整合相似性矩阵NDS更新A的每一列:
(10)
其中,ξd=∑dj∈N(di)NDS(di,dj),NDS(di,dj)表示疾病di与疾病dj之间的相似性,Acol(q)表示A矩阵的第q列,N(di)是疾病di的前n个邻居.接下来是通过lncRNA的整合相似性矩阵NLS更新A的每一行:
(11)
其中,ξl=∑lj∈N(li)NLS(li,lj),NLS(li,lj)表示lncRNAli与lncRNAlj之间的相似性,Arow(j)表示矩阵A的第j行,N(li)是lncRNAli的前n个邻居.之后,我们通过以下公式来更新A矩阵:
(12)
Aupdate=max(Aupdate,A)
(13)
3.3 多层线性投影
本文提出了一种多层线性投影模型,在线性投影模型中引入堆叠层策略,将上一层的输出结果作为下一层的输入,并且在下一层中对更新计算的lncRNA和疾病的相似性重新组合,在上一层计算结果的基础上进一步补全了lncRNA-疾病关联预测矩阵,以进一步提高关联预测性能.首先构造异质矩阵H如下[28]:
(14)
其中,zl和zd是平衡系数.假设lncRNA与疾病之间关联的可能性可以写成矩阵H和投影矩阵C的线性组合,通过投影矩阵C将H映射到P中,以便P可以近似于H:
P=HC
(15)

(16)
ε是一个控制阈值,用来表示H与P之间的相似程度.此外,通过对投影矩阵C引入L2正则化来最小化目标函数,同时避免过拟合:

(17)
其中,η是超参数,通过交叉验证确定.展开上述式子:
Q=ηTr[(H-HCT)(H-HC)]+Tr(CTC)=
ηTr(HTH-HTHC-CTHTH+CTHTHC)+Tr(CTC)
(18)
对C求导如下:
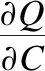
(19)

C*=η(ηHTH+I)-1HTH
(20)
P=HC*
(21)
接下来,提取出P矩阵左下角相应部分并与原始lncRNA-疾病关联矩阵A进行比较获得新的lncRNA-疾病关联矩阵P1output,如下所示:
P1output=max(A,P[(ρ+1):(ρ+),1:ρ]T)
(22)
为了便于描述,将第1层操作记为:
P1output=flayer(A,NLS,NDS,n,zl,zd)
(23)
参考先前的研究成果,zl和zd被设置为1.根据堆叠层策略,将P1output作为第2次迭代的输入,根据上述过程可以表示为:
P2output=flayer(P1output,NLS2,NDS2,n,zl,zd)
(24)
其中,NLS2和NDS2分别是经过第1层计算迭代后的lncRNA与疾病的整合相似性.以此类推,通过上一层推断的结果进行下一层的计算.而堆叠的层数k是根据模型的最佳性能来确定.
经过迭代更新,最终获得lncRNA-疾病关联预测评分矩阵Apredict=max(Pkoutput,Pk-1output).
4 结果和讨论
4.1 与其他方法的比较
为了进一步评估MLPLDA模型,我们将其与其他预测方法(RWRlncD,LRLSLDA,SIMCLDA和LLCPLDA)进行了比较. 首先在LOOCV实验中将MLPLDA与上述方法进行了比较(见图2).MLPLDA获得的AUC为0.8807,高于其他方法(LLCLPLDA为0.8712;LRLSLDA为0.8349;SIMCLDA为0.8298; RWRlncD为0.6448).然后,本文使用5-fold-CV框架进一步验证了MLPLDA的性能.如图3所示,MLPLDA的AUC值为0.8563±0.0045,优于其他方法(LLCLPLDA:0.8247±0.0046; LRLSLDA:0.8370±0.0041; SIMCLDA:0.8091±0.0062; RWRlncD:0.7015±0.0056).总而言之,MLPLDA在本文使用的数据集下优于其他方法且具有良好的性能.

图2 在LOOCV实验下评估的不同方法的性能Fig.2 Performance of different methods evaluated under the LOOCV framework

图3 在5-fold-CV实验下评估的不同方法的性能Fig.3 Performance of different methods evaluated under the 5-fold-CV framework
4.2 参数讨论
为了得到方法中的4个参数的最优值,一般的方法是构造一个四维矩阵,然后计算出所有情况下的AUC值,并找到最佳AUC值,从而得到参数的最优值.但是,该方法存在计算时间太长、计算机成本太高的缺点.由于本文算法的4个变量之间是相互独立的,因此,本文采用如下方法进行参数调优:首先,在对第1个参数进行调优时,其余3个参数的值分别取其调整范围的中间数.获得第1个参数得最优值后,固定其值,继而使用同样方法寻找第2个参数的最优值,其余参数按照上述步骤进行赋值.以此类推,重复上述步骤获得每个参数的最优取值.
本文方法引入了4个参数:权重参数α和β、邻居数n和平衡参数η.为了分析参数对整个模型的影响并获得最佳参数,实验中使用网格法进行参数选择并通过LOOCV框架和5-fold-CV框架进行验证.权重参数α和β的调整范围是0.1~0.9,步长为0.1;邻居数n的调整范围是2~20,步长为2;平衡参数η的调整范围是0.01~0.1,步长为0.01.如图4和图5所示,当α=0.8,β=0.1,n=2,η=0.02时,算法的预测性能最佳.

图4 参数α、β、n、η在LOOCV框架下对模型的影响Fig.4 Effect of parameters α,β,n,η on the method in LOOCV
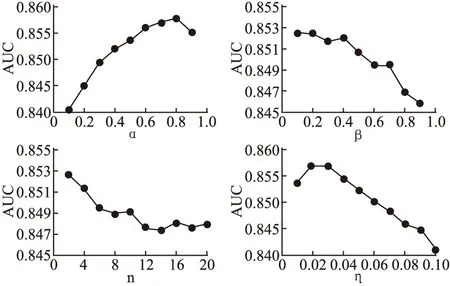
图5 参数α、β、n、η在5-fold-CV框架下对模型的影响Fig.5 Effect of parameters α,β,n,η on the method in 5-fold-CV
4.3 案例分析
为了评估MLPLDA模型的性能,本文对3种疾病(肺癌,宫颈癌和骨肉瘤)进行了案例研究.在实验验证中,使用所有已知的lncRNA-疾病关联作为训练集.当MLPLDA获得预测结果时,根据不同疾病获取了得分前10的llncRNA,并在Lnc2Cancer V2.0[29]和MNDR V2.0[30]中进行了验证.表1~表3显示了MLPLDA在肺癌,乳腺癌,骨肉瘤预测中未知关联的前10个lncRNA结果,分别达到90%,80%和100%的结果.

表1 预测与肺癌相关的IncRNA

表2 预测与乳腺癌相关的lncRNATable 2 Predicted lncRNAs associated with breast cancer
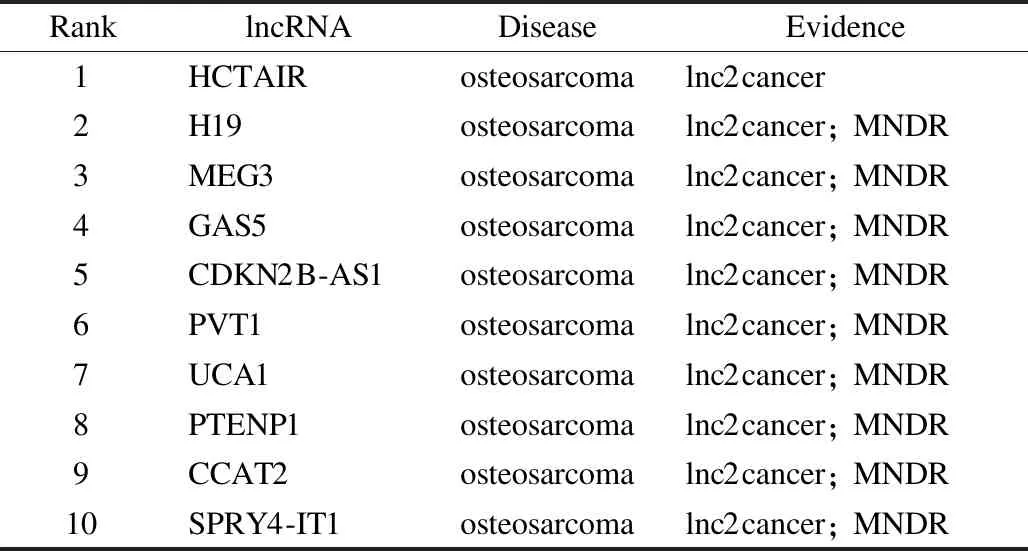
表3 预测与骨肉瘤相关的lncRNATable 3 Predicted lncRNAs associated with osteosarcoma
肺癌是肺组织中细胞的生长不受控制,通过移动到附近的组织或身体的其他部位,这些细胞可以生长并扩散到肺部以外[31].肺癌最常见的症状是咳嗽、血痰.长期吸烟是肺癌的主要原因(高达85%).从未吸烟的人患此病的机会通常为10%~15%,通常是由遗传因素,职业条件,电离辐射或空气污染引起的.Du等人[32]提到lncRNA CDKN2B-AS1调节p53信号通路,以预测患有特发性肺纤维病患者的肺癌. lncRNA GAS5在肺癌的发展中起抑制作用.此外,lncRNA UCA1可以调节肺癌细胞的增殖和侵袭并诱导细胞凋亡.因此,UCG1可能成为抑制肺癌的重要治疗靶点[33].
乳腺癌是女性中被诊断最多的癌症,也是癌症死亡的主要原因,占女性所有癌症的22%.传统的诊断基于肿瘤的大小,淋巴结状态和其他特征[34].近年来,越来越多的证据表明lncRNA在包括乳腺癌在内的癌症生物学中起着至关重要的作用.在乳腺癌的诊断和治疗中发现乳腺癌lncRNA与癌症的关系至关重要.研究表明,PTENP1在乳腺癌中起着至关重要的作用,它可以通过AKT和MAPK信号通路抑制乳腺癌细胞的增殖和迁移[35].研究表明lncRNA CCAT1在乳腺癌中属于高水平表达,并与总生存期和无进展生存期相关,是乳腺癌进展的潜在预后标志物[38].研究表明lncRNA TUG1[39]在乳腺癌组织和高侵袭性乳腺癌细胞系中得到显著表达.
骨肉瘤是最常见的骨恶性肿瘤类型.它从间充质细胞系发展而来.肿瘤的快速生长归因于肿瘤的骨样组织和通过肿瘤的软骨阶段直接或间接形成的骨组织.在外部因素(例如病毒)的影响下,下肢的负重骨骼导致细胞突变.骨肉瘤的突出症状是肿瘤部位的疼痛.这种疼痛是由肿瘤组织侵蚀和骨皮质溶解引起的.在骨肉瘤细胞中,CCAT2属于高水平表达.但是CCAT2沉默降低了MEG3细胞的活力,限制细胞增殖,迁移和侵袭,并促进细胞凋亡[36].lncRNA H19的下调可以抑制骨肉瘤细胞的迁移和侵袭[37].lncRNA UCA1[38]的沉默可抑制骨肉瘤细胞的增殖,促进细胞凋亡,并抑制细胞的侵袭和迁移.进一步的研究表明,它也可能促进骨肉瘤的发生和发展,并且是疾病和治疗靶标的预后标志物.
5 结 论
在本文提出了一种lncRNA与疾病关联预测的多层线性投影预测算法:1)MLPLDA分别通过组合加权整合lncRNA和疾病的两种相似性,以获得更完善的lncRNA和疾病相似性矩阵;2)使用整合的lncRNA相似性和疾病相似性的前n个邻居减少了原始关联矩阵的稀疏性;3)通过使用堆叠层策略的多层线性投影方法,有效利用了数据矩阵中的几何结构之间的联系,进一步捕获潜在的交互作用,最终得到lncRNA-疾病预测关联矩阵.为了更好地验证MLPLDA的预测性能,本文进行了留一交叉验证和五折交叉验证,结果表明本方法在lncRNA与疾病的关联预测中具有良好的性能.与此同时,对3种疾病的案例分析也充分表明了MLPLDA对lncRNA-疾病潜在关联具有可靠的预测效果.
尽管MLPLDA取得了相对较好的实验结果,但仍有一定局限性.例如MLPLDA需要在高质量、高可信度数据源下计算才能达到更好的效果,但目前可用且可靠的数据有限,并且数据中存在一定数量的噪声,在一定程度上影响了模型的性能.因此,结合多数据源,提高数据的质量和可信度也是我们下一步的研究方向.
