杜仲TCP转录因子鉴定及生物信息学分析
2022-09-20吴耀松任闪闪陈玉龙
刘 俊,李 龙,吴耀松,刘 燕,任闪闪,陈玉龙*
杜仲TCP转录因子鉴定及生物信息学分析
刘 俊1, 3, 4,李 龙2,吴耀松1,刘 燕1,任闪闪1,陈玉龙1*
1. 河南中医药大学中医药科学院 河南省中医方证信号传导重点实验室,河南 郑州 450046 2. 西北农林科技大学林学院,陕西 杨凌 712100 3. 国际竹藤中心 国家林业和草原局竹藤科学与技术重点开放实验室,北京 100102 4. 国际竹藤中心 安徽太平试验中心,安徽 黄山 245716
对杜仲TCP(teosinte branched 1/cycloidea/proliferating)基因家族进行筛选分析,以期为基因功能深入研究奠定基础。以杜仲基因组数据库为基础,通过生物信息学系统分析基因家族的理化性质、系统进化、基因结构、启动子顺式作用元件及其在杜仲叶片发育及胶形成中的表达水平。从杜仲基因组中共鉴定到14个,氨基酸数目介于139~492,理论等电点分布在5.54~9.72,相对分子质量区域为18 880~53 620,亚细胞定位预测在细胞核中,均为亲水性蛋白。系统进化分为PCF、CIN和CYC/TB1 3个亚家族,分别包含5、6、3个EuTCPs蛋白。表达模式分析显示,在杜仲叶片不同发育时期存在显著差异,每个发育时期表达量各不相同,正调控杜仲胶的形成。杜仲中TCP家族包含14个成员,各成员的分子特征和组织表达特异性存在差异,可能参与杜仲叶片发育及杜仲胶的形成。
杜仲;TCP转录因子;亚细胞定位;系统进化;表达分析
TCP(teosinte branched1,cycloidea,and proliferating cell factor family)蛋白一般由60个氨基酸组成,含有一个非典型的基本螺旋(basic helix-loop-helix,bHLH)结构和R结构域[1]。TCP基因家族分为Class I和Class II 2个亚家族,Class I又称为PCF亚家族,Class II进一步划分为CIN和CYC/TB1亚家族[2]。TCP转录因子参与植物胚胎发育[3]、侧枝形成[4-5]、开花调控[6]、胁迫响应[7]以及激素信号[5]等多种生物学过程。
TB1和TB1-like蛋白属于CYC/TB1亚家族,抑制拟南芥分枝发育[8]。通过诱导脂肪氧化酶()基因表达,促进茉莉酸(jasmonic acid,JA)生物合成和成熟叶片衰老[9],在花器官中抑制基因表达[10]。通过靶向生长素生物合成酶基因调控生长素生物合成[11],是PCF亚家族成员,在种子萌发过程中发挥负调控作用[12]。毛竹通过脱落酸(abscisic acid,ABA)依赖信号在抗旱中发挥正调控作用,以茉莉酸甲酯(methyl jasmonate,Me-JA)介导途径负调控侧根生长[13]。通过正调控影响水稻耐盐性[14],和是microRNA319的靶基因,在冷胁迫中发挥正调控作用[15]。在拟南芥中超表达诱导、、上调表达,下调表达,导致侧根数目减少,提高转基因植株胁迫耐受性[16]。
杜仲Oliver是我国特有单科、单属、单种的多年生第三纪孑遗树种,雌雄异株,具有橡胶和药材的双重性质[17]。广泛分布于温带及亚热带地区,适应能力强,在酸性或盐碱性的贫瘠土壤中均能生长良好[18]。其树皮、叶片及种子均可入药,富含多种活性成分,对多种疾病的预防及提高人体免疫力具有显著的效果,是我国二级珍稀保护植物。目前,对杜仲的研究与利用多局限于资源收集、药理学及形态学研究,分子遗传学方面的报道相对较少,严重制约了杜仲分子育种及相关生物学特性研究。目前TCP转录因子已在多个物种中进行了研究,然而杜仲TCP基因家族还未报道,本研究以杜仲基因组数据[19]为基础,从基因组水平对基因家族进行鉴定、理化性质、进化关系、保守基序以及表达模式分析,以期为进一步探索杜仲基因功能奠定基础。
1 材料与仪器
杜仲材料种植于西北农林科技大学苗圃(陕西杨凌),经西北农林科技大学李龙博士鉴定为杜仲Oliver。取生长正常、长势一致的2年生“秦仲1 号”(Qinzhong 1)杜仲幼苗的叶芽(茎尖)、生长叶(长叶片,3 cm)、幼叶(完全展开的新叶)、老叶(完全展开60 d叶片),每个样品3个重复,经液氮处理后−80 ℃冰箱保存,用于RNA提取。
2 方法
2.1 杜仲TCP基因家族鉴定及理化性质分析
从杜仲基因组数据库Genome Warehouse(https://bigd.big.ac.cn/gwh/Assembly/13/show)中下载TCP蛋白候选序列,利用NCBI的(Conserved Domain Search Service(CD Search))分析蛋白结构域,保留含有完整bHLH结构域的序列。通过在线软件ProtParam (http://web.expasy. org/protparam/)分析蛋白理化性质,使用Plant-mPLoc (http:// www.csbio.sjtu.edu.cn/bioinf/ plant-multi/)预测EuTCPs蛋白亚细胞定位,利用在线工具ExPASY(https://www.expasy.org/tools)预测EuTCPs氨基酸数量、相对分子质量(W)、理论等电点(PI),通过Expasy(https://web. expasy.org/protscale/)软件分析蛋白的亲疏水性,利用SOPMA(https://npsa- prabi.ibcp.fr/cgi-bin/npsa_automat.plpage=npsa_sopma.html)和Phyre 2(http://www.sbg.bio.ic.ac.uk/ phyre2/html/page. cgi?id= index)软件分别预测蛋白的二级结构和三级结构。
2.2 杜仲TCPs蛋白系统发育树构建
通过Clustal X1.83软件对杜仲、水稻、拟南芥和毛竹TCPs氨基酸序列进行多序列比对,利用MEGA 6.0的邻接法(neighbor-joining,N-J)构建系统发育树,重复次数设置为1000次,其他参数为默认值[20]。
2.3 保守基序及启动子分析
通过MEME(http://meme-suite.org/)在线软件对EuTCP基因家族成员进行基序分析(参数是:any number of Repetitions(anr),maximum number of Motifs=10,minimum width≥6,and maximum width ≤50)。为了解析启动子区域顺式作用元件,对基因ATG上游2000 bp序列进行查找分离,利用Plant CARE(http://bioinformatics. psb.ugent.be/webtools/plant care/htmL/)软件进行启动子顺式作用元件分析。
2.4 杜仲EuTCP基因家族表达模式分析
从NCBI的Short Read Arshive(SRA)数据库中下载杜仲叶片不同发育时期(叶芽、初生叶、幼叶、老叶)(版本号SRP218063)[21]及不同胶含量(低含量、高含量)(版本号SRP158357)[22]转录组数据,基因相对表达丰度用FPKM值表示,对该数值取对数(log2)进行统计分析,通过MeV4-9-0工具绘制基因表达图谱,表达量最高的用绿色方框表示,表达量最低的用黄色方框表示。
2.5 杜仲TCPs蛋白互作网络预测
利用STRING软件(https://string-db.org/)上传EuTCPs蛋白序列,选择拟南芥数据库进行序列比对,根据已知拟南芥蛋白互作关系,通过Blast比对杜仲同源蛋白,通过Cytoscape 3.7.0软件对EuTCP基因家族蛋白互作信息进行评估和预测[23]。
3 结果与分析
3.1 杜仲TCP基因家族鉴定及理化性质分析
通过Genome Warehouse数据库,从杜仲基因组中共鉴定到14个基因(表1),利用Pfam和NCBI的Conserved Domain Search在线分析软件对EuTCPs蛋白进行结构域验证。结果显示:14个EuTCPs蛋白均含有非典型的bHLH(BASIC、HELIX I、LOOP和HELIX II)特征结构域,分别命名为EuTCP1~EuTCP14。使用ExPASy工具,对EuTCP家族成员进行蛋白质理化性质分析,EuTCP14蛋白最长,编码492个氨基酸,EuTCP13蛋白最短,编码139个氨基酸,相对分子质量分布区域为18 880~53 620,等电点范围是5.54(EuTCP14)~9.72(EuTCP6)。亚细胞定位预测结果显示,EuTCPs均定位在细胞核中(表1)。
表1 杜仲TCP蛋白序列特征及亚细胞定位预测
Table 1 Amino acid sequence characteristics and predicted subcellular location of E. ulmoides TCP proteins
基因ID基因名拟南芥同源基因染色体定位CDS长度/bp蛋白质大小/aaMWPI亚细胞定位 EUC12473-RAEuTCP1AT2G37000Super-Scaffold_57 62720822 441.518.85细胞核 EUC20760-RAEuTCP2AT2G31070scaffold865_obj 78025929 028.368.48细胞核 EUC13147-RAEuTCP3AT4G18390Super-Scaffold_67131143647 303.138.82细胞核 EUC03320-RAEuTCP4AT3G27010Super-Scaffold_177 93030932 689.328.99细胞核 EUC22153-RAEuTCP5AT3G02150scaffold1183_obj102934238 298.696.90细胞核 EUC10434-RAEuTCP6AT5G23280Super-Scaffold_46 79826527 785.139.72细胞核 EUC05665-RAEuTCP7AT1G68800Super-Scaffold_28108336041 030.627.75细胞核 EUC03513-RAEuTCP8AT3G02150Super-Scaffold_174 87629132 694.719.44细胞核 EUC15623-RAEuTCP9AT1G67260scaffold1436_obj 74724827 907.408.86细胞核 EUC12970-RAEuTCP10AT3G47620Super-Scaffold_113112537440 069.247.26细胞核 EUC06461-RAEuTCP11AT1G53230scaffold166_obj 87929232 398.815.65细胞核 EUC10433-RAEuTCP12AT5G23280Super-Scaffold_46 79826527 785.139.72细胞核 EUC15622-RAEuTCP13AT1G67260scaffold1436_obj 50716918 878.146.41细胞核 EUC07713-RAEuTCP14AT3G15030Super-Scaffold_600147949253 623.465.54细胞核
3.2 杜仲TCPs蛋白结构分析
二级结构分析显示,EuTCPs蛋白主要由α螺旋、无规则卷曲、延伸链和β转角组成,顺序均是由无规则卷曲>α螺旋>延伸链>β转角,具体数值见表2。α螺旋在DNA结合基序中发挥重要作用,无规则卷曲易受侧链相互作用,构成活性部位和功能部位。EuTCPs蛋白三级结构非常相似,结果如图1所示,无规则卷曲所占比例最大,蛋白空间结构不同,决定功能的差异。
表2 杜仲TCP基因家族二级结构分析
Table 2 Secondary structures analysis of E. ulmoides TCP gene family
基因名最低值位点最高值位点α螺旋(占比/%)延伸链(占比/%)β转角(占比/%)无规则卷曲(占比/%) EuTCP1−2.733 122.056150 37(17.79) 27(12.98)15(7.21)129(62.02) EuTCP2−2.889 361.278232 39(15.06) 43(16.60)13(5.02)164(63.32) EuTCP3−3.400 351.700 340 71(16.28) 49(11.24) 9(2.06)307(70.41) EuTCP4−3.300 132.022 135 79(25.57) 31(10.03)14(4.53)185(59.87) EuTCP5−3.1443330.811 4827(7.89)27(7.89) 0(0.00)278(81.29) EuTCP6−2.944 191.778 57 72(27.17) 42(15.85)13(4.91)138(52.08) EuTCP7−3.789 2351.722 314129(35.83)25(6.94) 9(2.50)197(54.72) EuTCP8−2.522 1271.41122928(9.62) 34(11.68) 7(2.41)222(76.29) EuTCP9−2.756 841.200 166 84(33.87) 30(12.10)10(4.03)124(50.00) EuTCP10−3.533 2251.444 54 53(14.17) 46(12.30)17(4.55)258(68.98) EuTCP11−2.978 311.167 108 61(20.89) 40(13.70)10(3.42)181(61.99) EuTCP12−2.944 191.778 57 72(27.17) 42(15.85)13(4.91)138(52.08) EuTCP13−3.056 871.022 30 55(32.54) 17(10.06) 2(1.18) 95(56.21) EuTCP14−3.800 2261.300 452 87(17.68) 59(11.99)20(4.07)326(66.26)
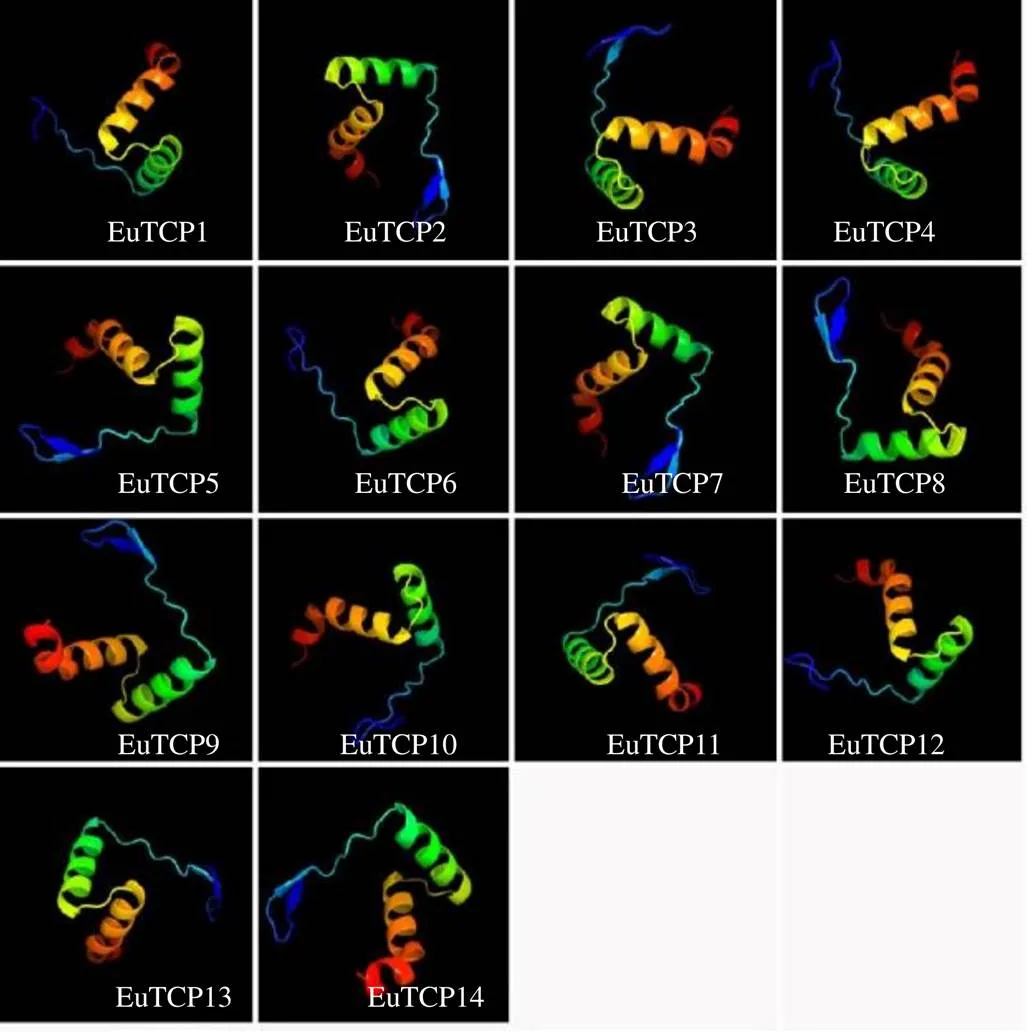
图1 杜仲TCPs三级结构预测
3.3 杜仲TCPs蛋白系统进化分析
利用MEGA 6.0分析软件,对14个杜仲EuTCPs,22个水稻OsTCPs[24],24个拟南芥AtTCPs[25]和19个毛竹PheTCPs[26]蛋白进行1000次重复搜索,构建系统进化树。结果如图2所示,79个TCPs蛋白分为Class I和Class II 2个大亚家族,其中Class I又称为PCF亚家族;Class II亚家族进一步划分为CYC/TB1和CIN亚家族。PCF亚家族所包含TCPs成员数量最多,含有40个TCPs蛋白,5个EuTCPs,13个AtTCPs,10个OsTCPs和12个PtTCPs,其次是CIN亚家族,由29个TCPs蛋白组成,CYC/TB1亚家族所含蛋白数量最少,仅包含10个TCPs蛋白,3个AtTCPs,3个EuTCPs,3个OsTCPs,1个PheTCPs。
3.4 杜仲TCPs结构域序列分析
为了分析杜仲TCP转录因子家族保守结构域序列特征,利用DNAMAN7.0工具对杜仲、拟南芥和水稻TCP家族成员氨基酸序列进行比对,结果如图3所示,杜仲TCP结构域氨基酸分布与拟南芥和水稻十分相似,14个杜仲TCP家族成员均含有完整的非典型性bHLH结构域,暗示杜仲TCP结构域高度保守性。BASIC区域由19个氨基酸组成,其中有9个氨基酸是高度保守的,HLH区域含有5个高度保守的氨基酸。杜仲TCP转录因子家族有5个蛋白(EuTCP1、EuTCP4、EuTCP6、EuTCP10和EuTCP12)BASIC区域缺少4个氨基酸,图2系统进化树结果显示,他们均属于Class I亚家族,其余蛋白均属于Class II亚家族,这与前人研究结果相一致[2],表明杜仲TCP转录因子家族进化的保守性。
3.5 杜仲TCP转录因子家族基序分析
为了解析基因结构多样性和进化关系,利用MEME在线软件对EuTCPs转录因子家族进行基序分析。结果如图4所示,EuTCPs转录因子分为3个亚家族(PCF、CYC/TB1和CIN),进化关系与图2结果相一致,20个保守基序,分别命名为Motif 1~Motif 20,14条EuTCPs蛋白均含有Motif 1基序,表明均属于TCP转录因子家族。同一亚家族EuTCPs蛋白基序具有高度相似性,CIN亚家族均含有Motif 1和Motif 2,PCF亚家族成员均含有Motif 1和Motif 19,表明同一亚家族EuTCPs可能具有相似的功能。不同亚家族基序存在显著差异,有些Motifs只在特定亚家族中存在,例如,Motif 3~Motif 6只存在于PCF亚家族,只有CYC/TB1亚家族含有Motif 7和Motif 9,暗示某些基序在亚家族功能特异性方面发挥重要作用。
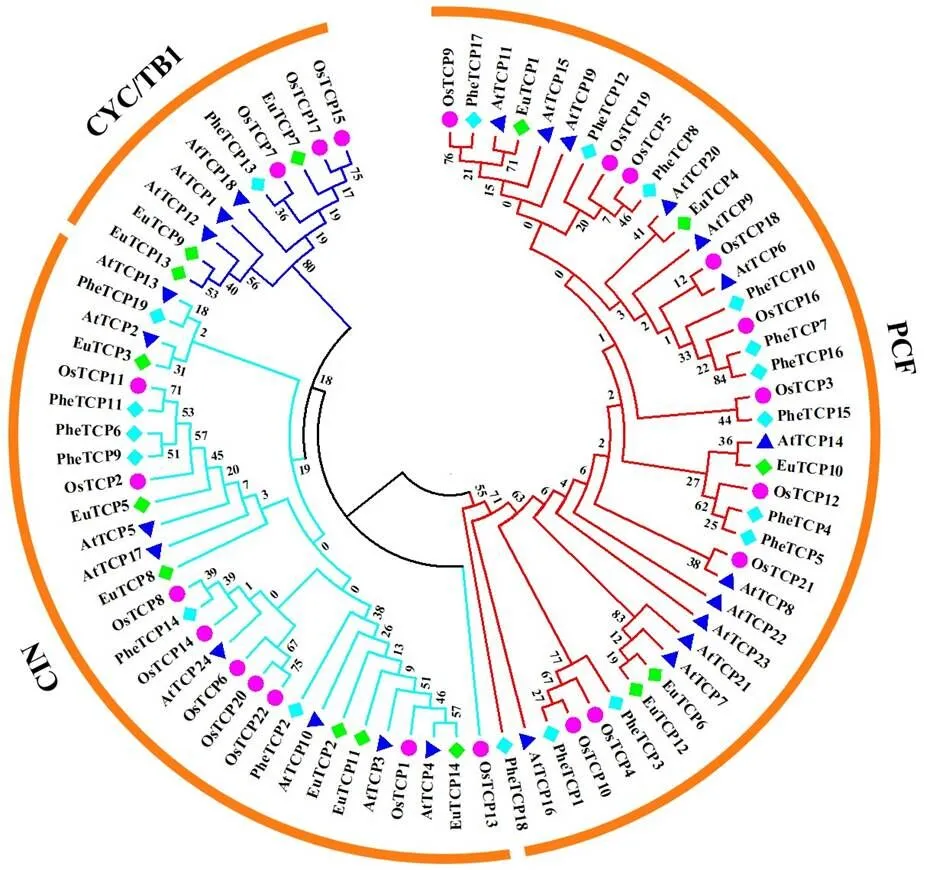
利用MEGA 6.0的邻接法构建系统进化树,不同物种TCPs蛋白用不同颜色标记,蓝色三角形代表拟南芥,红色圆形代表水稻,蓝绿色菱形代表毛竹,杜仲TCPs蛋白由绿色菱形标记

结构域名称在序列上方标记,黑色横线代表该区域的范围。黑色表示氨基酸是保守的,红色和蓝绿色表示氨基酸是相似的,无颜色表示氨基酸保守性较低。

A-利用MEGA 6.0的邻接法构建EuTCP基因家族系统进化树 B-EuTCPs基因结构分析 C-保守基序氨基酸分布
3.6 杜仲TCPs启动子顺式作用元件分析
为了探索基因功能和表达调控模式,利用Plant CARE在线分析软件对起始密码子(ATG)上游2000 bp序列进行顺式作用元件分析,结果如图5所示,启动子中不仅含有基本顺式作用元件,还存在4种类型元件:一、激素调控元件,如赤霉素响应元件:ABRE;生长素响应元件:AuxRR-core;水杨酸响应元件:CGTCA-motif;二、光响应元件,如Box 4、G-box、G-Box、GT1-motif、GATA-motif等,三、胁迫响应元件,如干旱胁迫响应元件:MBS,低温响应元件:LTR和厌氧胁迫相关元件:ARE等;四、生理响应元件,如O2-site、CAT-box等,推测可能在杜仲生长发育、胁迫响应、激素调节以及光周期调控中发挥重要作用。基因中光响应元件数量最多,共有135个,其中G-box元件有38个,含有28个Box 4元件,暗示s基因的转录可能受光周期调控。启动子区域含有44个ABRE元件和25个ARE元件(图5-B),表明可能参与ABA调节和厌氧调控。
3.7 杜仲TCP基因家族表达模式分析
为了解析基因在杜仲叶片不同发育时期及胶形成中的功能,根据转录组数据,检测基因的表达模式。结果如图6、7所示,不同基因在杜仲叶片不同发育时期及胶含量中表达丰度存在显著差异,大部分基因在杜仲叶片发育时期表达丰度较低,有2个基因(和)在杜仲叶片中未检测到,推测和在杜仲叶片中不发挥作用。基因在杜仲叶片不同发育时期表达趋势不尽相同,暗示在杜仲叶片发育中具有不同的功能,5个基因(、、、、)在杜仲叶芽中表达量最高,4个基因(、、、)在初生叶中表达丰度最高,3个基因(和)在老叶中转录水平最高。
为了预测在杜仲胶形成中的功能,利用不同胶含量的转录组数据,检测基因的转录水平。结果如图7所示,大部分在杜仲不同胶含量中表达水平较低,并且低胶含量叶片中的转录水平低于高胶含量叶片,例如,在高胶含量叶片中的FPKM平均值是17.25,在低胶含量叶片中为7.26,在高胶含量中的转录水平是24.26,在低胶中为5.05,推测在杜仲胶形成过程中发挥正调控作用。

A-每个EuTCP基因启动子区域顺式作用元件的数目 B-顺式作用元件数量和含有顺式作用元件EuTCPs基因数量
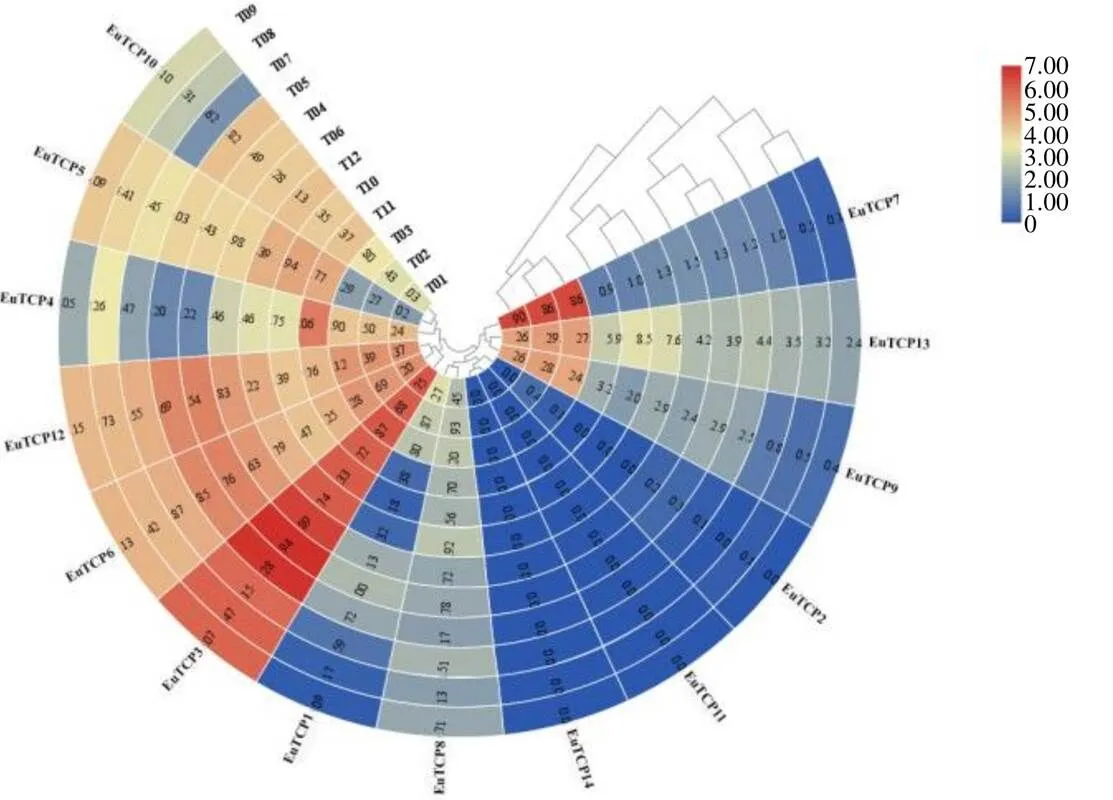
T01~T03-叶芽 T04~T06-初生叶 T3-1~T3-3-幼叶 T4-1~T4-3-老叶;热图右侧为色标,蓝色表示低表达丰度,红色表示高转录水平
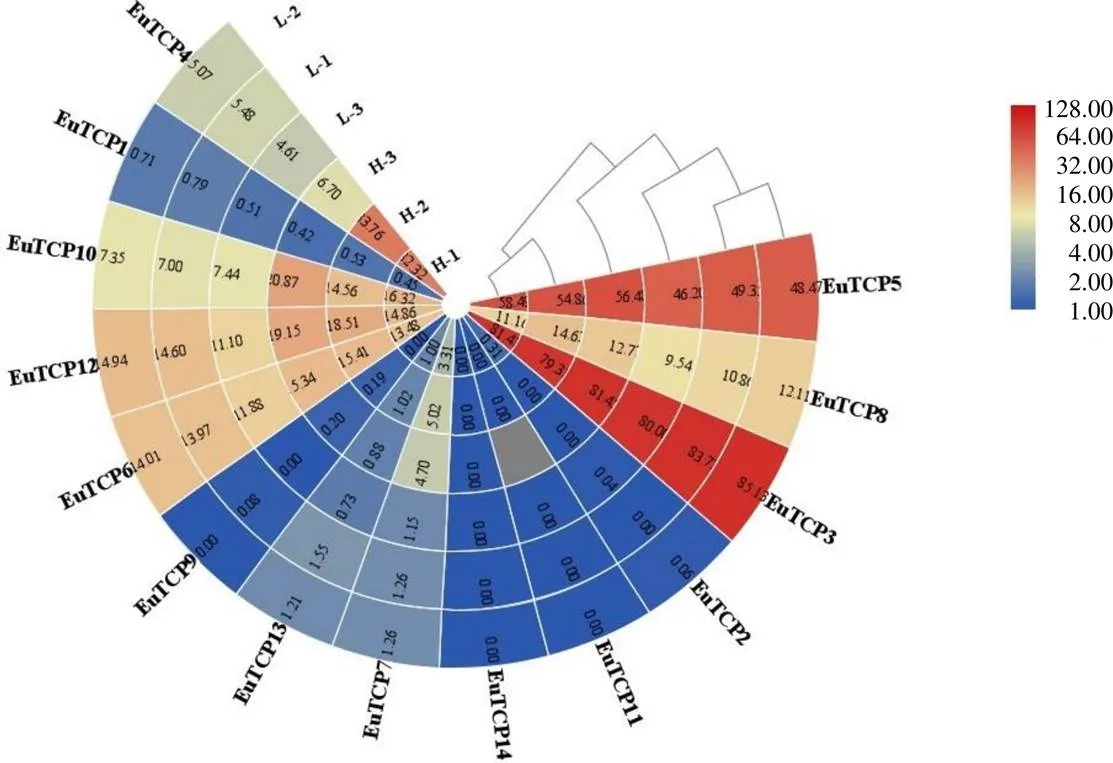
L-1~L-3-低胶含量叶片 H-1~H-3:高胶含量叶片。热图上方为色标,蓝色表示低转录水平,红色表示高转录水平
3.8 杜仲TCPs蛋白互作网络预测
表达模式结果显示,在杜仲叶片不同发育阶段和胶含量中表达量均较高,特别在初生叶中,FPKM值为111,暗示在杜仲叶片发育和胶形成过程中发挥重要作用。为了检测EuTCP3与其他蛋白的互作关系,利用STRING数据库,预测EuTCP3的互作蛋白。结果如图8所示,EuTCP3可以与10个蛋白发生相互作用,其中7个属于EuTCP基因家族,参与种子萌发、生物钟调节、细胞周期以及蛋白转录调控。MYB-like转录因子,在发育种子的原花青素积累中发挥关键的决定性作用,LOB转录因子AS2,是叶片正面细胞增殖负调控因子,调节叶片对称层的形成和脉络的建立,AN1-like(SAP11)锌指蛋白,参与植物非生物胁迫响应[27],推测EuTCP3可能与其他蛋白相互作用共同参与杜仲叶片生长发育以及胁迫响应。
利用Cytoscape 3.7.0软件,构建EuTCP转录因子家族共表达调控网络。结果如图9所示,调控网络中共有11个节点(代表EuTCPs蛋白)和35条边(代表蛋白质之间的相互作用),表明14个EuTCPs中有11个蛋白存在互作关系,其中EuTCP3、EuTCP5和EuTCP8蛋白互作数量最多,均与8个EuTCPs蛋白存在互作关系,暗示EuTCP3、EuTCP5、EuTCP8可能处于EuTCP基因家族核心位置,EuTCP1、EuTCP10、EuTCP11和EuTCP14与7个EuTCPs蛋白互作,EuTCP4和EuTCP6预测与5个EuTCPs蛋白互作,EuTCP7与4个EuTCPs蛋白互作,EuTCP9与2个EuTCPs蛋白发生互作关系。
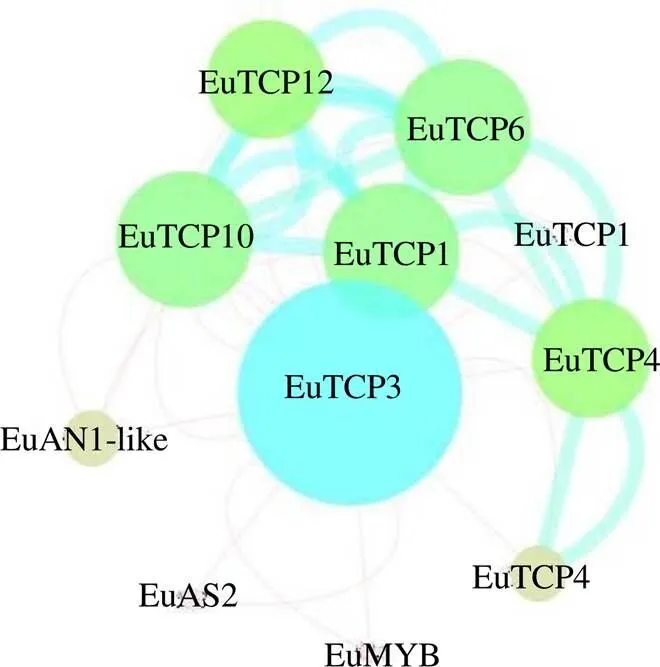
图8 EuTCP3蛋白互作网络
4 讨论
杜仲是绞木目(Garryales)杜仲科(Eucommiaceae)杜仲属L.中唯一现存的一个种,是我国主要天然橡胶树种,也是重要的木本油料和名贵中药材[28],主要分布于我国的陕西、湖南、贵州、云南、四川、江西等地区[29]。TCP蛋白是植物特有的参与生长发育、非生物胁迫、叶片衰老、侧枝形成、花粉发育等多种生物学过程的一类转录因子[13, 30]。目前为止,TCP基因家族已在多个物种中进行了报道,拟南芥中有24个AtTCPs[25],水稻含有22个OsTCPs[24],玉米中存在29个ZmTCPs成员[31],棉花中包含38个GrTCPs[32],番茄中有30个SlTCPs[33],黄瓜中存在22个CsTCPs[34],柑橘中含有20个CsTCPs[35],西瓜中有27个ClTCPs[36],葡萄中包含18个VvTCPs[37],苹果中52个MdTCPs[38],菠萝中有9个[39]、毛竹中存在19个PheTCPs[26]等。本研究以杜仲基因组数据为基础,利用生物信息学方法,从杜仲基因组中共鉴定到14个基因,分为3个亚家族,这与前人研究结果相一致。PCF亚家族包含5个EuTCPs蛋白,CYC/TB1亚家族含有3个,6个EuTCPs蛋白属于CIN亚家族。序列比对结果显示,14个EuTCPs与拟南芥、水稻TCP蛋白结构域具有高度的相似性,N末端均含有非典型的basic-helix-loop-helix结构域(图3),基序分析发现,Motif 1基序存在于所有EuTCPs转录因子(图4),表明他们均属于TCP转录因子家族,在基因进化过程中具有较保守性。根据保守结构域是否含有四种氨基酸残基,将TCP基因家族分为2个亚家族,Class I亚家族保守结构域含有16个氨基酸残基,Class II亚家族TCP结构域包含20个氨基酸,保守结构域外含有促进蛋白质相互作用的精氨酸富集R结构域[1],EuTCP3、EuTCP5、EuTCP7和EuTCP9均含有motif 8(R结构域),与AtTCPs、OsTCPs[24]、BpTCPs[40]、VvTCPs[37]、PmTCPs[41]和FvTCPs[42]等具有相似性,推测EuTCP3、EuTCP5、EuTCP7和EuTCP9可能具有相似的功能。
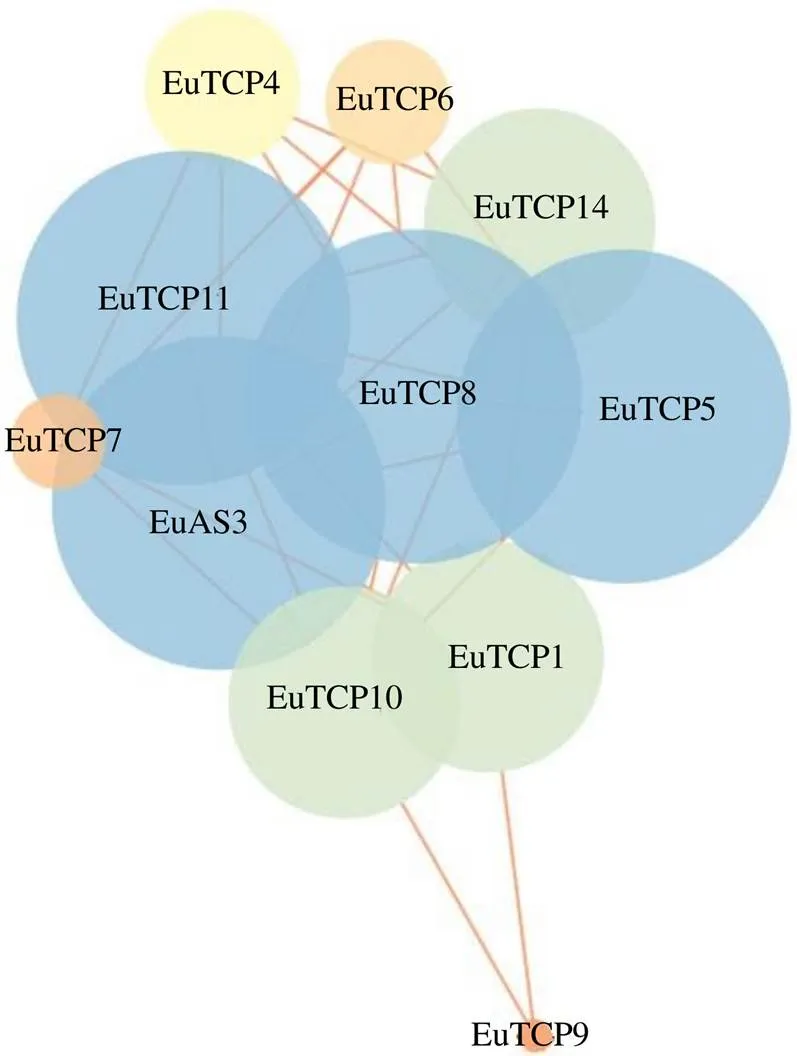
图9 EuTCP基因家族蛋白互作预测
聚类分析结果显示,与聚为同一分支,参与调节拟南芥细胞扩张、分裂和分化,在硝酸盐诱导的系统信号通路中发挥重要作用[43],菊花为的同源基因,超表达增加转基因菊花和拟南芥的侧根数量和长度[44]。是的同源基因,主要在胚胎维管组织中表达,促进幼嫩节间细胞增殖,抑制叶片细胞扩增,突变体提高种子萌发对外源脱落酸和多效唑的敏感性,参与激活拟南芥胚胎生长[45],异源转化拟南芥导致细胞周期相关基因下调表达,抑制器官大小,叶绿素和类胡萝卜素含量升高,延长花期,延缓叶片衰老[46]。和聚为同一分支的、、、和均在拟南芥幼嫩叶片中表达,与野生型相比,突变体增加叶片面积,四重突变体和五重突变体莲座叶数目减少,叶片增大[47]。为的同源基因,在幼苗子叶中表达,激活和的光响应表达,通过光敏色素相互作用因子(PIFs)调节与、和启动子的结合,黑暗条件下抑制与上述启动子的结合[48]。与(CO)启动子结合,黄昏前后诱导基因表达,与细胞核中另一个开花调节因子A()结合,诱导以依赖方式表达,调节发育和开花调控[49]。与互为同源基因,超表达导致转基因拟南芥在光依赖条件下下胚轴矮小,特别是蓝光条件下,与隐花色素蛋白(CRY1)互作,正调控和基因表达[50]。与亲缘关系最近,在阴暗条件下显著积累,及其同源基因和通过PIF依赖和独立途径诱导生长素生物合成,在介导阴暗诱导下胚轴伸长中发挥重要作用。白光下,三重突变体()表现出下胚轴缺陷;阴暗条件显著降低下胚轴伸长,与和的启动子结合,诱导生长素表达[51],推测EuTCPs转录因子在杜仲生长发育、激素调节和开花调控中发挥重要作用。
综上所述,本研究以杜仲全基因组数据为背景,对杜仲TCP转录因子家族进行了全面的生物信息学分析,共鉴定出14个,分为Class I与Class II 2大类,进一步划分为3个亚家族(PCF、CYC/TB1和CIN)。理化性质分析显示,EuTCPs编码139~492个氨基酸,PI分布于5.54~9.72,w区域为18 880~53 620,亚细胞定位于细胞核中,均为亲水性蛋白,二级结构以α-螺旋和无规则卷曲为主,均含非典型bHLH保守结构域。表达模式分析显示,EuTCPs基因家族在杜仲叶片发育中具有不同的表达模式,在高胶含量中的转录水平高于低胶含量,推测在杜仲胶形成中发挥正向调控作用,本研究为深入探索基因功能奠定理论基础。
利益冲突 所有作者均声明不存在利益冲突
[1] Cubas P, Lauter N, Doebley J,. The TCP domain: A motif found in proteins regulating plant growth and development [J]., 1999, 18(2): 215-222.
[2] Martín-Trillo M, Cubas P. TCP genes: A family snapshot ten years later [J]., 2010, 15(1): 31-39.
[3] Takeda T, Suwa Y, Suzuki M,. The OsTB1 gene negatively regulates lateral branching in rice [J]., 2003, 33(3): 513-520.
[4] Maurya J P, Singh R K, Miskolczi P C,. Branching regulator BRC1 mediates photoperiodic control of seasonal growth in hybrid aspen [J]., 2020, 30(1): 122-126.
[5] Wang M, le Moigne M A, Bertheloot J,. BRANCHED1: A key hub of shoot branching [J]., 2019, 10: 76.
[6] Yang X, Zhao X G, Li C Q,. Distinct regulatory changes underlying differential expression of teosinte branched1-cycloidea-proliferating cell factor genes associated with petal variations in zygomorphic flowers ofspp. of the family Gesneriaceae [J]., 2015, 169(3): 2138-2151.
[7] Guan P Z, Ripoll J J, Wang R H,. Interacting TCP and NLP transcription factors control plant responses to nitrate availability [J]., 2017, 114(9): 2419-2424.
[8] Finlayson S A.Teosinte Branched1-like 1 regulates axillary bud outgrowth and is homologous to monocot Teosinte Branched1 [J]., 2007, 48(5): 667-677.
[9] Danisman S, van der Wal F, Dhondt S,.class I and class II TCP transcription factors regulate jasmonic acid metabolism and leaf development antagonistically [J]., 2012, 159(4): 1511-1523.
[10] Rubio-Somoza I, Weigel D. Coordination of flower maturation by a regulatory circuit of three microRNAs [J]., 2013, 9(3): e1003374.
[11] Challa K R, Aggarwal P, Nath U. Activation of YUCCA5 by the transcription factor TCP4 integrates developmental and environmental signals to promote hypocotyl elongation in[J]., 2016, 28(9): 2117-2130.
[12] Rueda-Romero P, Barrero-Sicilia C, Gómez-Cadenas A,.DOF6negatively affects germination in non-after-ripened seeds and interacts with TCP14 [J]., 2012, 63(5): 1937-1949.
[13] Liu H L, Gao Y M, Wu M,. TCP10, a TCP transcription factor in moso bamboo (), confers drought tolerance to transgenic plants [J]., 2020, 172: 104002.
[14] Almeida D M, Gregorio G B, Oliveira M M,. Five novel transcription factors as potential regulators of OsNHX1 gene expression in a salt tolerant rice genotype [J]., 2017, 93(1/2): 61-77.
[15] Wang S T, Sun X L, Hoshino Y,. microRNA319 positively regulates cold tolerance by targeting OsPCF6and OsTCP21 in rice (L.) [J]., 2014, 9(3): e91357.
[16] Mukhopadhyay P, Tyagi A K. Erratum: OsTCP19 influences developmental and abiotic stress signaling by modulating ABI4-mediated pathways [J]., 2015, 5: 12381.
[17] 杜红岩. 中国杜仲图志 [M]. 北京: 中国林业出版社, 2014: 23.
[18] 刘建斌. 杜仲种子的化学成分研究 [D]. 西安: 西北大学, 2006.
[19] Wuyun T N, Wang L, Liu H M,. The hardy rubber tree genome provides insights into the evolution of polyisoprene biosynthesis [J]., 2018, 11(3): 429-442.
[20] Tamura K, Stecher G, Peterson D,. MEGA6: molecular evolutionary genetics analysis version 6.0 [J]., 2013, 30(12): 2725-2729.
[21] Li L, Liu M H, Shi K,. Dynamic changes in metabolite accumulation and the transcriptome during leaf growth and development in[J]., 2019, 20(16): 4030.
[22] Ye J, Han W J, Fan R S,. Integration of transcriptomes, small RNAs, and degradome sequencing to identify putative miRNAs and their targets related to Eu-rubber biosynthesis in[J]., 2019, 10(8): 623.
[23] Liu J, Cheng Z C, Li X Y,. Expression analysis and regulation network identification of the-like gene family in moso bamboo () under photoperiod treatments [J]., 2019, 38(7): 607-626.
[24] Yao X, Ma H, Wang J,. Genome-wide comparative analysis and expression pattern ofgene families inand[J]., 2007, 49(6): 885-897.
[25] Cubas P.[M] // CRC Press:: 2002: 247-266.
[26] 刘俊, 黄容, 程占超, 等. 毛竹TCP基因家族全基因组鉴定与分析 [J]. 基因组学与应用生物学, 2018, 37(12): 5388-5397.
[27] Giri J, Vij S, Dansana P K,. Rice A20/AN1zinc-finger containing stress-associated proteins (SAP1/11) and a receptor-like cytoplasmic kinase (OsRLCK253) interact via A20 zinc-finger and confer abiotic stress tolerance in transgenicplants [J]., 2011, 191(3): 721-732.
[28] Wang L, Jing T, Li T Z,. Identification and expression analysis of thefarnesyl diphosphate synthase gene family to reveal the key gene involved in rubber biosynthesis [J]., 2017, 40(1): 1-5.
[29] 张荣荣. 土壤干旱下杜仲截干苗生长和光合生理响应及引种栽培研究 [D]. 太原: 山西农业大学, 2016.
[30] Manassero N G, Viola I L, Welchen E,. TCP transcription factors: Architectures of plant form [J]., 2013, 4(2): 111-127.
[31] Chai W B, Jiang P F, Huang G Y,. Identification and expression profiling analysis of TCP family genes involved in growth and development in maize [J]., 2017, 23(4): 779-791.
[32] Ma J, Wang Q L, Sun R R,. Genome-wide identification and expression analysis of TCP transcription factors in[J]., 2014, 4: 6645.
[33] Parapunova V, Busscher M, Busscher-Lange J,. Identification, cloning and characterization of the tomato TCP transcription factor family [J]., 2014, 14: 157.
[34] 袁伟, 顾掌根, 褚伟雄, 等. 黄瓜转录因子TCP基因家族的鉴定及结构分析 [J]. 分子植物育种, 2014, 12(2): 287-295.
[35] 周延培, 张雅剑, 伊华林. 柑橘TCP家族生物信息学及表达谱分析 [J]. 果树学报, 2016, 33(5): 513-522.
[36] Shi P B, Guy K M, Wu W F,. Genome-wide identification and expression analysis of the ClTCP transcription factors in[J]., 2016, 16: 85.
[37] Leng X P, Wei H R, Xu X Z,. Genome-wide identification and transcript analysis of TCP transcription factors in grapevine [J]., 2019, 20(1): 786.
[38] 张士刚. 苹果TCP转录因子家族生物信息学分析 [J]. 山东农业科学, 2014, 46(5): 12-17.
[39] 阮城城, 胡福初, 罗志文, 等. 菠萝TCP基因家族的鉴定及成花诱导阶段的表达谱分析 [J]. 果树学报, 2020, 37(11): 1623-1635.
[40] Zhao M L, Peng X J, Chen N Z,. Genome-wide identification of thegene family inand functional analysis of BpTCP8, 14 and 19 in shoot branching [J].(), 2020, 9(10): 1301.
[41] Zhou Y Z, Xu Z D, Zhao K,. Genome-wide identification, characterization and expression analysis of thegene family in[J]., 2016, 7: 1301.
[42] Wei W, Hu Y, Cui M Y,. Identification and transcript analysis of the TCP transcription factors in the diploid woodland strawberry[J]., 2016, 7: 1937.
[43] Guan P Z, Wang R C, Nacry P,. Nitrate foraging byroots is mediated by the transcription factor TCP20 through the systemic signaling pathway [J]., 2014, 111(42): 15267-15272.
[44] Fan H M, Sun C H, Wen L Z,. CmTCP20 plays a key role in nitrate and auxin signaling-regulated lateral root development in[J]., 2019, 60(7): 1581-1594.
[45] Tatematsu K, Nakabayashi K, Kamiya Y,. Transcription factor AtTCP14 regulates embryonic growth potential during seed germination in[J]., 2008, 53(1): 42-52.
[46] Zhang T, Qu Y X, Wang H B,. The heterologous expression of aTCP-P transcription factor CmTCP14 suppresses organ size and delays senescence in[J]., 2017, 115: 239-248.
[47] Aguilar-Martínez J A, Sinha N. Analysis of the role ofclass I TCP genes AtTCP7, AtTCP8, AtTCP22, and AtTCP23 in leaf development [J]., 2013, 4: 406.
[48] Dong J, Sun N, Yang J,. The transcription factors TCP4 and PIF3 antagonistically regulate organ-specific light induction of SAUR genes to modulateopening during de-etiolation in[J]., 2019, 31(5): 1155-1170.
[49] Kubota A, Ito S, Shim J S,. TCP4-dependent induction of constans transcription requiresin photoperiodic flowering in[J]., 2017, 13(6): e1006856.
[50] He Z M, Zhao X Y, Kong F N,. TCP2 positively regulates HY5/HYH and photomorphogenesis in[J]., 2016, 67(3): 775-785.
[51] Zhou Y, Zhang D Z, An J X,. TCP transcription factors regulate shade avoidance via directly mediating the expression of boths and auxin biosynthetic denes [J]., 2018, 176(2): 1850-1861.
Identification and bioinformatics analysis of TCP family genes in
LIU Jun1, 3, 4, LI Long2,WU Yao-song1, LIU Yan1, REN Shan-shan1, CHEN Yu-long1
1. Henan key Laboratory of TCM Prescription and Syndrome Signaling, Academy of Chinese Medicine, Henan University of Chinese Medicine, Zhengzhou 450046, China 2. College of Forestry, Northwest Agriculture and Forestry University, Yangling 712100, China 3. International Center for Bamboo and Rattan, Key Open Laboratory of Bamboo and Rattan Science and Technology, China National Forestry and Grassland Administration, Beijing 100102, China 4. Anhui Taiping Experimental Station of International Center for Bamboo and Rattan, Huangshan 245716, China
To screen and analyze TCP (Teosinte branched 1/Cycloidea/Proliferating) gene family of, so as to lay a foundation for further study ofgene function research.Based ongenome database, the physicochemical properties, phylogeny, gene structure, promoter-acting elements and their expression levels in the leaf development and eu-rubber formation of the TCP gene family were analyzed by bioinformatics system.A total of 14were identified fromgenome, EuTCPs encoded 139—492 amino acids, and theoretical isoelectric points distribution were 5.54—9.72, molecular weight was between 18 880 to 53 620 by physicochemical analysis, all of EuTCPsproteins were hydrophilic proteins and expressed in nucleus. EuTCP gene family was divided into three subfamilies: PCF, CIN and CYC/TB1, which contained five, six and three EuTCPs proteins, respectively. Expression pattern analysis showed thatwere significantly different at different developmental stages ofleaves, and the expression levels ofwere different at each developmental stage.positively regulated the formation of eu-rubber.There are 14 members of TCP family identified in, and their molecular characteristics and tissue expression specificity are different, which may be involved in the development of leaves and eu-rubber formation.
Oliver; TCP gene family; subcellular location; phylogeny evolution; expression analysis
R286.12
A
0253 - 2670(2022)18 - 5813 - 12
10.7501/j.issn.0253-2670.2022.18.024
2022-01-09
河南省高等学校重点科研项目(22A360005);国家林业和草原局/北京市共建竹藤科学与技术重点实验室开放基金(ICBR-2020-05);国际竹藤中心安徽太平试验中心开放课题基金(1632021006-4);河南中医药大学博士科研基金资助项目(RSBSJJ2019-04)
刘 俊(1990—),女,助理研究员,博士,主要从事药用植物分子生物学研究。E-mail: liujun_0325@163.com
陈玉龙,男,教授,博士,主要从事中医药防治炎症与免疫。E-mail: cyl72621@163.com
[责任编辑 时圣明]
