基于生物信息学探讨骨关节炎miRNA-mRNA调控网络及其作用机制
2022-08-25陈锋陈涛章晓云陈跃平柴源
陈锋,陈涛,章晓云,陈跃平,柴源
(广西中医药大学附属瑞康医院骨科,广西 南宁 530011)
骨关节炎(osteoarthritis, OA)是一种慢性关节疾病,以软骨退化、骨赘形成及滑膜炎症为主要特征[1]。流行病学调查显示,全球OA总患病率高达16%,且随着人口老龄化的增加,OA的发病率也逐年上升[2]。OA引发的慢性疼痛和残疾严重影响着人们的生活质量,并造成巨大的经济负担,但其发病机制仍未明确。因此,寻找OA发病机制中的关键靶点及相关信号通路,并采用相应的措施进行干预,对于延缓甚至逆转OA进展具有重要意义。微小RNA(microRNA, miRNA)是一种内源性非编码RNA,可通过抑制mRNA的翻译或加速mRNA的降解来调控靶基因的表达[3]。近年研究证实,几乎所有细胞的生命过程都依靠miRNA的调控作用进行修饰,并对基因的表达覆盖了转录水平、转录后水平及表观遗传学水平的调控[4],其在OA的发生、预防和治疗中发挥着重要作用[5]。随着高通量技术不断应用于生物医学研究中,通过适当的方法和工具可有效分析OA的基因组学和蛋白质组学。因此本研究从GEO数据库检索分析OA的miRNA和mRNA基因表达谱数据,探讨OA中差异表达miRNAs的功能及调控的靶点,为靶向诊断、治疗OA提供一定的科学依据。
1 材料和方法
1.1 数据收集
以“Osteoarthritis”、“mRNA”、“miRNA”、“Human”为关键词,在GEO公共数据库中检索OA的相关芯片,获得OA患者和健康对照的miRNA和mRNA表达谱数据集。miRNA表达谱数据集芯片GSE105027所处平台为GPL21575,包含12例OA患者和12例健康对照的血清;mRNA表达谱数据集芯片GSE55235所处平台为GPL96,包含12例OA患者和12例健康对照的关节滑膜组织。
1.2 miRNAs和mRNAs数据的差异分析
利用Perl语言对数据进行基因重注释,获得OA患者和健康对照的基因表达谱数据集,并利用R语言limma包对基因进行校正,再进行差异分析。以P<0.05和|log2(fold change,FC)|>1作为过滤条件,筛选差异表达的miRNAs和mRNAs。
1.3 差异表达miRNAs转录因子预测及功能富集分析
FunRich是一个用于蛋白基因功能富集和相互作用网络分析的软件。为了解差异表达miRNAs的作用机制,利用FunRich软件对差异表达 miRNAs进行转录因子预测及功能富集分析。
1.4 miRNA-mRNA调控网络的构建
利用FunRich软件预测差异表达miRNAs的靶基因,将预测结果与步骤“1.2”所得的差异表达mRNAs取交集,整合得到差异表达miRNAs与差异表达mRNAs的调控网络,并根据两者的相关性筛选出mRNA交叉表达负调控的miRNA,最后将数据导入Cytoscape软件对miRNA-mRNA调控网络进行可视化处理。
1.5 蛋白互作网络构建
STRING是一款用来分析蛋白与基因间相互作用的数据库。为进一步探究差异表达mRNAs的作用机制,将基因导入STRING数据库,限定研究物种为“Homo Sapiens”,并设置连接评分>0.98,获得蛋白互作(protein-protein interaction, PPI)关系。将所得结果导入Cytoscape软件中,利用“NetworkAnalyzer”工具进行可视化处理,构建PPI网络,并利用“CytoHubba”工具根据度值筛选出Hub基因。
1.6 基因富集分析
利用R语言的clusterProfiler包对Hub基因进行GO功能富集分析,研究其主要生物功能;对Hub基因进行KEGG信号通路富集分析,研究其主要信号通路,P<0.05代表富集结果显著。最后利用R语言的ggplot2包和GOplot包进行可视化处理。
2 结果
2.1 差异表达miRNAs和mRNAs的鉴定
利用Perl和R语言对芯片进行重注释和数据校正后,再对其进行差异分析。结果显示,与健康对照相比,OA患者共存在265个明显改变的miRNAs,其中上调124个,下调141个;存在838个明显改变的mRNAs,其中上调449个,下调389个。将所得结果分别绘制成火山图,见图1,图中黑点代表无差异的基因,绿点代表下调的基因,红点代表上调的基因。
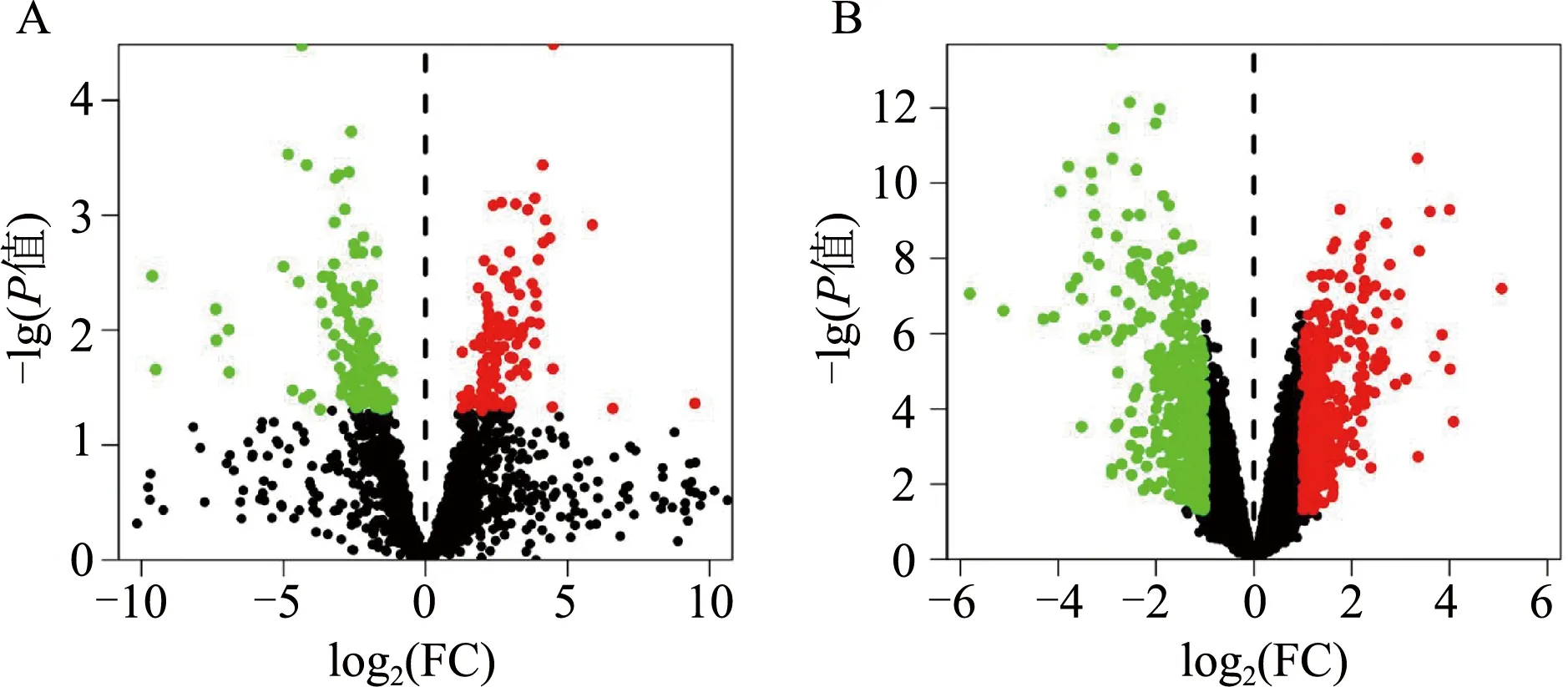
图1 差异表达miRNAs(A)和mRNAs(B)的火山图
2.2 转录因子-miRNA调控网络及富集分析结果
通过FunRich软件对265个差异表达的miRNAs进行转录因子预测,共得到203个转录因子,根据其所调控的基因数与P值,选取富集程度最显著的10个转录因子进行展示,见图2。差异最显著的10个转录因子分别为EGR1、SP1、POU2F1、SP4、MEF2A、NFIC、ZFP161、NKX6-1、FOXA1、RREB1,且其P值均<0.001,基本信息见表1;对265个差异表达的miRNAs进行功能富集分析,其生物过程主要涉及核碱基、核苷、核苷酸和核酸代谢的调控,调节细胞生长,转录等;细胞成分主要涉及细胞核、细胞质、内涵体等;分子功能主要涉及转录因子活性、转录调节活性、泛素特异性蛋白酶活性等,见图3。

图2 差异表达miRNAs的转录因子预测


图3 差异表达miRNAs的GO功能富集分析
2.3 miRNA-mRNA网络
利用FunRich软件对265个差异表达miRNAs的下游靶标进行预测,共得到3 572个靶基因,与838个差异表达mRNAs取交集获得194个候选靶标。然后,再根据差异表达miRNAs和差异表达mRNAs间的log2(FC)关系,筛选出交叉表达负调控的mRNAs 96个、miRNAs 25个。最后将数据导入Cytoscape软件对miRNA-mRNA调控网络进行可视化处理,见图4。图中红色节点表示基因上调,绿色节点表示基因下调,节点间的连线表示两者呈负调控关系。
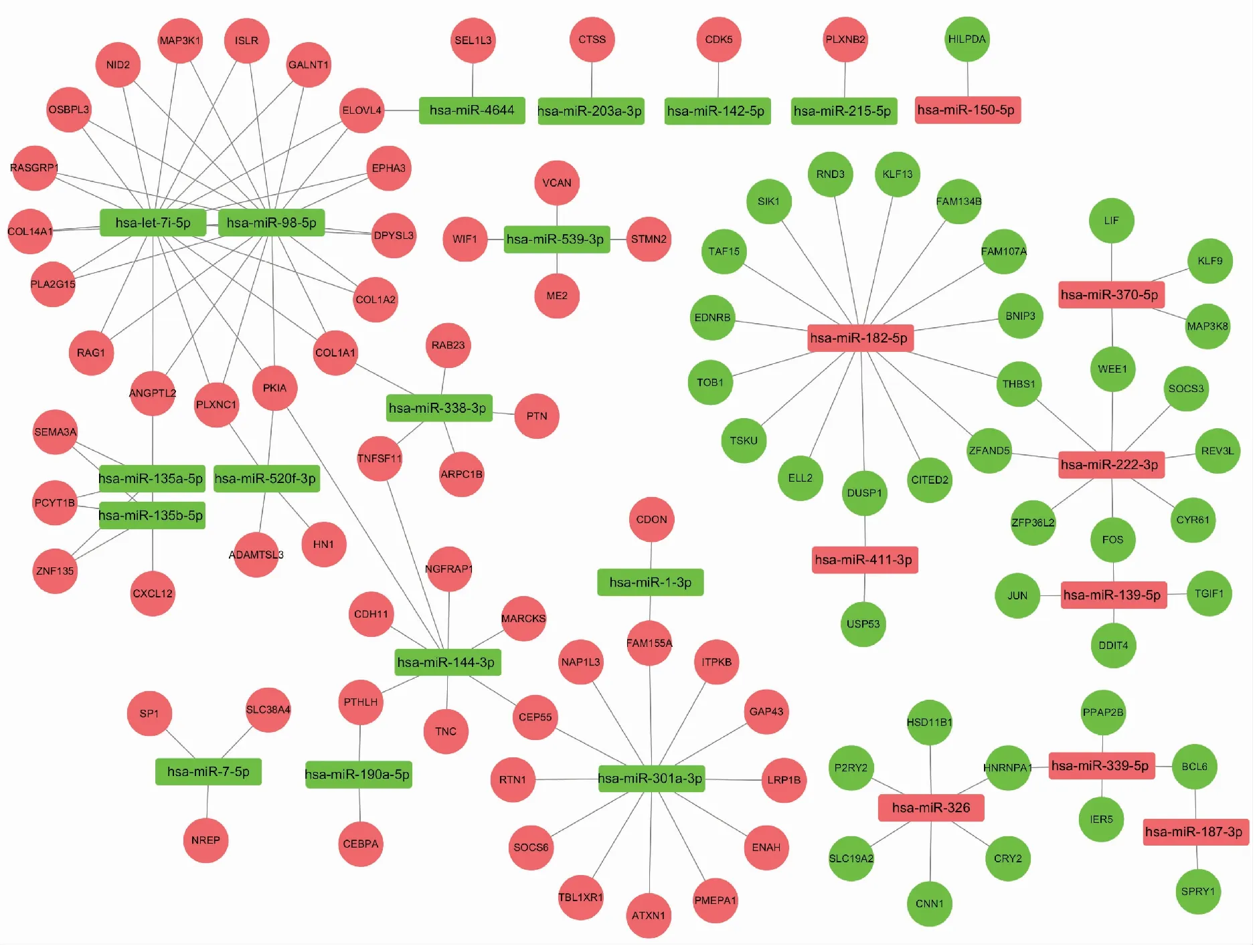
图4 miRNA-mRNA调控网络
2.4 PPI网络
借助STRING数据库和Cytoscape软件构建PPI网络,见图5。图中共涉及46个节点、62条边,节点代表mRNA,两节点的连线则说明两者间存在相互作用关系;节点越大、颜色越深则度值越大,边的粗细反映连接评分,边越粗,mRNA间的互作关系越紧密。根据度值筛选出排名前6的mRNAs为JUN、FOS、IL-6、TNF、IL-1β、CXCL8。这些度值较大的mRNAs在整个网络中起着关键作用,可能是OA发生发展的Hub基因,基本信息见表2。
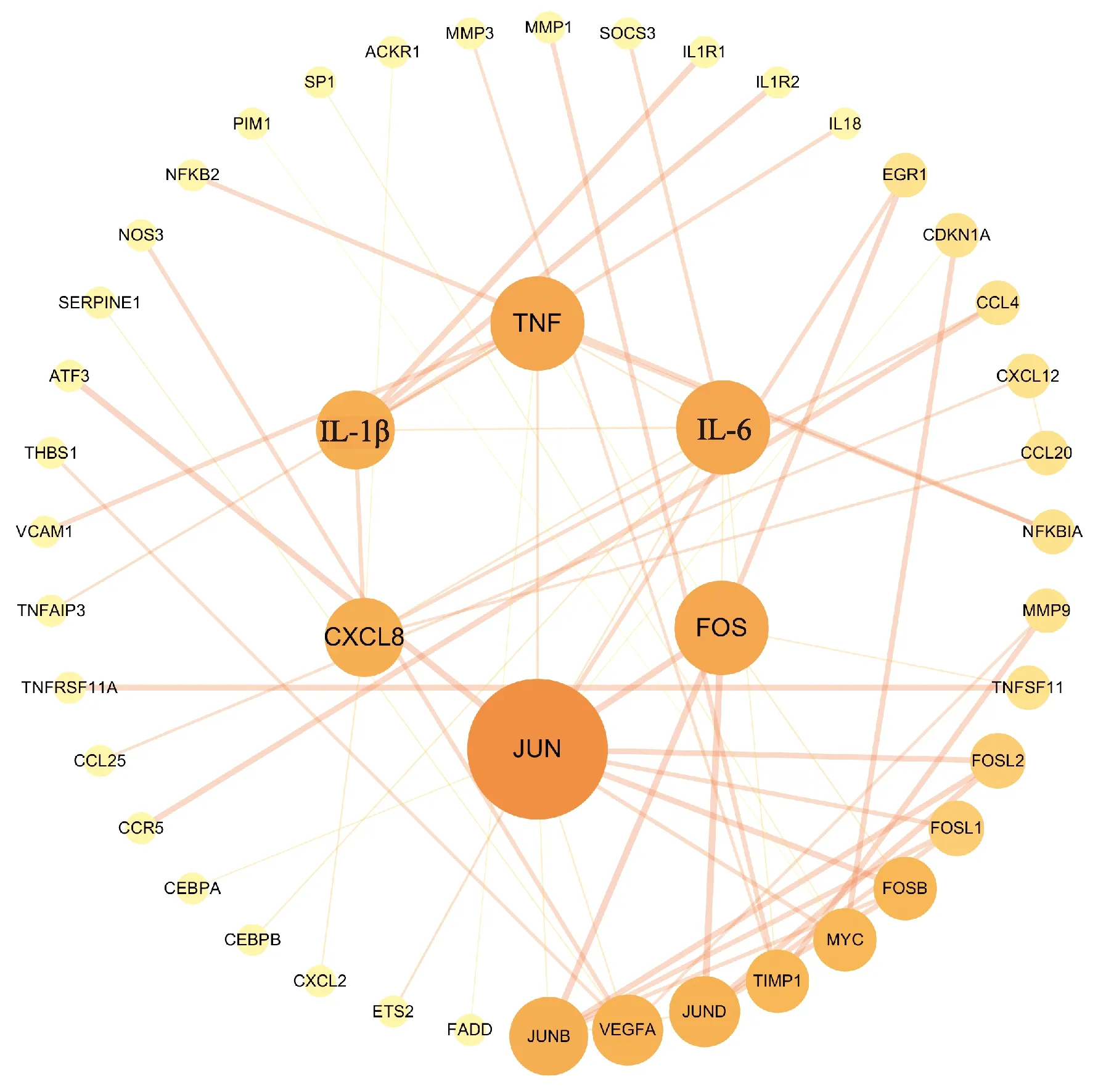
图5 mRNA的蛋白互作网络

表2 关键mRNA的基本信息
2.5 GO与KEGG富集分析
GO富集分析Hub基因的功能过程中,共确定了934个条目,其中包括908个生物过程、5个细胞成分和21个分子功能。根据P值,前10个富集的生物过程、细胞成分和分子功能如图6所示。生物过程主要涉及对脂多糖的反应、对细菌来源分子的反应、DNA结合转录因子活性的调节等方面;细胞成分主要涉及RNA聚合酶Ⅱ转录调节复合物、转录调节复合物、吞噬杯等;分子功能主要涉及细胞因子活性、细胞因子受体结合、受体配体活性等。KEGG富集分析Hub基因共确定了70个条目,主要涉及IL-17信号通路、Toll样受体信号通路、AGE-RAGE信号通路、TNF信号通路、NOD样受体信号通路,如图7所示。
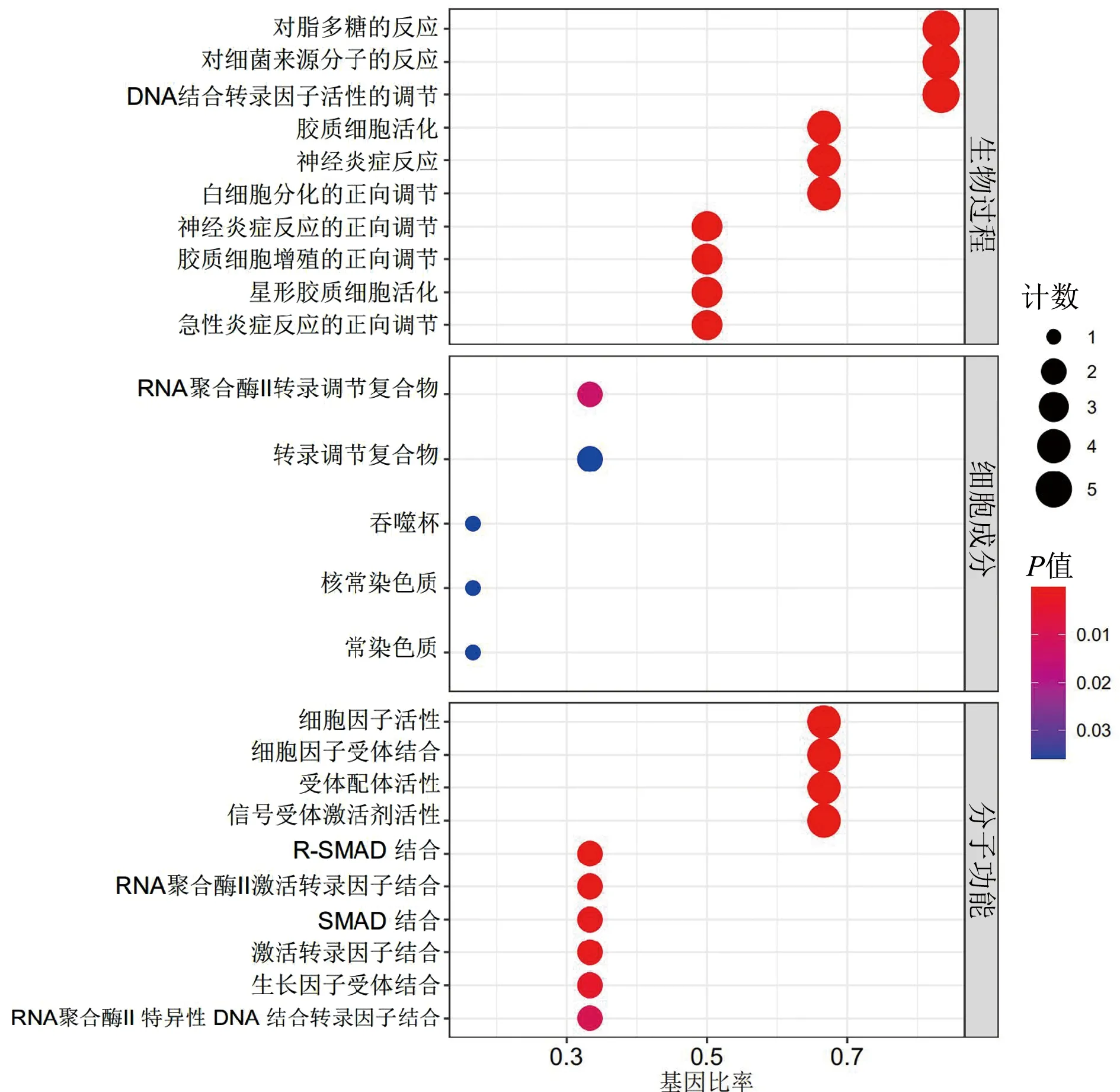
图6 Hub基因的GO富集分析气泡图

图7 Hub基因的KEGG富集分析气泡图
3 讨论
在OA的研究过程中,针对骨代谢、基质降解、氧化应激和免疫炎症反应等方面的治疗均已应用于临床,但这些方法的疗效并不理想[6]。越来越多的研究表明,OA的发生发展与miRNA密切相关,在最近的一项研究中发现[7],miR-206可调控补体因子H(complement factor H,CFH),干扰素诱导的含有解旋酶C域的蛋白1(interferon induced with helicase C domain 1,IFIH1),神经损伤诱导蛋白1(nerve injury-induced protein 1,NINJ1)等基因的转录水平,并显著抑制软骨细胞的增殖,同时促进分解代谢酶和凋亡诱导因子的表达。这表明通过敲低miR-206表达可有效抑制OA的软骨降解,并在介导OA发病机制中起着重要调控作用。通过基因测序技术更是发现在关节软骨损伤过程中表达量发生明显改变的miRNAs高达数百个。然而,针对OA尚缺乏完整的miRNA-mRNA调控网络模式。因此,深入研究miRNA在OA发生发展中的作用及其调控机制有重要意义。
之前的研究已经证实miRNA的表达受转录因子的调控,且这些转录因子在OA发生发展过程中发挥重要的调控作用,主要涉及滑膜炎症反应,软骨与骨组织的破坏[8]。本研究预测了调控OA的差异表达miRNAs的上游转录因子共203个,根据P值选出排名前10的转录因子分别为EGR1、SP1、POU2F1、SP4、MEF2A、NFIC、ZFP161、NKX6-1、FOXA1、RREB1,其中EGR1差异最显著。EGR1是一种核蛋白,具有转录调节功能,可潜在地调节miRNA的表达。研究表明,EGR1在miR-195上游启动子区有结合位点,可抑制miR-195的表达。而EGR1又可通过与Krüppel样因子5(Krüppel-like factor 5, KLF5)结合,减少其泛素化降解,从而上调基质金属蛋白酶的转录水平,最终导致软骨退变并诱发OA[9-10]。因此研究转录因子与miRNA组成的调控网络可对疾病的发病机制在系统层次上提供重要的线索。但这些转录因子在OA中的具体机制仍有待进一步验证。
对自身靶基因的调控是miRNAs在疾病发病过程中发挥作用的研究重点,本研究结果显示265个差异表达miRNAs共调控了194个差异表达mRNAs。对265个差异表达miRNAs进行功能富集分析显示其主要涉及核碱基、核苷、核苷酸和核酸代谢的调控,调节细胞生长,转录等过程,与既往研究完全符合[11]。而在miRNA-mRNA调控网络中,同属于原癌基因家族的JUN和FOS蛋白可在胞核中形成复合物,并与DNA内的转录因子激活蛋白1(activator protein 1, AP-1)调节位点结合,形成AP-1复合物来调节细胞生物过程。研究表明,TNF-α、IL-1、干扰素等炎症因子通过MAPK信号介导AP-1表达,诱导基质金属蛋白酶和胶原酶活化进而破坏软骨基质[12-13]。Luo等[14]研究表明,miR-139-5p过表达会抑制c-Jun的水平进而减少内皮因子表达,而内皮因子过表达诱导的血管异常增生和炎性反应是介导OA软骨退变、滑膜增厚及滑膜炎症的关键途径;miR-139-5p过表达可通过抑制c-Fos减少TNF-α、NF-κB的表达,而TNF-α在OA中是介导炎症反应和软骨降解的关键靶基因[15-16];miR-222-3p可通过抑制c-Jun表达减少心肌纤维化,而在OA中纤维化是软骨退变的病理变化之一[17-18]。因此,miRNA在OA的发病过程中与mRNA的互作对疾病进展影响重大,可为阐明OA的发病机制及后续研究提供依据。
为进一步探究差异表达mRNAs在OA中的作用机制,通过PPI网络还发现IL-6、TNF、IL-1β、CXCL8等Hub基因。这些基因的生物过程主要涉及对脂多糖的反应、对细菌来源分子的反应、DNA结合转录因子活性的调节等方面,与OA病情的发生发展密切相关[19]。研究表明,IL-6、TNF-α、IL-1β等炎症因子在OA中的表达显著上调,且三者间的协同作用共同导致了巨噬细胞去极化,滑膜炎症,软骨细胞凋亡及基质降解等病理改变[20-21]。而巨噬细胞受上述炎症因子刺激会产生趋化因子CXCL8,进而募集和激活相关炎症因子,加剧OA的病变程度[22]。对Hub基因进行KEGG富集分析,发现其主要富集在IL-17、Toll样受体、AGE-RAGE、TNF和NOD样受体信号通路上。研究发现,OA患者外周血中IL-17水平明显高于健康者,且与病情的严重程度呈正相关,通过降低血液中IL-17的水平能有效地控制OA患者的病情[23];Toll样受体和NOD样受体在体内水平与IL-1β、IL-6、TNF-α等炎症因子呈正相关,并会诱导破骨细胞分化并造成骨破坏,两者参与的信号通路均与OA有着紧密的联系[24-25];AGE-RAGE信号通路活化后会导致炎症因子的表达增多,进而引起细胞凋亡和组织损伤。通过抑制该通路可降低氧化应激与炎症反应,有效防止细胞损伤[26];TNF信号通路在炎症反应早期会释放大量TNF-α,进而诱导IL-1、IL-6等炎症因子的产生,与滑膜炎症及软骨细胞凋亡等病理改变过程密切相关[27]。因此,我们认为通过作用上述靶点与通路可有效调控血液中的炎症因子抑制机体炎症反应,同时也可调控软骨细胞和破骨细胞的增殖、分化与凋亡,从而缓解关节骨与软骨破坏情况,达到治疗OA的目的。
综上所述,本研究借助生物信息学成功筛选并构建了OA的miRNA-mRNA调控网络,探讨了可能与OA发生发展相关的致病机制。虽然该调控网络某些靶点的相关机制已阐明,但大多数miRNAs和mRNAs的致病机制仍不清楚,这对于挖掘新的机制和生物标志物很有意义,并可为今后阐明基因间的调控机制、协助预防和治疗OA提供关键的数据支撑和参考依据。此外本研究存在一些缺点,首先,由于筛选条件的限制,只能对关键基因、关键信号通路进行分析,在一定程度上使得研究结果具有局限性;其次,缺乏进一步的实验来验证miRNA-mRNA调控网络中基因间的互作关系。在后续的研究中,应通过相关实验进一步讨论和验证本研究的预测。
