梅花草属叶绿体基因组进化分析
2022-08-04夏铭泽张发起迟晓峰陈世龙
夏铭泽 张发起 迟晓峰 韩 霜 陈世龙*
(1. 中国科学院西北高原生物研究所高原生物适应与进化重点实验室,西宁 810001;2. 中国科学院大学,北京 100049)
梅花草属()物种为多年生小草本植物,全球60 余种,多分布于北温带高山地区,其中喜马拉雅及其毗邻地区是该属植物的分布和分化中心。梅花草属系统位置长期存在争议,多数学者认为梅花草属与虎耳草科(Saxifragaceae)亲缘关系最近。然而,也有学者认为梅花草属与其他科的亲缘关系较近,另有部分学者则支持梅花草属应处理为单独的科或目。近年来,梅花草属分子系统学研究结果认为该属和卫矛科(Celas‑traceae)亲缘关系最近,APG(Angiosperm Phyloge‑ny Group)Ⅳ综合分子系统学研究结果后,将梅花草属置于卫矛科之下。然而,部分研究结果将梅花草属作为卫矛科的姊妹类群;相反,也有一些研究认为梅花草属是卫矛科早期衍生的谱系而非姊妹类群。此外,梅花草属、核子木属()、假卫矛属()、砂纸木属()和卫矛科的关系仍存在争议。这些研究多基于序列数据(如-、-和ITS 序列)重建梅花草属与近缘类群间的系统发育关系,而近年来一些质体基因组结构的研究为物种进化适应提供了新的见解,这为解决梅花草属的系统发育关系问题提供了新的研究思路。
自1986年,烟草()完整叶绿体基因组被测序并报道后,越来越多的植物叶绿体基因组测序工作已经完成。随着二代测序技术的不断发展及测序成本的不断降低,叶绿体基因组序列变得更易获得。目前,已有超过2 000 个叶绿体基因组发布在National Center for Biotech‑nology Information(NCBI)上。先前研究表明,叶绿体基因组结构较保守,一般由大单拷贝区(large single copy,LSC)、小单拷贝区(small single copy,SSC)和2 个反向重复区(inverted repeat sequence,IR)4 个区域组成,但仅有少数植物的叶绿体基因组结构较特殊,如鹰嘴豆()、牻牛儿苗属()和豌豆()丢失1 个反向重复区;篦子三尖杉()丢失2 个反向重复区;黑松()仅有495 bp 的反向重复区,叶绿体基因组仅为119 kb。叶绿体基因组结构和序列变异研究,如反向重复区收缩和扩张、基因丢失和假基因化、密码子偏好性和核苷酸替换率等,有助于了解相关物种的进化过程,同时为近缘类群系统学研究提供了新的研究思路。
本研究选取梅花草()、鸡肫梅花草()、白耳菜()、甘肃梅花草()、三脉梅花草()、德格梅花草()和青铜钱()共7个梅花草属物种作为研究对象。同时,选取卫矛科双花假卫矛()、圆叶南蛇藤()、白杜()和永瓣藤()4 个物种作为外类群,通过统计比对叶绿体基因组的结构特征和基因情况,分析密码子使用模式,探讨梅花草属叶绿体基因组的进化趋势。
1 材料与方法
1.1材料
三脉梅花草(Chen2013132)采自四川炉霍(31°10′N,100°53′E),青铜钱(Chen2013134)采自四 川色 达(31°44′N,100°45′E),双 花 假 卫 矛(Zhang2019720)采自 广 西 恭 城(24°53′N,111°0′E),圆叶南蛇藤(Zhang2019713)采自广西金秀(24°10′N,110°22′E),白杜(Zhang2019763)采自青海西宁(36°37′N,101°45′E)。野外采集的新鲜幼嫩叶片,硅胶干燥后带回中国科学院高原生物适应与进化重点实验室,-20 ℃保存。凭证标本存放于中国科学院西北高原生物研究所青藏高原生物标本馆(HNWP)内。梅花草、鸡肫梅花草、白耳菜、甘肃梅花草、德格梅花草和永瓣藤的叶绿体基因组数据下载于NCBI数据库(见表1)。
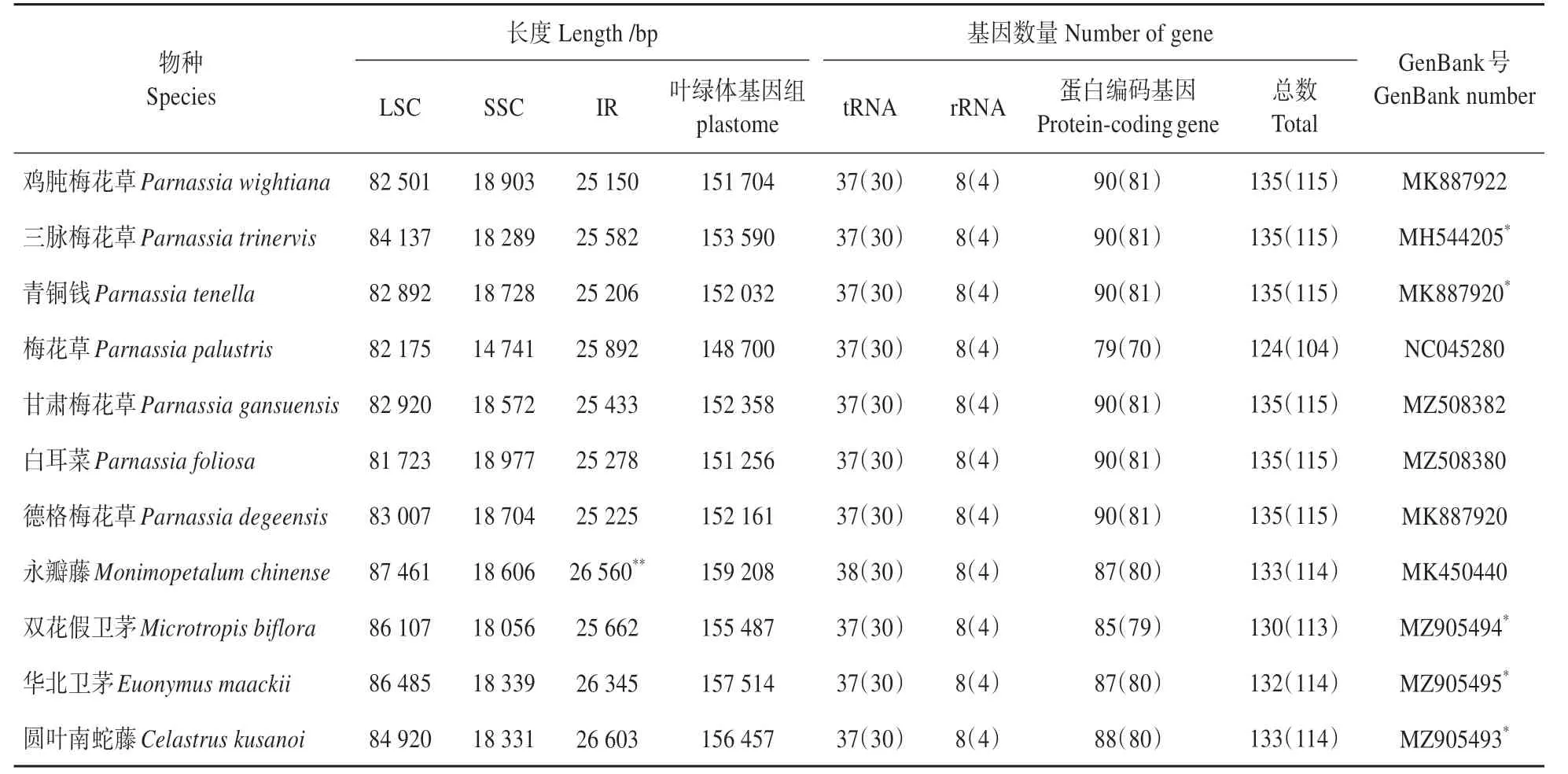
表1 叶绿体基因组结构信息和GenBank登录号Table 1 Structure information and GenBank number of chloroplast genomes
1.2 DNA 提取和测序
采用改良的CTAB 法从约10 mg 干燥叶片中提取总DNA,1%琼脂糖凝胶电泳检测后送至北京诺禾致源科技股份有限公司进行全基因组小片段文库构建;利用Illumina HiSeq 2500(Illumina Inc.,San Diego,California,USA)测序平台进行长度为150 bp的双端测序,获得下机数据(Raw reads);使 用Trimmomatic v. 0.33(参 数:PE-phred33-trimlog seq.log-threads 4 SLIDINGWINDOW:5:20 LEADING:5 TRAILING:5 MINLEN:50)和FastQC v.0.11.8对下机数据进行质量控制和筛选,获得高质量的序列数据(Clean reads)供后续分析。
1.3叶绿体基因组组装及注释
使用GetOrganelle v. 1.7.5 组装叶绿体基因组;使 用 在 线 工 具GeSeq(http://chlorobox.mpimp-golm.mpg.de/geseq.html)进行叶绿体基因组注释;BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)后,应用软件Sequin v. 15.50 手动增删和调整基因起始/终止密码子及内含子/外显子位置。
1.4叶绿体基因组特征统计
注释后的叶绿体基因组结构特征进行对比,统计各个物种基因和内含子。以三脉梅花草为参考物种,利用mVISTA(https://genome.lbl.gov/vista/index.shtml)(参数:LAGAN)软件对梅花草属及近缘属11个种的叶绿体基因组相似度进行可视化比对,结合DnaSP v.6.10.01 软件对核苷酸多样性进行分析绘图(参数:400 bp window length and 200 bp step size);利用Scalable Vector Graphics v.2.8.6在Perl v.5.16.3(https://dev.perl.org/)中绘制各物种IR/SC区域连接处基因分布对比图。
1.5同源基因信息比对分析
筛选梅花草属及近缘属11个物种叶绿体基因组的共有基因,将其分为14 个数据集:(fatty acid synthesis)、(ATP synthase)、(carbon metabolism)、(proteolysis)、(translational initiation factor)、(RNA processing)、(NADPH dehydrogenase)、(cytochrome b/f com‑plex)、(photosystem Ⅰ)、(photosystem Ⅱ)、(rubisco)、(large subunit of ribosome)、(DNA dependent RNA polymera)、(small subunit of ribosome)。采用密码子比对策略,对所有数据集使 用MAFFT v.7.409(参 数:--thread 3--auto--inputorder)进行比对,比对后使用PAML v.4.9j软件中的CodeML 选项(数据类型设为密码子、平衡密码子频率设为F3×4、其他参数为默认)计算各数据集的同义替代率(synonymous,dS)和非同义替代率(nonsynonymous,dN);使用CodonW v.1.4.2(http://codonw.sourceforge.net/)计算各物种及数据集密码子偏好性相关指数:相对同义密码子使用度(relative synonymous codon usage,RSCU),有效密码子数(effective number of codon,ENc),密码子第一、第二和第三位置碱基含量(GC1、GC2、GC3),同义密码子第三位GC 含量(GC3s),密码子适应指数(Codon Adaptation Index,CAI)等。有效密码子数是展示同义密码子偏好程度的重要指标,取值在20~61,数值低于35 则表明密码子具有高偏好性。密码子适应指数指编码蛋白的同义密码子与最佳密码子使用频率的符合程度,该值越大表明符合度越高,范围在0~1。根据RSCU、ENc和GC3s 值,在R v.4.0.1 中使用ggplot2 绘制密码子使用偏好分析图。
1.6系统发育分析
为了探讨梅花草属和近缘属间的系统发育关系,以绿玉树()为外类群,使用共有基因数据集重建系统发育关系。利用IQTREE v.1.6.8(参数:-st CODON11-m TEST-alrt 1000-bb 5000-bcor 0.90-wbt-nt AUTO)中的ultra‑fast bootstrap 功能构建最大似然树;使用FigTree v.1.4.3(http://tree.bio.ed.ac.uk/software/figtree/)对最终树进行可视化和编辑。
2 结果与分析
2.1叶绿体基因组结构特征
三脉梅花草、青铜钱、双花假卫矛、圆叶南蛇藤和白杜的叶绿体基因组均为四分体结构,由大单拷贝区(large single copy,LSC)、小单拷贝区(small single copy,SSC)和2 个反向重复区(invert‑ed repeats,IR)构成。所研究的物种叶绿体基因组总长相差较大,介于148 700 bp(梅花草)到159 208 bp(永瓣藤)之间;但不同物种间各分区长度差异较小,仅梅花草SSC 区较其他物种相对较短(见表1)。本研究中获得的5 个物种叶绿体基因组序列已提交至NCBI数据库。
除梅花草(105 种基因)外,梅花草属6 个物种的基因种类均为115 个;所有物种tRNA 基因均为30 种,rRNA 基因为4 种(见表1)。与其他物种相比,梅花草基因家族的、、、、、、、和基因全部缺失,仅含和基因;而基因在永瓣藤、白杜、南蛇藤和双花假卫矛的叶绿体基因组中缺失。对基因进行统计后发现共18个基因具内含子(见表2),包括12 个蛋白质编码基因(和)和6 个tRNA 基因(----和-)。除了和基因外,其余具内含子基因为本研究中所有物种共有。永瓣藤叶绿体基因组具内含子基因最多,有18个。此外,与本研究的其他物种相比,梅花草属物种的基因均无内含子。

表2 叶绿体基因组基因外显子和内含子长度Table 2 Exon and intron length(bp)of chloroplast genes
2.2叶绿体基因组变异分析
为了比较梅花草属与其近缘属叶绿体基因组的整体相似度,以三脉梅花草叶绿体基因组为参考,利用mVISTA程序进行多序列相似度可视化展示。如图1 所示,物种间编码区序列高度保守,非编码区序列差异较大。此外,核苷酸多样性折线图(见图2)显示,梅花草属与近缘属物种叶绿体基因组的反向重复区序列较单拷贝区序列更为保守。
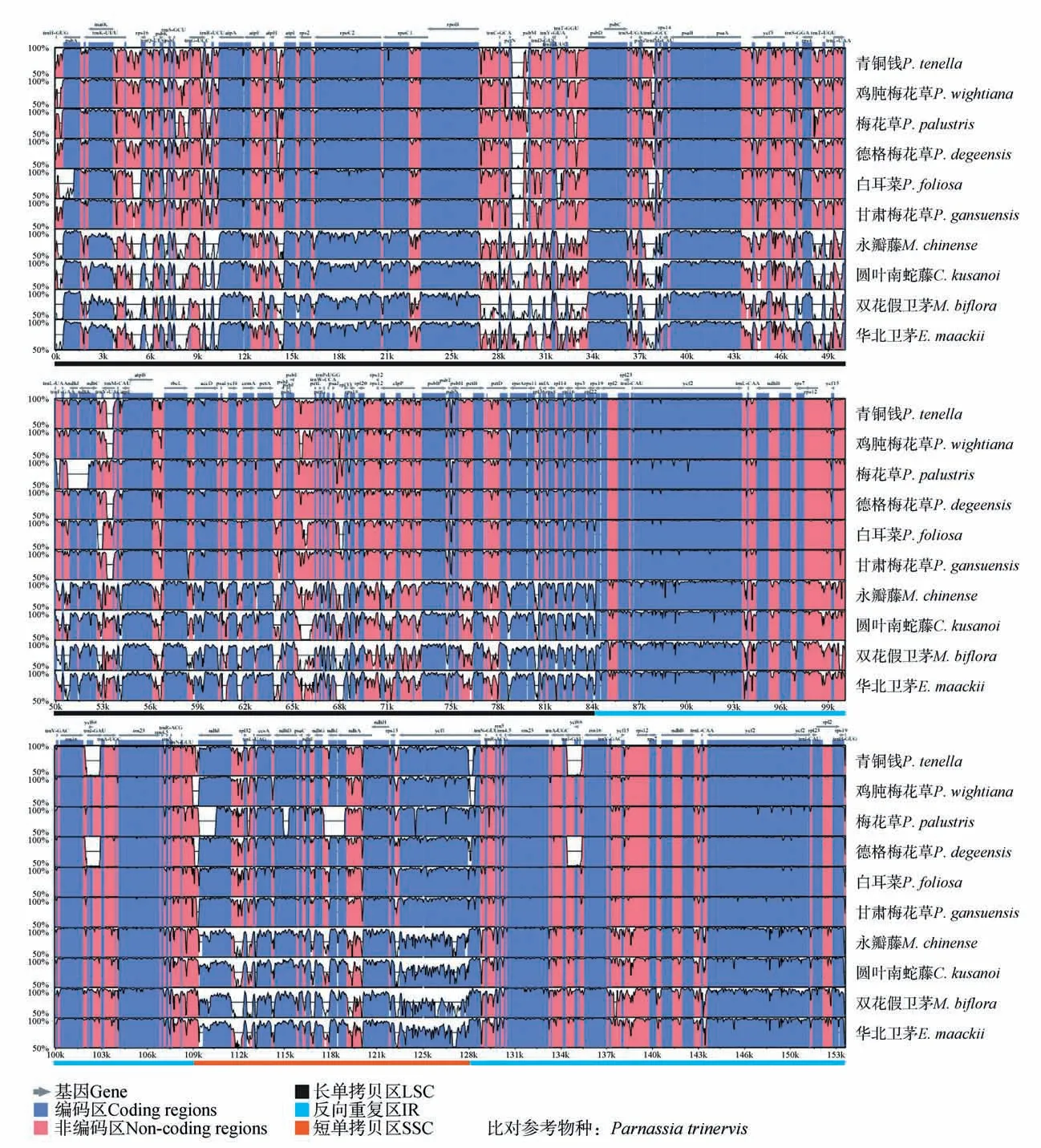
图1 梅花草属与其近缘属叶绿体基因组比对Fig.1 Comparison of chloroplast genome between Parnassia and allied genera
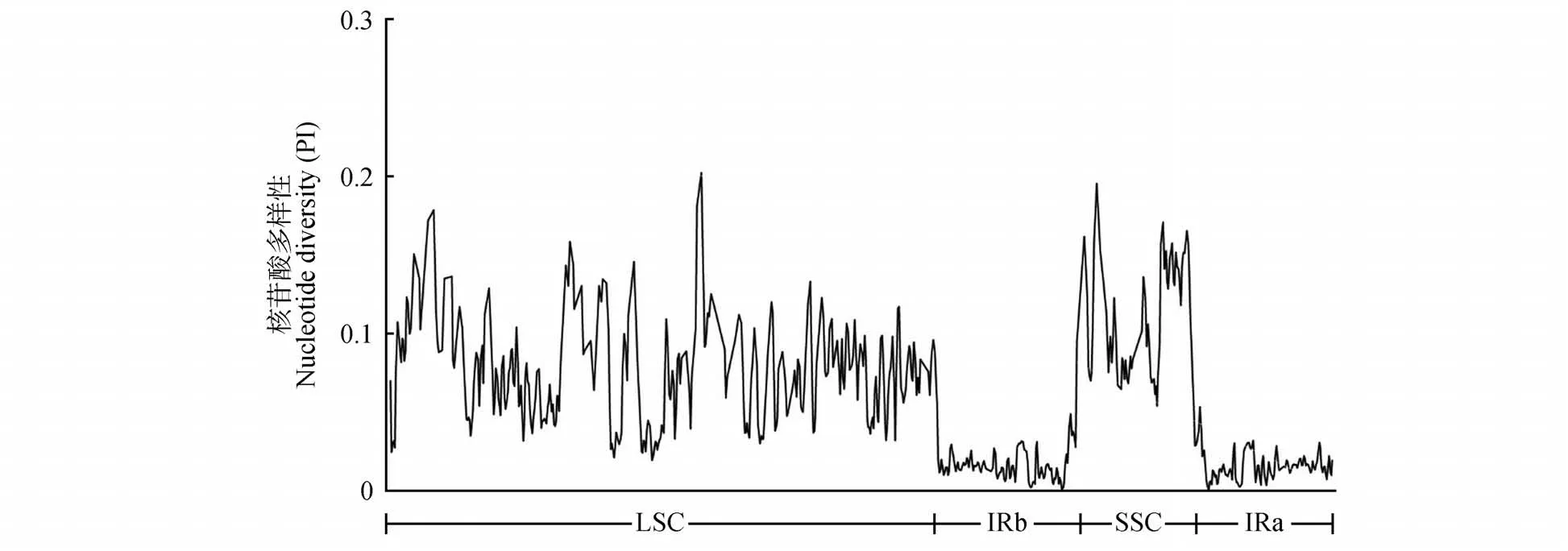
图2 梅花草属与其近缘属叶绿体基因组核苷酸多样性Fig.2 Nucleotide diversity of chloroplast genome of Parnassia and allied genera
梅花草属与其近缘属叶绿体基因组IR/SC 连接区比对结果显示,所有物种叶绿体基因组均为四分体结构,物种间各个分区长度接近(见图3)。梅花草属所有物种LSC 与IRb 边界左右两端基因分别为基因和基因,与南蛇藤属和永瓣藤属保持一致,而卫矛属和假卫矛属物种基因相对向LSC 区移动,使LSC 与IRb边界两端基因变为基因和基因。IRb、SSC 和IRa 边界两端均为和基因,在所有物种中保持一致(梅花草除外,该物种和基因丢失)。值得注意的是,梅花草的SSC 区最短,仅有14 741 bp,这也导致该物种SSC 区两端基因为和基因。此外,所有物种的基因跨过SSC 和IRa 的边界处,仅青铜钱的基因整体位于SSC 区,且与SSC 边界相距439 bp。南蛇藤属和梅花草属LSC 与IRa 边界左右两端的基因均为和基因,且除南蛇藤属和假卫矛属外,所有物种的基因跨过IRa与LSC边界处(见图3)。

图3 梅花草属与近缘属叶绿体基因组分区连接处比较JLB指LSC区与IRb区连接处;JSB指SSC区与IRb区连接处;JSA指SSC区与IRa区连接处;JLA指LSC区与IRa区连接处Fig.3 Comparison of boundaries among SC and IR regions of chloroplast genomes of Parnassia and allied generaJLB refers to the connection between LSC region and IRb region;JSB refers to the connection between SSC region and IRb region;JSA refers to the connection between SSC region and IRa region;JLA refers to the connection between LSC region and IRa region
2.3核苷酸替代率
为了检测蛋白质水平上受到的选择压力,首先将梅花草属和近缘属叶绿体基因组共有的65蛋白编码按照基因家族分为14 个数据集:、(、、、、和)、、、、(和)、(、、、和)、(、、、和)、(、、、、、、、、、、、、和)、、(、、、、、、、和)、(、、、)和(、、、、、、、、、、和),然后对这14 个数据集的同义替代率(dS)、非同义替代率(dN)以及dN/dS值进行估计。结果显示,所有基因均有较低的非同义替换率,仅基因的非同义替换率稍高(见图4)。所有基因dN/dS 值均小于0.4,其中、、和的dN/dS 值较大,为0.25~0.40,其余基因的dN/dS值均小于0.25。
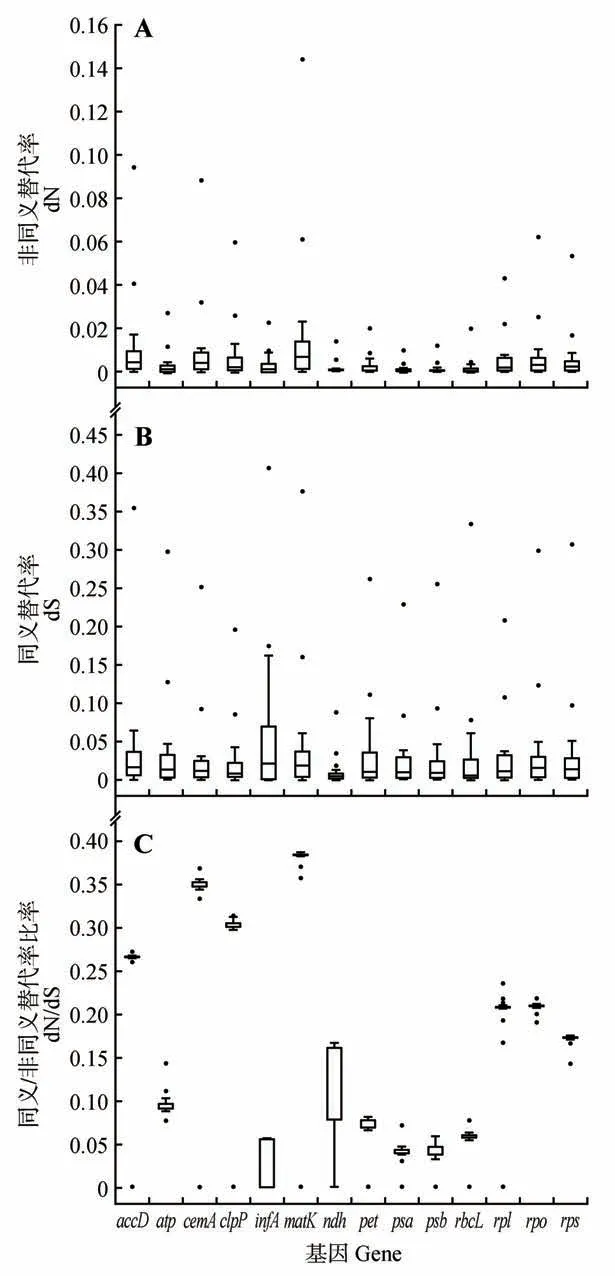
图4 蛋白质编码基因的非同义替代率(dN)(A)和同义替代率(dS)(B)及其比率(dN/dS)(C)Fig.4 Comparison of nonsynonymous(dN)(A)and synonymous substitution rates(dS)(B)and their ratios of protein-coding genes(dN/dS)(C)
2.4密码子使用偏好性分析
对叶绿体蛋白编码基因的密码子偏好性进行统计(见表3),结果显示,物种间总的GC 含量、第一密码子GC 含量和第二密码子GC 含量相差不大,差值未超过0.5%。第三密码子GC含量相对而言差别较大,双花假卫矛含量最低(28.97%),白杜含量最高(29.95%)。所有物种第三位密码子GC含量不超过30%,说明该位点A/T 碱基使用较多。密码子适应指数在0.170~0.175,有效密码子数在49.02~49.74(见表3)。

表3 叶绿体蛋白编码基因的密码子偏好性Table 3 Codon usage of chloroplast protein-coding genes
绘制所有个体14个蛋白质编码基因数据集的ENc-GC3s 散点图(见图5),结果显示,大部分基因的点在曲线上或者接近曲线,、、和基因的点离曲线较远。和基因的点在曲线附近较为分散,其中双花假卫矛和三脉梅花草的基因以及双花假卫矛和南蛇藤的基因离曲线较远。
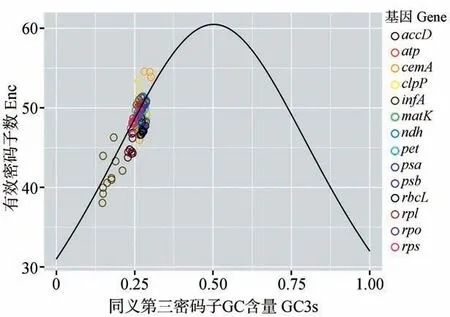
图5 蛋白质编码基因的有效密码子数(ENc)与同义第三密码子GC含量(GC3s)散点图Fig.5 ENc plotted against GC3s of protein-coding genes
对叶绿体蛋白质编码基因的64个同义密码子进行分析(见图6),有29个同义密码子的RSCU 值大于1,且均以碱基A 和U 结尾(UUG 除外)。编码亮氨酸的UUA的RSCU值最高(1.93~2),其次是编码丙氨酸的GCU(1.81~1.88)。RSCU 值最低的同义密码子为编码亮氨酸的CUC(0.35~0.39)和CUG(0.35~0.44),以及编码酪氨酸UAC(0.36~0.42)。物种间的相对同义密码子使用情况略有差异,聚类结果显示,梅花草属的个体聚在一起,然后和((卫矛属,南蛇藤属),永瓣藤属)形成一个分支,最后假卫矛属与其余属聚在一起。
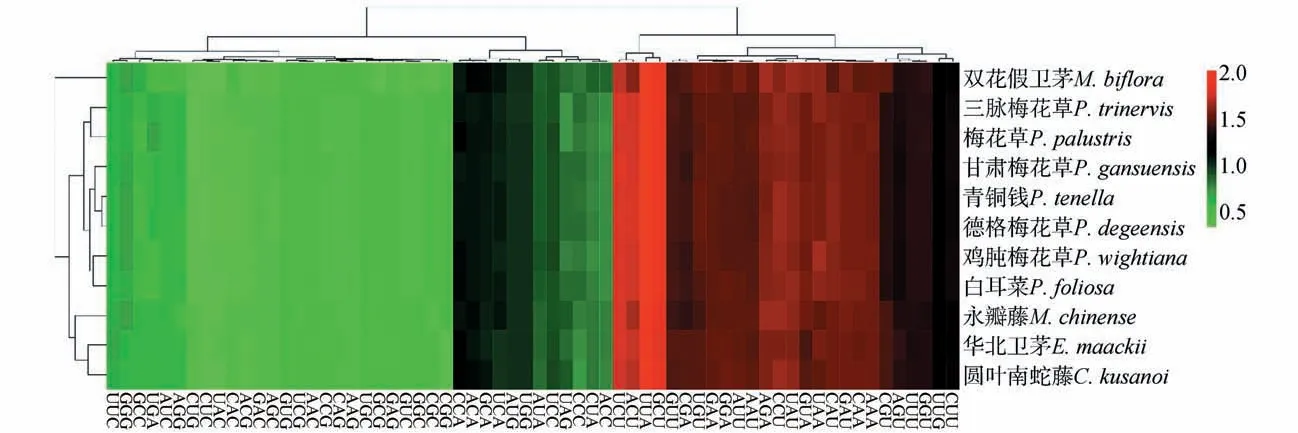
图6 叶绿体基因相对同义密码子使用度热图Fig.6 Heat map of relative synonymous codon usage(RSCU)values of chloroplast genes
2.5梅花草属及其近缘属的系统发育关系
基于叶绿体基因组蛋白编码序列的梅花草属及其近缘属系统发育分析发现,所有分支都有较高的支持率。其中,梅花草属物种单独聚为一支,卫矛科卫矛属、南蛇藤属和永瓣藤属聚为一支,这两个分支互为姊妹类群,与假卫矛属形成一个大的分支(见图7)。
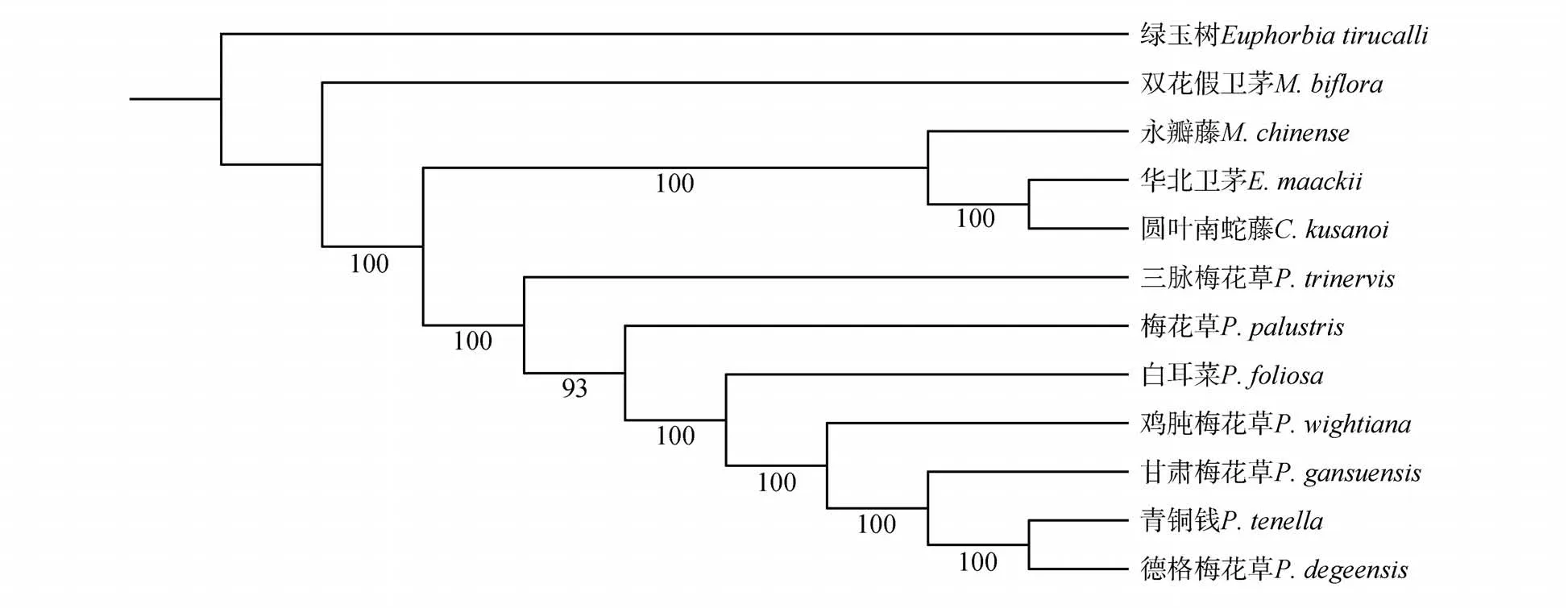
图7 基于叶绿体基因组蛋白编码序列构建的梅花草属与其近缘属的系统发育树系统发育树分支上的数字代表自展支持率Fig.7 Phylogenetic tree of Parnassia and allied genera based on protein-coding sequences of chloroplast genomesThe numbers on the branches of the phylogenetic tree represent bootstrap support rate
3 讨论
自Shinozaki 等发布第一个叶绿体基因组后,越来越多的植物叶绿体基因组相关研究相继被发表,这些研究提高了我们对植物叶绿体基因组的认识。近年来,多个研究利用叶绿体基因组序列数据解决了系统学疑难问题,展现了其在系统学研究上的巨大价值。而植物叶绿体基因、核苷酸替换率和密码子偏好性等特征,可能预示着质体进化的动力和方向,从另一角度揭示物种间的系统发育关系。我们将讨论梅花草属叶绿体基因组的结构和密码子偏好性等特征,期望利用比较基因组学的研究结果,为该属的系统发育研究提供新的思路。
本研究所涉及11个物种的叶绿体基因组结构高度保守,均为四分体结构。梅花草属7个物种的叶绿体基因组总长度在148 700~153 590 bp,其中梅花草叶绿体基因组长度最短,并且该物种叶绿体基因数目也最少。梅花草tRNA 基因和rRNA 基因与同属物种保持一致,蛋白质编码基因缺少11个,其中9 个是家族基因(,,,,,,,和)。基因与叶绿体的呼吸过程相关,而最近对质体基因丢失和进化的多个研究结果显示,是最易丢失的基因之一,推测该基因在当前环境中的生物学意义可能有限。丢失的基因可能转移到细胞核或线粒体中,缺失的功能可由核编码蛋白替代。本研究中梅花草属仅梅花草丢失基因,后续仍需增加该属的采样覆盖度,确认基因的缺失是否代表梅花草属某一分支物种的共同特征。而梅花草属的近缘属,假卫矛属、南蛇藤属、卫矛属和永瓣藤属的物种叶绿体基因组中,均缺少基因。物种在自然选择的过程中会发生基因丢失和获得,这会极大地促进性状进化。结合此前研究中所证实的本属的单系性,我们认为,梅花草属物种中均拥有基因是共同祖先遗传的结果。此外,较本研究中的其他物种而言,仅假卫矛属的双花假卫矛丢失了基因。结合系统发育分析的结果,我们认为,这可能预示着梅花草属与南蛇藤属、卫矛属和永瓣藤属的亲缘关系更近。内含子基因分析结果与该观点相一致,本研究中仅双花假卫矛的基因具有一个内含子,其余物种的基因均无内含子。梅花草属所有物种基因的内含子消失,代表梅花草属这一分支物种的共有特征。需注意的是,本研究中来源自NCBI 数据库的永瓣藤叶绿体基因组序列或存在问题,表现在两个IR 区长度的不一致、序列中出现大片段的重复(直接导致-基因在LSC 区中出现了一个重复)等。由于原始文献中并未发布该物种的测序原始数据,不能确认该情况是物种叶绿体基因组中真实存在,还是序列组装过程中的人为操作失误所致。
梅花草属及其近缘属的叶绿体基因组的整体相似度和核苷酸多样性比对,结果显示,所有物种IR 区比SC 区遗传多态性更低,编码区序列比非编码区序列更为保守。这与大多数被子植物此前的研究结果相类似,变异率高的区域被认为是系统发育分析的潜在分子标记材料。此外,蛋白质编码基因的非同义/同义替代比率已经广泛用于物种进化动力推断等研究。某基因非同义替代率和同义突变替代率值相等,表明自然选择对该基因的适合度没有影响。而dN
作为一个重要的进化特征,密码子偏好性在不同物种之间、同物种不同基因之间存在显著差异。叶绿体基因组蛋白编码基因的碱基含量分析结果显示,物种间基因的GC 含量相差不大,且不同密码子位置的GC 含量具有相同趋势(GC1>GC2>GC>GC3),而在兰科植物核基因密码子的GC 含量分析中也发现了类似的趋势。梅花草属及近缘属叶绿体基因的ENc 值均大于35,表明其密码子偏好性不强。ENc-GC3s 散点图(见图5)可以更好地展示叶绿体基因组的密码子使用偏好性,当基因仅受突变压力作用时,ENc 值应在图中所示的曲线上,ENc 值偏离该曲线则表明基因受到选择压力的作用。根据ENc-GC3s 散点图的结果,大部分基因的点在曲线上或者接近曲线,表明这些基因主要受突变的影响。而偏离该曲线的基因(、、和基因)主要受选择的影响,类似结果在白刺科研究中也有发现。
叶绿体基因组蛋白编码基因的RSCU值比对和聚类分析结果显示,卫矛属、南蛇藤属和永瓣藤属聚在一起,和梅花草属聚成一个分支,最终与假卫矛属聚成一个大支。尽管过去部分研究表明,密码子偏好性的聚类结果与物种的亲缘关系之间存在较大差异。然而在本研究中,该密码子偏好性的聚类结果与蛋白编码序列重建的系统发育关系结果相一致,说明在本研究涉及的类群中,密码子偏好性的部分特征或可反应真实的物种系统发育关系。
梅花草属及其近缘属的系统发育分析结果显示,梅花草属为单系类群,与过去的研究结果一致。而梅花草属与卫矛属、南蛇藤属和永瓣藤属聚为一支,然后该分支与假卫矛属聚在一起,支持梅花草属或为卫矛科早期的衍生谱系而非姊妹类群。而假卫矛属的系统学位置目前仍处于争论之中,相关研究展现的结果并不相同。仍需增加卫矛科物种采样覆盖度,解决梅花草属的系统发育关系问题。
本研究测序并组装了三脉梅花草、青铜钱、双花假卫矛、圆叶南蛇藤和白杜5种植物的叶绿体基因组。结合近缘物种相关信息,揭示了本属的叶绿体基因组结构和密码子偏好性等特征,探讨了本属叶绿体基因组的进化趋势。该属叶绿体基因可能经历纯化选择作用,选择压力在叶绿体基因组蛋白编码基因进化过程中或发挥作用。后续仍需增加该属物种取样覆盖度,以进一步支持和完善梅花草属叶绿体基因组的特征和进化趋势研究。
