幼年特发性关节炎10p11.21 遗传位点敲除的单克隆细胞系的建立
2022-08-02张四鹏李津
张四鹏,李津
(天津医科大学基础医学院细胞生物学系,天津 300070)
自身免疫性疾病是指人体的免疫系统错误地攻击自身的组织、器官,造成自身损伤。目前已经发现了超过80 种自身免疫疾病,总发病率为3%~5%[1]。其中儿童自身免疫疾病严重影响了儿童的身心健康,对个人及家庭带来了重大的经济负担。儿童自身免疫疾病危险因素复杂多样,多项研究显示遗传因素对疾病的发生发展有重要的影响[2]。因此,开展遗传学研究对于儿童自身免疫疾病的预防和精准治疗具有一定的指导作用。
近20 年来,全基因组关联分析(genome-wide association study,GWAS)聚焦于基因组的单核苷酸多态性(single nucleotide polymorphism,SNP),无偏地系统性地研究了与复杂疾病和性状相关的遗传位点,取得了丰硕的成果,已有数千遗传位点在文献中报道[3]。但是关于这些位点的作用机制的研究却很少[4]。
一项关于10 种儿童自身免疫疾病的研究报道了位于ANKRD30A 的内含子区域的SNPrs7100025与幼年特发性关节炎(Juvenile idiopathic arthritis,JIA)显著相关[5]。但是对于这一位点如何影响疾病的发生、发展缺少相应的实验研究。因此,本研究聚焦于对ANKRD30A 的内含子位点的功能研究,通过生物信息学分析和实验方法精细定位这一位点的因果变异,利用CRISPR/Cas9 技术构建删除因果变异所在基因组区域的单克隆细胞系,为后续机制研究奠定基础。
1 材料与方法
1.1 材料 实验所用细胞人HEK293T 以及人Jurkat 细胞均购自ATCC 细胞库,DMEM 培养基、1640培养基、胎牛血清、青霉素链霉素双抗、胰酶均购自美国Gibco 公司,使用的BsmbⅠ酶购自NEB 公司,KpnⅠ、HindⅢ酶和T4 连接酶购自Thermo 公司,Dual-Luciferase Reporter Assay System 试剂盒购自promega 公司,PCR 酶和同源重组酶购自诺维赞公司。
1.2 方法
1.2.1 精细定位因果变异 PICS 平台(https://pubs.broadinstitute.org/pubs/finemapping/)是一个对GWAS遗传位点进行因果变异精细定位的在线分析网站[6]。该算法整合1 000 G 基因组数据中SNP 间的连锁不平衡信息和各种免疫细胞中转录和顺式调控元件,鉴定GWAS 遗传位点最有可能导致疾病的因果变异,即PICS 概率(PICS_Probability)最高的SNP。使用此分析工具,输入10p11.21 位点最相关的SNP(称为index SNP)rs7100025 和疾病关联强度P 值的负对数值-log10(P 值)=10.075 7。rs7100025与JIA 的关联分析P 值=8.4E-11,所以其-log10(P值)=10.075 7。Ancestry 选择EUR,可以得到这个位点的 SNPs 的 PICS 排序以及这些 SNPs 与rs7100025 之间的连锁不平衡关系r2。
1.2.2 PheWAS 数据 PheWAS(Phenome-wide Association Studies,全表型关联研究)是对一个特定的SNP 在全表型组中找到与它关联的表型的一种研究方法,与GWAS 相反,被称为反向GWAS。在Open Targets Genetics 数据库中[7-8],输入rs7100025位点,得到与该位点相关的疾病信息。
1.2.3 细胞培养 HEK293T 细胞使用DMEM 完全培养基培养。DMEM 完全培养基由DMEM 空培加10%胎牛血清以及1%青霉素链霉素双抗组成。Jurkat细胞使用1640 完全培养基培养。1640 完全培养基由1640 空培加10%胎牛血清以及1%青霉素/链霉素双抗组成。两种细胞均在37℃,5%CO2浓度的恒温细胞培养箱中培养。
1.2.4 荧光素酶报告基因质粒构建 在rs7100025位点上下游各250 bp 设计引物。考虑到荧光素酶报告基因的方向效应,在其反方向上也同样设计引物。引物序列见表1。以HEK293T 细胞的基因组DNA 为模板,用PCR 酶将其扩增。PCR 程序为:95℃3 min,95℃15 s 58℃15 s 72℃1 min 35 个循环,72℃5 min。使用的载体为PGL4.23,使用的酶切位点分别是HindⅢ和KpnⅠ。将PCR 产物和酶切产物同时跑胶纯化,然后用同源重组酶将两个产物进行无缝连接,用DH5α 感受态细胞进行转化,铺板。最后挑取单克隆进行摇菌扩增,选择Sanger测序结果正确的质粒进行实验。
构建rs7100025 次等位基因型(minor allele)即突变型的荧光素酶报告基因质粒,使用的引物序列见表1。以构建完成的野生型质粒为模板,PCR 程序为:95℃3 min,95℃15 s 58℃15 s 72℃7 min 30个循环,72℃5 min,PCR 产物用DpnⅠ酶37℃消化2 h,然后用DH5α 进行转化、铺板,挑取单克隆测序,选取测序结果正确的质粒进行实验。构建的突变型质粒同样经过Sanger 测序验证。

表1 质粒构建引物序列Tab 1 Primer sequences for plasmid construction
1.2.5 荧光素酶报告基因实验 将得到的rs7100025 位点的4 个质粒(野生型正向、野生型反向、突变型正向、突变型反向),各取500 ng 分别和50 ng PGL4.74 转染6 孔板中生长的HEK293T 细胞,同时使用500 ng 空载的PGL4.23 与50 ng PGL4.74 作为对照组。转染试剂使用的是PEI。转染的时候使用无血清培养基,6 h 后更换为完全培养基,48 h 后收细胞,并使用promega 公司的Dual-Luciferase Reporter Assay System 试剂盒以及GloMax 20/20 仪器检测荧光强度。
1.2.6 rs7100025 位点 gRNA 质粒构建 在rs7100025 位点上下游区域分别构建sgRNA,sgRNA1 序列: 5′-GCTTCTACAAGTCTTTTTCG-3′,sgRNA2 序列:5′ -AGTGGGATTGCCATGTTATT -3′ ,sgRNA3 序列:5′-CAGTAATTATCTATCGAGTG-3′,sgRNA4 序列:5′-GTTTTGCATCGTAGTTACAC-3′。将这些sgRNA oligo 退火形成双链,退火的条件为95℃10 min,缓慢降温至室温2 h,同时以lenti-cas9-V2 质粒为载体,用BsmbⅠ55℃酶切2 h 回收。用T4连接酶将退火形成的sgRNA 双链和回收后的载体片段连接,构建质粒,质粒均经Sanger 测序正确后使用。
1.2.7 rs7100025 位点敲除细胞系构建 将4 个构建好的rs7100025 位点sgRNA 质粒各2.5 μg,与PAX2 质粒6.5 μg,VSVG 质粒3.5 μg,在空培中混匀,并缓慢加入等体积的混有80 μg PEI 的空培,轻轻混匀后静置20 min,加入到空培培养的10 cm 培养皿的HEK293T 细胞中,6 h 后将空培换成完全培养基。收取其培养48 h 后产生的病毒液,感染Jurkat细胞24 h,然后换取新鲜的完全培养基,加入终浓度2 μg/mL 的嘌呤霉素培养3 d,然后更换为新鲜的完全培养基。培养2 d 后,收集部分细胞,提取基因组DNA,PCR 进行基因型鉴定。PCR 条件:95℃3 min,95℃15 s 58℃15 s 72℃1 min 35 个循环,72℃5 min。
1.2.8 rs7100025 位点敲除细胞系的单克隆的筛选 对经鉴定产生rs7100025 位点敲除的细胞进行单克隆筛选。将细胞稀释到100 个/mL 的浓度后,在显微镜下将单个细胞挑取到96 孔板中,每天观察细胞的生长状况,待细胞长满96 孔板后将其传至24 孔板内,待细胞量足够后,收取部分细胞提取基因组DNA,然后做基因型鉴定。将PCR 产物进行琼脂糖电泳实验,选取有敲除的条带进行Sanger 测序。将测序结果显示rs7100025 位点有敲除的单克隆细胞进行传代培养。
1.3 统计学处理 数据分析和作图采用GraphPad Prism 7.0 软件。两组间比较采用t 检验,P<0.05 差异具有统计学意义。
2 结果
2.1 生物信息学发现10p11.21 位点因果变异 表2 为PICS 值前5 的SNPs。rs7100025 的PICS 值为0.705 7,远远大于同一连锁不平衡区域(Linkage disequilibrium,LD)的其他SNPs。
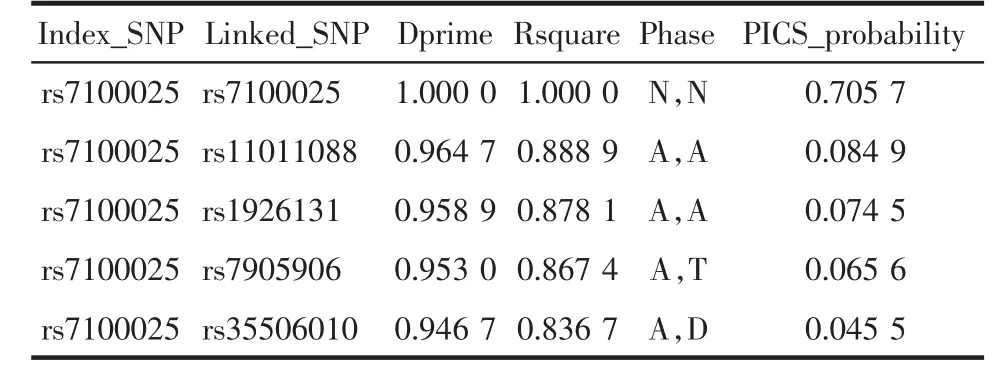
表2 10p11.21 位点的SNPs 为幼年特发性关节炎因果变异的PICS 后验概率Tab 2 PICS posterior probability of being causal variants in juvenile idiopathic arthritis for SNPs at 10p11.21 locus
2.2 SNPrs7100025 是一个多效性变异 针对rs7100025 位点的PheWAS 研究显示,rs7100025 与众多疾病有着密切的关系,例如典型的心脏病、免疫系统疾病,rs7100025 是一个多效性变异(pleiotropic variant),对许多疾病有着重要影响,见图1。
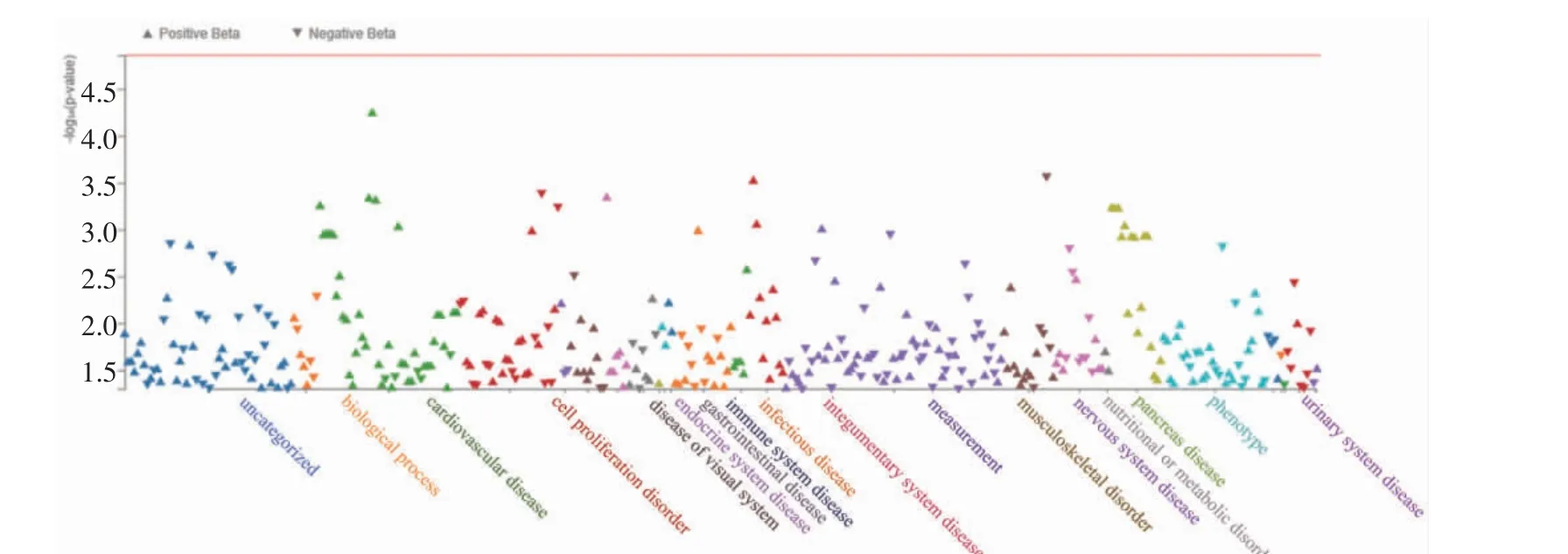
图1 rs7100025 位点PheWAS 结果图Fig 1 The results of PheWAS at rs7100025
2.3 PGL4.23-rs7100025 质粒构建 将rs7100025位点附近一段500 bp 的序列克隆到PGL4.23 载体上,如图2 所示,rs7100025 位点正向和反向序列的PCR 产物为545 bp,载体用Hind Ⅲ和KpnⅠ双酶切之后的长度为4 236 bp,通过同源重组技术将PCR 纯化产物和载体纯化产物连接。质粒构建成功后Sanger 测序无误。
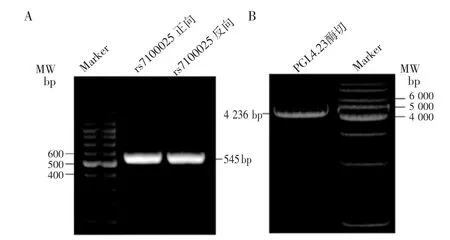
图2 重组质粒PGL4.23-rs7100025 的构建Fig 2 Construction of recombinant plasmid PGL4.23-rs7100025
2.4 荧光素酶报告基因实验检测rs7100025 位点的功能 双荧光素酶报告基因实验显示,正向插入包含G 等位基因和A 等位基因的rs7100025 序列可以提高荧光素酶报告质粒转录活性,并且G 等位基因效果更加明显;反向插入包含C 等位基因和T等位基因的rs7100025 序列对基因的调控与空载对照相比无明显差异,见图3。
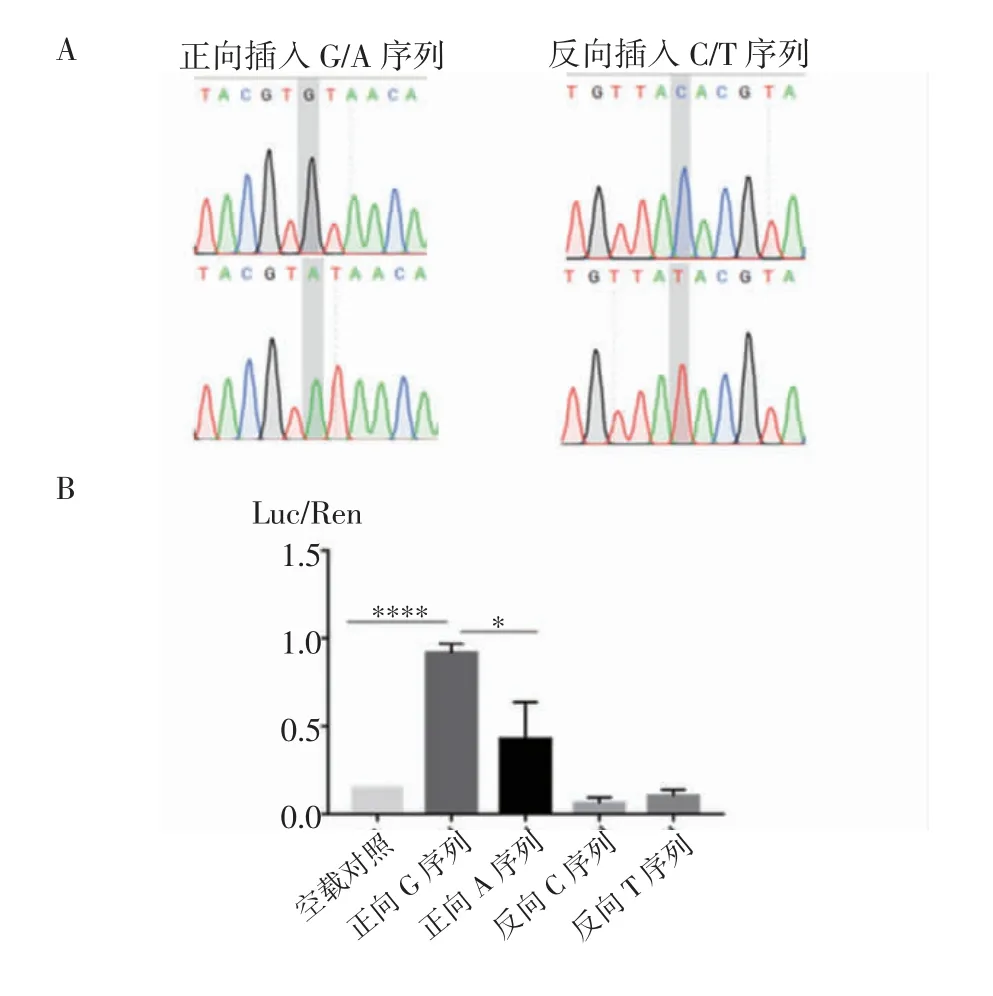
图3 荧光素酶报告基因实验检测rs7100025 的转录调控功能Fig 3 Luciferase reporter gene assay detects the transcription regulatory activity of rs7100025
2.5 rs7100025 位点gRNA 质粒构建 Lenti-Cas9-V2 质粒经BsmbⅠ酶切之后的产物,琼脂糖凝胶电泳回收12 988 bp 大小的条带(图4B),与退火形成的sgRNA 双链用T4 连接酶连接,其中sgRNA 在基因组上的位置如图4A 所示,构建好的质粒均经过Sanger 测序验证正确。2.6 rs7100025 位点敲除的Jurkat 细胞系构建 利用构建的sgRNA 质粒以及慢病毒介导的感染技术,将rs7100025 位点在Jurkat 细胞系中进行敲除,并经过嘌呤霉素筛选,筛选出单克隆。单克隆细胞基因型鉴定的结果如图所示(图5A)。选取3 号单克隆做Sanger测序,测序结果如图所示(图5B)。Jurkat 细胞chr10:37303388 至chr10:37303844 的基因组DNA 序列(456 bp)被敲除。SNPrs7100025(chr10:37303610)就位于这段序列之中。因此,成功建立了幼年特发性关节炎10 号染色体上遗传位点因果变异删除的单克隆细胞。
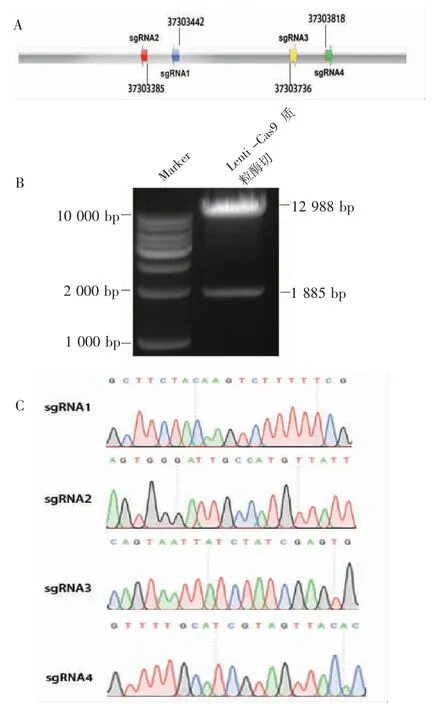
图4 rs7100025 位点敲除质粒的构建Fig 4 Construction of plasmids with rs7100025 genomic region being deleted
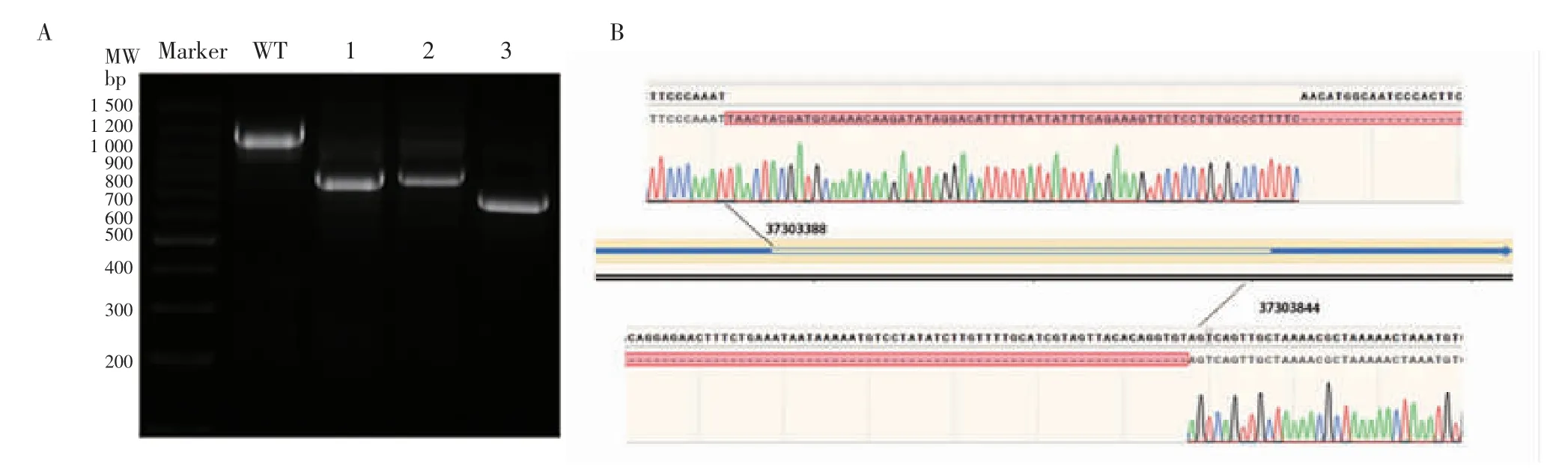
图5 rs7100025 位点敲除的Jurkat 细胞系构建Fig 5 Construction of Jurkat cell line with rs7100025 region being deleted
3 讨论
在过去十多年中,GWAS 已经对人类基因组中数百万个遗传变异进行了基因型-表型关联测试,研究SNP 与疾病的相关性,报道了众多复杂疾病的遗传位点[4,9]。GWAS 中所发现的遗传位点具有以下3 个特点。
(1)GWAS 研究发现的indexSNP 不一定是因果变异。由于LD 的存在,遗传变异通常会与染色体上的一些临近变异高度连锁。因此,当一个区域中的一个或多个变异,对人类复杂特征或疾病产生因果效应时,该区域中的许多非因果变异也与疾病等表型呈现显著相关性。目前已经有许多统计方法和基因组功能注释被广泛应用于确定因果变异[10-12],例如贝叶斯分析、ANNOVAR 注释软件、VEP 注释软件等等。本研究使用了自身免疫病致病变异的遗传和表观遗传精细定位平台进行生物信息学分析确定了rs7100025 为10p11.21 位点最为可能的因果变异。
(2)无论是GWAS 的indexSNP 还是因果变异,大约93%的SNP 都位于基因组的非编码区,例如基因的内含子和基因间的区域[13]。因此,GWASSNPs对疾病发生、发展的影响是通过其对靶基因的转录调控实现的。通过荧光酶素实验证明了rs7100025具有调控转录的能力。
(3)GWAS 位点所调控的靶基因不一定是距离其最近的基因。有可能是eQTL 基因或者Hi-C 基因[14],与GWAS 因果变异在基因组分布上有一定的距离。由于染色质的高度折叠,GWAS 因果变异与靶基因的启动子区相反应,实现其对靶基因的远端调控作用。
由于基因组编辑的困难,GWAS 位点的功能研究远远落后于GWAS 的遗传学发现。CRSPR/Cas9 技术的发明和应用为进行GWASSNP 的功能研究提供了新的方向。在肾癌易感性遗传位点的功能研究中,研究者在人肾细胞癌细胞中敲除因果变异rs35252396,获得了单克隆细胞。进一步与野生型细胞相比,发现其所调控的靶基因及所影响的信号通路[15]。在另一项对多种自身免疫病的遗传学研究中,作者利用CRISPR/Cas9 技术将rs558245864 位点敲除获得了两个杂合细胞系,并与野生型细胞系相比,发现这一位点对染色质可及性及基因转录的影响[16]。因此,建立因果变异删除的单克隆细胞系是对GWAS 位点进行功能研究的重要环节。
GWAS 单一SNP 对疾病的作用力相对较小,体现为其OR 一般仅为1.1~1.2。此外,GWAS 位点的作用往往由多个变异共同驱动,对单个变异进行编辑可能无法表现出足够的作用力以产生相应的细胞或生化水平的变化,因此对整个疾病遗传位点的删除可能是一种更为有效的研究策略[17-19]。同样在技术上,对单一SNP 的编辑目前相对较难。因此在GWAS 位点的功能学研究中,往往对GWAS 位点进行敲除。敲除的片段大小从几十个碱基到几百个碱基不等。当然对单一SNP 编辑,更能精准地研究这一SNP 的功能和作用。在一项关于5 种血管系统疾病(vascular diseases)的研究中,研究者在内皮细胞和血管平滑肌细胞中特异性敲除了rs9349379 位点附近的88bp 区域,通过RNA-seq 实验发现这一缺失导致内皮素-1(EDN1)基因的表达水平升高。然后再对rs9349379 位点进行单碱基的编辑,得到rs9349379的次等位基因型(G/G)的细胞系,验证了EDN1 为rs9349379 位点的靶基因[20]。
利用CRSPR/Cas9 技术对免疫细胞进行基因组编辑的实验存在着许多技术难点。与肿瘤细胞系的贴壁生长不同,作为自身免疫疾病的研究模型Jurkat 细胞是悬浮细胞。对其进行转染效率低,因此采用了慢病毒感染的方法。通过获得高滴度的病毒液来感染悬浮细胞,从而使悬浮细胞的基因组编辑成功进行。应用该技术成功敲除了Jurkat 细胞中rs7100025 所在的基因组区域,为寻找这一位点的靶基因以及后续的相关功能研究奠定了基础。
