基于CRISPR/Cas系统的DNA碱基编辑研究进展
2022-07-22赖昕彤王柯岚由雨欣谭俊杰
赖昕彤 王柯岚 由雨欣 谭俊杰
(南京农业大学农学院 江苏省植物基因编辑工程研究中心,南京 210095)
基因编辑是一种新兴的能对特定基因组进行插入、删除、替换等各种修饰的基因工程技术。CRISPR/Cas系统[1],跟传统基因编辑工具如锌指核酸酶(zinc finger nucleases,ZFNs)[2]和类转录激活因子效应物核酸酶(transcription activator-like effector nucleases,TALENs)[3]等相比,具有设计简单、成本低、效率高等优点,已成为当下基因组编辑主导技术,推动了各行各业的发展。
CRISPR/Cas是细菌和古细菌进化出来的用来抵御噬菌体及外源DNA的适应性免疫系统[4]。目前使用最广泛且技术开发最成熟的是来源于化脓性链球菌(Streptococcus pyogenes)并经过改造的CRISPR/Cas9系统。该系统主要包括单向导RNA(single guide RNA,sgRNA)及Cas9两 个 元 件。CRISPR/Cas9完成基因编辑主要分为两步(图1):(1)Cas9核酸酶在sgRNA的引导下利用其包含的两个核酸酶结构域RuvC和HNH分别对靶向DNA的两条链在距离PAM(protospacer adjacent motif)上游大约3 bp处进行剪断,引起DNA双链断裂(doublestrand break,DSB);(2)DSB内源修复。其修复途径主要包括非同源末端连接(non-homologous end joining,NHEJ)[5]和同源重组修复(homology-directed repair,HDR)[6]。前者介导的DSB修复简单、高效,是大部分细胞修复DSB的最主要方式,但其出错率高,往往引发难以预测的核苷酸插入或删除(insertions and deletions,indels),导致靶基因功能丧失;HDR方式保真度高,但是依赖于外部提供的修复模板,且在大部分细胞中效率极低。因此,虽然CRISPR/Cas9介导的基因编辑简单高效,但因其对DSB的高度依赖,缺乏对编辑结果的控制,可能导致被编辑后的细胞产生无法预测的紊乱[7-8]。实现高效且不依赖DSB的精准编辑将是未来基因编辑的发展趋势。
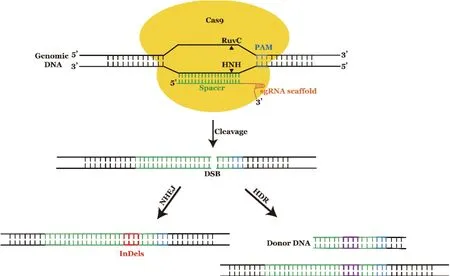
图1 CRISPR/Cas9介导的基因编辑Fig. 1 CRISPR/Cas9-mediated gene editing
1 碱基编辑系统简介
点突变是自然界中最常见的基因突变类型。根据人类基因组变异数据库ClinVar分析,超过半数的人类遗传疾病是由点突变引起[9]。在作物遗传育种上,点突变也是许多重要农艺性状遗传变异的基础。将高效、精准的核苷酸替换应用于基因治疗,可以从根本上治愈点突变引起的人类遗传疾病;应用于作物育种,可以简化并加快育种进程,可见开发高效精准的碱基替换工具十分重要。
2016年,哈佛大学David R. Liu团队在原有CRISPR/Cas9基础上首次开发了实现单碱基替换的新系统-碱基编辑器(base editor,BE)[10]。其将无催化活性的Cas9(catalytically inactive Cas9,dCas9)或者Cas9切口酶(Cas9 nickase,nCas9)与胞嘧啶脱氨酶APOBEC1以及尿嘧啶糖基化酶抑制剂 (uracil glycosylase inhibitor,UGI)进行融合,通过sgRNA将融合蛋白带至靶位点,从而在不引入DSB的前提下,实现对单个碱基的精准替换(图 2)。跟原有CRISPR/Cas9相比,碱基编辑器既不引入DSB,也无需提供外源修复模板即可进行高效精准的基因编辑。该工具的开发使CRISPR/Cas系统从切割特异DNA的分子“剪刀”变成可以重写碱基的“修正器”,打开了精准基因编辑的大门。
由于基于胞嘧啶脱氨酶的碱基编辑器(cytosine base editor,CBE)只能催化C到T的转变,为了扩展编辑类型,该团队随后尝试将自然界中的各种腺嘌呤脱氨酶与nCas9融合,基于CBE类似工作原理,腺嘌呤脱氨酶可以将腺嘌呤(A)转变为次黄嘌呤(I),其在DNA复制中常被识别为鸟嘌呤(G),因此,I与胸腺嘧啶(T)产生I∶T错配后,通过DNA修复途径或细胞复制,完成A到G的转变(图 2)。考虑到自然界中腺嘌呤脱氨酶只能以RNA为底物,该团队随后将大肠杆菌中tRNA腺嘌呤脱氨酶(tRNAspecific adenosine deaminase,TadA)与Cas9切口酶 融合,并进行多轮人工进化,最终获得了编辑效率最高的腺嘌呤碱基编辑器(adenine base editor,ABE)ABE7.10[11](图 2)。
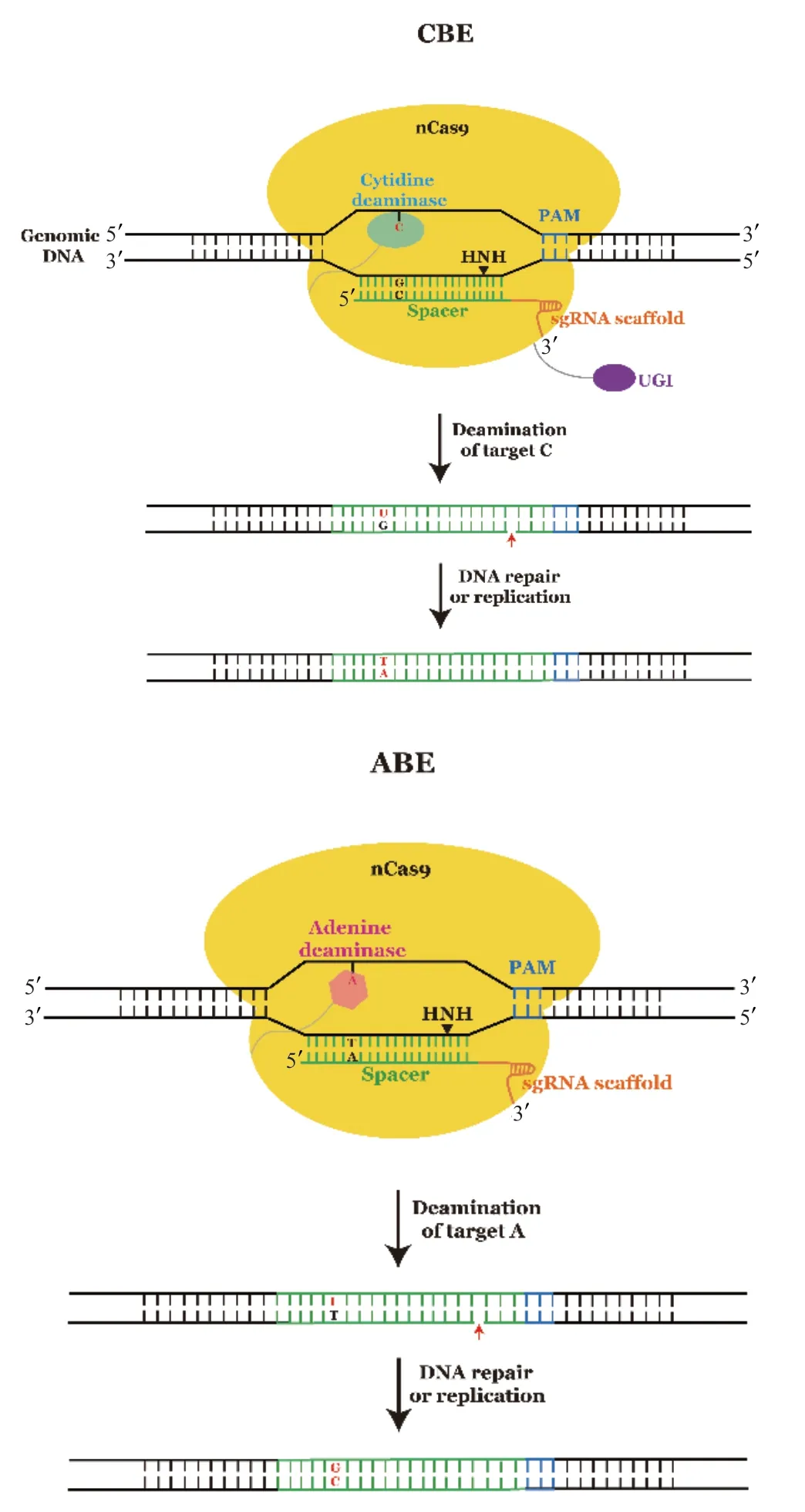
图2 CBE和ABE介导的碱基编辑Fig. 2 CBE and ABE-mediated base editing
CBE和ABE能实现全部单碱基转换(base transition),但不能有效实现单碱基之间的颠换(base conversion)。随后研究人员也在积极探索除CBE和ABE之外的可以实现其他类别的碱基编辑技术。2020年,张学礼和J. Keith Joung两团队首次独立报道了能实现碱基颠换的新型碱基编辑器 GBEs(glycosylase base editors) 和CGBE1(C-to-G base editors)[12-13]。其工作原理和CBE很接近,都是首先将胞嘧啶转换成尿嘧啶,不同的是,GBE接下来直接用融合在nCas9另一端的尿嘧啶 DNA 糖基化酶(uracil DNA N-glycosylase,UNG)将U移除,并引发碱基切除修复,提供C碱基向其他碱基突变的机会,在大肠杆菌中主要是C→A,而在哺乳动物细胞中主要是C→G[12]。为了在大多数位点上实现高效、高纯度的C→G碱基编辑,David R. Liu团队通过CRISPRi筛选、靶点数据库分析并联合机器学习开发了更加有应用前景的新型CGBE[14]。
这些DNA碱基编辑工具目前已成功应用于动物[15-17]、植物[18-20]和微生物[21],在点突变引起的人类疾病治疗和作物精准育种上有巨大的应用 潜能[22]。
2 碱基编辑系统优化
2.1 提高编辑效率
第一版碱基编辑器(BE1)通过将大鼠的胞嘧啶脱氨酶rAPOBEC1简单地融合到dCas9的氨基端,并用一段16 aa的XTEN蛋白基序连接。虽然APOBEC1可以催化C到U的转变,但由于细胞内存在尿嘧啶糖基化酶UNG,会不断去除脱氨后产生的U,从而导致C→T转换效率低下。针对此问题,第二版碱基编辑器(BE2)通过融入尿嘧啶糖基化酶抑制剂UGI,从而保护U不被移除,使在人体内的编辑效率提高了3倍。第三版碱基编辑器(BE3)主要是用Cas9切口酶替代dCas9,通过在非编辑链引入切口,从而激活碱基错配修复(mismatch repair,MMR)机制,并以编辑链为模板进行修复,促进了编辑产物的形成,该策略将编辑效率提高了2-6倍,平均编辑效率达到37%,同时indel的产生比例仅有约1.1%[10]。第四版碱基编辑器BE4主要是通过融入更多的UGI,最大程度的减少了编辑副产物,提高了C→T突变的效率[23]。此外,通过在编辑器两端融入密码子优化的双分型核定位信号(bipartite NLS,bpNLS),产生了编辑效率最大化的BE4max以及ABEmax[24]。在腺嘌呤碱基编辑器ABE7.10的基础上,研究人员利用腺苷脱氨酶变体库对其进行了进一步的优化,获得效率更高的ABE8版本,与ABE7.10相比,其在A5-A7处的编辑效率提高了约1.5倍,A3-A4和A8-A10处约3.2倍,非NGG PAM变体的总体目标编辑效率提高约4.2倍[25]。David R. Liu团队对ABE7.10进行多轮定向进化,获得了ABE8e,与ABE7.10相比,该版本在TadA上引入了8个额外突变,活性更是提高了590倍,且能与各种Cas9 或Cas12同源蛋白广泛兼容[26],ABE8e的晶体结构解析表明,其催化 DNA 脱氨的速度比早期的ABEs快约1 100 倍[27]。除了哺乳动物,ABE8e在编辑其它物种上同样具有超高的编辑效率,比如多个研究团队几乎同时报道ABE8e在编辑水稻基因组上比原来ABEmax表现出更高的编辑效率,在诸多靶位点的编辑效率更是达到了几乎100%[28-29]。此外,周焕斌团队通过比较分析ABE8.17、ABE8.20以及ABE8e,开发了全新版本ABE9,其编辑水稻基因组的能力甚至高于ABE8e,尤其是对于原来难以编辑的位点,ABE9表现出更佳的编辑能力[28]。
其它的效率优化策略还包括在脱氨酶与Cas蛋白之间插入非特异的单链DNA结合蛋白基序,增强脱氨酶与底物的互作[30]。这些策略极大提高了碱基编辑器在哺乳动物以及植物细胞中的编辑效率,推动了其进一步的应用。
2.2 改变编辑窗口宽度
Cas9-sgRNA复合体与目标DNA链结合后,随后与互补的DNA序列退火形成“R-loop” 结构[31],其中暴露的单链DNA即为脱氨酶的底物。碱基编辑器在单链DNA上具有一定靶向范围,该范围内的碱基均能被脱氨酶有效脱氨,该编辑范围称为碱基编辑器的编辑窗口或活性窗口[10]。由于融合蛋白与目标DNA序列结合后掩盖了PAM相邻的核苷酸,因此BE的活性窗口通常位于PAM的远端[10,32-33]。例如,BE3的活性窗口为远离PAM端的第4-8位碱基(C4-C8)。
编辑窗口的宽度受多种因素的影响,包括脱氨酶与Cas蛋白的类型、碱基编辑器的空间构造 等[10-11,15,33-35]。表1 总结了目前主要的碱基编辑器及其编辑窗口宽度、序列偏向等特征。

表1 主要碱基编辑器及其特征Table 1 Major base editors and their characteristics
靶向碱基的编辑依赖于脱氨酶和底物之间的互作,因此不同种类的脱氨酶会影响活性窗口宽度[34]。例如,通过把BE3中的APOBEC1替换为源于海鳗(Petromyzon marinus)的脱氨酶CDA1,构建出新的碱基编辑器CDA1-BE3,其编辑窗口变为C1-C7。研究学者利用不同种类的胞嘧啶脱氨酶构建了多样化的碱基编辑编辑器,主要包括哺乳动物APOBEC家 族 蛋 白,如AID[36-37]、APOBEC1和APOBEC3(A3A-H)[38-42],以及七鳃鳗CDA1家族蛋白,如CDA1[33-35,43-45]。另外,改变脱氨酶的催化或结合活性也会影响编辑窗口的宽度。例如,通过突变rAPOBEC1中与底物结合或催化相关的氨基酸残基,产生一系列BE3变体YE1/YE2/YEE-BE3,大大缩小了碱基编辑窗口宽度[46]。在APOBEC3A中引入点突变N57G,产生工程化的eA3A-BE3,增强了对TCR基序的偏好性,间接缩短了编辑窗口宽度[38]。将F148A突变引入人工进化的TadA*,所组成的ABE7.10F148A也大大缩小了编辑窗口[47]。此外,通过同时融合多个相同或不同的脱氨酶,使R-loop中底物核苷酸在局部高浓度脱氨酶的环境中更易暴露,从而达到拓宽编辑窗口的目的[48],还可使编辑产物多样化或提高编辑效率[49-52]。大多数脱氨酶对其底物DNA的序列表现出一定的偏好,这也间接影响着编辑窗口的宽度。例如,APOBEC1对底物序列的偏好 性 为TC≥CC≥AC>GC[10],而APOBEC3G偏好CC基序中的第2个C[41-42]。目前,包含CDA1、AID或A3A脱氨酶的碱基编辑器对底物DNA序列的依赖性较小,可能是因为这些脱氨酶的活性相对较高[33,37-40,53]。
自然界天然存在或人工进化的Cas变体可以识别不同的PAM基序,且其组成的碱基编辑器可能拥有不同的编辑窗口。例如SaCas9介导的碱基编辑器支持更宽的编辑窗口:其介导的CBE编辑窗口为C3-C12,ABE编辑窗口为A4-A12[46];基于Cfp1/Cas12a的碱基编辑窗口为C8-C13[54];由微型Cas12f变体(CasMINI)衍生的ABE具有A3-A4的窄窗口[55];而由环状排列的Cas9变体(Cas9-CP)组成的CBE和ABE改变了脱氨酶在R-loop中的定位,从而在更宽的编辑窗口中实现了更有效的靶向碱基[56-57]。
碱基编辑器内部构象也会影响编辑窗口宽度,包括脱氨酶与Cas蛋白的融合位点、连接两者的接头类型和长度等。例如,将PmCDA1分别融合到Cas9切口酶的两端,形成n/cCDA1-BE3,两者呈现出不同的编辑窗口(nCDA1-BE3,C1-C7;cCDA1-BE3,C2-C5)[34]。对于nCas9介导的碱基编辑器,将脱氨酶融合在N端通常会比融合在C端产生更宽的编辑窗口[10,34,44],这可能是由于N端融合会使脱氨酶更易接近R-loop内的ssDNA。除在两端融合之外,最近还报道了脱氨酶可以嵌入Cas9中,所产生的一系列碱基编辑器拥有独特的活性窗口,其宽度取决于它们在Cas9中的插入位点[58-59]。此外,碱基编辑器中Cas9和脱氨酶的连接长度也会影响编辑窗口宽度。例如,适当缩短Cas9与CDA1之间的连接长度,甚至去掉脱氨酶C端不必要的序列,可以很大程度的缩小编辑窗口宽度至1-2 nt[34]。
总之,碱基编辑窗口的多样化可以满足碱基编辑器不同的应用需求,从而推动其进一步的发展和应用。
2.3 拓展编辑范围
碱基编辑需要靶位点包含PAM,比如基于SpCas9的碱基编辑器识别的PAM序列为NGG,这种特定的PAM要求限制了可编辑的区域,严重阻碍了其广泛应用。为了扩大碱基编辑的范围,常采用的方式是利用识别不同PAM的天然Cas蛋白或人工改造的Cas9变体。比如,陈佳团队使用Cas12a (Cpf1)开发了新型的CBE系统,识别的PAM 序列为TTTV(V可以是A、C或G),这一系统适用于富含T的基因组DNA[54]。David R. Liu团队利用来自金葡菌(Staphylococcus aureus)的Cas9蛋白,构建了靶向包含NNGRRT的PAM序列的碱基编辑器SaBE3[46]。此外,通过Cas变体的替换,获得了各种PAM松弛型的碱基编辑变体,如VQRBE3(NGA)、VRER-BE3(NGCG)、SaBE3-KKH(NNNRRT)[46]。尤其是利用Nureki实验室开发的SpCas9-NG(NGN)[60]以及Kleinstiver实验室开发的SpG(NGN)和SpRY(NRN 或NYN)[61]等Cas9变体所获得的碱基编辑器,几乎能够突破PAM的限制,靶向所有NNN的位点。值得注意的是,尽管这些变体的利用拓展了碱基编辑器的编辑范围,但却大大降低了其编辑效率,也增加了对靶位点的依赖性。因此,如何在维持松弛型PAM的识别下进一步提高碱基编辑器的效率值得进一步的研究。此外,这些PAM松弛型的碱基编辑变体的应用具有一定的自编辑活性,尤其是基于SpRY的CBE和ABE,其能够识别sgRNA骨架上与靶序列相连的GTT PAM 序列,因此会对自身的sgRNA进行编辑,从而一方面降低其编辑效率,另一方面改变的sgRNA可能会产生非靶向编辑,导致脱靶效应[62-64]。如何减少自编辑活性仍是基因编辑领域目前亟待解决的难题之一。
2.4 提高编辑特异性和精准度
在细胞中应用碱基编辑器时,会出现DNA及RNA水平上的脱靶。其中DNA水平上的脱靶分为sgRNA依赖型和非sgRNA依赖型。前者是由于sgRNA错误地引导了Cas蛋白,使之与相似的DNA序列相结合[65-67]。研究表明,使用工程化的高保真Cas变体[68-71]或截短的sgRNA[72-73],或递送碱基编辑器与sgRNA形成的核糖核蛋白复合体(ribonucleoproteins,RNPs)[74]等策略均可以降低此类脱靶效应;而后者是由于脱氨酶的过度表达,使全基因组特别是基因富集区域发生了随机点突变。值得注意的是,这类脱靶只出现在CBEs中,在ABEs中并未发生,其原因可能是ABE中的脱氨酶来源于RNA特异性编辑的腺嘌呤脱氨酶,经人工进化改造后表现出了较高的特异性,从而未表现出过度的DNA碱基编辑[75-76]。大量研究表明,通过工程化改造脱氨酶可以减少这类脱靶现象。例如,利用人工改造的rAPOBEC1变体YE1建立YE1-BE3碱基编辑系统,可以大大减少非sgRNA依赖型的脱靶[77-78]。通过筛选153种胞嘧啶脱氨酶,并进一步优化其靶标位点和非靶标位点的编辑,研究人员发现其中4种脱氨酶变体(AmAPOBEC1,PpAPOBEC1,RrA3F,SsAPOBEC3B)显示出最小的非sgRNA依赖型的DNA脱靶活性[79]。另有研究表明,将脱氨酶精准嵌入Cas蛋白结构中,也可以减少全基因组范围的DNA和RNA脱靶[58-59]。拥有较窄编辑窗口的碱基编辑器一般伴随着较少的脱靶现象,因为这种情况下,无论是目标还是非目标区域能编辑的碱基都变少了,从而导致脱靶也变少或消失[35,77]。
除了DNA脱靶,由于CBE和ABE所使用的均是能作用于RNA的脱氨酶,因此两者还会出现全基因组范围内的RNA脱靶[47,80-82]。尽管RNA脱靶突变存在的时间很短,但是如果持续诱导,仍存在很高的风险,特别是在遗传疾病治疗上。与非sgRNA依赖的DNA脱靶类似,目前消除此类RNA脱靶效应的策略是在CBE和ABE中设计合适的脱氨酶变体。例如,通过工程化改造脱氨酶,获得了3个高 保 真 的CBE变 体(BE3W90Y/R126E,A3AR128A-BE3,A3AY130F-BE3)和一个ABE变体(ABE7.10F148A),它们不仅可以高效地靶向目标DNA,还能有效减少RNA的脱靶编辑[47]。另有研究人员通过筛选一系列BE3和ABE变体,发现两个BE3变体(rAPOBEC1;R33A,R33A/K34A)和 两 个ABE变 体(TadA*;K20A/R21A,V82G)具有较低的RNA编辑活性[80-81]。而且,他们还发现含有eA3A(eA3A-BE3)、hAID(hAID-BE3)和CDA1(target-AID)的CBE可以减少RNA脱靶。此外,通过V106W的替换或R153的缺失突变,获得了工程化改造的TadA*变体,该变体构成的ABE大大减少了RNA脱靶[82-83]。
除了在DNA和RNA水平上发生非预期的脱氨,当编辑窗口内含有多个非目标碱基C时,目标碱基C被编辑的同时也伴随着非目标碱基C的编辑,从而产生大量的编辑副产物,给碱基编辑尤其是基因治疗上的应用带来了潜在的风险。此外,在植物碱基编辑方面,尽管发生旁侧编辑的植株可以在后期中筛掉,但大量的编辑副产物不仅会使后续的筛选工作更加繁琐,还导致了对前期转化编辑效率的过度依赖。因此,缩小碱基编辑器的编辑窗口,提高其编辑精度,变得尤为重要。大量相关研究表明,缩小碱基编辑窗口的策略主要有以下3种:(1)通过突变碱基编辑器的脱氨酶,适当降低其催化和结合活性。比如,通过突变脱氨酶APOBEC1催化及结合相关的关键氨基酸残基W90Y、R126E或R132E,开发一系列缩小碱基编辑窗口的BE3变体YE1/YE2/YEE-BE3[46]。虽然这3个变体的编辑窗口均有缩小,但其编辑效率也随之降低,尤其是编辑窗口最窄的YEE-BE3,在许多位点缺乏编辑活性。(2)通过突变脱氨酶,增强其对底物的偏好性。比如,通过在人类APOBEC3A引入N57G突变,构建工程化的碱基编辑器eA3A-BE3,实现选择性的识别TCR底物基序[38]。相似的,通过工程化产生的eA3G-BE3,增强对CC底物基序识别的偏好性[41-42]。该方法虽然在包含特定基序的编辑位点上使编辑窗口变窄,但缩小程度有限且极大依赖于底物序列。(3)调整碱基编辑器的内部空间构造。比如,通过降低脱氨酶与Cas9之间的接头长度,改变碱基编辑器的空间构造,从而让脱氨酶的活性区域在靶向DNA上维持于一个更加精准的定位[34-35]。而且该策略不改变氨基酸的催化及结合活性,只是让脱氨酶在非靶碱基处“英雄无用武之地”。尽管该方法目前只测试了少数脱氨酶及其构成的碱基编辑器,但由于该策略产生的碱基编辑器在大大缩短编辑窗口的同时并未降低目标碱基的催化活性,因此值得进一步的研究和发展。
3 碱基编辑应用
3.1 碱基编辑用于疾病治疗
根据人类基因组变异数据库ClinVar分析,超过半数的人类遗传疾病是由点突变引起[9]。而碱基编辑器能够高效实现单个碱基的替换,因此其主要的潜在应用之一是用来纠正点突变引起的人类遗传疾病。例如,把BE3转入小鼠星形胶质细胞,可以将与阿尔茨海默病相关的等位基因APOE4转变为低风险的APOE3r,也可以纠正乳腺癌细胞中与癌症相关的p53基因突变(Y163C)[10]。利用ABE将与镰状细胞病(SCD)相关的致病基因型HBBS转化为造血干细胞中的良性等位基因型HBBG,并成功将镰状细胞病小鼠的血液学参数恢复到接近正常水平[84]。利用脂质纳米颗粒将ABE递送到食蟹猴肝脏细胞中,并对PCSK9基因实现碱基突变,将其体内的坏胆固醇含量降低约60%[85]。其他应用碱基编辑治疗的疾病包括Duchenne肌营养不良症[86]、白化病[86]、Hutchinson-Gilford早衰综合征[87]和其他代谢疾病[88-89]。
3.2 碱基编辑用于作物遗传改良
与传统育种相比,碱基编辑器能够快速高效地对植物基因组进行精准修饰,加速了作物育种进程。近年来,许多植物研究者通过密码子优化,构建了适用于植物的碱基编辑系统,由此实现了对水稻、小麦等主要农作物的重要农艺性状相关基因定点突变,快速获得改良品种。例如,Zong等[90]构建了植物BE系统PBE(Plant base editor),并在原生质体和再生的水稻、小麦和玉米植株中,实现了C3-C9位点的碱基编辑,效率高达43.48%。随后他们又通过融合nCas9和人类APOBEC3A构建了A3A-PBE,并成功在小麦、水稻和马铃薯中产生高效C→T的靶向突变。A3A-PBE实现了在17 nt的编辑窗口内对内源基因组位点和不同序列的高效碱基替换[40]。此外,Hua等[91]利用人工进化的SpCas9和SaCas9变体,并构建了相应BE变体,在水稻中大大扩展了编辑范围。ABE和CBE在改变作物株型、提高作物抗性和养分吸收效率方面均发挥重要作用。Lu等[92]利用大鼠APOBEC1基因开发了水稻碱基编辑系统,并有效编辑了水稻的两个重要基因NRT1.1B和SLR1,编辑效率分别为3.7%和13.3%。SLR1编码DELLA蛋白,其TVHYNP基序及其附近的氨基酸发生置换,导致植株高度降低,获得了半矮秆型材料。Li等[93]将nCas9、胞苷脱氨酶和UGI进行融 合,在水稻中产生目标点突变,成功在OsPDS(编码一种植物烯去饱和酶),OsSBEIIb(编码水稻淀粉分支酶IIb)的3个目标位点上引入了精准编辑。
值得注意的是,BE 在人类和作物上的应用有所不同,作物基因编辑上强调特异性外,也强调多样化,产生更多的变异。该特征通常用于植物内源基因定向进化,通过对靶基因区域进行碱基编辑,产生大量和多样化的突变,随后进行人工筛选获得提高农艺性状的目标变异。最显著的例子是植物抗除草剂位点的发掘,例如通过合用CBE和ABE,以及针对靶基因的sgRNA文库,在水稻中对除草剂内源靶标基因OsALS1进行近乎饱和突变,从而成功鉴定出OsALS1抗除草剂新位点,并创制出具有除草剂抗性的水稻新种质[94]。高彩霞团队通过同时融合两种不同类型脱氨酶设计了双碱基编辑器STEMEs,并利用STEME-1和STEME-NG对水稻OsACC基因进行定向进化,获得了抗除草剂的水稻新种质[50]。为了提高定向进化效率,未来碱基编辑器需要拥有更宽的活性窗口、更大的编辑范围和更多碱基转换类型,从而为后续的人工筛选产生更加多样化的编辑产物。
3.3 CBE引入终止密码子实现基因敲除
CBE可以通过C→T单碱基转换,将4种密码子(正义链上的CAA、CAG、CGA和反义链上的TGG)转换为终止密码子(TGA、TAG、TAA),实现在不引入DSB的情况下,在目标位点处提前引入终止密码子来敲除蛋白质编码基因。Jin Soo Kim团队率先利用BE3在小鼠胚胎中实现了对靶基因Dmd或Tyr的高效终止密码子引入,并成功在小鼠中建立了肌营养不良以及白化病的相关疾病模型[86]。此外,利用BE3引入无义突变的原理,Adli团队以及Ciccia团队分别建立了使基因失活的CRISPRStop[95]和iSTOP[96]系统,后者同时在包括人类、小鼠和拟南芥等8个物种建立了sgRNA文库用于配合BE3使用,可在全基因组范围内失活真核基因,并模拟癌症相关的无义突变。
与CRISPR介导的移码突变相比,基于CBE终止密码子引入方法的最大优点是无需产生DSB。作为最严重的细胞损伤,DSB会激活广泛的细胞损伤修复机制或者细胞应激甚至死亡,相比之下,CBE介导的基因失活策略更加温和、安全。为了更深入了解二者的差别,Dang等[97]针对肿瘤抑制基因,设计了两个靶向TP53位点的sgRNA,利用BE3介导的iSTOP和传统的CRISPR/ Cas9对其进行敲除。实验表明,BE3产生的基因型更少,对染色体结构和邻近基因影响不大。此外二者对于两个相邻基因的双敲除效率存在差异,BE3介导的基因敲除对相邻基因的副作用更小,更能安全、有效地敲除两个近距离的基因。
此外,该策略形成的编辑结果精准可控,利用该策略在第一代获得纯合突变体的概率也会更高。将其应用于在植物中,可以加速获得纯合突变体,从而应用于反向遗传或作物育种过程。Ren等[98]开发了高效的A3A/Y130F-CBE_V01系统,并用于植物的多重编辑。他们在多个控制种子形状相关的基因中提前引入终止密码子,成功敲除OsGS3、OsGW2等基因,并快速获得纯合的多突变体,有效增加籽粒的长度和宽度。
总之,碱基编辑器在动、植物中已获得了广泛的应用,除了上述主要应用外,随着人们对碱基编辑器的深入了解,其更多的未知应用将被逐一挖掘。
4 总结和展望
碱基编辑器由于其设计简单、高效、结果可预测等优点,在基因治疗以及作物精准育种上有着无限的应用潜能。本文初步概括了DNA碱基编辑的研究进展。这些研究进展一方面为碱基编辑器进一步的发展奠基了基础;另一方面对碱基编辑的未来发展提出了新的问题和挑战:(1)如何进一步提高碱基编辑器的精度以及在维持碱基编辑器的高精度下,如何进一步平衡编辑范围、效率以及特异性。尽管目前许多研究报道了提高碱基编辑器精度的方法,但目前仍然没有办法仅仅编辑单个碱基而不影响旁侧同类碱基。此外,编辑精度的提高也伴随靶向区域减小,以及对PAM依赖的增加。随着各种Cas9变体的开发,尤其是较少依赖PAM的Cas9-NG、Cas9-SpRY等的出现,维持编辑精度且突破靶向范围变得可能,但Cas9变体的引入牺牲了部分的编辑效率,此外,随着精准碱基编辑范围的扩展,其特异性是否受到影响仍待评估。其背后的科学问题,比如碱基编辑器中脱氨酶脱氨的结构机理、sgRNA识别Cas9的分子机理以及碱基编辑器脱靶的分子机制等值得进一步的探究,解决这些技术瓶颈背后的科学问题,为研发高效、广适性和特异性的精准碱基编辑工具提供坚实的理论依据。(2)如何进一步拓宽能够被编辑的碱基类型。目前的碱基编辑器主要实现碱基转换而不能进行碱基颠换,虽已有少量报道C到G(或A)的碱基编辑器,但其编辑产物依赖被编辑的细胞类型,且会使indel出现的频率增加,因此还需要进一步的优化。(3)如何开发更强大的基于碱基编辑器的人工定向进化工具。碱基编辑器可以模拟自然界在设定的靶向DNA区域内随机产生大量的点突变,后续利用适当的筛选压力鉴定出所需的有益变异。扩增编辑窗口宽度、拓展可同时编辑的碱基类型以及提高编辑效率都是未来优化的方向。
