质谱法定量生态物种基因组DNA低甲基化
2022-07-19胡俊杰吴毅聪陈桂连高圆圆兰善红吕小梅
胡俊杰,吴毅聪,陈桂连,高圆圆,赵 桐,余 广,兰善红,吕小梅*
质谱法定量生态物种基因组DNA低甲基化
胡俊杰1,吴毅聪1,陈桂连1,高圆圆2,赵 桐3,余 广1,兰善红1,吕小梅1*
(1.东莞理工学院生态环境与建筑工程学院,广东 东莞 523808;2.东莞市生态环境技术中心,广东 东莞 523009;3.长江大学资源与环境学院,湖北 武汉 434023)
为了定量生态相关物种基因组中低丰度的5-甲基-2-脱氧胞苷(5-mdC),建立了一种采用高效液相色谱串联质谱(HPLC-MS/MS)的无标记方法.全基因组DNA甲基化计算公式5-mdC(mole)/(5-mdC(mole)+dC(mole)可以转化为1/(1+dC(mole)/5-mdC(mole)),然后通过HPLC-MS/MS得到DNA样品中5-mdC和dC的物质的量比(dC(mole)/5-mdC(mole))即可得到基因组DNA甲基化值.此外,还对HPLC条件进行了优化使正常核苷和修饰核苷基线分离,消除分析物的交叉干扰.本方法避免了使用昂贵的稳定同位素标记内标,在等度高效液相色谱条件下实现了正常和修饰核苷基线分离,5-mdC和dC的保留时间分别为5.50和3.06min.5-mdC和dC的定量限分别为14.2和19.1pg/mL.日内和日间偏差和准确性(相对误差)都≤10%.该方法测定的小牛胸腺DNA和大型蚤及秀丽线虫的全基因组DNA甲基化值分别为(6.44 ± 0.25)%,(0.097 ± 0.010)%和(0.025 ± 0.002)%.采用HPLC-MS/MS技术建立并验证了一种快速、高精密度和灵敏度的DNA样品全基因组DNA甲基化测定方法,适用于评估环境污染物对生态学相关物种的潜在表观遗传风险.
全基因组DNA甲基化;无标记;电喷雾质谱;表观遗传效应;生态物种
DNA甲基化是表观遗传学的重要内容,涉及DNA的构象和染色体结构的改变,以及DNA和转录因子间相互作用.DNA甲基化在基因表达调节和基因组印记中发挥着重要作用[1].不良健康效应,如致癌发生、生殖异常和糖尿病等都与DNA甲基化图谱的变化密切相关[2-5].已有研究发现,金属和有机污染物可导致环境物种中DNA甲基化状态异常[6].因此,DNA甲基化被认为是一个有前途的生物监测标志物,用于评估环境因素的不良表观遗传效应[7].在一些环境物种中,如海洋生物和大型蚤,这些物种的全基因组中5-甲基-2-脱氧胞苷(5-mdC)的丰度很低[8],对全基因组DNA甲基化精密定量提出了挑战.因此,建立测量低丰度基因组DNA甲基化的方法是表观遗传学在毒害效应评估研究中的先决条件.
到目前为止,现有的DNA甲基化检测方法包括高效液相色谱(HPLC)[9]、气相色谱串联质谱(GC-MS/MS)[10]、液相色谱串联质谱(HPLC-MS/ MS)[11-12]、毛细管电泳[13-14]、免疫定量[15]、LINE1测序[16]和荧光甲基化检测[17].这些方法可以提供具有高准确性和可重复性的全基因组DNA甲基化值,但有存在不足[18].例如,GC-MS/MS具有高灵敏度,但需要用衍生剂对核苷进行繁琐而耗时的衍生[10].荧光法甲基化检测适用于没有参考基因组序列信息的任何物种,而它只能检测CCGG序列内的甲基化状态,这导致基因组中其他CpG位点的信息丢失.目前的大多数方法适用于测定具有高丰度DNA甲基化的样品.因此,需要开发一种高灵敏度、省时简便的方法来评估低丰度全基因组DNA甲基化的生态物种的表观遗传风险.
HPLC-MS/MS技术可以高灵敏度和选择性地检测修饰核苷,近年来已经开发了多种该类方法.然而,大多数方法均缺乏合适的5-mdC和胞苷(dC)的内标来校正质谱信号的偏差.一些研究使用了同位素标记的化合物,如2H4-5-甲基2-脱氧胞苷[12]、Cyt13C15N2[19]和15N3-2'-脱氧胞苷[20],作为内标或替代内标.然而,这些同位素标记的化学品需要定制合成,而且往往价格昂贵.因此,一些研究人员使用DNA中5-mdC与dG的物质的量比作为全基因组DNA甲基化值[21].此外,修饰核苷与正常核苷的共同洗脱会干扰目标物的电离效果,特别是那些低含量的目标物[22].因此,优良的色谱条件,将5-mdC与正常核苷进行基线分离,对于低丰度5-mdC的定量至关重要.正常核苷和修饰核苷的梯度分离需要更多的时间对色谱柱进行修饰.选择合适的分离和检测条件以达到所需的选择性和灵敏度非常重要.
因此,本文开发一种新的低成本和高准确性的方法,利用无标记HPLC-MS/MS对生态相关物种的DNA样品中的全基因组DNA甲基化进行定量分析.研究通过HPLC-MS/MS测定DNA样品中5-mdC (mole)/dC(mole)的摩尔比,然后通过Methylation%= 1/(1+dC(mole)/5-mdC(mole))×100%的公式计算出全基因组DNA甲基化值,以期与现有方法相比.
1 材料与方法
1.1 材料和试剂
HPLC级乙腈、甲醇和甲酸(德国默克公司).乙酸铵和碳酸氢铵(美国杜邦公司).核酸酶P1(来自柠檬青霉菌)、磷酸二酯酶I(来自Crotalus adamanteus)和小牛肠道碱性磷酸酶与10×孵化缓冲液(500mM乙酸钾;200mM乙酸三钠;100mM乙酸镁;1mg/mL BSA)(美国Sigma公司).小牛胸腺DNA(北京鼎国生物技术有限公司).5-mdC、2-脱氧腺苷(dA)、2-脱氧鸟苷(dG)、2-脱氧胞苷(dC)、A、G、C、T和U(美国Amresco公司).去离子水采用纯水机制备(美国Millipore公司).其他化学试剂均为分析纯.
两种21碱基DNA寡核苷酸,其序列为5-ACT CAC TCA CGC AGT GAG AGA-3(7A:6C:5G:3T)和5-A(5mC)T (5mC)A(5mC) T(5mC)A(5mC) AGT GAG AGA GAG-3(7A:6mC:5G:3T)从上海生工生物技术有限公司采购.
5-mdC和dC溶于水制备1mg/mL浓度的储备液.5-mdC(1、10、100和1000ng/mL)和dC(50、100、500和1000μg/mL)的标准工作溶液通过去离子水进一步稀释储备液制备.
1.2 DNA的酶促水解
基因组DNA和寡核苷酸酶解成核苷的分析方法如前所述[21].将约1μg DNA溶解在3μL去离子水中,然后100℃下孵育3min,并立即在冰中冷却以变性.加入1μL(2U)核酸酶P1和1μL 0.1mol/L乙酸铵(CH3COONH4)(pH 5.3)后,将混合物在45℃孵育3h.继续加入1μL(0.002U)蛇毒磷酸二酯酶I和1μL 1M碳酸氢铵(NH4HCO3),混合物在37℃孵育2h.5μL (0.5U)碱性磷酸酶,1μL 10×孵育缓冲液和1.5μL去离子水,37℃孵育1h.最终的DNA水解物在Microcon 10kDa离心过滤装置(Microcon YM-10; Millipore, MW cut-off 10000)上以12000×g4℃离心30min进行纯化.
1.3 HPLC-MS/MS测定
仪器分析采用岛津LC20A系列HPLC系统(日本岛津公司)(配备真空脱气机、自动进样器、四级泵和紫外检测器)和TurbolonSpray源与API 4000Q-trap质谱仪(美国Applied Biosystems).色谱分离使用Hypersil-HyPURITY C18反相柱(150mm× 2.1mm, 5μm 粒径)(美国Thermo Fisher 公司),由C18保护柱(20mm×2.1mm, 5μm粒径)(美国Phenomenex公司)保护.流动相为甲酸、甲醇和水(0.1:5:95,//),流速为0.3mL/min.样品进样量为10μL.
使用电喷雾电离(ESI)三重四极杆获得5-mdC和dC的MS/MS特征.溶解在去离子水中的20ng/mL的分析物通过注射泵注入ESI探针,流速为10μL/min,以优化采集参数.质谱仪在ESI+MS/MS模式下运行.优化后参数为:喷雾电压4000V;涡轮气体温度430℃;解聚电位45V;入口电压7V;碰撞能量18V;碰撞池出口电位8V;气体1(设置32)和气体2(设置55);气帘气(设置20).5-mdC和dC的多反应监测(MRM)离子对分别是242.2>126.1和228.0>112.2.此外,C、dA、A、G、dG、T和U的离子对为/244.1>112.2,/252.3>135.9,/268. 1>136.3,/284.1>152.3,/268.1>152.3,/243.3>127.2,/245.3>112.9.每个离子对驻留时间为200ms.四极杆Q1和Q3分辨率被设定为单位质量分辨率. Analyst®软件1.3版(Applied Biosystems)用于数据采集和处理.
1.4 环境应用
实验是按照经济合作与发展组织(OECD)的标准进行的[23].为了更好地观察各种参数的变化,将大型蚤暴露在NaAsO2(0–10μmol/L)中,每个浓度设置5个平行样品(包括对照组).温度控制在(25±1)°C,光照强度为(1500±100)lx,光照与黑暗时间循环为16h:8h.采用半静态试验方法,用新鲜的网格喂养大型蚤,每天更换一次培养液.
野生型N2秀丽线虫采用固体生长培养基,20℃的孵化器中黑暗中培养(除了操作过程中)[24].将L4秀丽线虫转移到K培养基中,并分成24孔板,每孔约50个秀丽线虫.在每个孔中加入不同浓度的NaAsO2溶液(=5).在空白对照组中加入等量K培养基.在暴露期间,每天重复喂毒计划.
暴露后,使用天根基因组DNA提取试剂盒(北京,中国)从大型蚤和秀丽线虫中分离出基因组DNA.使用Nanodrop测定基因组DNA的浓度和纯度,用去离子水将DNA样品调整到500μg/mL的最终浓度,-80℃保存.
1.5 统计学分析
数据以平均值±标准差表示. 统计分析采用SPSS19,统计学显著性水平设定为<0.05.
2 结果和讨论
2.1 公式推导和测量误差分析
全基因组DNA甲基化值被定义为5-mdC与总dC的摩尔比,通过公式(1)计算.
甲基化率= 5-mdC(mole)/(5-mdC(mole) +
dC(mole)) × 100% (1)
式中:5-mdC(mole)是5-mdC核苷的物质的量量; dC (mole)是DNA样品中dC核苷的物质的量量.
根据式1提出一个变型方程,如式(2)所示,用于计算全基因组DNA甲基化率.
甲基化率=1/(1+dC(mole)/5-mdC(mole))×100% (2)
因此,只要通过HPLC-MS/MS准确地确定DNA样品中5-mdC(mole)/dC(mole)的摩尔比值,即可利用公式(2)获得全基因组DNA甲基化值.该方法避免了对DNA样品中5-mdC和dC含量的分别独立测量.减少了分析误差,提高了准确性.
2.2 HPLC-MS/MS条件的优化
合适的流动相和固定相是实现正常和修饰核苷基线分离的先决条件.首先以甲醇和水(30:70,/)的混合物作为流动相进行分离测试, 结果显示正常核苷和修饰核苷的保留时间很短, 且存在部分分析物的共洗脱.流动相中水的比例增加到80%(/),尽管保留时间增加, C和U与目标分析物共同洗脱.因此,流动相的水溶液比例进一步增加到95%(/),尽管所有的核苷酸都相互分离,但dC的峰不对称.为了纠正这种情况,流动相中加入0.1%(/)的甲酸.结果表明在0.3mL/min的流速下,所有目标物质都实现了基线分离,典型的色谱峰如图1所示.正常和修饰核苷间实现基线分离,5-mdC和dC保留时间分别为5.50和3.06min(图2).可见,在优化条件下5-mdC、dC和其他分析物的测定不会交叉干扰.
已经有文献报道正常和修饰核苷的HPLC分离方法[25-26].其中,Song等[25]和Magana等[26]讨论了DNA样品被RNA污染时的色谱性能.Song等[25]提出的总离子色谱图中,5-mdC和U之间的峰有重叠现象.Magana等[26]开发的分离方法实现了所有核苷酸的基线分离,但在色谱条件中使用了三种流动相,其中一个是磷酸盐缓冲液.
此外,流动相的梯度很复杂,需要更多的时间实现柱子的重新平衡.Zhang等[20]使用2.5mmol/L甲酸铵和乙腈(:=7:93)作为流动相.该方法线性关系好,但由于使用不易挥发的盐溶液作为流动相,容易堵塞LC-MS/MS管路和色谱柱.与以往报道的方法相比,本文提供的色谱分析时间短,采用含有0.1%甲酸的水和甲醇作为流动相有利于ESI电离.本研究方法避免了RNA和其他物质的污染,且使用方便,节省时间.
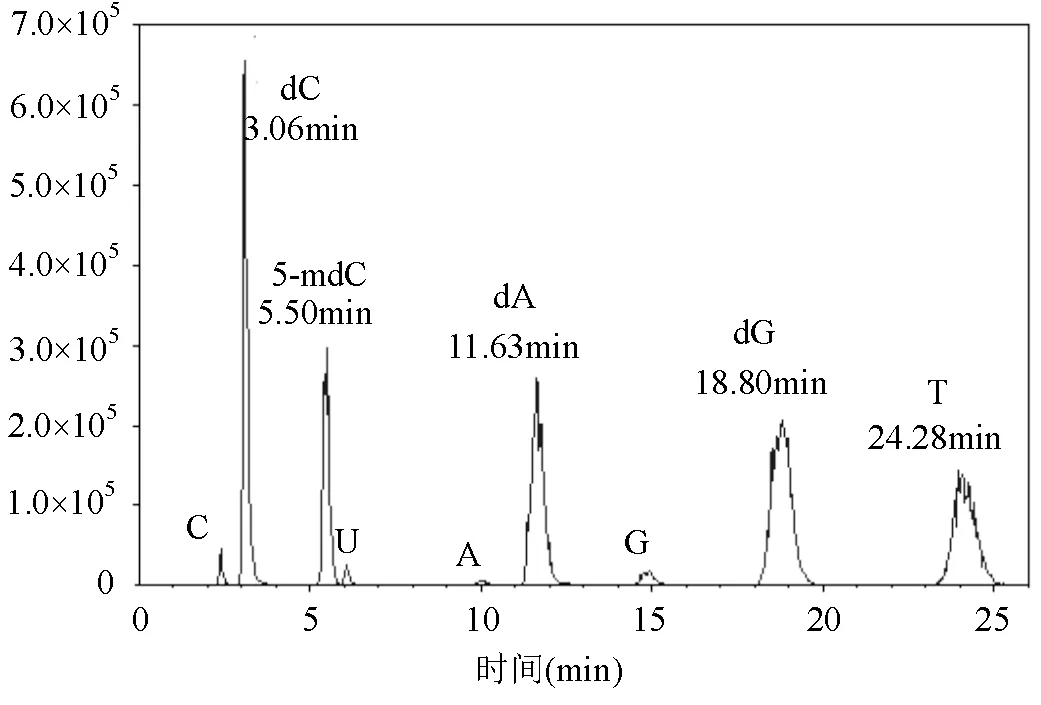
图1 正常核苷和修饰核苷的典型总离子色谱
DNA提取过程中没有加入RNase以消除RNA
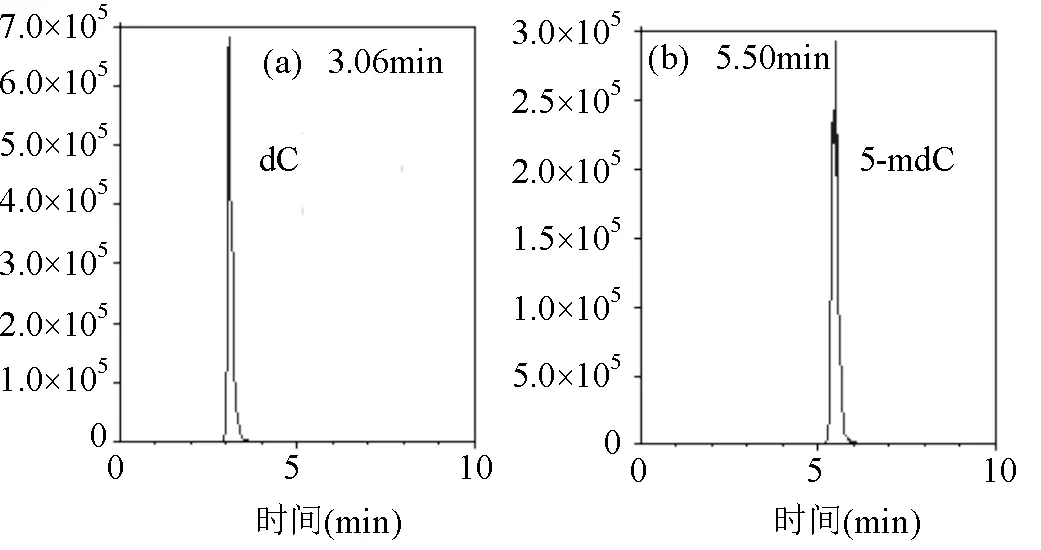
图2 水解基因组DNA样品产生的典型MRM色谱
2.3 校准曲线和灵敏度
将不同浓度的5-mdC(0.1,0.5,2.5,5,10,15,20,25, 30,35,40,45,50和55ng/mL)加入500ng/mL dC中,制备新鲜校准标准溶液,5-mdC/dC的物质的量比范围为0.02%~10.31%.通过绘制5-mdC与dC的MRM/MS信号比和5-mdC与dC的物质的量比来获得校准曲线.该方法的线性方程为=3.482,连续5d的测试均获得了良好线性关系,相关系数2= 0.9998(图3).结果表明所开发的方法在浓度范围内具有良好线性关系.
5-mdC和dC(/=3)的检测限(LOD)分别为3.5和7.4pg/mL.5-mdC和dC(/=10)的定量限(LOQ)分别为14.2和19.1pg/mL.
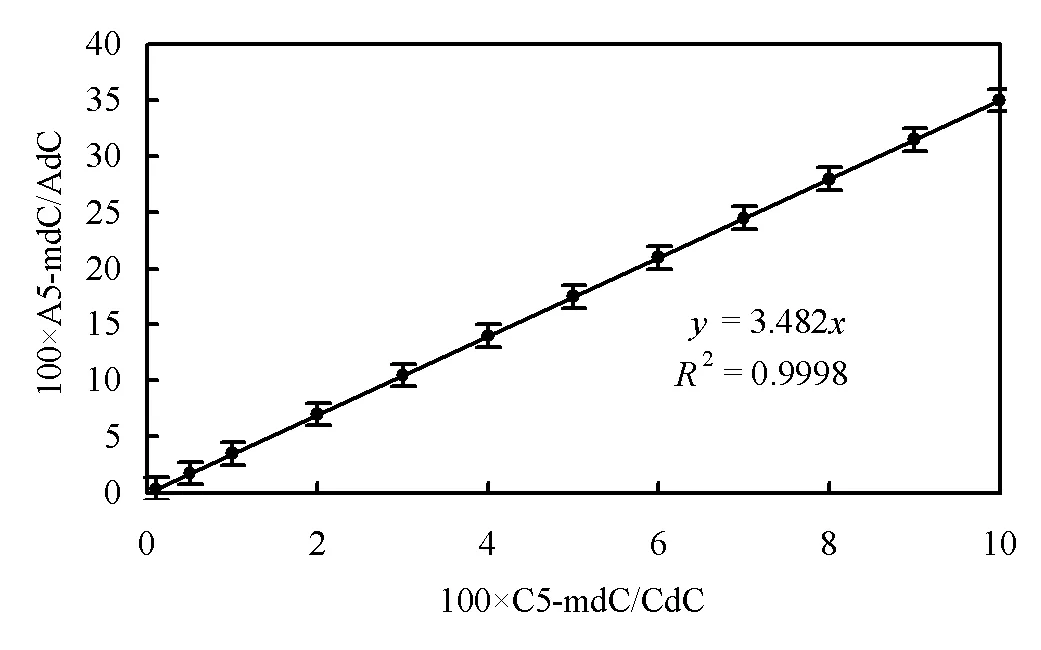
图3 DNA甲基化校准曲线
2.4 基质效应评估
将两个目标物溶解在水和酶制剂缓冲液中以评估酶解缓冲液对5-mdC和dC MS/MS信号的影响.酶解缓冲液含有0.1mol/L CH3COONH4(pH 5.3)、1mol/L NH4HCO3、10×孵化缓冲液和水,体积比为1 : 1 : 1 : 7,5-mdC的最终浓度为0.5,5,50和100ng/mL, dC的最终浓度为5,10,50和100ng/mL.采用优化的HPLC-MS/MS条件对其进行分析,并通过比较加在酶解缓冲液中的分析物与溶解在水中的分析物的峰面积来评估基质效应.酶解缓冲液对5-mdC和dC的MS/MS信号没有明显的基质效应(数据未提供).
2.5 准确度和精密度
制备质量控制(QC)样品评估方法的准确性和精密性,QC样品5-mdC/dC物质的量比为0.5%、1%、5%和10%.日内精密度通过在同一天的不同时间测试QC样品来评估(=3).日间精度通过连续五d每天3次测试质控样品来评估(=15).日内精密度为98.4%~107.5%(=3),而日间精密度为97.2%~ 109.6%(表1).在所有样品中,使用所开发的方法得到的变异系数<10%,表明使用本方法测定DNA甲基化值是可靠的.
使用两种21碱基DNA寡核苷酸,其序列为5-ACT CAC TCA CGC AGT GAG AGA-3(7A:6C: 5G:3T)和5-A(5mC)T (5mC)A(5mC) T(5mC)A(5mC) AGT GAG AGA GAG-3(7A:6mC:5G:3T)制备校准溶液,分别含有0,5,2.5,5和10%的DNA甲基化率.寡核苷酸消化后,样品用Microcon超滤装置(Microcon YM-10;Millipore,截留分子量10000)进行超滤.滤液用HPLC-MS/MS进行分析,以估计本方法的准确性和精密度.日间测量的分析精度为94.0%~101.4%(=5),而日间测量的分析精度为92.0%~102.0%(=5).标准偏差<10%,表明该方法具有良好的准确性(表2).
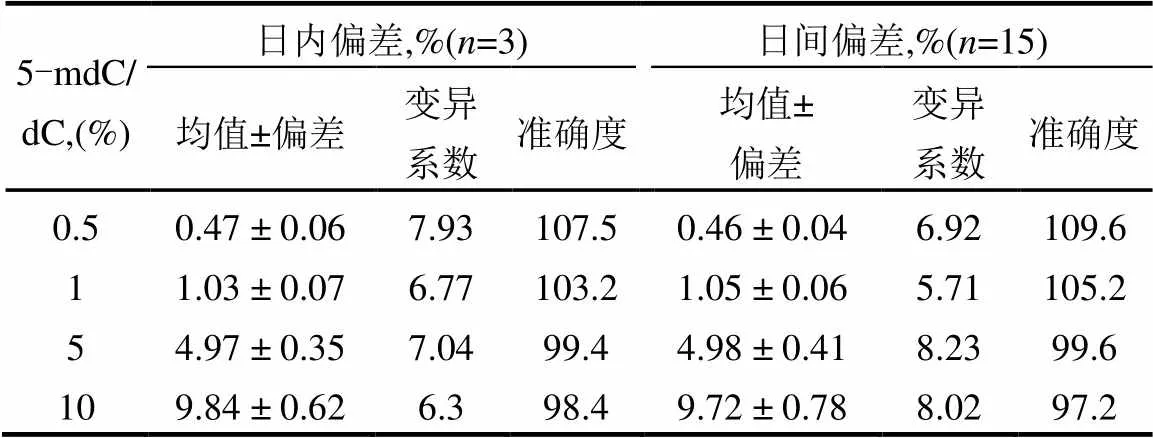
表1 HPLC-MS/MS测定流动相中添加的5-mdC和dC百分比的准确度和精密度
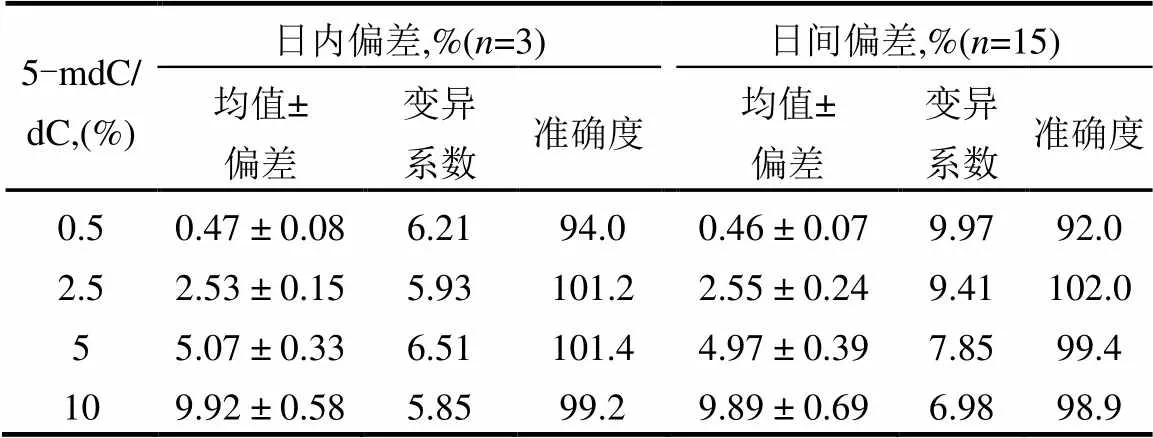
表2 HPLC-MS/MS测定合成寡核苷酸混合物中5-mdC和dC百分比的准确性和精密度
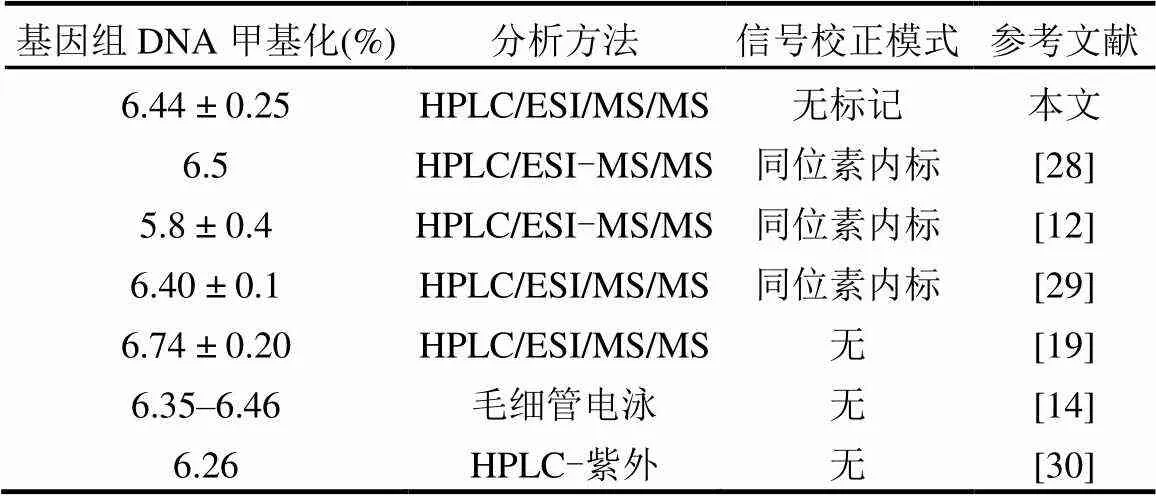
表3 HPLC-MS/MS测定小牛胸腺DNA的甲基化值与文献值比较
通过检测小牛胸腺DNA基因组DNA甲基化值验证本方法.根据Zinellu等[27]方法水解小牛胸腺DNA.采用本方法测得小牛胸腺DNA基因组DNA甲基化值为(6.44±0.25)%(=5),这与其他报道的数据一致(表3).为了探索DNA量对全基因组DNA甲基化检测的影响,评估了25,50,100,250和500ng采用小牛胸腺DNA的基因组DNA甲基化值,结果分别为(6.43±0.21)%、(6.47±0.19)%、(6.46± 0.13)%、(6.41±0.25)%和(6.49±0.27)%.可见,本方法在亚微克DNA量时可以提供准确的基因组DNA甲基化值(图4).结果表明,本方法具有良好的准确性和良好的方法可替代性前景.
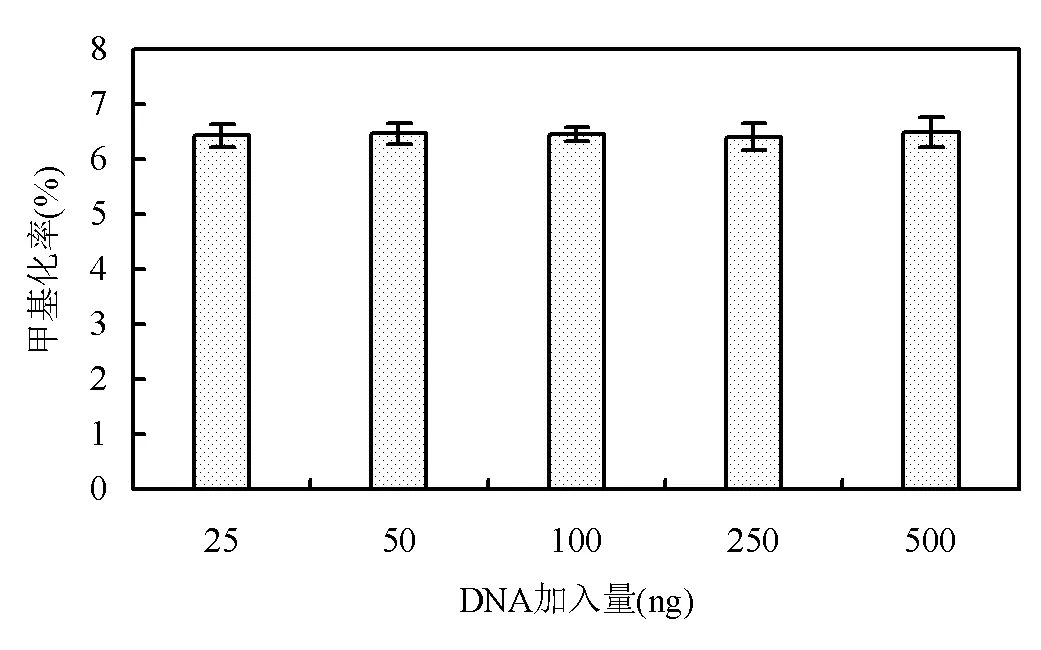
图4 不同数量小牛胸腺DNA样品中的全基因组DNA甲基化(n = 3)
2.6 生物样品分析
大型蚤和秀丽线虫是DNA甲基化程度低的物种.采用本方法检测了大型蚤和秀丽线虫的全基因组DNA甲基化值,分别为(0.097±0.010)%、(0.025± 0.002)%.与文献中大型蚤(0.146±0.0126)%和秀丽线虫(0.035±0.003)%的数据要低(~30%)[8,31].由于没有标准物质来确证这种差异的来源,有可能与大型蚤和秀丽线虫的培养环境有关.
环境中的一些有机污染物和金属可诱发全基因组DNA甲基化.本文分析了NaAsO2对大型蚤和秀丽线虫基因组DNA甲基化的影响(图5).暴露于NaAsO2会诱发大型蚤和秀丽线虫的DNA去甲基化.大型蚤的全基因组DNA甲基化值为0.071%~ 0.104%,甲基化值随着NaAsO2浓度的增加而明显下降.同样,秀丽线虫的全基因组DNA甲基化值随着NaAsO2浓度的增加而下降,其含量为0.020%~ 0.027%,低于大型蚤的水平.总之,本研究提供了一个节省成本,易于使用且可以为低丰度DNA甲基化生物物种提供高精密度结果的方法,可以用于评估环境污染物对生态物种表观遗传毒性风险.
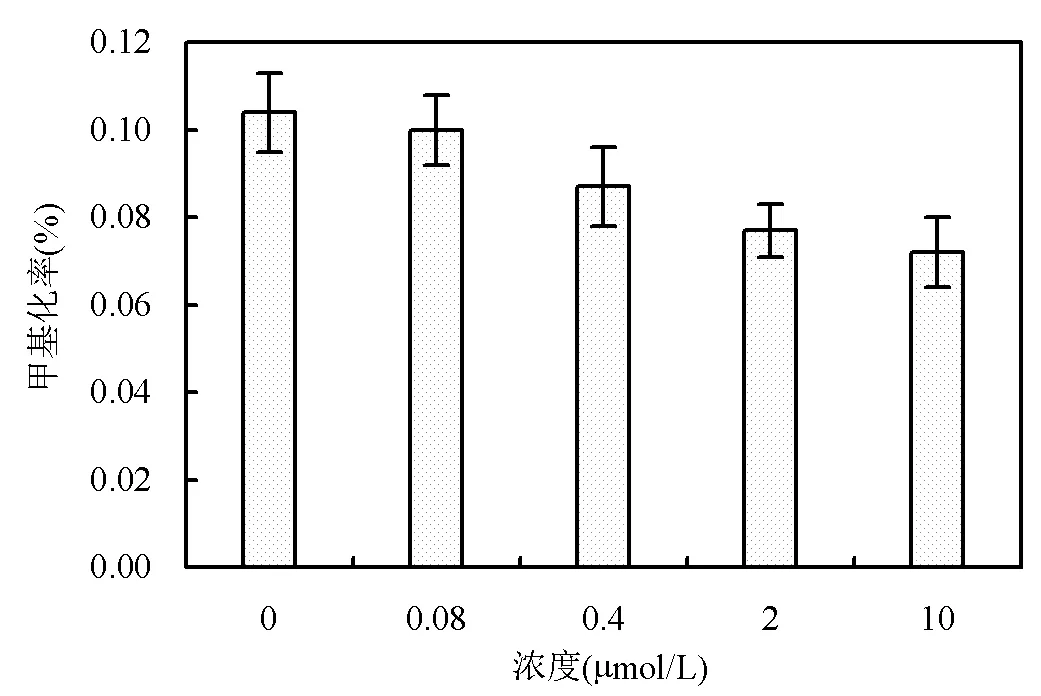
3 结语
本文提出了一种用于全基因组DNA甲基化检测的无标记且准确的HPLC-MS/MS方法.该方法通过HPLC-MS/MS检测样品中5-mdC(mole)/dC (mole)的物质的量比,然后利用甲基化率公式计算出全基因组DNA甲基化值.5-mdC和dC的LOQ分别为14.2和19.1pg/mL.5-mdC和dC的日内和日间相对标准偏差均小于10%.通过检测大型蚤和秀丽线虫的全基因组DNA甲基化值,验证了本方法的可行性.
[1] Schulz H, Ruppert A K, Herms S, et al. Genome-wide mapping of genetic determinants influencing DNA methylation and gene expression in human hippocampus [J]. Nature Communications, 2017, 8:1511–1521.
[2] Hu J, Yang Y, Lv X, et al. Dichlorodiphenyltrichloroethane metabolites inhibit DNMT1activity which confers methylation-specific modulation of the sex determination pathway [J]. Environmental Pollution, 2021,279:116828.
[3] Yang Z, Qi W, Sun L, et al. DNA methylation analysis of selected genes for the detection of early-stage lung cancer using circulating cell-free DNA [J]. Advances in Clinical and Experimental Medicine, 2019,28:355–360.
[4] 周新文,朱国念,Jilisa Mwalilino, et al. Cu、Zn、Pb、Cd及其混合重金属离子对鲫鱼()DNA甲基化水平的影响[J]. 中国环境科学, 2001,21(6):549–552.
Zhou Xinwen, Zhu guonian, Jilisa Mwalilino, et al. Influence of Cu、Zn、Pb、Cd and their heavy metalion mixture on the DNA methylation level of fish () [J]., 2001,21(6):549-552.
[5] 吴思寒,吴雨濛,王双杰,等.全氟辛烷磺酰胺在小麦和蚯蚓中的富集与转化[J]. 中国环境科学, 2021,41(5):2434–2440.
Wu S H, Wu Y M, Wanf S J, et al. Bioaccumulation and biotransformation of perfluorooctane sulfonamide in wheat and earthworms [J]. China Environmental Science, 2021,41(5):2434–2440.
[6] Hu J, Liu J, Li J, et al. Metal contamination, bioaccumulation, ROS generation, and epigenotoxicity influences on zebrafish exposed to river water polluted by mining activities [J]. Journal of Hazardous Materials, 2021,405:124150.
[7] De Paoli-Iseppi R, Deagle B E, Polanowski A M, et al. Age estimation in a long-lived seabird (Ardenna tenuirostris) using DNA methylation-based biomarkers [J]. Molecular Ecology Resources, 2019,19:411–425.
[8] Hearn J, Plenderleith F, Little TJ. DNA methylation differs extensively between strains of the same geographical origin and changes with age in Daphnia magna [J]. Epigenetics Chromatin, 2021,14:4.
[9] Rastegar L, Mighani H, Ghassempour A. A comparison and column selection of hydrophilic interaction liquid chromatography and reversed-phase high-performance liquid chromatography for detection of DNA methylation [J]. Analytical Biochemistry, 2018,557:123–130.
[10] Tang Y, Gao X D, Wang Y, et al. Widespread existence of cytosine methylation in yeast DNA measured by gas chromatography/mass spectrometry [J]. Analytical Chemistry, 2012,84:7249.
[11] Nakagawa T, Wakui M, Hayashida T, et al. Intensive optimization and evaluation of global DNA methylation quantification using LC- MS/MS [J]. Analytical and Bioanalytical Chemistry, 2019,411:7221– 7231.
[12] Hu C W, Lee H, Chen J L, et al. Optimization of global DNA methylation measurement by LC-MS/MS and its application in lung cancer patients [J]. Analytical and Bioanalytical Chemistry, 2013, 405:8859–8869.
[13] Zinellu A, Sotgiu E, Sotgia S, et al. A capillary electrophoresis UV detection-based method for global genomic DNA methylation Assessment in human whole blood [J]. Methods in Molecular Biology, 2019,1972:213–219.
[14] Zinellu A, Sotgia S, De Murtas V, et al. Evaluation of methylation degree from formalin-fixed paraffin-embedded DNA extract by field- amplified sample injection capillary electrophoresis with UV detection [J]. Analytical and Bioanalytical Chemistry, 2011,399:1181–1186.
[15] Bhattacharjee R, Moriam S, Nguyen NT, et al. A bisulfite treatment and PCR-free global DNA methylation detection method using electrochemical enzymatic signal engagement [J]. Biosensors and Bioelectronics, 2019,126:102–107.
[16] Childebayeva A, Jones TR, Goodrich JM, et al. LINE-1and EPAS1DNA methylation associations with high-altitude exposure [J]. Epigenetics, 2019,14(1):1–15.
[17] Luttropp K, Sjöholm L K, Ekström T J. Global analysis of DNA 5-methylcytosine using the luminometric methylation assay, LUMA [J]. Methods in Molecular Biology, 2015,1315:209.
[18] Chowdhury B, Cho I H, Irudayaraj J. Technical advances in global DNA methylation analysis in human cancers [J]. Journal of Biological Engineering, 2017,11:10.
[19] Hu J J, Zhang W B, Ma H M, et al. Simultaneous determination of 8-hydroxy-2'-deoxyguanosine and 5-methyl-2'-deoxycytidine in DNA sample by high performance liquid chromatography/positive electrospray ionization tandem mass spectrometry [J]. Journal of Chromatography B, 2010,878:2765–2769.
[20] Zhang J J, Zhang L, Zhou K, et al. Analysis of global DNA methylation by hydrophilic interaction ultra high-pressure liquid chromatography tandem mass spectrometry [J]. Analytical Biochemistry, 2011,413:164–170.
[21] Hu J, Liu J, Lv X, et al. Assessment of epigenotoxic profiles of Dongjiang River: A comprehensive of chemical analysis, in vitro bioassay and in silico approach [J]. Environmental Pollution, 2021,282: 116961.
[22] Sun Y, Stransky S, Aguilan J, et al. High throughput and low bias DNA methylation and hydroxymethylation analysis by direct injection mass spectrometry [J]. Analytica Chimica Acta, 2021,1180:338880.
[23] 杨晓凡,陆光华,刘建超,等.环境相关浓度下的药物对大型蚤的多代慢性毒性[J]. 中国环境科学, 2013,33(3):538–545.
Yang X F, Lu G H, Liu J C, et al. Multigenerational chronic effects of pharmaceuticals onat environmentally relevant concentrations [J]. China Environmental Science, 2013,33(3):538-545.
[24] 张绪超,陈 懿,胡 蝶,等.生物炭中持久性自由基对秀丽隐杆线虫的毒性[J]. 中国环境科学, 2019,39(6):2644–2651.
Chen Y, Hu D, Zhao L, et al. Neurotoxic effect of environmental persistent free radicals in rice biochar to Caenorhabditis elegans [J]. China Environmental Science, 2019,39(6):2644–2651.
[25] Song K, James S R, Kazim L, et al. Specific method for the determination of genomic DNA methylation by liquid chromatography-electrospray ionization tandem mass spectrometry [J]. Analytical Chemistry, 2005,77:504–510.
[26] Magaña A A, Wrobel K, Caudillo Y A, et al. High-performance liquid chromatography determination of 5-methyl-2'-deoxycytidine, 2'- deoxycytidine, and other deoxynucleosides and nucleosides in DNA digest [J]. Analytical Biochemistry, 2008,374:378–385.
[27] Zinellu A, Sotgiu E, Assaretti S, et al. Evaluation of global genomic DNA methylation in human whole blood by capillary electrophoresis UV detection [J]. Journal of Analytical Methods in Chemistry, 2017,2017:4065892.
[28] Kok R M, Smith D E C, Barto R, et al. Global DNA methylation measured by liquid chromatography-tandem mass spectrometry: analytical technique, reference values and determinants in healthy subjects [J]. Clinical Chemistry and Laboratory Medicine, 2007,45: 903–911.
[29] Rocha M S, Castro R, Rivera I, et al. Global DNA methylation: comparison of enzymatic- and non-enzymatic-based methods [J]. Clinical Chemistry and Laboratory Medicine, 2010,48:1793–1798.
[30] Jatinderpal S, Balvinder K, Christine A, et al. Determination of 5-methyl-2’-deoxycytidine in genomic DNA using high performance liquid chromatography-ultraviolet detection [J]. Journal of Chromatography B, 2009,877:1957–1961.
[31] Thaulow J, Song Y, Lindeman LC, et al. Epigenetic, transcriptional and phenotypic responses in Daphnia magna exposed to low-level ionizing radiation [J]. Environmental Research, 2020,190:109930.
HPLC-MS-MS method for low level DNA methylation value determination in ecological species.
HU Jun-jie1, WU Yi-chong1, CHEN Gui-lian1, GAO Yuan-yuan2, ZHAO Tong3, YU Guang1, LAN Shan-hong1, Lü Xiao-mei1*
(1.School of Environment and Civil Engineering, Dongguan University of Technology, Dongguan 523808, China;2.Environmental Technology Center of Dongguan, Dongguan 523009, China;3.College of Resources and Environment, Yangtzeu University, Wuhan 434023, China)., 2022,42(7):3385~3391
DNA methylation patterns which associated with normal tissue development and disease initiation could be affected by environmental contaminants and related toxicity. For the requirement of quantification low abundant of 5-methyl-2’-deoxycytidine (5-mdC) in the genome of ecologically relevant species, a label-free and accurate method using high performance liquid chromatography tandem mass spectrometry (HPLC-MS/MS) was developed and validated. The global DNA methylation calculation formula of 5-mdC(mole)/(5-mdC(mole)+dC(mole)) could be transformed as 1/(1+dC(mole)/5-mdC(mole)), then the molar ratio of 5-mdC and dC (dC(mole)/5-mdC(mole)) in DNA samples was obtained by HPLC-MS/MS. The important features of the provided method are as follows: (1) avoiding the usage of expensive stable isotope labeled internal standards; (2) isocratic separating normal and modified nucleosides and eliminating cross-interferences of analytes; (3) providing data with high accuracy even detecting low levels of methylated DNA samples. The limits of quantification for 5-mdC and dC were 40 and 60pg/mL, respectively. The intra-day and inter-day relative standard deviations (£10%) and accuracy (relative errors) were satisfactory. The results obtained from the analysis of calf thymus DNA and genomic DNA from Daphnia magna and elegans demonstrated the feasibility of the new developed method in evaluation of the potential epigenetic risk of environmental pollutions on ecologically relevant species.
global DNA methylation;label-free;electrospray tandem mass spectrometry;epigenetic risk;ecological species
X172
A
1000-6923(2022)07-3385-07
胡俊杰(1984-),男,湖北武汉人,特聘副教授,博士,主要从事环境毒理学和环境监测方面研究.发表论文10余篇.
2022-12-20
广东省自然科学基金资助项目(2019A1515110750);东莞市社会科技发展重点项目(20211800905532,20211800904652,2020507140150)
* 责任作者, 讲师, 2017158@dgut.edu.cn
