生防菌NCD-2 菌株定量检测体系的建立及其在棉花根际定植检测中的应用
2022-07-07苏星苏振贺宣立锋李社增王培培郭庆港马平
苏星,苏振贺,宣立锋,李社增,王培培,郭庆港,马平*
(1.河北农业大学植物保护学院,河北 保定 071000;2.河北省农林科学院植物保护研究所/ 河北省农业有害生物综合治理中心/ 农业农村部华北北部作物有害生物综合治理重点实验室,河北保定 071000;3.河北省农林科学院石家庄果树研究所,石家庄 050061)
由大丽轮枝菌(Verticillium dahliae)、尖孢镰刀菌萎蔫专化型 (Fusarium oxysporumf. sp.vasinfectum)和立枯丝核菌(Rhizoctonia solani)引起的棉花(Gossypium)土传病害给棉花生产造成严重的经济损失。 近年来,很多研究报道表明,利用微生物杀菌剂是防治作物土传病害行之有效,且环境友好的措施之一[1-3]。 以枯草芽孢杆菌(Bacillus subtilis)为代表的生防细菌之所以能够在作物土传病害防控中发挥作用,主要是因为这类生防菌能够有效定植在寄主的根际土壤中,通过与病原菌进行营养竞争或空间位点竞争而降低病原菌的侵染,同时产生抑菌活性物质有效抑制病原菌的生长[4-5]。 监测外源生防菌在植物根际的定植情况,对于生防菌的合理、高效利用具有重要的指导价值。
土壤微生物常用的定量检测方法主要包括菌落计数法、聚合酶链式反应(polymerase chain reaction,PCR)法、免疫测定法、实时PCR 法等[6-7]。土壤中存在庞大的微生物群体,多种微生物具有相似的培养特性,因此,传统的菌落计数法难以准确反映根际土壤中靶标微生物的数量。 免疫测定法适用于快速鉴定靶标微生物的存在,但无法对菌体数量进行定量分析[8]。 随着PCR 技术的普及,PCR 技术已被广泛应用于土壤中微生物的鉴定和检测[9]。 常规PCR 为在菌株水平上鉴定微生物提供了1 种准确的方法,但无法准确定量菌体数量[10-12]。 随着实时PCR 技术的快速发展,该技术已被广泛用于土壤微生物的定量检测,具有省时、 省力、 高通量并能准确定量微生物的优点[13-14]。王川等[15]建立了灵敏、高效的实时PCR 快速检测体系, 在盆栽条件下检测了马铃薯(Solanum tuberosum)黑痣病的生防菌QHZ11 在马铃薯根际土壤中的定植情况,结果表明,在接种后60 d 时QHZ11 菌株在土壤中定植量达到峰值。 Sa 等[16]利用实时PCR 技术对枯草芽孢杆菌菌株N6-34 进行了定量检测, 发现其在杨树(Populus)植株及根际土壤中的定植量呈先增加后稳定再逐渐降低的趋势。
枯草芽孢杆菌NCD-2 菌株能有效防治棉花黄萎病和棉花立枯病,以该菌株为主要活性成分的微生物杀菌剂“10 亿活芽孢/ 克枯草芽孢杆菌可湿性粉剂” 已于2006 年获得农药注册登记[17]。前期研究表明, 生防菌NCD-2 菌株对棉花立枯病的防治效果和NCD-2 菌株的生物膜产生能力呈正相关[18]。目前已获得NCD-2 菌株全基因组序列(GenBank ID:CP023755.1)。 本研究设计了NCD-2 菌株的特异性引物, 进一步构建了NCD-2 菌株的实时PCR 定量检测技术, 并定量检测了NCD-2 菌株在棉花根际的定植动态,同时也检测了NCD-2 菌株在防治棉花立枯病时的根际定植数量, 以期为NCD-2 菌株在寄主根际的定植情况提供直接证据, 为高效使用NCD-2菌株提供科学指导。
1 材料与方法
1.1 供试培养基
Luria-Bertani(LB)液体培养基:5 g·L-1酵母提取物、10 g·L-1胰蛋白胨、5 g·L-1NaCl, 调pH至7.0。
LB 固体培养基:5 g·L-1酵母提取物、10 g·L-1胰蛋白胨、5 g·L-1NaCl,15 g·L-1琼脂,调pH至7.0。
马铃薯葡萄糖琼脂培养基 (potato dextrose agar,PDA):200 g·L-1马铃薯(浸出液)、20 g·L-1葡萄糖、15 g·L-1琼脂。
立枯丝核菌选择性培养基:K2HPO41 g、MgSO4·7H2O 0.5 g、KCl 0.5 g、FeSO4·7H2O 10 mg、NaNO20.2 g、氯霉素0.05 g、琼脂20 g、蒸馏水1 000 mL。 制备完成后于121 ℃灭菌20 min,在培养基冷却到50~55 ℃时加入0.1 g·L-1链霉素、0.035 g·L-1孟加拉红、0.08 g·L-1甲霜灵和0.007 g·L-1咪鲜胺。
1.2 供试菌株与培养条件
本研究所用细菌菌株见表1 和表2。 细菌菌株于-80 ℃长期保存,使用前在LB 培养基上活化。 立枯丝核菌菌株AG-4 由河北省农林科学院植物保护研究所植物病害生物防治实验室保存,使用前在PDA 培养基上活化。
1.3 DNA 提取
利用天根生化科技(北京)有限公司细菌DNA 提取试剂盒进行细菌DNA 的提取, 利用FastDNA Spin Kit for Soil(MP Biomedicals,LLC,美国)提取土壤总DNA,具体提取步骤按照说明书操作。 利用NanoDrop 2000(Nano Drop Technologies,美国)测定所提取DNA 的质量浓度。
1.4 NCD-2 菌株实时PCR 检测体系中特异性引物的设计与验证
从NCBI(http://ncbi.nlm.nih.gov)数据库中下载11 株芽孢杆菌标准菌株B. subtilis168、B. subtilisBSn5、B. subtilisRO-NN-1、B. subtilissubsp.spizizeniiW23、B. subtilissub sp.spizizeniiTU-B-10、B.velezensisFZB42、B.amyloliquefaciensDSM7、B. licheniformisATCC14580、B. pumilusSAFR-032、B. atrophaeus1942 及B. thuringiensisATCC10792 的基因组序列, 利用BLAST+ 软件将NCD-2 菌株的全基因组序列与标准菌株的全基因组序列进行序列比对, 获得NCD-2 菌株的特异性基因序列。 根据特异性基因序列, 利用Primer 5.0 软件设计针对NCD-2 菌株的特异性引物。 在NCBI 网站上初步分析该引物的特异性, 然后利用普通PCR 技术优化引物的扩增条件, 并利用22 株芽孢杆菌野生型菌株进行引物特异性检测。
1.5 NCD-2 菌株实时PCR 体系的特异性检验
利用实时PCR 技术验证NCD-2 菌株引物的特异性。 以NCD-2 菌株DNA 为阳性对照,以20株芽孢杆菌参考菌株DNA(表1)和22 株芽孢杆菌野生型菌株DNA(表2)为阴性对照,以无菌水为空白对照, 同时检测土壤总DNA 以及土壤中加入NCD-2 菌株后所提取的DNA 的扩增结果。实时PCR 所用仪器为ABI Step OneTM(Applied Biosystems Inc,美国),反应体系25 μL,其中包含SYBR Green real-time PCR Mix(北京全式金生物技术股份有限公司)10 μL,上下游引物(10 μmol·L-1)各2 μL,模板DNA 约1 ng,用双蒸水补足至25 μL。 反应条件:95 ℃30 s;95 ℃10 s,62 ℃15 s,72 ℃10 s,40 个循环; 以0.1 ℃·s-1的速度从65 ℃递增到95 ℃,在每一循环的退火阶段采集荧光,实时监测荧光信号的变化,得出扩增产物的熔解曲线。

表1 本研究所用标准型芽孢杆菌菌株Table 1 Standard Bacillus strains used in this study
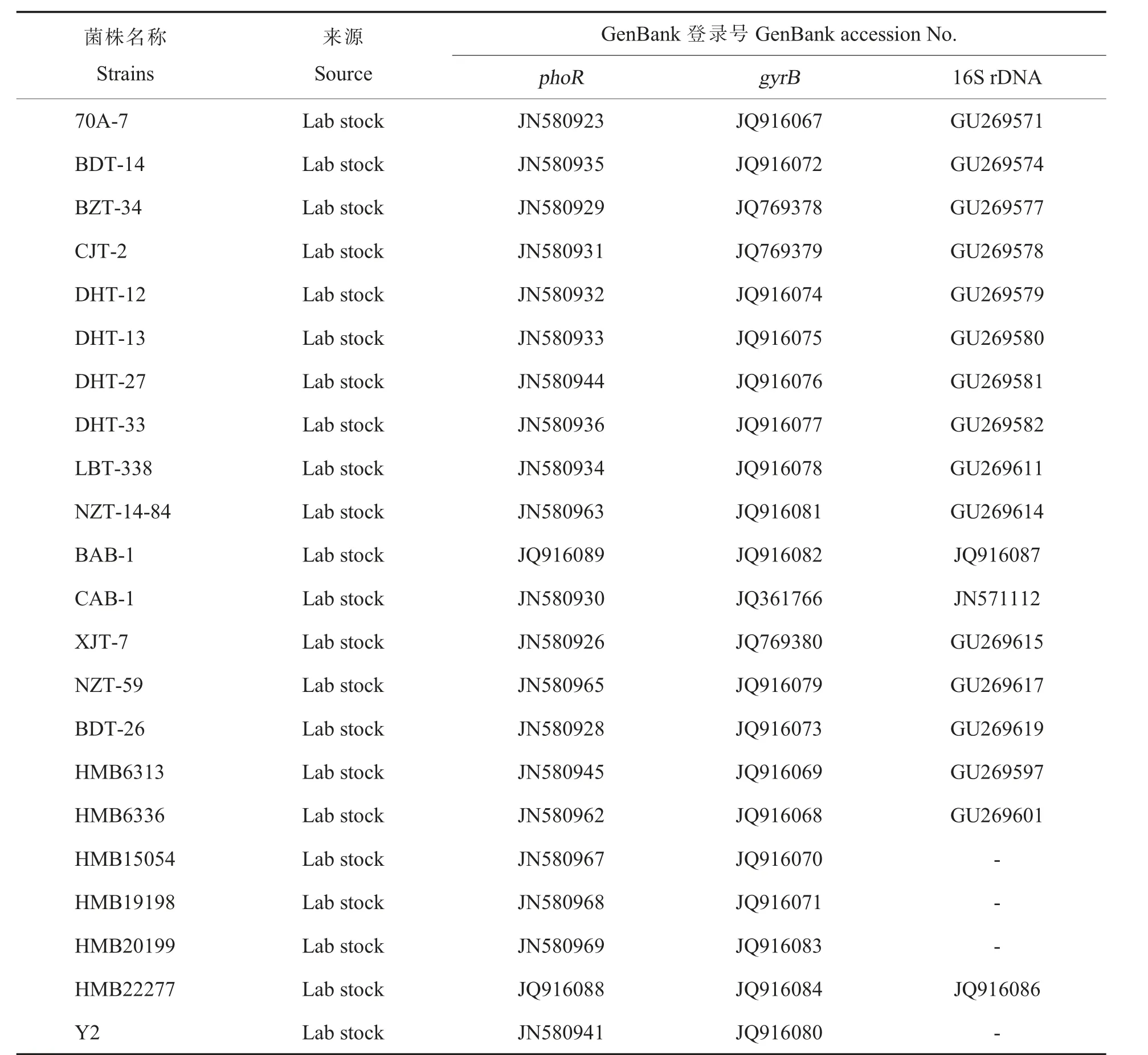
表2 野生型芽孢杆菌菌株phoR、gyrB 和16S rDNA 登录号Table 2 The accession numbers of phoR, gyrB and 16S rDNA of wild type Bacillus strains
1.6 NCD-2 菌株实时PCR 检测体系的灵敏性检验
将NCD-2 菌株DNA 以10 倍为梯度稀释至质量浓度为0.8 μg·L-1~8.0 mg·L-1,取2 μL DNA 稀释液为模板进行实时PCR 扩增。以DNA质量浓度(ρDNA)以10 为底的对数(lgρDNA)为横坐标,以检测的循环阈值(Cycle threshold,Ct)为纵坐标,绘制标准曲线,计算扩增效率(EP=101/k-1,k为标准曲线斜率), 检测实时PCR 体系的灵敏性。 每个质量浓度设3 次重复。
为了检测土壤中的抑制因子对实时PCR 检测的影响, 从出苗2 周的棉苗根部切下200 mg附有根际土壤的根,将其置于装有10 mL 无菌水的离心管中, 并加入NCD-2 菌株使根中NCD-2菌株的有效活菌数(以下简称为“含量”)分别为5×103、5×104、5×105、5×106和5×107g-1。将离心管置于超声波 (90 W) 中超声处理1 min,10 000 r·min-1下离心30 min 收集沉淀物。 利用土壤DNA 提取试剂盒提取沉淀物中总DNA。 取2 μL DNA 为模板进行实时PCR 扩增,对照样品中不添加NCD-2 菌株。设3 次重复。以提取的土壤总DNA 质量浓度(ρDNA)以10 为底的对数(lgρDNA)为横坐标,以Ct 值为纵坐标,绘制标准曲线,计算扩增效率,检测土壤中抑制因子对扩增效率的影响。
1.7 NCD-2 菌株在棉花根际土中定植能力的检测
NCD-2 菌株于200 mL LB 液体培养基中30 ℃、160 r·min-1震荡培养48 h,12 000 r·min-1下离心30 min 收集菌体,用无菌水调节菌液菌体含量至109mL-1。将表面消毒的冀棉11 种子于25 ℃过夜催芽,挑选露白的棉种在100 mL 菌悬液中浸泡30 min,然后播种于灭菌土壤中,置于25~28 ℃温室中培养,光照为自然光,定期浇水,以无菌水浸泡的棉籽为对照,每个处理48 株,每个处理进行3 次重复。播种后8 d 和16 d,从不同处理的棉苗中各随机取9 株并用力抖落附着在棉苗根部的土壤,收集棉苗根际土壤,充分混匀后取250 mg 风干土壤利用土壤DNA 提取试剂盒提取DNA。利用NCD-2 菌株特异性引物,以提取的土壤总DNA 为模板,利用实时PCR 技术检测NCD-2 菌株在棉花根际的定植数量。 同时利用菌落计数法检测NCD-2 菌株在棉花根际的定植数量。 取250 mg 根际土壤悬浮于30 mL 0.9%(质量分数)NaCl 溶液中,充分混匀后进行10 倍系列稀释,取100 μL 稀释液涂布在LB 培养基上,51 ℃培养12 h 后, 记录菌落数量, 挑取与NCD-2 菌株表型相似的菌落用NCD-2 菌株特异性引物进行PCR 扩增, 根据阳性菌落数占总菌落数的比值计算NCD-2 菌株的数量。
1.8 NCD-2 菌株对棉花立枯病的防治效果及对根际立枯丝核菌定植数量的影响
将立枯丝核菌接种于PDA 培养基中,25 ℃黑暗培养7~10 d, 收集菌丝并将菌丝打碎与自然土按一定比例(土壤、蛭石、菌丝质量比为2.0∶1.0∶0.1)充分混匀,制备含有病原菌的土壤。 将露白的鄂荆一号棉籽在NCD-2 菌悬液(109mL-1)中浸泡30 min,播种于含有病原菌的土壤中,以清水处理的棉种为对照。 每个处理48株,3 次重复。 播种后置于温室中培养,温度25~28 ℃,光照为自然光,定期浇水。 播种后16 d 取棉苗根际土壤, 检测根际立枯丝核菌及NCD-2菌株的数量。 取200 mg 根际土置于装有20 mL无菌水的锥形瓶中, 充分混匀后进行系列稀释,取100 μL 稀释液涂布在立枯丝核菌选择性培养基中,28 ℃培养24 h, 根据立枯丝核菌的菌落形态统计立枯丝核菌的菌落数[19]。取根际土250 mg,利用土壤DNA 提取试剂盒提取总DNA, 使用NCD-2 菌株特异性引物通过实时PCR 分析根际NCD-2 菌株的含量。
播种后16 d,调查NCD-2 菌液处理和清水对照的棉花死苗数,计算防治效果(E)。公式:E(%)=(N0-N1)/N0×100%。 式中,N0为清水对照处理的死苗数,N1为NCD-2 菌株处理的死苗数。
1.9 统计分析
菌落数以实际计数以10 为底的对数来表示。 利用SAS for Windows Ver 9.0 软件对试验数据进行相关性分析(P≤0.05),利用Origin 7.0 对试验数据进行作图。
2 结果与分析
2.1 NCD-2 菌株实时PCR 检测体系特异性引物设计
将NCD-2 菌株全基因组序列与其他11 株芽孢杆菌标准菌株的全基因组序列进行序列比对,发现NCD-2 菌株中存在1 个特异性基因簇,该基因簇位于gabp和yeaB基因之间(图1),全长8 883 bp,含有8 个开放阅读框(open reading frame,ORF)。其中ORF7 的序列与枯草芽孢杆菌UD1022 菌株(美国,CP011534.1)基因组序列同源性为99%, 与贝莱斯芽孢杆菌 (B.velezensisRD7-7)菌株(韩国,CP016913.1)基因组序列同源性为93%, 与芽孢杆菌SDLI1 菌株 (美国,CP013950.1)序列同源性为91%。 根据ORF7 的序列,设计针对NCD-2 菌株的特异性引物qNCDORF7-F:5'-AGGCAGCATTCAAGCACCAG-3',qNCD-ORF7-R:5'-AGCCAGCGATCATTCCCATC-3'。 引物特异性检测发现,该引物与UD1022菌株序列一致,但该特异性引物仍可在一定区域内开展NCD-2 菌株的定量检测。

图1 NCD-2 菌株特异性基因簇示意图Fig. 1 Schematic diagram of strain NCD-2 specific gene cluster
2.2 NCD-2 菌株实时PCR 检测体系特异性检测
通过常规PCR 技术检测引物对NCD-2 菌株的特异性,结果(图2)表明,以qNCD-ORF7-F/qNCD-ORF7-R 为引物, 以NCD-2 菌株DNA以及加入NCD-2 菌株的土壤总DNA 为模板,能特异性地扩增出目的片段, 而以其他22 株野生型芽孢杆菌菌株DNA 以及土壤总DNA 为模板,不能扩增出目的片段。 进一步通过实时PCR 技术检测引物qNCD-ORF7-F/qNCD-ORF7-R 对NCD-2 菌株的特异性, 熔解曲线结果 (图3)表明, 以NCD-2 菌株DNA 以及加入NCD-2 菌株的土壤总DNA 为模板,在81.4 ℃出现特异性的熔解曲线峰, 而以其他22 株野生型芽孢杆菌菌株DNA、20 株芽孢杆菌参考菌株DNA 以及土壤总DNA 为模板,未出现熔解曲线峰。以上结果表明,引物qNCD-ORF7-F/qNCD-ORF7-R 对NCD-2菌株具有较高的特异性, 以该引物构建的实时PCR 体系可用于定量检测NCD-2 菌株。

图2 NCD-2 菌株引物特异性检测Fig. 2 Detection of strain NCD-2 by strain specific primers
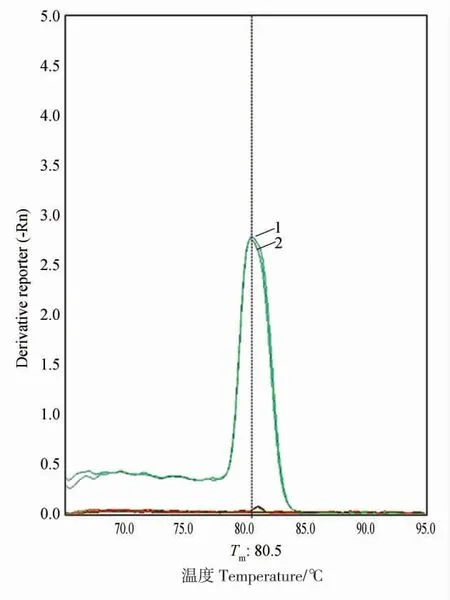
图3 实时荧光PCR 熔解曲线图Fig. 3 Dissolution curve of real-time PCR
2.3 NCD-2 菌株实时PCR 检测体系标准曲线的建立
以NCD-2 菌株DNA 作为模板, 利用实时PCR 体系构建标准曲线,结果(图4)显示,模板质量浓度在0.8 μg·L-1~8.0 mg·L-1范围内与Ct值呈线性关系(r=0.995)。 土壤中定量加入不同含量的NCD-2 菌株后,以提取的土壤总DNA 为模板,构建标准曲线,结果(图4)显示,土壤中NCD-2 菌体含量在5×103~5×107g-1范围内,菌体含量与Ct 值呈良好的线性关系(r=0.995),表明土壤DNA 对NCD-2 菌株的定量检测结果没有显著影响。 因此,该方法可用于定量检测土壤样品中NCD-2 菌株的数量。
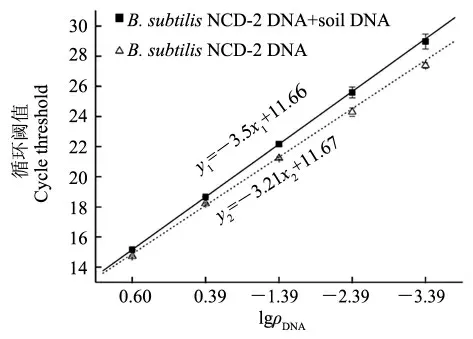
图4 实时PCR 标准曲线Fig. 4 Standard curve of real-time PCR
2.4 NCD-2 菌株实时PCR 检测体系对其在棉花根际土壤中定植能力的检测结果
实时PCR 检测结果(图5)表明,播种后8 d和16 d 时,NCD-2 菌株在棉花根际土壤中的菌体含量分别为1.14×105和9.5×104g-1。 菌落计数法检测结果(图5)显示,播种后8 d 和16 d 时,NCD-2 菌株在棉花根际土壤的菌体含量分别为2.15×105g-1和2.45×105g-1。 通过2 种方法检测NCD-2 菌株在棉花根际的定植数量具有较高的相关性, 在8 d 时2 种方法检测的相关系数为0.99,在16 d 时两者相关系数为0.95。
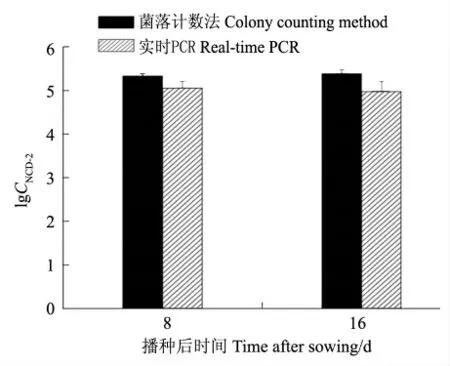
图5 实时PCR 和菌落计数法对棉花根际土壤中NCD-2 菌株的定量检测Fig.5 The population of Bacillus subtilis strain NCD-2 in cotton rhizosphere soil detected by real-time PCR and colony counting method
棉花立枯病调查结果(表3)表明,109mL-1的NCD-2 菌液浸种处理在播种后16 d 时对棉花立枯病的防治效果达到67.9%;此时NCD-2 菌株在棉花根际土壤的菌体含量为7.6×105g-1,棉花立枯丝核菌在根际土壤中含量为416 g-1,而对照中立枯丝核菌在棉花根际土壤中的含量为3 011 g-1。

表3 NCD-2 菌株对棉花立枯病的防治效果及其与棉花立枯丝核菌在棉花根际土壤中的含量Table 3 Control effect of NCD-2 strain on cotton damping-off and contents of NCD-2 strain and Rhizoctonia solani in cotton rhizosphere soil
3 讨论
定量检测生防微生物在田间的消长动态不仅有助于研究生防菌的防病效果,同时也是评价生防菌环境安全性的重要指标[19]。 以往常利用抗生素驯化技术获得抗生素抗性菌株,然后根据抗生素筛选以定量检测特定微生物在环境中的数量[20]。抗性菌株在环境中容易丧失抗性,加上其他抗性微生物的干扰, 往往造成检测结果不准确[20-21]。 近年来,实时PCR 已被广泛应用于土壤中真菌[22]、细菌[23]、病毒[24]的定量检测。 获得针对靶标微生物的特异性引物或探针是建立基于实时PCR 技术定量检测的关键, 目前针对病原菌建立的定量检测体系较多,但针对特定生防菌株建立的实时PCR 定量检测体系较少, 主要原因是缺乏针对单个菌株的特异性引物[25]。近年来,随着高通量测序技术的广泛应用,获得了越来越多的微生物全基因组序列。 通过全基因组序列比对,获得某个菌株特异性基因序列,据此设计针对特定菌株的特异性引物已成为可能。 Zhang等[26]通过对木霉菌(Trichoderma guizhouense)NJAU4742 设计特异性引物,建立实时PCR 检测体系监测菌株在土壤中的定植动态。 枯草芽孢杆菌是一类重要的生防细菌,目前超过350 株芽孢杆菌获得全基因组序列。 本研究通过将生防枯草芽孢杆菌NCD-2 菌株全基因组序列与已知枯草芽孢杆菌类细菌全基因组序列进行比对, 发现NCD-2 菌株中有1 个功能未知且特异性较强的基因簇, 根据该基因簇序列设计了针对NCD-2菌株的特异性引物, 建立了定量检测NCD-2 菌株的检测体系, 并开展了NCD-2 菌株在棉花根际的定植动态检测。 然而,随着数据库中提交的微生物全基因组序列越来越多,可能会发现不同地理起源的菌株具有相似或一致的基因序列。 通过GenBank 数据库比对发现,本研究中获得的特异性基因簇, 与美国起源的枯草芽孢杆菌UD1022 菌株同源性非常高(99%),可能会影响NCD-2 菌株的定量检测。 但是,如果类似菌株相隔很远,或者在不同地域范围进行检测,这种相对特异的基因序列仍然可用于菌株特异性引物的开发,实现对一定区域内靶标微生物的检测。
土壤中含有丰富的有机酸和腐殖酸, 尽管目前土壤总DNA 提取试剂盒可最大限度地排除土壤中的有机物, 但残存的腐殖酸仍然会抑制PCR 的扩增效率[27-28]。 本研究建立了2 条标准曲线, 一条是以纯化的NCD-2 菌株DNA 为模板,另外一条是将系列稀释的NCD-2 菌液添加到土壤中,然后提取土壤总DNA,系列稀释后作为模板建立的标准曲线。 通过比较这2 条标准曲线, 发现土壤中的抑制因子的确降低了PCR 的扩增效率。 本研究通过菌落计数法和实时PCR 技术分析了棉花根际NCD-2 的数量,结果发现,实时PCR 法检测的菌群数量低于菌落计数法得到的结果, 推测是由于土壤中的有机物抑制PCR 扩增效率造成的[29],这与任海英等[30]的研究结果一致。 但由于实时PCR 技术和菌落计数法检测结果具有较高的相关性, 而且实时PCR 技术具有准确且检测迅速等优势,因此, 实时PCR 技术可用于NCD-2 菌株的定量检测及实际应用。
生防菌防治作物土传病害的机理包括抑菌作用、竞争作用和诱导抗性,其中通过竞争作用有效定植在寄主根际并抑制根际病原菌的生长是生防菌防治土传病害的重要作用机理。 Samaras等[31]通过共聚焦激光扫描显微镜发现,枯草芽孢杆菌MBI600 能够定植在黄瓜(Cucumis sativus)的根际,定性分析认为该菌株的根际定植能有效防治由尖孢镰刀菌和腐霉菌(Pythium aphanidermatum)引起的枯萎病和根腐病。本研究前期工作发现, 丧失生物膜形成能力的NCD-2 菌株突变体在棉花根际的定植能力下降,同时对棉花立枯病的防治效果也显著下降, 间接证明NCD-2 菌株的根际定植能力与其对棉花立枯病的防效呈正相关。 本研究通过实时PCR 技术可准确检测NCD-2 菌株在棉花根际的实际定植情况,不但可以评估NCD-2 菌株在寄主根际的有效定植数量, 而且可用于棉花病害防治中NCD-2 菌株和病原菌相互关系的研究。
4 结论
通过获得枯草芽孢杆菌NCD-2 菌株的特异性引物, 建立了基于实时PCR 技术的NCD-2 菌株检测体系, 利用该技术能够快速准确检测NCD-2 菌株在棉花根际的数量,明确其在棉花根际土壤中的实际定植能力,为检测该菌株防治作物土传病害的效果提供科学依据。
