棉花SRS 基因家族的全基因组鉴定及生物信息学分析
2022-07-07张雪孙瑞斌马聪聪马丹张晓睿刘志红刘传亮
张雪,孙瑞斌,马聪聪,马丹,张晓睿,刘志红,刘传亮
(中国农业科学院棉花研究所/ 棉花生物学国家重点实验室,河南 安阳, 455000)
短链相关序列(SHI-related sequence,SRS)基因,也称为短节间(short internodes, SHI)或者STY(STYLISH)基因,编码一类植物特有的转录因子, 在调节植物生长发育中发挥重要作用[1-2]。SRS基因家族具有2 个高度保守结构域,分别为环状锌指结构域和IGGH 结构域[2-3]。 侧根原基1(lateral root primordium1,LRP1)是拟南芥中发现的第一个SRS基因,通过调节生长素的合成对侧根发育起负调控作用[4-5]。
在拟南芥中已有10 个SRS基因被报道:SHI(At5G66350),STY1(At3G51060),STY2(At4G36260),SRS3~8(At2G21400,At2G18120,At1G75520,At3G54430,At1G19790,At5G33210)以及LRP1(At5G12330)[5-10],家族成员间功能冗余。 SRS 转录因子可以通过调节激素的生物合成、信号转导等参与拟南芥根系的形成、叶片及花等器官的发育过程[10-13]。 例如:拟南芥LRP1,可以通过诱导生长素合成基因YUCCA4(又称YUC4)的表达调控侧根的发育,过表达LRP1导致生长素水平升高、侧根密度减少[5-6]。 LRP1 可能与SHI、STY1、SRS3、SRS6 和SRS7 形成复合物,推测这些基因在侧根发育过程中功能冗余[5]。在玉米中,LRP1在根分生组织中特异性表达,通过RUM1 (rootless with undetectable meristem 1)介导的生长素信号通路调节根系发育[14]。据报道,STY1 可以直接激活生长素合成基因YUC4和YUC8的表达,从而调控拟南芥生长素的稳态[3,5]。拟南芥中STY1 与STY2 部分功能冗余, 通过调节生长素的稳态影响雌蕊发育过程,促进柱头的形成,也可以影响维管发育[11,15]。 进一步研究发现,STY1基因突变后会造成雌蕊发育缺陷,在STY1单基因突变基础上敲除家族内的其他成员,发现突变体雌蕊的发育缺陷程度增强,叶片的发育也受到影响[2,8]。 拟南芥的shi突变体表型与赤霉素(gibberellin,GA)生物合成缺陷突变体的表型相似,表明SHI可能参与GA 信号通路的转导[9]。 在观赏植物长寿花和一品红中异源过表达拟南芥SHI基因,其转基因材料出现株型紧凑的表型[16-17]。SRS7在拟南芥茎尖和花中高水平表达,参与绒毡层开裂的调控;srs7突变体表皮细胞长度明显较短、对GA 的敏感性显著降低;过表达SRS7可能通过中断茉莉酸信号通路导致花药开裂延迟[18]。 SRS5 通过与光形态发生促进基因的启动子直接结合调控植物光形态建成[13]。 除了拟南芥之外,水稻中的OsSHI1 可以通过抑制理想株型调控因子1 (ideal plant architecture 1,IPA1)的转录激活活性增加分蘖数并减少穗分枝数[19]。 将大豆的GmSRS18基因转入拟南芥中异源表达后能影响与胁迫相关的生理指标,包括叶绿素含量、 脯氨酸含量和相对电解质泄漏等,从而对耐旱性和耐盐性产生负调控作用[20]。 在大麦中,SHI/STY基因家族的2 个成员LKS2和VRS2可以调节麦芒伸长、 雌蕊形态和花序图案等[21-22]。 在杨树中,沉默SHI基因能促进杨树茎和根的伸长[23]。 然而,还没有关于棉花SRS的全基因组分析和功能鉴定的相关报道。
棉花是一种重要的经济作物,棉纤维是纺织业的主要天然纤维原料,棉籽可以用作食用油的原料。我国主要种植陆地棉(Gossypium hirsutum)和海岛棉(G.barbadense)2 个栽培种。 陆地棉和海岛棉基因组测序的完成, 为棉花基因组学研究奠定了基础[24-26]。 本研究基于陆地棉与海岛棉基因组数据, 利用生物信息学的方法对棉花SRS基因进行全基因组鉴定, 并对其系统进化及基因保守基序进行研究, 对家族基因之间的进化关系进行共线性分析, 并分析SRS基因在棉花不同发育时期的胚珠和纤维、 不同组织及在多种胁迫处理下的表达模式, 相关研究结果有助于进一步研究棉花SRS 蛋白在生长发育过程中的作用。
1 材料与方法
1.1 棉花SRS 基因家族成员的全基因组鉴定
为了鉴定SRS基因家族成员,从公共数据库(http://cotton.hzau.edu.cn/EN/download.php)下 载棉花基因组序列和基因注释文件[26]。 在Pfam[27]数据库(http://pfam.xfam.org/)中下载SRS 蛋白结构域的隐马尔可夫模型 (PF05142), 利用HMMER v.3.1b2[28]软件对SRS 的同源蛋白进行搜索。同时利用已经报道的拟南芥中SRS 蛋白的氨基酸序列在BLAST+v.2.6.0[29]进行相似性搜索(E值<1×10-5,一致性大于50%)。 把在2 种方法鉴定结果中均出现的候选基因保留下来,利用InterProScan v.5.32-71.0[30]软件进行蛋白特征结构域(IPR006510)预测,把具有SRS 特征结构域的候选基因保留下来。 通过在线软件ExPASy(http://web.expasy.org/protparam/)对棉花SRS 蛋白质的分子质量、氨基酸数量和等电点等基本理化性质进行预测分析。
1.2 棉花SRS 基因系统发育树的构建及保守基序分析
将棉花SRS基因家族成员的氨基酸序列和已经报道的拟南芥SRS基因家族成员相结合,在MUSCLE[31]上进行多重序列比对,利用IQTREE[32]构建系统发育树,采用最大似然统计法并自动选择最佳替代模型,自展值(bootstrap value)设为1 000。利用MEME Suite 5.1.0[33]在线软件预测棉花SRS基因的保守基序,最大发现数设为20。 利用TBtools[34]软件可视化棉花SRS基因的保守基序分布情况。
1.3 棉花SRS 基因家族成员的染色体定位和共线性分析
利用R 语言工具包RIdeogram[35]绘制基因在染色体上的分布。通过MCScanX[36]软件对海岛棉和陆地棉中SRS基因进行共线性分析,鉴定基因复制事件,用Circos v.0.69[37]对共线性分析结果进行可视化,参数均使用软件默认参数。
1.4 陆地棉SRS 基因的表达模式分析
在浙江大学棉花数据库(http://cotton.zju.edu.cn/index.htm)下载陆地棉不同发育时期的胚珠和纤维、 不同组织及多种胁迫下的转录组数据,将数据进行标准化处理,用lg(TPM+1)(TPM 是每千个碱基的转录每百万映射读取的读长数,transcripts per kilobase of exon model per million mapped reads)计算SRS基因的表达水平,使用R语言pheatmap 软件包对表达谱进行可视化。 分析SRS基因在不同发育时期的胚珠和纤维,在根、茎、叶、花托、花瓣、雄蕊、雌蕊等器官以及在低温、高温、盐、干旱等不同胁迫处理的棉苗中的表达模式。
1.5 陆地棉总RNA 的提取和实时定量聚合酶链式反应
使用天根多糖多酚植物总RNA 提取试剂盒提取陆地棉TM-1 不同发育时期的胚珠和纤维样品的总RNA,用全式金反转录试剂盒合成第1 链cDNA, 用NCBI primer-BLAST 设计实时定量聚合酶链式反应 (quantitative real-time polymerase chain reaction,qRT-PCR)引物(表1)。 将获得的cDNA 模板稀释5 倍后进行扩增,qRT-PCR 的反应体系为10 μL,包含cDNA 模板1 μL、正向和反 向 引 物 各0.5 μL、qRT-PCR 反 应 混 合 物(TransStartTop Green qPCR Super Mix)5 μL 以及双蒸水(ddH2O)3 μL。 所有的反应都设3 个技术重复,并在罗氏光循环480 仪器上运行。 扩增反应程序为:95 ℃预变性5 min;95 ℃变性10 s,60 ℃退火10 s,循环40 次;72 ℃延伸15 s。 利用熔融曲线来检测扩增产物的特异性。 以GhHis3(GenBank 登录号:AF024716)为内参基因,荧光定量数据采用2-ΔΔCt 方法计算, 用本地软件Origin 8 生成柱状图。
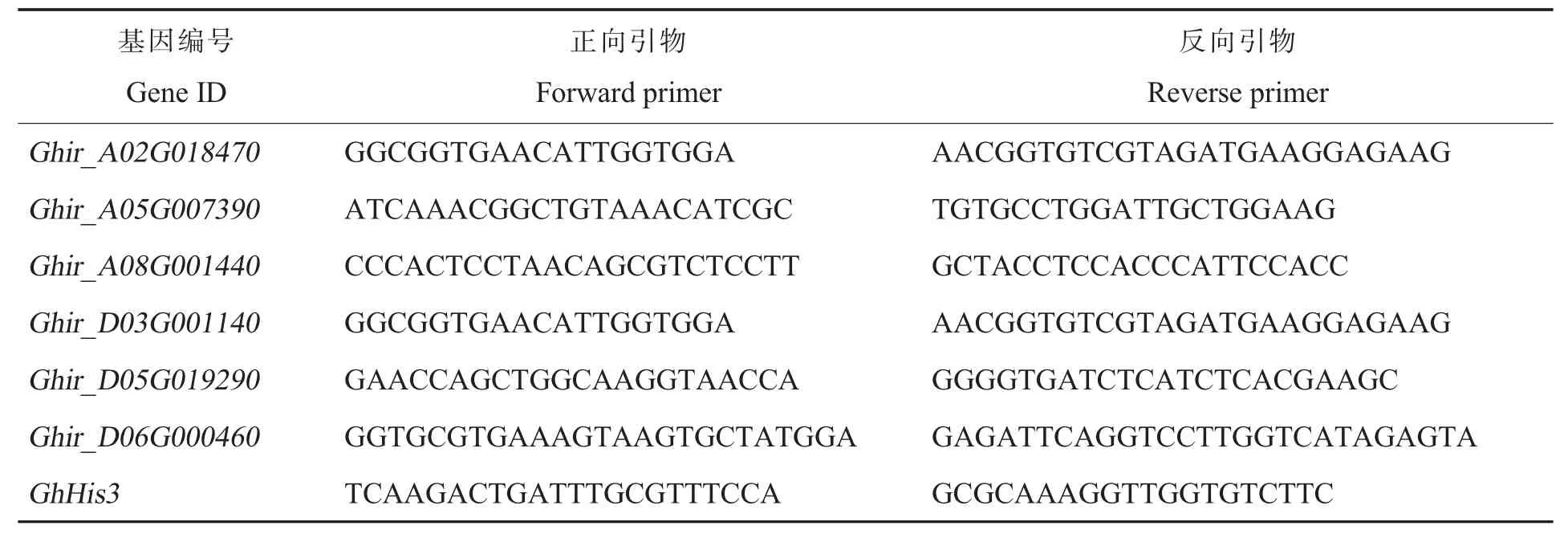
表1 qRT-PCR 反应引物列表Table 1 List of primer sequences for qRT-PCR
2 结果与分析
2.1 棉花SRS 基因家族成员的鉴定
利用拟南芥的10 个SRS 蛋白的氨基酸序列,通过HMMER 和BLAST 这2 种比对方法把棉花基因组中具有SRS 特征结构域的候选基因保留下来。 结果显示,在陆地棉基因组中共鉴定出27 个SRS基因, 在海岛棉基因组中共鉴定出26 个SRS基因, 所有基因编码的蛋白均定位在细胞核中。 对这些基因所编码蛋白的氨基酸数量、 蛋白质分子质量及等电点等进行统计分析(表2)。 结果显示,陆地棉中SRS 蛋白的氨基酸数量为194~433; 分子质量为21.959~45.074 ku;等电点变化范围比较大,其中Ghir_D13G003130编码的蛋白等电点最大, 为8.933,Ghir_A02G018470编码的蛋白等电点最小, 为5.197,表明陆地棉中SRS 蛋白质从酸性到碱性都有分布。 海岛棉中SRS 蛋白的氨基酸数量为195~536;分子质量为21.944~57.555 ku;等电点变化范围相比陆地棉较小,其中Gbar_A13G003030编 码的蛋白等电点最大,为9.095,Gbar_D11G019330编码的蛋白等电点最小,为6.267,表明海岛棉中SRS 蛋白质大部分为中性或碱性。
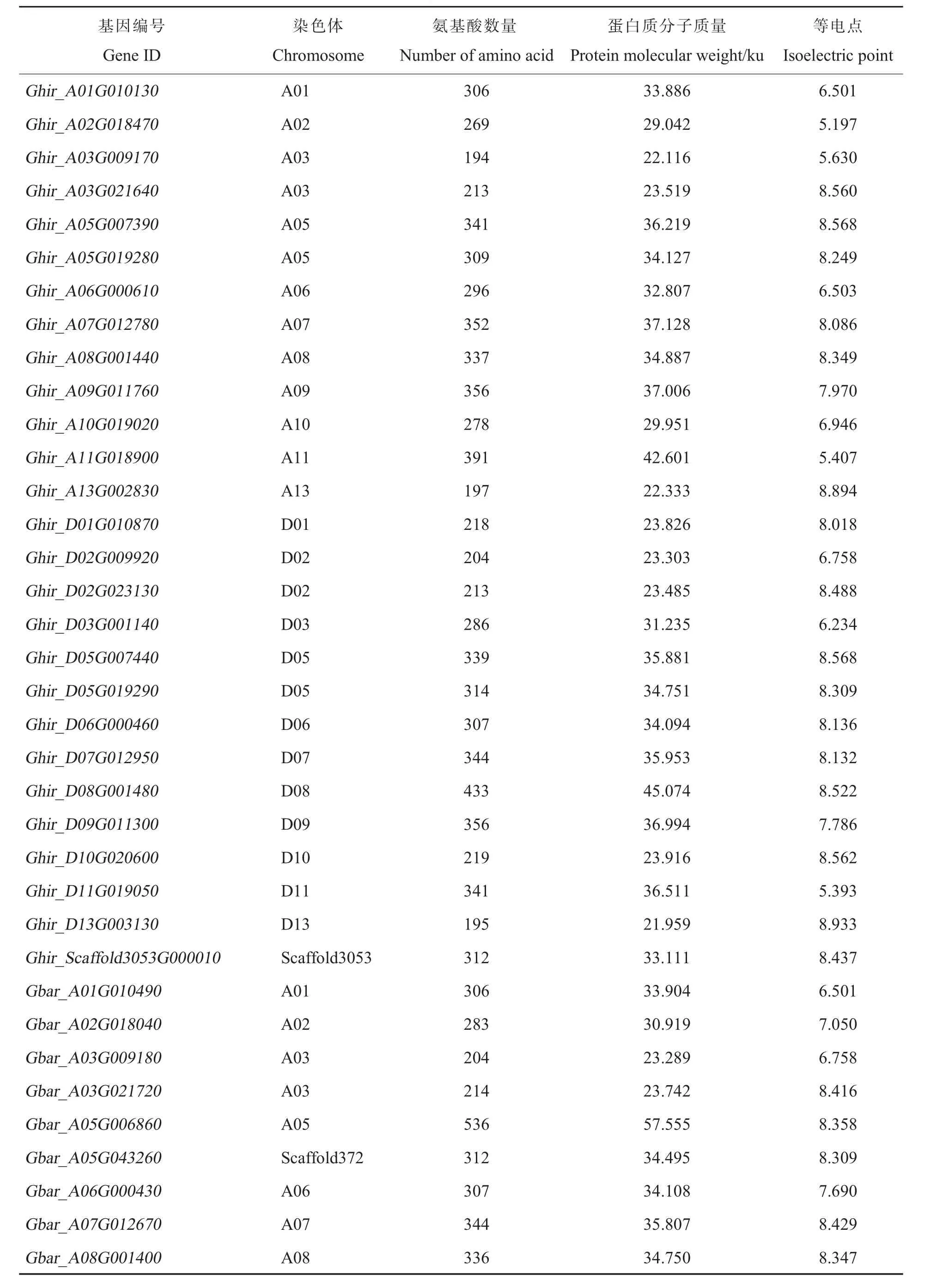
表2 陆地棉和海岛棉中SRS 基因家族成员鉴定Table 2 Identification of SRS genes in G. hirsutum and G. barbadense
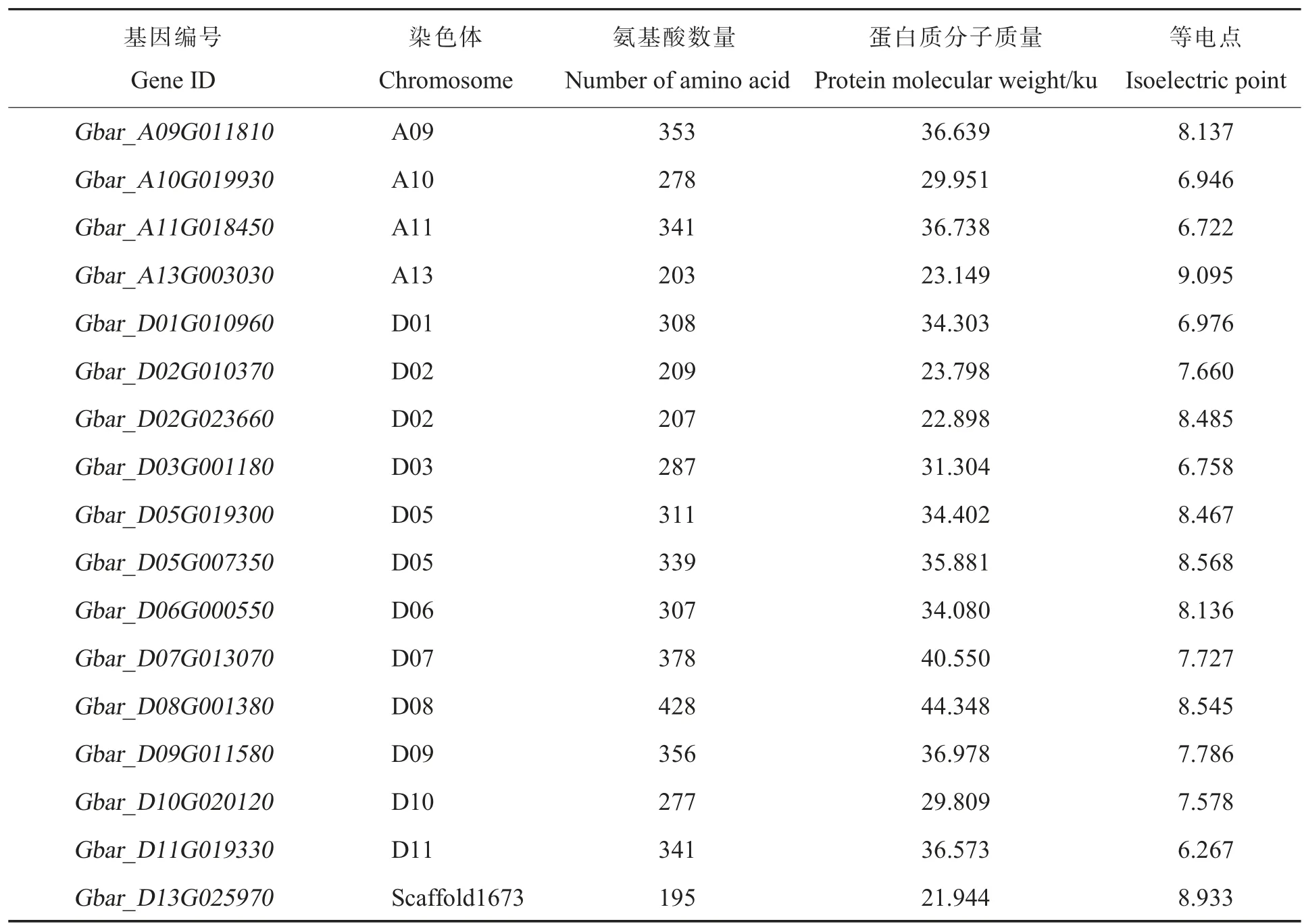
表2 (续)Table 2 (Continued)
2.2 棉花SRS 系统进化分析和保守基序分析
以53 个棉花SRS 和10 个AtSRS 氨基酸序列构建棉花SRS 家族成员的系统发育树。 如图1A 所示,所有的SRS 可分为5 个分支,每个分支成员数量不等, 分支Ⅰ和分支Ⅱ分别有3 个和2个成员,全部来自拟南芥。 分支Ⅲ有23 个成员,2个来自拟南芥,11 个来自陆地棉,10 个来自海岛棉。 分支Ⅳ有27 个成员,来自陆地棉和海岛棉的各12 个,拟南芥的3 个。 分支Ⅴ有8 个成员,陆地棉和海岛棉各4 个。
分析拟南芥、陆地棉和海岛棉中SRS 的保守基序(图1B),将预测到的20 种基序依次命名为基序1~20。 同一分支的蛋白序列呈现相似的保守基序排列方式,不同SRS 蛋白所包含的保守基序数目及种类存在差异,基序种类最少的1 个蛋白仅含有3 种基序,最多的含有15 种基序,说明不同的家族成员间可能存在功能分化。 在63 个SRS 蛋白中,基序1~4 存在于大多数SRS 蛋白,说明这些基序比较保守,可能是SRS 蛋白功能的重要元件。 基序18 出现了5 次,基序16 出现了13 次,且这2 个基序只存在于分支Ⅲ中,说明基序18 和基序16 可以作为该分支的标志序列,可能与这个分支基因的功能有关。
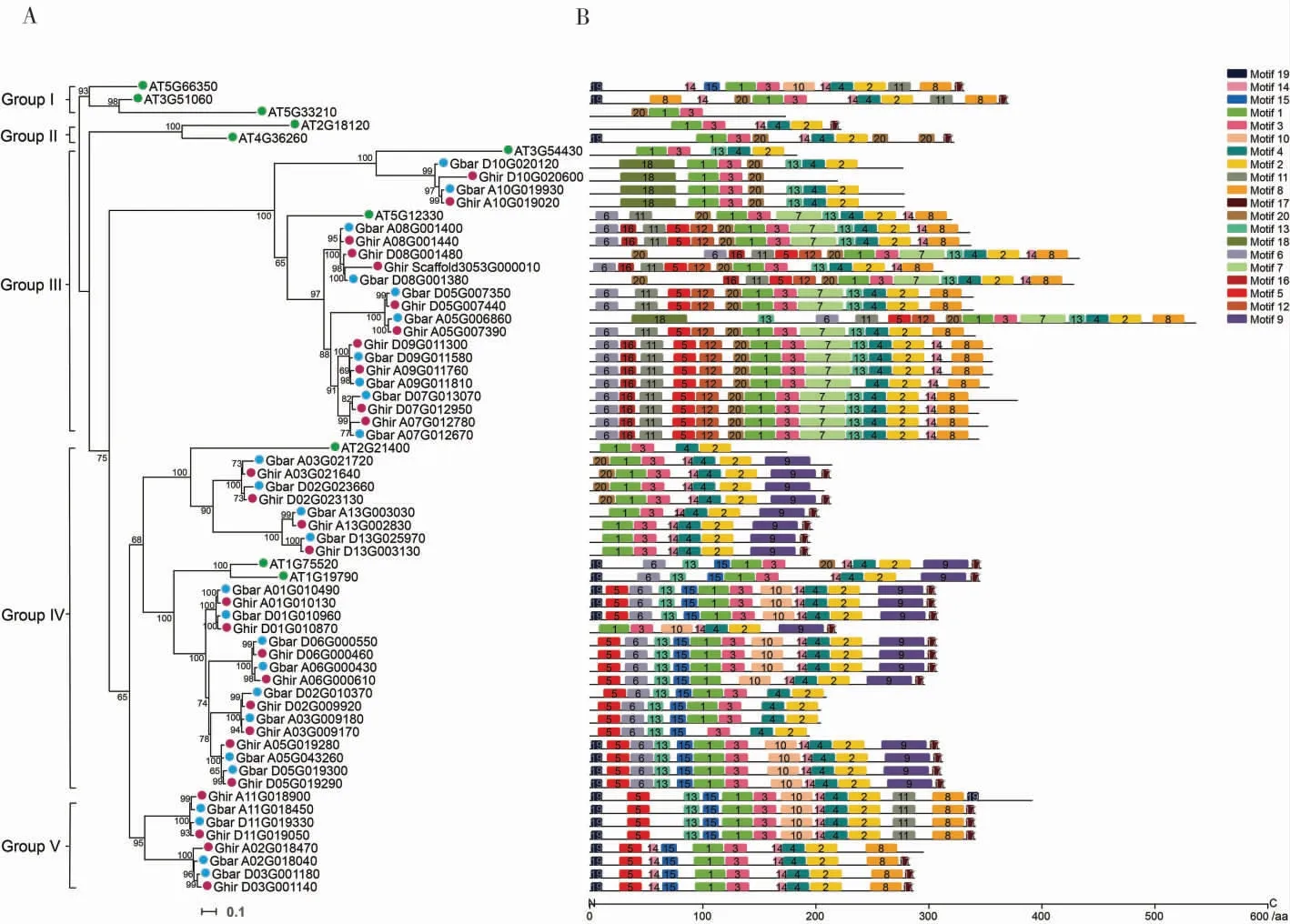
图1 拟南芥、陆地棉和海岛棉SRS 基因家族的系统进化和保守基序分析Fig. 1 Phylogenetic and conserved motif analysis of SRS genes in A. thaliana, G. hirsutum and G. barbadense
2.3 棉花SRS 基因家族成员的染色体定位和共线性分析
图2 为基于基因坐标信息绘制的陆地棉及海岛棉SRS基因家族在染色体上的分布图。如图所示, 陆地棉的27 个基因定位在除A04、A12、D04、D12 以外的染色体上,其中A03、A05、D02、D05 号染色体上均有2 个SRS基因,有1 个基因位于没有拼接到染色体的scaffold。 海岛棉中的24 个SRS基因也都可以定位在染色体上, 有2个基因位于没有拼接到染色体的scaffold, 分别是Gbar_A05G043260和Gbar_D13G025970,其中A03、D02、D05 号染色体上均有2 个SRS基因。
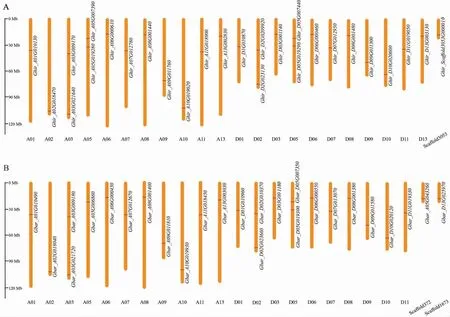
图2 陆地棉(A)和海岛棉(B)中SRS 基因在染色体上的分布Fig. 2 Chromosomal distribution of SRS genes in G. hirsutum (A) and G. barbadense (B)
为进一步探索棉花SRS基因家族的进化关系, 对陆地棉和海岛棉中SRS基因进行共线性分析。 如图3 所示,发现陆地棉中无串联重复复制,有13 对基因是直系同源,9 对基因为旁系同源。 分支Ⅲ中有5 对均为直系同源基因;分支Ⅳ中有6 对直系同源基因、7 对旁系同源基因,主要集中在A01、A03、A05、A06、D02、D05、D06 和D11、D13 号染色体上; 分支Ⅴ中有2 对直系同源基因、2 对旁系同源基因,主要在A02、A11 和D03、D11 号染色体上。 在海岛棉中,同样也没有串联重复复制,有11 对为直系同源基因、9 对为旁系同源基因。 分支Ⅲ中有5 对直系同源基因、2 对旁系同源基因, 集中在A07、A09 和D07、D09 号染色体上; 分支Ⅳ中有4 对直系同源基因、5 对旁系同源基因,主要集中在A01、A03、A06、A13、D01、D02、D05 和D06 染色体上;分支Ⅴ中有2 对直系同源基因、2 对旁系同源基因,主要在A02、A11、D03 和D11 号染色体上。
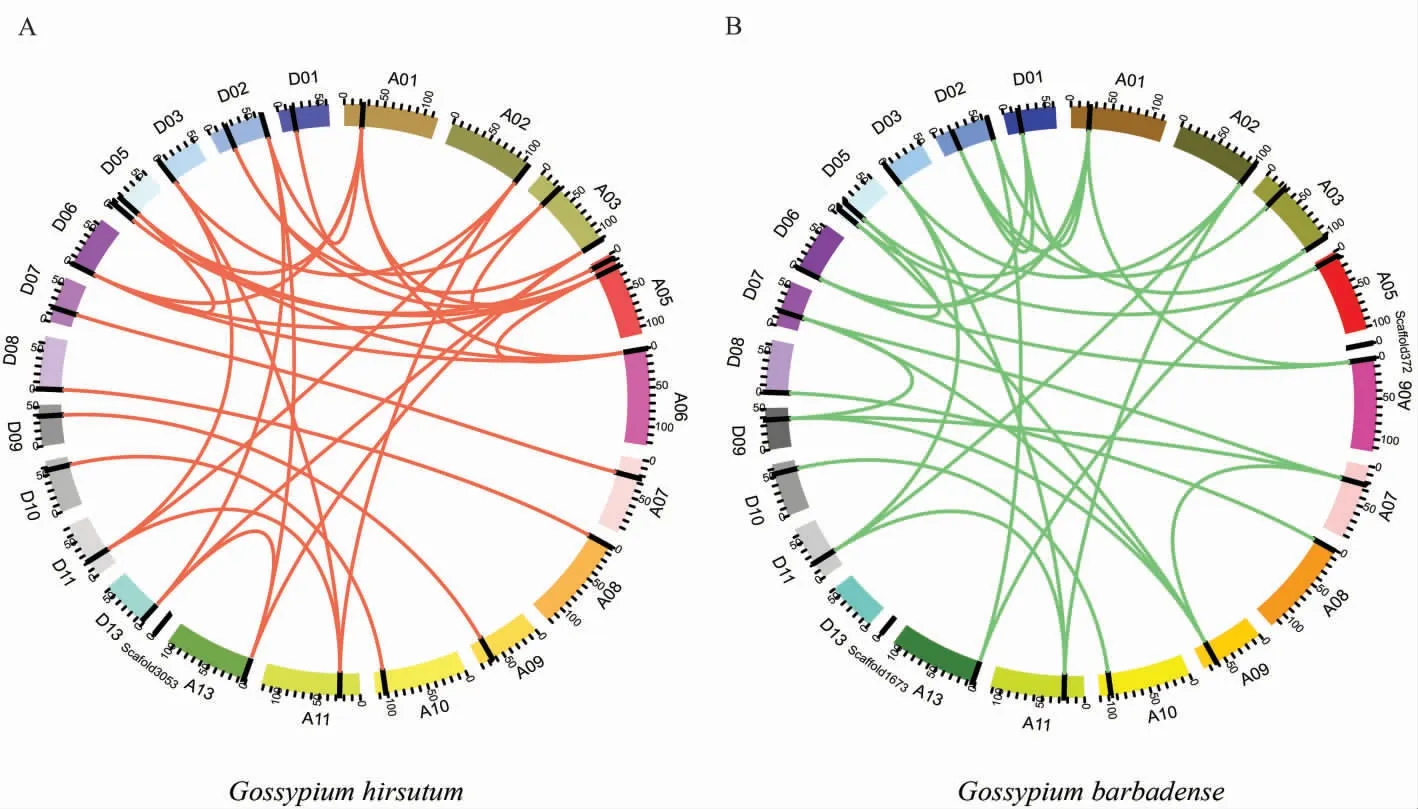
图3 陆地棉(A)和海岛棉(B)中SRS 基因在染色体上的共线性分析Fig. 3 Collinearity of SRS genes in G. hirsutum (A) and G. barbadense (B)
2.4 陆地棉SRS 基因家族表达模式分析
为探究陆地棉SRS基因在棉花生长发育过程中的生物学功能,利用已发表的陆地棉转录组数据分析了27 个SRS基因在不同发育时期的胚珠和纤维、 以及不同器官中的表达模式。 如图4所示,根据表达谱中各基因在同一时期的表达量及同一基因不同时期的表达量比较分析,发现有13 个基因在不同发育时期的胚珠和纤维中活跃表达。其中Ghir_D05G019290基因在胚珠和纤维发育的不同时期及不同器官中均有表达,Ghir_A02G018470和Ghir_D03G001140是 同 源基因,Ghir_A02G018470基因在不同发育时期的棉花胚珠和纤维及不同组织中都有表达,相较于其他基因,该基因在花丝、花瓣、雌蕊中表达量较高;Ghir_D03G001140基因在-3~20 DPA(开花后天数,Days post anthesis) 的胚珠及在根、 茎、叶、 花托等组织中的表达量较其他SRS基因高,而在20 DPA、25 DPA 的纤维中几乎不表达。Ghir_A08G001440基因在-3~1 DPA 胚珠的表达量相对于其他时期较高, 在20 DPA、25 DPA 的纤维和花药、 花丝、 叶中表达量较低。 而Ghir_D08G001480和Ghir_Scaffold3053G000010在胚珠和纤维发育的20 DPA、25 DPA 和叶中表达量低,在花药、花丝、雌蕊、萼片等器官中表达量相对较高。Ghir_A03G021640在棉花的花药、花瓣、 花托和萼片的表达量较其他器官高,Ghir_A05G007390基因在胚珠发育初期的0 DPA和1 DPA、茎和花托中表达量高,在胚珠发育后期、 纤维发育不同时期和其他组织中表达量低。Ghir_D06G000460基因在纤维发育各个时期的表达量高于其他基因在同时期的表达量。Ghir_A13G002830基因在胚珠发育初期的-3 DPA、0 DPA、1 DPA 和3 DPA 的表达量高于其他时期。
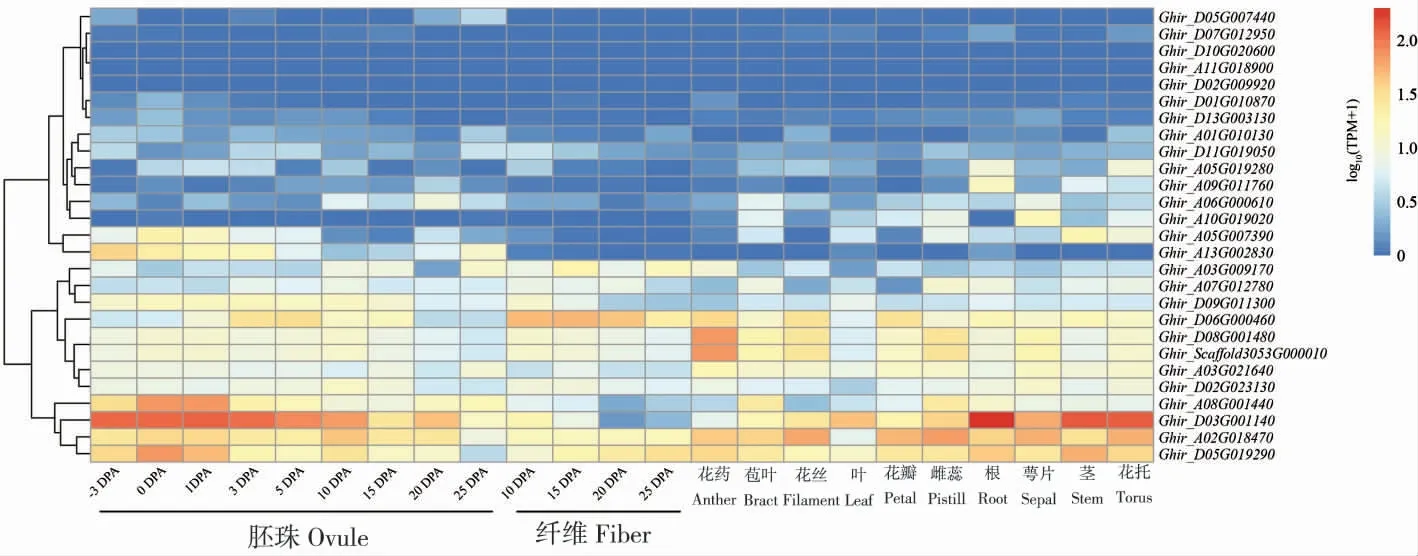
图4 陆地棉SRS 基因在不同器官、不同发育时期的胚珠和纤维中的表达谱Fig. 4 Expression profiles of SRS genes in different organs, ovule and fiber at different developmental stages in G. hirsutum
为进一步探究陆地棉SRS基因的生理生化及生物学功能,分析了SRS基因在多种胁迫下的表达水平变化情况。 与对照相比,Ghir_Scaffold3053G000010和Ghir_D08G001480基 因在低温胁迫、高温胁迫、盐处理和干旱胁迫处理3 h 的表达量较高, 之后有所下调, 表明这2个基因参与了多种胁迫应答。 相较于对照,Ghir_A10G019020基因在3~24 h 低温胁迫处理下表达量较低,Ghir_A05G007390基因在盐处理和干旱胁迫处理6 h 后表达量较高, 可能是受到外界环境刺激后才被激活。 与对照相比,Ghir_A07G012780、Ghir_D07G012950、Ghir_A11G018900和Ghir_D11G019050基因在盐处理和干旱胁迫12 h 的样本优势表达, 但Ghir_A11G018900和Ghir_D11G019050基 因 在处理24 h 的样本中表达量下降。 与对照相比,Ghir_A08G001440基因在低温胁迫、 盐胁迫24 h 的样本中表达量降低。
根据棉花SRS基因在胚珠和纤维发育不同时期的表达谱数据,从分支Ⅲ、Ⅳ、Ⅴ中各筛选出2 个差异表达的SRS基因,分别为Ghir_A05G007390、Ghir_A08G001440、Ghir_D05G019290、Ghir_D06G000460、Ghir _ A02G018470、Ghir _D03G001140,利用qRT-PCR 分析其在-3 DPA、0 DPA、1 DPA、3 DPA、5 DPA、10 DPA 和20 DPA的胚珠,以及10 DPA、15 DPA 和20 DPA 纤维中的表达情况。 结果如图6 所示,qRT-PCR 的结果与转录组学数据分析的结果相似:来自分支Ⅲ的Ghir_A05G007390和Ghir_A08G001440基 因在胚珠发育初期的表达量略高于胚珠发育后期和纤维发育时期;来自分支Ⅳ的Ghir_D05G019290基因在胚珠和纤维发育的不同时期都有表达,其中在0 DPA 胚珠中表达量最高, 在15 DPA 和20 DPA 纤维中的表达量高于10 DPA 纤维;Ghir_D06G000460基因在20 DPA 纤维的表达量低于在其他时期纤维的表达量; 来自分支Ⅴ的Ghir_A02G018470基因在整个胚珠发育时期都有表达;Ghir_D03G001140基因在20 DPA 纤维中的表达量低于其在其他时期胚珠及纤维中的表达量。以上结果表明部分SRS基因在棉花胚珠和纤维发育的不同时期都有表达,可能参与调控相关的生长发育过程。
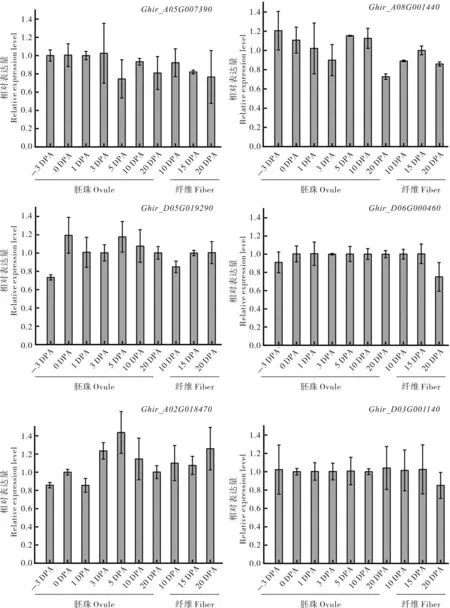
图6 陆地棉SRS 基因在胚珠和纤维不同发育时期的相对表达量Fig. 6 Relative expression level of cotton SRS genes in the ovule and fiber at the different developmental stages
3 讨论
棉花基因组测序的完成为加速棉花基因功能的研究奠定了基础。 近几年,调控棉花开花[38-40]、产量形成和纤维品质[41-43]等基因的研究被相继报道。SRS基因家族是植物中特有的转录因子[2],在调节植物生长发育和逆境响应中发挥重要作用,该家族基因已有部分成员的功能在拟南芥[8-10]、玉米[14,44]、杨树[22]、小立碗藓[45]等植物中被挖掘,但棉花中尚未见相关报道。 本文基于棉花基因组数据[24,46],采用生物信息学方法对棉花SRS基因家族成员进行鉴定, 对其蛋白质基序、系统进化关系及其在胚珠、纤维不同发育时期和不同组织中的表达模式等进行分析。
从陆地棉和海岛棉中分别鉴定了27 和26个SRS基因,进化分析显示位于同一进化树分支的成员具有相似的保守基序,表明他们可能具有相似的进化关系或功能(图1)。 例如:21 个棉花SRS基因与拟南芥LRP1和SRS6来自同一分支,LRP1和SRS6在根和花中活跃表达[15],并且LRP1可以通过调节生长素水平来影响侧根发育[2,5],推测棉花中的同源基因可能发挥类似的生物学功能。 大多数SRS基因中都含有基序1~4,表明这4 个基序可能是SRS基因家族成员发挥正常功能所必需的元件(图1B)。 大部分棉花SRS基因都比来自拟南芥的同源基因多了几个额外的基序, 在A10 和D10 号染色体上的基因比拟南芥SRS6多了基序18 和基序20,A03、D02 和A13、D13 号染色体上的基因比拟南芥SRS3多了基序9、14、17、20。 另外,基序16 和基序18 只存在于分支Ⅲ中, 表明这些基序可能与基因特有的功能相关。
SRS基因在陆地棉中的表达模式存在差异,说明这些基因可能参与不同的生物学过程或发挥不同的生物学功能。对陆地棉SRS基因在胚珠和纤维发育不同时期、不同组织的表达模式分析发现(图4),除了Ghir_D03G001140和Ghir_D05G019290基因外, 其他基因在叶片中几乎不表达,这与之前拟南芥、水稻中的研究结果一致。一些拟南芥SRS基因在花和根中高表达,但在叶片中不表达[2,15]。 水稻OsSHI1在根和穗中检测到较高表达量,但在叶片中未检测到[18]。 研究表明,LRP1 可能在拟南芥早期侧根发育过程中与SHI/STY 共同参与生长素介导的发育调控[4-5],并且LRP1和SRS6基因在花中均有表达, 可能也在开花时间的调节上发挥重要作用[2,8]。Ghir_D08G001480和Ghir_Scaffold3053G000010基因是LRP1和SRS6的同源基因, 在胚珠发育初期的-3 DPA、0 DPA、1 DPA 和3 DPA 阶段表达量低,在花药、花丝、雌蕊、萼片等组织中表达量相对较高,此外在低温胁迫、高温胁迫、盐处理和干旱胁迫处理中均有响应(图5),推测这2 个基因不仅参与胁迫响应还可能参与开花等植物发育过程的调控。
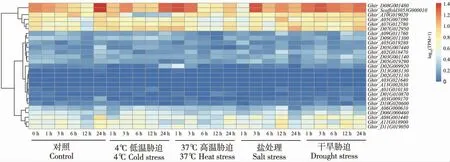
图5 陆地棉SRS 基因在多种胁迫处理下的表达Fig. 5 Expression profile of SRS genes under various abiotic stresses in G. hirsutum
4 结论
本研究在陆地棉和海岛棉中分别鉴定出27个和26 个SRS基因, 系统进化分析将其划分为3 个分支,不同分支都具有相似的保守基序。共线性分析显示棉花SRS基因家族中存在大量的同源基因。大部分SRS基因在胚珠发育初期表达量较高,一些基因也参与多种非生物胁迫响应。 以上结果为进一步探究SRS基因在棉花中的生物学功能特性奠定了基础并提供理论依据,但在调控棉花生长发育过程中的分子机制还需要进一步研究。
