全基因组测序和real-time PCR法检测食源性沙门氏菌parC、gyrA基因突变特征
2022-07-07毕旺来赵巍薇
毕旺来,赵巍薇,马 达,李 睿,*,周 敏
(1.武汉轻工大学生命与科学技术学院,湖北 武汉 430023;2.武汉轻工大学食品科学与工程学院,湖北 武汉 430023)
沙门氏菌(spp.)是主要的食源性致病菌之一,也是食品污染的常见指示菌,可通过动物性食品、蔬菜及水果等传播引起人类感染发病,引发肠胃炎、菌血症和伤寒等疾病。据报道,全球每年有超过1亿 人感染沙门氏菌,其中大多数人会在短期内自行恢复,但也有大约15万感染者死亡。我国的细菌性食物中毒有70%~80%是由沙门氏菌引起,抵抗力低下的人群,老年、婴幼儿尤其容易感染。沙门氏菌造成的食品安全问题在我国很常见。在不发达地区或者农村地区,由于当地人民卫生意识差,由聚餐引发的沙门氏菌食物中毒事件屡见报端。
除此之外,沙门氏菌的耐药现象也很严重。近年来,全国各地屡屡从鸡蛋、水产品、禽肉制品等食物中分离到多重耐药沙门氏菌,对食品安全防控造成了严重威胁。沙门氏菌耐药机制复杂,主要涉及灭活酶及钝化酶的产生、基因突变、细菌主动外排作用、可移动基因元件介导和生物膜的形成等,这些机制相互作用共同决定沙门氏菌的耐药水平。由于抗生素滥用、细菌耐药基因水平传播等原因,加剧了食品中耐药性致病菌的潜在风险。2020年11月20日,多个联合国机构共同宣布成立一体化卫生抗微生物药物耐药性领导人小组,以促进全球重视并采取行动,避免微生物耐药性造成的灾难性后果。
喹诺酮类药物是人畜通用的抗生素,包括第1代药物萘啶酸,第3代药物环丙沙星,第4代药物莫西沙星等。喹诺酮类耐药主要由喹诺酮耐药决定区(quinolone resistance-determining region,QRDR)基因突变导致。喹诺酮类药物的作用靶位为DNA促旋酶和拓扑异构酶IV,前者由2个GyrA亚基和2个GyrB亚基组成,分别由和基因编码;后者由2个ParC亚基和2个ParE亚基组成,分别由和基因编码。DNA促旋酶和拓扑异构酶IV的QRDR发生氨基酸突变可引起酶的结构与功能改变,影响喹诺酮类药物与靶位的结合,降低细菌对喹诺酮类药物的敏感性,从而导致耐药菌株的出现。此外质粒上的喹诺酮类耐药(plasmid mediated quinolone resistance,PMQR)基因也可介导喹诺酮类耐药,PMQR基因主要是编码Qnr蛋白的基因。
鉴于细菌耐药性日趋严重的现实,查明喹诺酮类耐药株QRDR和PMQR状况,有助于更好地了解沙门氏菌耐药性的产生和传播途径,为预防和控制沙门氏菌提供理论依据。目前国内外对耐药基因突变的研究还是以基因测序为主,耗费大、时间长。基于以上背景,本实验筛查食源性沙门氏菌喹诺酮类耐药株PMQR分布状况,并设计一种real-time PCR方法,结合全基因组测序方法对QRDR突变进行real-time PCR检测和测序验证。本实验结果将为耐药基因突变位点筛查提供新的准确快捷的检测方法。
1 材料与方法
1.1 菌株与试剂
大肠杆菌()ATCC25922、沙门氏菌()ATCC14028,神农架林区检验检测中心提供。120 株待检食源性沙门氏菌分离株2017—2020年间分离获得,其中92 株分离自江西南昌市冰冻禽肉制品,28 株分离自神农架林区冰冻禽肉制品。
细菌基因组提取试剂盒(TIANamp bacteria DNA Kit离心柱型) 天根生化科技有限公司;Gold view核酸染料 北京博大泰克生物基因技术有限公司;2×realtime PCR Mix 武汉楚诚正茂生物工程有限公司;PCR产物纯化试剂盒、DNA Marker 2000、TaKaRa试剂盒宝生物(大连)有限公司;引物由生工生物工程(上海)股份有限公司合成。
1.2 仪器与设备
DYY-10C电泳仪 北京六一仪器厂;恒温培养箱上海新苗医疗器械制造有限公司;梯度PCR仪 德国Biometra公司;台式高速冷冻离心机 德国Eppendorf公司;ABI7500型荧光定量PCR仪 美国应用生物系统公司;GBox-HR-E-M凝胶图像系统 英国Syngene公司。
1.3 方法
1.3.1 药敏实验
采用药敏纸片扩散法(K-B法)测定细菌药物敏感性,参考2018年临床和实验室标准协会(Clinical and Laboratory Standards Institute,CLSI)的《抗菌药物敏感性试验执行标准》进行。即用无菌棉拭子蘸取菌液,涂布MH平板表面。用无菌镊子将萘啶酸(5 μg/片)、环丙沙星(5 μg/片)药敏纸片贴在琼脂表面。纸片贴完15 min内,将平板倒置于37 ℃生化培养箱培养16~18 h。测量各个药敏纸片的抑菌圈直径。用大肠杆菌ATCC25922作质控株同步做质量控制。将各抗生素药敏纸片的抑菌圈直径与CLSI标准进行比对,报告待测细菌对该抗生素是否敏感(S)、中介(I)或耐药(R)。
1.3.2 全基因组二代测序
选取4 株南昌来源食源性沙门氏菌,菌株编号为3、4、31、62。将菌液制备成甘油管送上海美吉生物公司,采用Illumina HiSeq X-10平台进行二代测序、序列组装和基因功能注释。对4 株菌耐药基因进行深入挖掘。采用抗生素耐药基因数据库(http://arpcard.Mcmaster.ca,Version 1.1.3)和Resfinder 4.1(https://cge.cbs.dtu.dk/services/ResFinder/)对耐药基因进行搜索预测。
1.3.3 细菌核酸提取
将细菌用LB培养基37 ℃培养过夜,用细菌基因组提取试剂盒提取DNA。提取的DNA利用超微量分光光度仪测定DNA的含量,利用1%琼脂糖凝胶电泳检测DNA完整性,-20 ℃保存备用。
1.3.4基因PCR筛查
PCR扩增检测、、,引物参照文献[17]合成,序列见表1。PCR扩增体系(25 μL)为:DNA模板1 μL,上游引物和下游引物(10 μmol/L)各1 μL,TaKaRa r酶0.15 μL,10×Buffer 2.5 μL,dNTPMix(2.5 mmol/L)2 μL,ddHO补足总反应体系到25 μL。PCR扩增条件:94 ℃预变性5 min;94 ℃变性45 s,预定温度退火45 s,72 ℃延伸1 min,32个循环;72 ℃延伸10 min。其中、退火温度为53 ℃,退火温度为55 ℃。扩增得到的产物进行1.5%琼脂糖凝胶电泳,紫外灯下观察产物片段大小进行验证。基因产物大小516 bp;基因产物大小417 bp;基因产物大小469 bp。
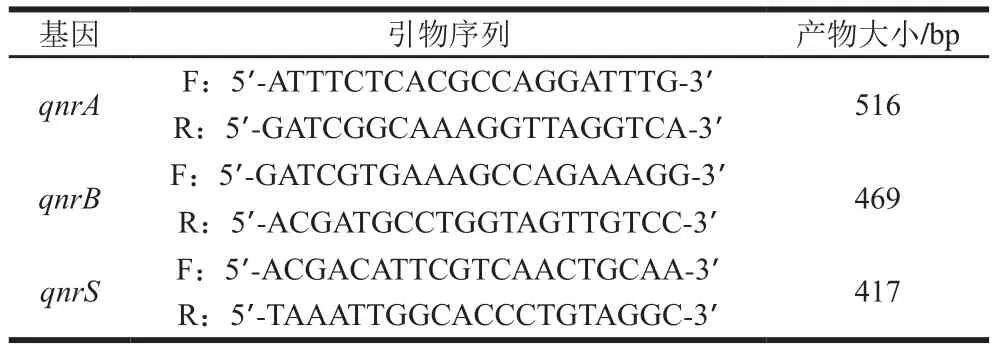
表1 qnr基因PCR检测引物Table 1 PCR primers for qnr gene
1.3.5 real-time PCR引物设计
将4 株测序的沙门氏菌进行序列组装后,分析、基因序列及基因突变位点的信息,食源性沙门氏菌包含有4个常见的突变位点,分别为Asp87Tyr,核苷酸GAC→TAC;Asp87Asn,核苷酸GAC→AAC;Thr57Ser,核苷酸ACC→AGC;Ser80Ile,核苷酸AGC→ATC。使用在线软件对4个突变位点设计特异性引物(http://cedar.genetics.soton.ac.uk/public_html/primer1.Html)。上游引物3’末端为待检验的突变位点,用下划线标识。为提高引物特异性,在上游序列靠近3’末端人为引进碱基错配,以减少引物和模板间形成的二级结构自由能值。碱基错配也用下划线标识。引物序列见表2。

表2 基因突变位点real-time PCR检测引物Table 2 Real-time PCR primers for mutation sites
1.3.6 real-time PCR扩增
对提取得到的沙门氏菌基因组DNA分别进行Asp87Tyr、Asp87Asn、Thr57Ser和Ser80Ile四个位点real-time PCR扩增筛查,以不含以上突变位点ATCC14028菌株作为阴性对照,以含有上述突变位点的测序菌株作为阳性对照。扩增结束后查看熔解曲线和扩增曲线。预实验中扩增得到的产物进行琼脂糖凝胶电泳,紫外灯下观察产物片段大小。根据4个位点设计的real-time PCR引物见表2。
PCR体系(25 μL):DNA模板1 μL、上游引物和下游引物(10 μmol/L)各0.5 μL、2×SYBR Green realtime PCR Mix 12.5 μL、ddHO 10.5 μL。PCR扩增条件:95 ℃预变性2 min;95 ℃变性10 s,预定温度退火15 s,72 ℃延伸30 s,40个循环。Asp87Tyr位点检测的退火温度为56.2 ℃,Asp87Asn位点检测的退火温度为55.5 ℃,Thr57Ser位点检测的退火温度为61.6 ℃,Ser80Ile位点检测的退火温度为56.8 ℃。
1.3.7 QRDR位点的测序验证
将待验证的10 株沙门氏菌分别进行、基因全长扩增,上游引物为CGCCTACTTAAACTACTCCA,下游引物为GCCGACCACCTTCTGTA;上游引物为TGCCCGTGTCGTTGGT,下游引物为GTCGCACGAGACTTGG。
PCR扩增体系(25 μL)为:DNA模板1 μL,上游引物和下游引物(10 μmol/L)各1 μL,TaKaRa r酶0.1 μL,10×Buffer 2.5 μL,dNTPMix(2.5 mmol/L)2 μL,ddHO 17.4 μL。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s,52 ℃退火45 s,72 ℃延伸1 min,32个循环;72 ℃延伸10 min。1.5%琼脂糖凝胶电泳检测PCR产物,然后用PCR产物纯化试剂盒纯化,送生工生物工程(上海)股份有限公司进行双向测序验证QRDR位点。将测序原始数据采用DNASTAR软件进行拼接,查找并验证real-time PCR检测出来的QRDR位点。
2 结果与分析
2.1 喹诺酮类药物敏感性实验和qnr基因筛查
药敏实验结果证实,120 株食源性沙门氏菌分离株有45 株菌耐喹诺酮类抗生素萘啶酸,其中11 株来源于神农架,34 株来源于江西南昌。3 株神农架株、8 株南昌株同时耐环丙沙星,环丙沙星耐药率分别为10.7%(3/28)、8.7%(8/92)。神农架株对喹诺酮类药物耐药率为39.3%(11/28),南昌株对喹诺酮类药物耐药率为37%(34/92)。两地菌株喹诺酮类药物耐药率非常接近。
将4 株南昌株送样进行二代测序,序列组装、基因功能注释后进行耐药基因检测,结果仅4号菌株检出携带基因,未检出其他亚型。以余下的41 株喹诺酮耐药沙门氏菌为研究对象,PCR筛查喹诺酮耐药基因、、。图1、2为部分沙门氏菌DNA提取和基因检测PCR产物电泳检测结果。南昌株有8 株PCR检出耐药基因,检出率为23.5%(8/34);神农架株有6 株检出耐药基因,检出率为54.4%(6/11)。而、耐药基因检出率均为0%。
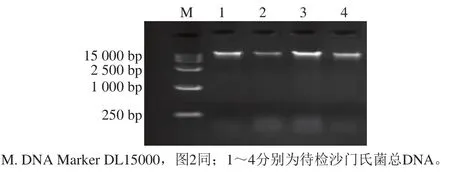
图1 沙门氏菌总DNA电泳结果Fig. 1 Agarose gel electrophoresis of total DNA extracted from Salmonella isolates
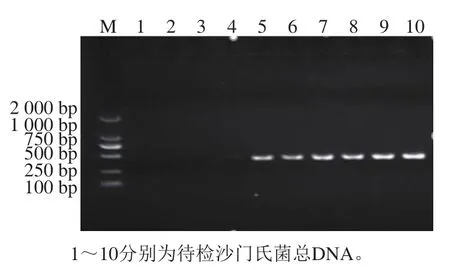
图2 qnrS基因PCR扩增结果Fig. 2 PCR amplification products of qnrS gene
2.2 real-time PCR扩增检测gyrA和parC基因突变
对本课题组测序的4 株南昌株进行QRDR位点分析。通过与Resfinder数据库比对,发现4 株沙门氏菌和没有突变位点,但和基因均有数个QRDR突变位点。其中有4个突变位点是文献常报道的QRDR位点。除了测序的3号菌不含有上述4个QRDR位点,4号菌携带Thr57Ser突变,31号菌携带Asp87Tyr突变,62号菌同时携带Thr57Ser+Ser80Ile+Asp87Asn突变。
根据这4个QRDR位点设计real-time PCR检测引物。经过PCR筛查,45 株耐喹诺酮的沙门氏菌中有31 株未检出基因,推断这些菌株和基因应存在QRDR位点突变,从而导致喹诺酮耐药。因此对这31 株菌采用real-time PCR法检测和基因QRDR位点。
real-time PCR检测的熔解曲线图和扩增曲线见图3~6。样品熔解曲线均形成单一峰,说明无非特异性扩增。样品real-time PCR扩增Ct值在16~28之间,扩增效率良好。阴性对照Ct值大于35,说明real-time PCR无污染,无假阳性。二代测序的4 株菌进行real-time PCR检测后,根据测序所得和基因QRDR位点,与realtime PCR结果进行分析比对,结果表明这4个菌real-time PCR扩增结果和测序结果吻合,说明real-time PCR特异性良好。
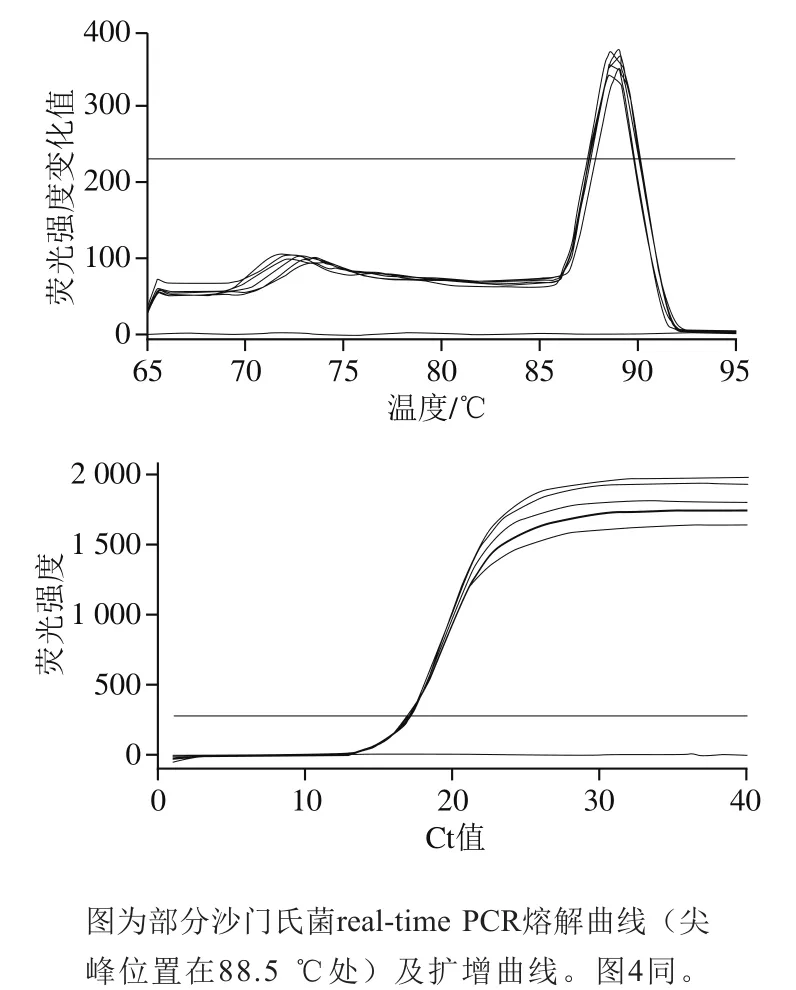
图3 real-time PCR检测parC Thr57Ser突变位点Fig. 3 Real-time PCR detection of parC Thr57Ser
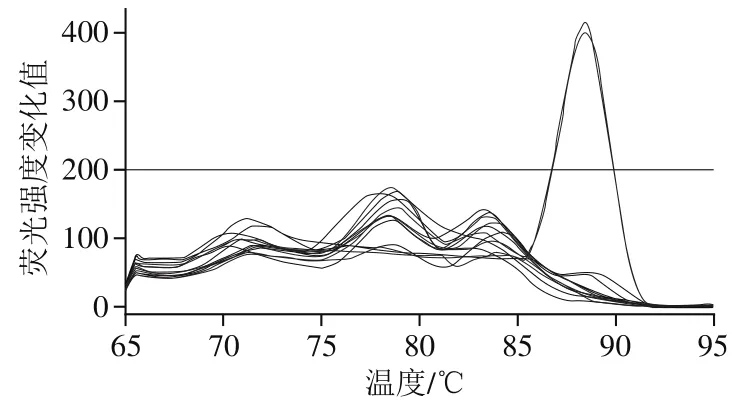
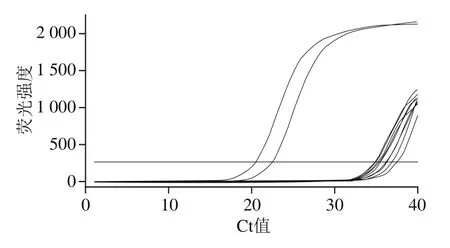
图4 real-time PCR检测parC Ser80Ile突变位点Fig. 4 Real-time PCR detection of parC Ser80Ile
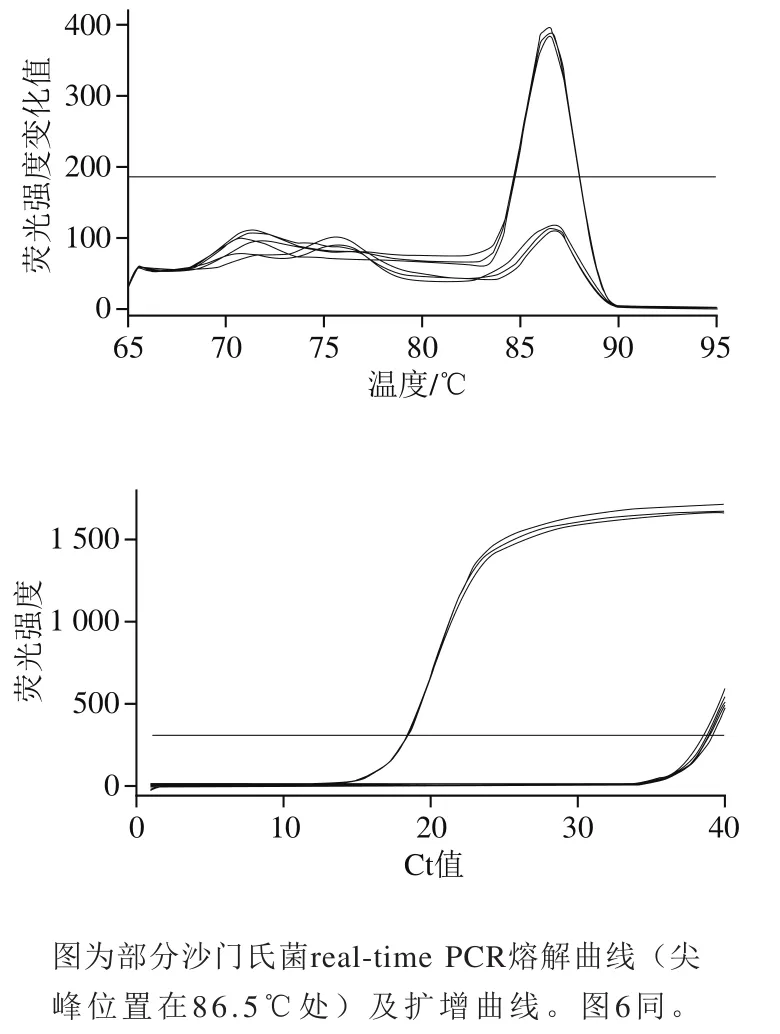
图5 real-time PCR检测gyrA Asp87Asn突变位点Fig. 5 Real-time PCR detection of gyrA Asp87Asn
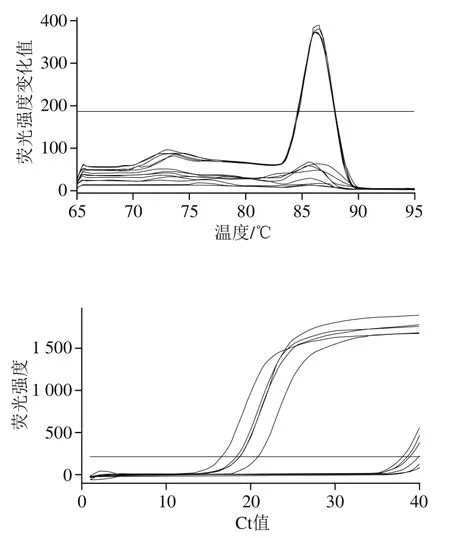
图6 real-time PCR检测gyrA Asp87Tyr突变位点Fig. 6 Real-time PCR detection of gyrA Asp87Tyr

表3 31 株沙门氏菌基因突变位点检测结果Table 3 Gene mutation sites of 31 Salmonella isolates
如表3所示,31 株沙门氏菌除2 株未检出QRDR突变外,其他菌株均有检出。其中Thr57Ser+Asp87Asn型突变最常见,菌株检出数目最多。
为进一步验证real-time PCR的准确性,挑选10 株real-time PCR阳性菌株分别扩增和全长,纯化后送生物公司双向测序。部分PCR产物如图7所示。将测序数据进行拼接比对,找寻和基因突变位点,测序结果与real-time PCR结果高度吻合,进一步证实了realtime PCR的可靠性。
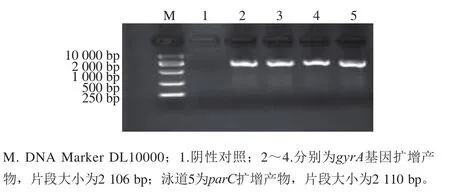
图7 parC、gyrA基因全长PCR扩增结果Fig. 7 PCR amplification of the complete sequences of parC and gyrA
3 讨 论
沙门氏菌耐药日益严重,相对于其他抗生素类药物,沙门氏菌对喹诺酮类药物敏感性相对较高。本实验发现因此喹诺酮类三代药物如环丙沙星仍是沙门氏菌感染治疗的推荐药物之一。
沙门氏菌对喹诺酮类耐药主要由QRDR突变和PMQR介导引起。目前基因单核苷酸多态性(single nucleotide polymorphism,SNP)的检测方法可以分为两类:一类是以单链构象多态性、等位基因特异性PCR等为代表的以凝胶电泳为基础的传统方法。另一类是以直接测序、变性高效液相色谱、高分辨率溶解曲线等为代表的高通量检测方法。近年来部分学者研发了一些筛查QRDR突变的新技术,比如单链构象多态性分析、变性高效液相色谱、高分辨率溶解曲线法等。这些技术操作要求高,费用高,而且主要是检测已知的基因突变位点。目前多数文章研究QRDR突变采取的还是基于测序的方法。Takashi等采取Illumina平台二代测序方式研究了107 株伤寒沙门氏菌QRDR突变,识别出2 461个SNP位点。其中高耐药菌含有3个QRDR突变,Ser83Phe、Asp87Asn和Ser80Ile。游兴勇等采取二代全基因组测序分析了2018年江西省临床分离的58 株非伤寒沙门菌,这些菌存在不同程度的QRDR突变。其中突变率最高(31.03%,18/58),且全部是Thr57Ser突变,其次是(27.59%,16/58)突变,最常见的有Asp87Asn(4 株)和Asp87Tyr(8 株)突变。葛琨等采取测序的方法研究了30 株乌鲁木齐牛羊肉源沙门氏菌QRDR基因突变状况,发现14 株沙门氏菌发生Ser83Phe基因突变,25 株发生了Thr57Ser基因突变。刘贵深等发现16 株耐环丙沙星沙门氏菌常见突变类型是Ser83Phe和Asp87Gly变异,同时伴随Ser80Arg变异。
以上文献结果与本实验有很多相似之处,也有不同之处,比如发现2 株南昌株携带了Ser80Ile突变。游兴勇等研究的58 株南昌临床株均未发现该突变。本实验发现Thr57Ser和Asp87Asn很常见,南昌株有22 株发生Thr57Ser突变,15 株发生Asp87Asn突变,这个结果和游兴勇等的研究相似。在本实验中,120 株沙门氏菌中有45 株对萘啶酸耐药,其中仅14 株菌检出基因。所有沙门氏菌均未检出、两种亚型。游兴勇等发现58 株江西来源临床株PMQR基因携带率最高为(29.31%,17/58),有3 株菌检出(5.17%,3/58)。这一结果和本实验不同。沙门氏菌耐药状况复杂,因此耐药株来源、分离时间、样本数量等,都会对研究结果造成一定影响。
本实验没有对阳性的菌株进行real-time PCR检测,有的文献报道部分沙门氏菌菌株同时具有QRDR和PMQR,如果加上这部分菌株,发生QRDR突变的菌株检出率会更高。此外QRDR突变比较复杂多样,比如经测序发现3号菌株虽然没有Asp87Tyr、Asp87Asn、Thr57Ser和Ser80Ile这4个文献常报道的QRDR突变,但具有Thr748Met、Thr255Ser、Asn395Ser、Ala620Thr和Ser469Ala突变。这些突变是否能影响菌株对喹诺酮药物的敏感性尚需验证。
在耐药机制研究上全基因组测序具有较大的优势,可识别新的耐药基因及其突变。但单纯采用全基因组测序的方法跟踪研究耐药基因费用昂贵、时间长。本实验建立了一种real-time PCR的方法检测QRDR突变。对31个无PMQR的喹诺酮耐药株筛查Asp87Tyr、Asp87Asn、Thr57Ser和Ser80Ile这4个文献常报道的QRDR突变,采取已经二代测序的4 株菌作为对照,并随机挑选有real-time PCR阳性产物的10个菌扩增基因和基因全长,结果这14个菌的realtime PCR结果和测序结果完全吻合。说明本实验建立的real-time PCR方法特异性强、准确可靠。采取本实验的引物设计思路还可以建立筛查其他各种耐药基因突变的real-time PCR方法。采用全基因组测序和real-time PCR联用的方法既可以发现新的耐药基因及其突变,又可以筛查大量的样本,成本低、耗时短。
沙门氏菌耐药机制复杂,目前全球处于新冠后疫情时代,各地大规模消毒可能会诱导环境中细菌产生新的耐药性。因此对环境-食品-人这一链条上细菌耐药性变化的监控力度应该加大。全基因组测序结合realtime PCR检测,可应用于跟踪细菌耐药基因突变的研究,更快的识别新出现的耐药危害,有助于及时制定公共卫生的风险控制策略。
