基于Cas9靶向富集的纳米孔高通量基因测序技术的应用进展
2022-06-20谢水莲万绍贵冷小敏
杨 影,谢水莲,万绍贵,冷小敏
(1. 赣南医学院2021级硕士研究生;2. 赣南医学院2020级硕士研究生;3. 赣南医学院基础医学院;4. 赣南医学院基础医学院分子病理中心,江西 赣州 341000)
高通量测序技术已广泛应用于遗传性疾病和癌症的研究,但因成本相对较高阻碍了其在基因组水平上大规模基因变异检测的应用[1]。为提高基因检测效率,需提高测序深度和全面变异检测的方法,通过针对感兴趣的区域(Region of interest,ROI)或基因进行靶向富集然后再高通量测序的方法应运而生[2]。当前高通量测序的靶向富集方法主要有两种方式:第一是杂交捕获法,主要利用探针杂交富集目标片段,适用于基因组目标区域的全面检测,但依赖于成百上千个寡核苷酸探针的设计、复杂的微阵列芯片制造和较长的杂交时间[3];第二是多重PCR 扩增法,其核心是引物设计,先通过PCR扩展富集目标片段,再进行文库构建,适用于研究的目标区域相对较小,对于拷贝数较低的模板DNA,可产生足够数量用于测序的扩增子,这种方法能明显提高效率,节约时间,降低经济成本,不足之处在于存在引物互相干扰和非特异性扩增等问题[4]。这两种方法不仅过程繁琐、成本昂贵,还受PCR 扩增效率影响导致基因组的复杂区域和高GC区域会难以覆盖。近年来,CRISPR/Cas9 靶向富集的出现扩大了高通量测序技术在临床方面的应用范围,通过对ROI 进行Cas9 特异性靶向切割富集,提高了ROI 的覆盖深度,显著减少背景基因的测序数据,从而达到了便于分析后续数据的目的[5]。该方法具有无PCR 扩增、保留了碱基修饰的信息、实现了高测序深度、低错误率和低成本的长读长测序等优点。基于Cas9 靶向富集纳米孔基因测序技术通过对样本目标区域的研究,已被应用于获得高覆盖度和高质量特定基因组区域的序列信息,利于发现和证明与疾病相关的候选基因和位点,在遗传变异和全基因组测序等方面已得到广泛应用。本文对Cas9 靶向富集纳米孔测序技术的原理和应用进行了回顾,并重点阐述该技术在融合基因检测中的最新进展。
1 基于Cas9 靶向富集纳米孔基因测序技术的基本原理
CRISPR/Cas(Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR-associated)是 细菌中一种应对噬菌体和质粒等外来DNA 的防御系统[6]。在某些细菌基因组中存在“规律间隔成簇短回文重复序列”(Clustered Regularly Interspaced Short Palindromic Repeats,CRISPR),这些序列被转录成为RNA,也被称为导向RNA(guide RNA,gRNA)。gRNA 能和细菌体内Cas 蛋白质形成复合体,并对Cas 蛋白有导向作用。当复合体检测到入侵的DNA 和gRNA 序列一致时,Cas 蛋白就能切割降解入侵的DNA,达到防御的目的[7]。CRISPR/Cas9是基于CRISPR/Cas 构建的一种基因编辑技术,在基因编辑方面具有高效、快速、简单等特点。该系统是一个Cas9 核糖核蛋白(ribonucleoprotein,RNP),包含Cas9 蛋白和sgRNA(crRNA-tracrRNA 的 嵌合体)[8]。其中tracrRNA 具有茎环结构,与Cas9 蛋白结合;crRNA 引导Cas9定位到要编辑的DNA 序列附近。在进行基因编辑时,crRNA 先与基因组上的靶序列结合,然后RNP 识别并切割靶序列[8]。RNP 识别和剪切位点附近具有前间隔序列邻近基序(Protospacer Adjacent Motif, PAM),它是三个紧挨着的碱基序列,最常用的是NGG(N 是任意一种核苷酸序列,G是鸟嘌呤)。RNP识别出与crRNA 互补的原间隔序列(PAM序列),然后解开DNA双链,形成R-loop环,使crRNA与互补链碱基配对,另外一条单链仍然保持游离的状态;接着Cas9中的HNH(His-Asn-His)活性位点剪切与crRNA互补配对的DNA单链,RuvC活性位点剪切非互补链;最终使DNA 双链断裂,形成磷酸化的切割端口[6-7,9]。
纳米孔测序技术采用“边解链边测序”的方法,基于电信号的变化识别相应的碱基序列。纳米孔是该技术的核心,它是一个外表面由脂质双分子层且两端分别各有一对电极的跨膜蛋白构成的纳米孔通道。双链DNA 分子在马达蛋白的作用下解螺旋并在其牵引作用下通过纳米孔,不同的碱基会产生不同的偏转电流,最后通过电流的变化特征实现对不同碱基的判定[10-11]。Cas9靶向富集纳米孔基因测序技术是Cas9 和纳米孔基因测序技术的新结合,该技术利用Cas9靶向切割ROI上游(5')和下游(3')的侧翼序列进行纳米孔测序[12]。CRISPR/Cas9 系统的特异性靶向切割和富集ROI 片段的主要步骤分为:⑴对样本DNA 进行去磷酸化处理以避免后续的非目标区域连接;⑵使用Cas9 复合物对选定ROI 进行切割,露出可连接目标端;⑶对所有的3'端均进行加A尾,与此同时将测序接头只连接在切割端上;⑷将整个文库添加到测序芯片上进行纳米孔测序(图1)[13-15]。这样,对应ROI 将会得到富集,降低测序成本,加之纳米孔测序技术测序片段长和快的特点,使得Cas9 靶向富集纳米孔基因测序技术在基础和应用研究中发挥了巨大作用。

图1 Cas9靶向切割富集示意图
2 基于Cas9 靶向富集纳米孔基因测序技术的主要应用场景
Cas9 靶向富集纳米孔基因测序能捕获并检测到多个区域中未经扩增的DNA 片段,在融合基因、结构性变异和癌症驱动基因的甲基化检测等方面发挥了非常重要的作用(表1)。为了评估TP53、KRAS 和BRAF 的单核苷酸位点变异(Single-nucleotide variants,SNVs),GILPATRICK T 等[16]使用纳米孔Cas9靶向测序(nanopore Cas9-targeted sequencing,nCATS)不需扩增的策略,检测了淋巴母细胞系和乳腺细胞系,结果表明,nCATS 不仅可对目的基因DNA 片段进行富集和长读长测序,且可用于SNVs的检测。在检测基因的甲基化方面,nCATS 也展现了非常明显的优势,该团队通过该方法评估三种乳腺细胞系中一系列靶基因发生甲基化的能力,角蛋白家族成员基因KRT19 作为乳腺癌向淋巴结微转移的标记物,常用于循环肿瘤的检测,KRT19的甲基化表达具有显著差异,在非致瘤性MCF-10A 细胞系中保持高甲基化;在致瘤性MCF-7 和MDA-MB-231乳腺细胞系中为低甲基化[16]。WONGSURAWAT T等[17]利用nCATS 同时检测4种人类胶质母细胞瘤(GBM)细胞系和8份新鲜的人类脑瘤样本中的脱氢酶(IDH)基因突变状态和O6-甲基鸟嘌呤-DNA 甲基转移酶(MGMT)启动子的甲基化水平,nCATS 在36 小时内准确检测IDH1 和IDH2 突变,结果与Sanger 和Illumina 测序数据一致。检测到的MCMT甲基化百分比与Illumina 的结果存在显著的一致性,并与质谱分析检测的CpG 位点相同[17]。另外,GABRIELI T 等[18]使用Cas9 靶向染色体片段(Cas9-Assisted Targeting of Chromosome,CATCH)的方法捕获一个200 kb 的侧翼区域和调控区域大的DNA 片段进行测序,检测出NGS 未检测到的乳腺癌和卵巢癌基因BRCA1中存在的单核苷酸多态性(Single-nucleotide polymorphisms,SNPs)。由此可见,这些先前描述的研究表明,将nCATS 作为癌症精准医疗的临床工具有很大的潜力,该方法只需约3µg 的基因组DNA,可在一次实验中靶向富集大量的位点,实现细胞和组织的低成本、低基因组起始量检测目标区域的碱基修饰和基因组结构的改变。由于融合基因的检测在临床诊疗方面具有重要的指导意义,我们将重点介绍Cas9 靶向富集纳米孔基因测序对融合基因的检测应用。
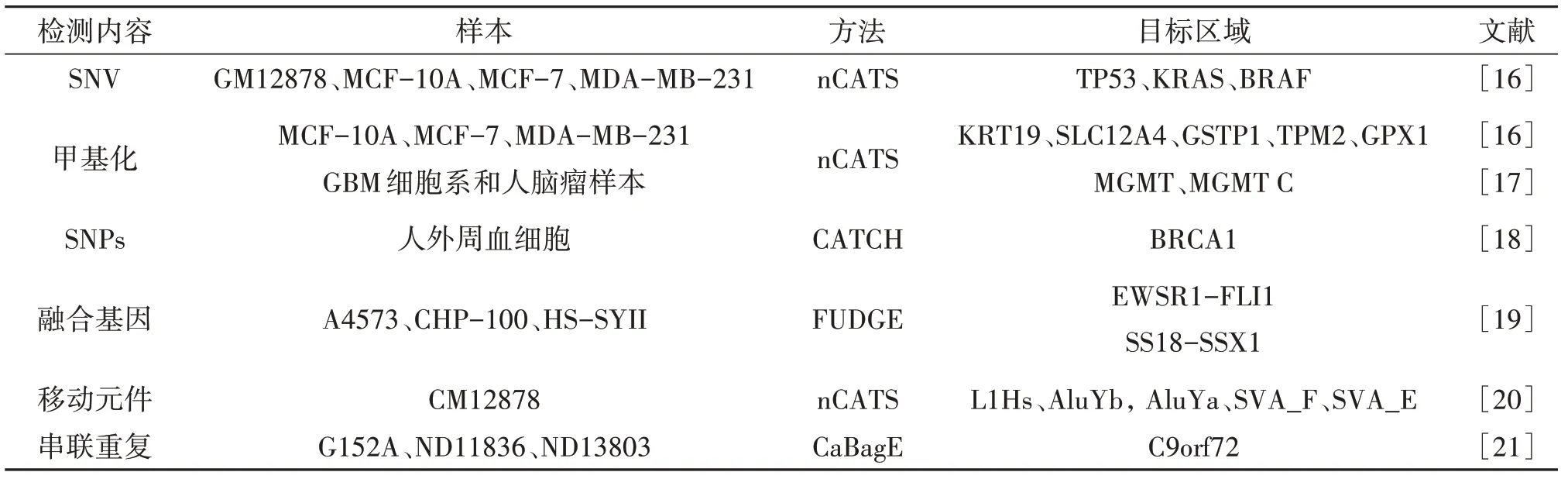
表1 基于Cas9靶向富集纳米孔基因测序技术的应用
3 基于Cas9 靶向富集纳米孔基因测序技术检测融合基因
染色体倒置、串联重复、间质缺失或易位将不同的、独立的基因或基因片段串联在一起形成融合基因。作为癌症的常见驱动因素,约20%癌症导致的死亡与融合基因有关。因此,融合基因可作为多种癌症诊疗的标志物[22-23]。例如,CCDC6/RET 融合基因的过度表达会促进乳头状甲状腺癌的发生[24];编码酪氨酸激酶受体的基因(ALK[25]、ROS1[26]等)与其他基因的融合(EML4/ALK、CD47/ROS1 等)会激活下游细胞信号通路,引发细胞无限增殖,最终导致细胞癌变[27-28]。临床上已将一些与癌症发生发展有关的融合基因用于临床诊疗参考,其中最著名的是慢性粒细胞白血病(chronic myelocytic leukemia,CML)中的BCR/ABL1 融合基因的发现,临床开发出了靶向该融合基因的小分子抑制剂甲磺酸伊马替尼,该抑制剂在CML 患者中有显著疗效,有效延缓了CML 患者病情发展[29-30]。此外,靶向实体瘤中致癌基因融合治疗也取得了成功,例如抑制非小细胞肺癌中的ALK和ROS1融合基因,NTRK抑制剂拉罗替尼已被FDA 批准用于NTRK1/2/3 所有类型的融合阳性实体瘤[31]。目前临床上经典的融合基因检测手段包括:免疫组化(Immunohistochemistry,IHC)[32]、荧光原位杂交(Fluorescence in situ hybridization,FISH)[33]、反转录-聚合酶链反应(Reverse transcription-polymerase chain reaction,RT-PCR)[34]、第二代基因测序技术(Next-generation sequencing,NGS)等[35]。
对于融合位点和融合伴侣高度可变的融合基因、长片段的串联重复序列等,经典的检测手段不能准确识别。其中IHC染色是在蛋白质水平上对融合蛋白进行检测,但对于小间隙缺失或倒置检测不敏感,无法直接检测融合基因;FISH、RT-PCR、NGS则是在核酸水平上对融合基因进行检测,FISH 虽然敏感性较好,但操作复杂、技术要求高、只针对阳性融合基因检测,这种检测比较浪费时间[36];RT-PCR的不足之处是只能研究一个融合基因,且只能检测已知的融合基因[35,37]。尽管NGS常用于新的融合基
因检测,但其测序读长短、依赖于PCR 扩增、受GC碱基含量影响大,在复杂的融合基因检测中仍存在很大的困难[4]。Cas9靶向富集纳米孔基因测序技术通过提高测序覆盖度和深度可发现肿瘤中新的融合基因,且能用于复杂结构变异的解析和识别发现新的变异[16,38]。STANGL C 等[19]开发的基因富集融合 检 测(Fusion Detection from Gene Enrichment,FUDGE)技术通过基因靶向富集与纳米孔测序相结合,能在48 小时内快速定位融合伴侣和断点位置,完成融合基因的检测。该技术不仅可检测已知的融合基因及其断裂点,基于其中一个伴侣基因的信息,还能检测出尚未发现的融合伴侣和断点。并且基于共有的融合片段去设计切割靶点的引物,该技术实现了同时检测多个伴侣的融合基因[16]。该方法已成功应用于AML、Ewing 肉瘤、结肠癌等癌症中融合基因的快速识别[16]。为了鉴定新的融合基因,研究人员将该技术应用于检测已知融合基因的肿瘤细胞系,检测的细胞与对应的融合基因分别为尤文氏肉瘤(Ewing's sarcoma,ES)细胞株A4573 和CHP-100 中的EWSR1-FLI1 融合基因、滑膜肉瘤HS-SYII 细胞系中的SS18-SSX1 融合基因[39]。在对A4573、CHP-100 和HS-SYII 的检测中,靶向切割融合基因所在的目标区域后覆盖率分别增加到了81x(A4573)、66x(CHP-100)和11x(HS-SYII)[18]。同时,EWSR1-FIL1融合基因的不同断点位置得到了新的鉴定。通过设计序列crRNA来跨越PAX3和PAX7的断点区域进行测序,检测出了新的融合基因和断点:FOXO1-PAX3 和DRICH1-BCR[18]。FISH 可 诊 断 出KMT2A 存在融合,但无法检测出融合伴侣。NGS 可识别包括启动子融合在内的所有融合基因和断点位置,但受限于测序读长短、样本使用量大和周转时间长。而FUDGE能在48小时内检测出MTTL6基因为融合伴侣。且可使用低通量的细胞和单个样品进行检测,无需PCR 扩增,从而减少了工作的流程,显著降低了检测成本[18]。
4 总结与展望
随着高通量测序技术的不断发展,纳米孔测序技术发展迅速且具有读长长、实时数据监测、简单便捷等特点,在基因修饰识别、表观遗传学研究、微生物检测、癌症诊断及相关医学检测方面,发挥着越来越重要的作用。CRISPR/Cas9 靶向富集测序技术的出现,使单次快速读取更长分子的碱基序列和大规模样本的基因组目标区域测序成为现实。该技术有针对性地测序使研究人员能富集ROI,显著降低测序成本和劳动力。在通过对感兴趣的基因组区域获得高覆盖度数据,该技术使研究人员可快速识别异质样本中的突变频率和甲基化模式。尤其是在只知道其中一个融合伴侣的保守序列情况下,该技术可同时检测多个融合伴侣的断裂位点,并对相应位点进行其结构分析。然而,Cas9 靶向纳米孔测序在检测方法和后续的生物信息学方面仍需完善,例如:对于重复序列的测序,如何得到较短的等位基因片段且不会产生偏差;如何利用更少的细胞样本进行DNA 水平的分析;对于ROI,怎样使Cas9 切割的脱靶效率降低;如何使生物信息学工具更易于使用。随着三代测序技术的应用推广和检测方法的推陈出新,Cas9 靶向富集纳米孔基因测序将在临床检测应用方面拥有更广阔的前景。
