基于高通量测序的带鱼肌肉组织转录组微卫星信息分析
2022-06-15刘玉萍王棋黄新芯徐开达王忠明高天翔杨天燕
刘玉萍 王棋 黄新芯 徐开达 王忠明 高天翔 杨天燕
摘要:【目的】通過高通量测序平台对带鱼肌肉组织进行转录组测序,从海量数据中查找微卫星(SSR)位点并进行生物信息学分析,为科学制定带鱼种质资源保护对策和管理措施提供参考依据。【方法】基于Illumina HiSeqTM 2500高通量测序平台对采自浙江舟山近海的带鱼肌肉组织进行转录组测序,经FastQC和Trimmomatic进行数据质量评估和过滤后,使用Trinity组装、去冗余得到Unigenes,再运用Micro-Satellite(MISA)挖掘SSR信息,并以Excel 2010计算SSR的数目、发生频率、出现频率、分布距离与密度、重复基元类别及重复区段长度等信息。【结果】带鱼肌肉组织转录组测序共获得40424018条Raw reads,经Trinity从头组装获得70113条Transcripts,去冗余后得到50482条Unigenes,其总长度为33886190 bp,平均长度为671.25 bp。使用MISA进行筛选,结果共发现18873个SSR位点,且这些SSR位点仅分布在其中的13082条Unigenes上,发生频率为25.91%,出现频率为37.39%。SSR按核苷酸重复类型进行分类,可分为单核苷酸重复、二核苷酸重复、三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复6种类型,以单核苷酸重复SSR数最多(10763个),出现频率高达21.32%;带鱼肌肉组织转录组SSR中共检测出173种重复基元,以四核苷酸重复基元种类最多(66种)、单核苷酸重复基元种类最少(4种)。单核苷酸重复中以A碱基的数量最多,有5167个(占48.09%);二核苷酸重复基元以TG为主,有692个(占19.55%);三核苷酸重复基元中以GAG为主,有206个(占10.08%);四核苷酸重复基元出现频率较高的有AAAC、ATGG、ATGT、CTGT、CTTT和TCCA,出现频率均为3.64%;五核苷酸重复和六核苷酸重复的基元类型数量分布较均匀,无明显优势重复基元。SSR基元重复次数主要分布在5~6次和10~12次,共9864个,占SSR总数的52.27%。【结论】经高通量测序获得的带鱼肌肉转录组SSR可用性高且具有较高的多态性潜能,在此基础上可有针对性进行引物设计,为带鱼遗传多样性评价、遗传结构分析及功能基因克隆等研究提供有效的分子标记,进而为其种质资源的保护与利用提供遗传学数据资料。
关键词: 带鱼;肌肉组织;微卫星(SSR);转录组;高通量测序
中图分类号: S917.4;S965.326 文献标志码: A 文章编号:2095-1191(2022)03-0725-10
Bioinformatic analysis of microsatellite loci in the muscle transcriptome of Trichiurus lepturus based on
high-throughput sequencing
LIU Yu-ping WANG Qi HUANG Xin-xin XU Kai-da WANG Zhong-ming GAO Tian-xiang YANG Tian-yan
(1 Fisheries College, Zhejiang Ocean University, Zhoushan, Zhejiang 316022, China; 2 Marine Fishery Research
Institute of Zhejiang Province, Zhoushan, Zhejiang 316021, China)
Abstract:【Objective】Transcriptome sequencing of Trichiurus lepturus muscle tissue was carried out based on high-throughput sequencing platform. Microsatellite (SSR) loci were searched from massive data and bioinformatics analysis was conducted to provide references for germplasm resources protection and management of T. lepturus. 【Method】The Illumina HiSeqTM 2500 high-throughput sequencing platform was used to sequence the muscle transcriptome of T. lepturus collected from the coastal waters of Zhoushan, Zhejiang Province. The quality of high throughput sequencing data was evaluated and filtered by software FastQC and Trimmomatic. Unigenes were obtained through assembly and redundancy removal using software Trinity. Micro-Satellite (MISA) tool was performed to explore the SSR information. Excel 2010 was used to calculate the number, occurring frequency, appearing frequency, distribution distance and density, repeat motif and repeat length of SSR. 【Result】A total of 40424018 raw reads were generated from muscle transcriptome sequencing. About 70113 transcripts were obtained by de novo assembly using Trinity, and 50482 unigenes were retained after deduplication, with a total length of 33886190 bp and an average length of 671.25 bp. A total of 18873 SSR loci distributing among 13082 unigenes were screened by MISA, with the occurrence frequency of 25.91% and the occurrence frequency of 37.39%, respectively. SSR was classified according to the types of nucleotide repeats, which could be divided into six types:Mononucleotide repeat, dinucleotide repeat, trinucleotide repeat, tetranucleotide repeat, pentanucleotide repeat and hexanucleotide repeat. The number of mononucleotide repeat was the largest (10763), with the occurrence frequency up to 21.32%. A total of 173 repeat motifs were detected, in which the number of tetranucleotide repeat motifs (66) was the largest and the number of mononucleotide repeat motifs (4) was the smallest. Among the mononucleotide repeats, the number of A was the largest (5167, 48.09%); TG was the main dinucleotide repeat (692, 19.55%); GAG was the main trinucleotide repeat (206, 10.08%); AAAC, ATGG, ATGT, CTGT, CTTT and TCCA were the most frequent repeats with the same frequency of 3.64%. Pentanucleotide repeats and hexanucleotide repeats were evenly distributed, and no obvious dominant repeat motifs were observed. The number of repeat motifs (9864) was mainly distributed in 5-6 and 10-12 times, accounting for 52.27% of the total number of SSR. 【Conclusion】The SSRs obtained by high-throughput sequencing have higher availability and polymorphism potential. On this basis, researchers can targetedly design primers and provide effective molecular markers for the studies of genetic diversity evaluation, genetic structure analysis and functional genes cloning of T. lepturus, and then provide genetic data for further protection and utilization of its germplasm resources.FF6A4387-7446-4F61-9047-3F7726652A6C
Key words: Trichiurus lepturus; muscle tissue; microsatellite (SSR); transcriptome; high-throughput sequencing
Foundation items: Zhejiang Key Research and Development Project(2019C02056); Zhoushan Science and Technology Project (2022C41022); Open Foundation for Marine Sciences of Zhejiang Ocean University in the First-Class Subjects of Zhejiang (20180017)
0 引言
【研究意义】带鱼(Trichiurus lepturus)又称刀鱼或肥带等,隶属于鲈形目(Perciformes)带鱼科(Tri-chiuridae)带鱼属(Trichiurus),生活在温暖水域,我国的渤海、黄海、东海及南海海区均有分布,曾与曼氏无针乌贼(Sepiella maindroni)、大黄鱼(Larimichthys crocea)和小黄鱼(L. polyactis)并称为四大海产,是我国单鱼种产量最高的经济鱼类(严利平等,2005;林龙山等,2006;李发凯等,2016)。但自20世纪80年代以来,由于过度捕捞和产卵场环境遭到破坏,东海带鱼种群一度出现小型化、低龄化和低质化现象(徐汉祥等,1997;周永东等,2002)。近年来,随着东海产卵带鱼保护区、东海带鱼国家级水产种质资源保护区和伏期休渔制度的建立,其资源衰退趋势有所减缓,但整体形势仍不容乐观(杜萍等,2020)。因此,开展带鱼种质资源现状及遗传背景的调查研究,对于带鱼种群资源的恢复和开发管理具有重要意义。【前人研究进展】至今,国内外有关带鱼的研究主要集中在资源变动、渔业生物学及种群生态学等方面(徐汉祥等,2003;凌建忠等,2004;林龙山等,2006;陈云龙等,2013;金鑫等,2014)。关于带鱼遗传多样性和种群鉴别的常见技术手段有同工酶(王可玲等,1994;杨天燕和高天翔,2007)、随机扩增DNA多态性(蒙子宁等,2003)、線粒体DNA(张继民等,2009;郑文娟等,2015;卞光明等,2017;吴仁协等,2019)及微卫星(Yang et al.,2007;An et al.,2010;毕金贞,2010)等。其中,微卫星DNA(Microsatellites DNA)又称为简单重复序列(Simple sequence repeat,SSR),是由1~6个核苷酸组成的串联重复片段所构成,每个单元长度在1~10 bp,而单个微卫星DNA的总长度多在200 bp内(赵彦花等,2019;周康奇等,2020)。SSR分子标记广泛分布于真核生物和少许原核生物基因组中,具有多态性丰富、稳定性良好、PCR扩增重复性高、杂合率高且遵循孟德尔遗传定律呈共显性遗传等优点,已成为鱼类种群遗传多样性、遗传连锁图谱构建、增殖放流效果评估等研究领域的重要分子标记之一(何平,1998;朱滨和常剑波,1999;郭宝英等,2007)。近年来,随着高通量测序技术的发展,基于转录组(RNA-Seq)数据大规模开发SSR分子标记的研究已十分成熟和便捷(肖韵铮等,2020;杨利艳等,2020)。Mikheyev等(2010)从斑蝶(Euphydryas editha)转录组中快速挖掘出大量SSR位点,有效解决了鳞翅目(Lepidoptera)昆虫从基因组中开发微卫星困难的问题。Yuan等(2014)对列入世界自然保护联盟(IUCN)名录中的濒危物种棘腹蛙(Quasipaa boulengeri)进行转录组测序,成功分离获得32个SSR位点,与采用传统的富集文库法开发SSR分子标记相比更便利。Hu等(2015)利用高通量测序技术从中国特有植物麻核桃(Juglans hopeiensis)的叶片、花蕾和果实组织转录组中开发出25对多态性SSR引物,并对2个地理种群的植株进行遗传差异分析。Zhao等(2019)从我国北方常见淡水小型虾类中华小长臂虾(Palaemonetes sinensis)转录组中检测出16对多态性SSR引物,进而对7个地理种群的319尾个体进行遗传多样性和遗传结构研究。此外,章霞等(2019)对日本带鱼(T. japanicus)肝脏转录组SSR特征进行统计,并开发出10对可于今后多态性分析和验证的SSR引物。【本研究切入点】传统的SSR分子标记开发多采用生物信息法、链霉亲和素磁珠富集法、硝酸纤维素膜杂交法等方式,其开发过程复杂、费时费力,且多态位点数量不多,能揭示的遗传信息十分有限,难以满足精准的种群遗传结构分析和种质资源评估(贾小平等,2009)。因此,亟待通过高通量测序技术大量开发带鱼SSR分子标记,为其资源保护与利用提供遗传学数据资料。【拟解决的关键问题】基于Illumina HiSeqTM 2500高通量测序平台对采自浙江舟山近海的带鱼样本肌肉组织进行转录组测序,从海量数据中查找SSR位点并进行生物信息学分析,以期为科学制定带鱼种质资源保护对策和管理措施提供参考依据。
1 材料与方法
1. 1 试验材料
用于转录组测序的10尾带鱼样品于2020年3月采自浙江近海的东海带鱼国家级水产种质资源保护区(东经123°15′,北纬30°10′)。剪取适量背部肌肉装入含RNAhold保存液(北京全式金生物技术股份有限公司)的2 mL冻存管(美国Corning公司)中,置于-80 ℃超低温冰箱中冷冻保存。
1. 2 RNA提取及转录组测序
采用常规TRIzol法提取总RNA(美国Invitrogen公司),使用Agilent 2100(美国Agilent公司)和NanoDrop 2000(美国Thermo Fisher公司)检测RNA浓度和纯度。合格的RNA样品送至生工生物工程(上海)股份有限公司构建cDNA混合文库,然后基于Illumina HiSeqTM 2500平台进行转录组测序。测序获得的带鱼肌肉组织转录组Raw reads经FastQC和Trimmomatic进行质量评估和剪切,去除接头、样品标识序列、低质量的Reads及带N碱基较多的Reads,以获取高质量的Clean reads(Bolger et al.,2014)。FF6A4387-7446-4F61-9047-3F7726652A6C
1. 3 数据拼接组装及SSR位点筛选和统计
使用Trinity对Clean reads进行De nove组装以获得转录本(Transcript),参数设为“min_kmer_cov 2”,其余默认(Haas et al.,2013),去冗余后取每个转录本聚类中最长的转录本作为Unigenes用于后续分析。以Micro-Satellite(MISA)对带鱼Unigenes中潜在的SSR进行搜索(Beier et al.,2017),筛选条件参数设为:基元长度1~6 bp,单核苷酸重复次数不小于10次,二核苷酸重复次数大于6次,三核苷酸、四核苷酸、五核苷酸、六核苷酸重复次数至少5次。若2个SSR间的距离小于100 bp,则记为1个复合微卫星。使用Excel 2010统计并计算SSR的数目、发生频率、出现频率、分布距离与密度、重复基元类别及重复区段长度等信息。具体统计公式如下:
SSR发生频率(%)=含SSR的Unigenes总数/Unigenes总数×100
SSR相对丰度(个/Mb)=筛选获得的SSR总数/Unigenes总长度
SSR平均距离(bp)=Unigenes总长度/筛选获得的SSR总数
SSR出現频率(%)=检测所得SSR总数/Unigenes总数×100
2 结果与分析
2. 1 转录组测序、拼接及组装结果
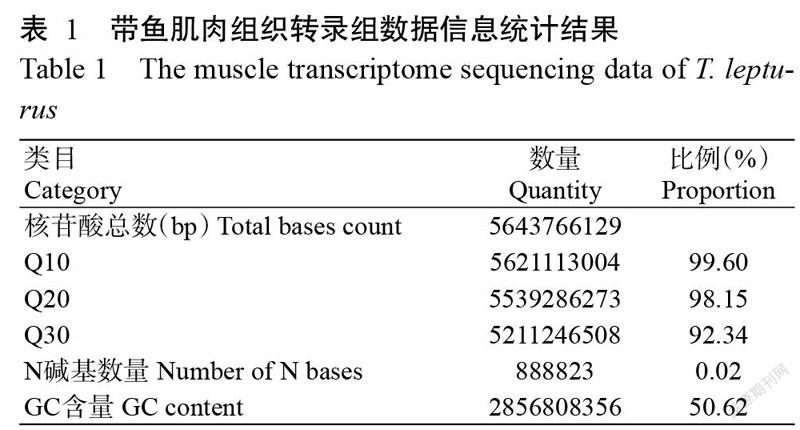
基于Illumina HiSeqTM 2500平台测序共获得41886302条Raw reads,经质控后得到40424018条Raw reads,共计5643766129个核苷酸,平均为139.61 bp。GC含量为50.62%,N碱基数量为888823个,所占比例仅为0.02%。Q10、Q20和Q30分别为99.60%、98.15%和92.34%(表1),以上数据表明获得的测序结果较好。
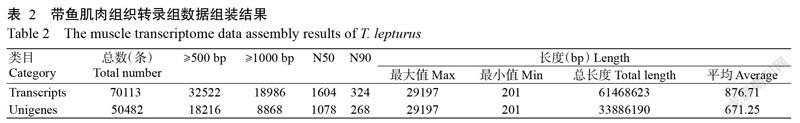
经Trinity从头组装获得70113条Transcripts,去冗余后得到50482条Unigenes,总长度为33886190 bp,平均长度为671.25 bp。N50和N90(累加转录本长度为不小于总长50%或90%的拼接转录本长度)分别为1078和268 bp,其中最大长度为29197 bp、最小长度为201 bp。长度大于1000 bp的Unigens有8868条,占总Unigenes的17.57%;长度在500 bp以上的有18216条,占比36.08%(表2)。
2. 2 转录组SSR重复类型及其分布情况
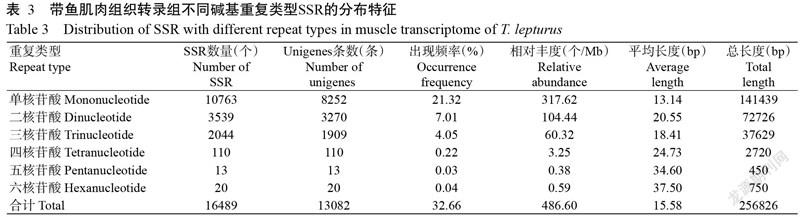
使用MISA对总长33886190 bp的50482条Unigenes进行筛选,结果共发现18873个SSR位点,且这些SSR位点仅分布在其中的13082条Unigenes上,发生频率为25.91%,出现频率为37.39%。将这些SSR位点按核苷酸重复类型进行分类,可分为单核苷酸重复、二核苷酸重复、三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复6种类型。其中,以单核苷酸重复SSR数最多,有10763个,存在于8252条Unigenes上,出现频率最高(21.32%);然后依次是二核苷酸重复、三核苷酸重复、四核苷酸重复、六核苷酸重复和五核苷酸重复SSR,出现频率分别为7.01%、4.05%、0.22%、0.04%和0.03%。SSR位点相对丰度排序为单核苷酸重复(317.62个/Mb)>二核苷酸重复(104.44个/Mb)>三核苷酸重复(60.32个/Mb)>四碱基重复(3.25个/Mb)>六核苷酸重复(0.59个/Mb)>五核苷酸重复(0.38个/Mb);SSR平均长度以六核苷酸重复最长,为37.50 bp,然后依次为五核苷酸重复(34.60 bp)、四核苷酸重复(24.73 bp)、二核苷酸重复(20.55 bp)、三核苷酸重复(18.41 bp)和单核苷酸重复(13.14 bp)(表3)。此外,以复合形式存在的SSR位点有2384个,出现频率为4.72%。
2. 3 转录组SSR重复基元类型
带鱼肌肉组织转录组SSR中共检测出173种重复基元,其中以四核苷酸重复基元种类最多,有66种,然后依次为三核苷酸重复(58种)、六核苷酸重复(20种)、五核苷酸重复(13种)和二核苷酸重复(12种),单核苷酸重复因为受碱基数量的限制,重复基元种类最少(仅4种)。在66种四核苷酸重复基元类型中,数量和分布特征变化较小,SSR的优势重复基元为AAAC、ATGG、ATGT、CTGT、CTTT和 TCCA等6种。在重复基元中,单核苷酸重复依旧在数量上占据绝对优势,其中A、T碱基重复基元占单核苷酸重复SSR位点数量的94.50%;相对而言,五核苷酸重复和六核苷酸重复的基元类型与数量分布均较少。
从不同重复基元类型的含量(表4)来看,单核苷酸重复中以A碱基数量最多,有5167个(占48.09%),而C碱基数量最少,为251个(仅占2.33%);二核苷酸重复基元以TG为主,有692个(占19.55%),其次是GT(19.02%)、AC(17.77%)和CA(15.91%)等3种重复基元,而CG和GC的数量分别只有4和5个;在三核苷酸重复基元中,所占比例最高的是GAG,有206个(占10.08%),其次是TCC(5.19%)、CAG(4.55%)、CCT(4.40%)和GGA(4.31%),而CGA的数量仅有2个(占0.10%);四核苷酸重复基元出现频率较高的有AAAC(3.63%)、ATGG(3.63%)、ATGT(3.63%)、CTGT(3.63%)和TCCA(3.64%);五核苷酸重复和六核苷酸重复的基元类型数量分布较均匀,无明显的优势重复基元(图1)。
在带鱼肉组织转录组SSR中,以10次重复的SSR数量最多,达3659个,占SSR总数的19.39%;其次是11、12、6和5次重复,SSR数量在1000~2300个,约占总SSR数目的52.27%。其中,单核苷酸重复次数分布在10~50次,主要集中在10~25次,占单核苷酸重复总数的97.71%;二核苷酸重复次数分布在6~42次,且主要集中在6~12次,共2887个,占该类型核苷酸重复总数的81.58%;三核苷酸重复次数分布在5~22次,其中2~8次重复占三核苷酸重复总数的93.74%;四核苷酸重复次数分布在5~24次,其中5~6次重复占四核苷酸重复总数的85.45%;五核苷酸重复、六核苷酸重复次数均在15次以内,以5~10次居多(表5)。FF6A4387-7446-4F61-9047-3F7726652A6C
综上所述,SSR基元重复次数主要分布在5~6次和10~12次,共9864个,占SSR总数的52.27%;其次是7~9次和13~20次,共5772个,占SSR总数30.58%;重复次数大于25次的SSR相对较少,主要由单核苷酸和二核苷酸重复基元组成,共342个,占SSR总数的1.81%,且单核苷酸和二核苷酸基元无重复5次的相关数据。此外,SSR基元重复次数增至10次后,随着重复次数的增加SSR数量依次递减,而单核苷酸重复所占的比例逐渐增加。
2. 4 转录组SSR长度分布及多态性评价结果
带鱼肌肉组织SSR序列长度区间跨度较大,范围在10~96 bp。其中,六核苷酸重复SSR长度变化最小,在30~60 bp;二核苷酸重复SSR长度变化范围最大,为12~84 bp。单核苷酸重复、三核苷酸重复、四核苷酸重复、五核苷酸重复的SSR长度范围分别为10~51 bp、15~66 bp、20~96 bp和25~65 bp。根据SSR长度可为两大类:第一类是重复序列长度在20 bp及其以上的高度多态性I型,第二类是重复序列长度在12~20 bp的中度多态性II型(Wang et al.,2010)。本研究中,I型SSR仅2558条,约占SSR总数的18%;II型SSR共8663条,约占SSR总数的50%(图2)。
3 讨论
充分了解和掌握渔业对象的生物学特征和遗传学背景,是开展渔业资源科学管理及可持续开发利用的前提(Ward,2020)。带鱼作为我国重要的海洋经济鱼类,线粒体和SSR分子标记仍是其种群遗传学研究的主要手段。已报道的带鱼SSR分子标记筛选方法主要有链霉素磁珠捕获法和富集文库法,但这2种方法不仅耗时长、花费高,开发的SSR分子标记数量还十分有限。随着高通量测序技术的发展和测序成本的降低,基于转录组学数据大量开发具有高多态性的带鱼SSR位点变得更加高效、便捷。本研究对采自浙江舟山近海的带鱼肌肉组织进行转录组高通量测序,使用SSR识别工具MISA在50482条Unigenes中搜索出18873个SSR位点,出现频率为37.39%,与其他海洋鱼类转录组中SSR的出现频率相比,高于牙鲆(Paralichthys olivaceus,27.12%)(李超等,2015)、刀鲚(Coilia nasus,8.76%)(Fang et al.,2015)、银鲳(Pampus argenteus,2.62%)(刘磊等,2016)和龙头鱼(Harpadon nehereus,18.99%)(黄新芯等,2021),但略低于黄姑鱼(Nibea albiflora,39.30%)(龚诗琦等,2016),说明带鱼属于转录组中SSR数量较丰富的鱼类,带鱼转录组测序质量和SSR含量均处于较高水平,能为后续群体遗传学研究提供充足的序列资源。相对于带鱼肝脏转录组测序数据中的发生频率(40.95%)(章霞等,2019),本研究所得的数据略低,且与肝脏中单核苷酸重复~六核苷酸重复SSR的出现频率(53.79%、30.63%、14.34%、1.12%、0.08%和0.03%)相比也偏低。此外,带鱼肝脏转录组SSR中单核苷酸和二核苷酸在重复次数分别达23和11次时突然增加,而在肌肉组织中单核苷酸重复~六核苷酸重复的出现次数随着重复次数的增加依次递减,并无突增现象,究其原因可能与选择的组织不同有一定关系,但不排除測序平台及检索参数设置不同带来的影响。
相对于序列长度长且重复性低的串联重复单元,序列长度短且重复性高的串联重复单元则表现出更高的突变率,其多态性比例也较高(Kelkar et al.,2008),但重复序列长度小于12 bp时SSR多态性反而降低,因此,长度在20 bp及其以上的I型SSR和长度在12~20 bp的II型SSR是可用性较高的潜在分子标记(Temnykh et al.,2001)。从核苷酸重复次数来看,本研究中带鱼肌肉组织SSR核苷酸重复次数在5~50次,剔除易发生错配的单核苷酸重复类型,重复数最高可达50次。从SSR长度来看,重复长度在12~20 bp的SSR为2558个,重复长度大于20 bp的SSR有8663个,即超过75%的SSR具有中度及以上水平的多样性。可见,带鱼肌肉转录组SSR可用性高且具有较高的多态性潜能,在此基础上可有针对性进行引物设计,为带鱼的种质鉴定、遗传差异检测及功能基因定位等研究提供理论依据和实践基础。
二核苷酸重复是带鱼基因组SSR的主要类型(张浩冉等,2019),而本研究发现带鱼肌肉组织转录组中的SSR重复类型以单核苷酸重复为主。已有研究表明,二核苷酸重复类型的SSR主要位于非编码区(虞杭等,2018)。由于简化基因组测序是采取对基因组特定区域进行测序以反映物种全基因组信息的策略,能检测到广泛分布于编码区、非编码区和调控区的SSR位点信息,而真核生物在转录本RNA加工过程中会对内含子进行剪切,丢失部分非编码区序列信息。因此,在依托转录组和基因组数据开发的SSR中其主导重复类型和重复数量存在一定差异。考虑到基因组信息有助于转录组注释,而转录组数据也可对基因组序列进行补充和校正,在今后开展带鱼遗传多样性分析时可将这2种技术手段结合起来,为分子标记的挖掘和应用提供更全面的参考信息。Harr和Schl?tterer(2000)对黑腹果蝇(Drosophila melanogaster)基因组SSR结构和组成的研究发现,复杂的多基元重复SSR出现频率较高,通常暗示着物种进化水平较低或具有较低的变异频率。在本研究中,带鱼肌肉组织转录组中低级重复基元(单核苷酸重复~三核苷酸重复)约占SSR总数的86.61%,出现频率也较高(32.38%),而高级重复基元SSR的多态性相对较低,表明带鱼可能具有较长的进化历史,且积累了较多的遗传变异。
4 结论
经高通量测序获得的带鱼肌肉转录组SSR可用性高且具有较高的多态性潜能,在此基础上可有针对性进行引物设计,为带鱼遗传多样性评价、遗传结构分析及功能基因克隆等研究提供有效的分子标记,进而为其种质资源的保护与利用提供遗传学数据资料。FF6A4387-7446-4F61-9047-3F7726652A6C
参考文献:
毕金贞. 2010. 带鱼微卫星标记的开发和微卫星标记在牙鲆选择育种中的应用[D]. 青岛:中国海洋大学. [Bi J Z. 2010. The development of microsatellite markers from cutlassfish and the application of SSR in the selective bree-ding project of Japanese flounder[D]. Qingdao:Ocean University of China.] doi:10.7666/d.y1828271.
卞光明,王娜泠,胡则辉,王跃斌,胡成硕,柴学军. 2017. 舟山带鱼线粒体COI基因SNP位点分析[J]. 浙江海洋学院学报(自然科学版),36(1):19-24. [Bian G M,Wang N L,Hu Z H,Wang Y B,Hu C S,Chai X J. 2017. Analysis on mitochondrial COI gene SNP loci of Trichiurus lepturus from Zhoushan Sea Area[J]. Journal of Zhejiang Ocean University(Natural Science),36(1):19-24.]
陈云龙,单秀娟,戴芳群,金显仕. 2013. 东海近海带鱼群体相对资源密度、空间分布及其产卵群体的结构特征[J]. 渔业科学进展,34(4):8-15. [Chen Y L,Shan X J,Dai F Q,Jin X S. 2013. Relative stock density and distribution of hairtail Trichiurus lepturus and its spawning stock structure in coastal waters of the East China Sea[J]. Progress in Fishery Sciences,34(4):8-15.] doi:10.3969/j.issn. 1000-7075.2013.04.002.
杜萍,陈全震,李尚鲁,颜云榕,叶文建,俞存根. 2020. 东海带鱼资源变动及其栖息地驱动因子研究进展[J]. 广东海洋大学学报,40(1):126-132. [Du P,Chen Q Z,Li S L,Yan Y R,Ye W J,Yu C G. 2020. Advances in the Trichiu-rus lepturus changes and habitat driving factors in the East China Sea[J]. Journal of Guangdong Ocean University,40(1):126-132.] doi:10.3969/j.issn.1673-9159.2020.01. 017.
龚诗琦,王志勇,肖世俊,林爱强,谢仰杰. 2016. 黄姑鱼转录组SSR的开发与验证[J]. 集美大学学报(自然科学版),21(4):241-246. [Gong S Q,Wang Z Y,Xiao S J,Lin A Q,Xie Y J. 2016. Development and verification of SSR based on transcriptome of yellow drum,Nibea albiflora[J]. Journal of Jimei University(Natural Science),21(4):241-246.] doi:10.3969/j.issn.1007-7405.2016.04.001.
郭寶英,谢从新,熊冬梅. 2007. 微卫星DNA标记技术及其在鱼类中的应用[J]. 水利渔业,27(4):5-9. [Guo B Y,Xie C X,Xiong D M. 2007. mtDNA marking technique and their application in fishes[J]. Reservoir Fisheries,27(4):5-9.]
何平. 1998. 真核生物中的微卫星及其应用[J]. 遗传,20(4):42-47. [He P. 1998. Abundance,polymorphism and applications of microsatellite in eukaryote[J]. Hereditas,20(4):42-47.] doi:10.16288/j.yczz.1998.04.013.
黄新芯,蒋艳琳,蒋小姿,杨天燕. 2021. 基于高通量转录组测序技术的龙头鱼微卫星信息分析[J]. 浙江海洋学院学报(自然科学版),40(3):189-197. [Huang X X,Jiang Y L,Jiang X Z,Yang T Y. 2021. Analysis of microsatellite markers in Harpadon nehereus based on transcriptome sequencing Illumina HiSeqTM 2500[J]. Journal of Zhejiang Ocean University(Natural Science),40(3):189-197.] doi:10.3969/j.issn.1008-830X.2021.03.001.
贾小平,王天宇,黎裕,宋燕春,石云素. 2009. 微卫星开发技术研究进展[J]. 安徽农业科学,37(24):11439-11440. [Jia X P,Wang T Y,Li Y,Song C Y,Shi Y S. 2009. Research development of microsatellite exploitation techno-logies[J]. Journal of Anhui Agricultural Sciences,37(24):11439-11440.] doi:10.3969/j.issn.0517-6611.2009.24.037.FF6A4387-7446-4F61-9047-3F7726652A6C
金鑫,朱清澄,陳丙见,王晓杰,张衍栋,路伟. 2014. 缅甸外海大西洋带鱼基础生物学特性初步研究[J]. 南方农业学报,45(1):112-117. [Jin X,Zhu Q C,Chen B J,Wang X J,Zhang Y D,Lu W. 2014. Biological characteristics of Trichiurus lepturus in Andaman Sea of Myanmar[J]. Jour-nal of Southern Agriculture,45(1):112-117.] doi:10.3969/ j:issn.2095-1191.2014.1.112.
李超,侯吉伦,王桂兴,张晓彦,刘永富,童爱萍,刘海金. 2015. 基于牙鲆RNA-seq数据中SSR标记的信息分析[J]. 海洋渔业,37(2):122-127. [Li C,Hou J L,Wang G X,Zhang X Y,Liu Y F,Tong A P,Liu H J. 2015. Bioinformatic analysis of SSR markers in transcriptomic sequencing Paralichthys olivaceus[J]. Marine Fisheries,37(2):122-127.] doi:10.3969/j.issn.1004-2490.2015.02.004.
李发凯,田中荣次,岩田繁英,俞存根. 2016. 应用年龄结构产量模型评估东黄海带鱼资源[J]. 浙江海洋学院学报(自然科学版),35(2):91-98. [Li F K,Tanaka E,Iwata S,Yu C G. 2016. Stock assessment of hairtail Trichiurus lepturus in the East China Sea and Yellow Sea using age structured production model[J]. Journal of Zhejiang Ocean University(Natural Science),35(2):91-98.] doi:10.3969/ j.issn.1008-830X.2016.02.001.
林龙山,张寒野,李惠玉,程家骅. 2006. 东海带鱼食性的季节变化[J]. 中国海洋大学学报(自然科学版),36(6):932-936. [Lin L S,Zhang H Y,Li H Y,Cheng J H. 2006. Study on seasonal variation of feeding habit of hairtail (Trichiurus japonicus) in the East China Sea[J]. Periodical of Ocean University of China,36(6):932-936.] doi:10.16441/j.cnki.hdxb.2006.06.017.
凌建忠,程家骅,任一平,林龙山. 2004. 东海带鱼主要体征与个体繁殖力的关系[J]. 中国水产科学,11(2):116-120. [Ling J Z,Cheng J H,Ren Y P,Lin L S. 2004. Relationships between main physical characters and individual fecundities of hairtail Trichiurus japonicus in the East China Sea[J]. Journal of Fishery Sciences of China,11(2):116-120.] doi:10.3321/j.issn:1005-8737.2004.02.006.
刘磊,彭士明,高权新,张晨捷,施兆鸿. 2016. 基于银鲳RNA-seq数据中SSR标记的信息分析[J]. 安徽农业科学,44(28):102-105. [Liu L,Peng S M,Gao Q X,Zhang C J,Shi Z H. 2016. Bioinformatic analysis of SSR markers based on RNA-seq of Pampus argenteus[J]. Journal of Anhui Agricultural Sciences,44(28):102-105.] doi:10. 3969/j.issn.0517-6611.2016.28.033.
蒙子宁,庄志猛,金显仕,唐启升,苏永全. 2003. 黄海带鱼、小带鱼RAPD和线粒体16S rRNA基因序列变异分析[J]. 自然科学进展,13(11):1170-1176. [Meng Z N,Zhuang Z M,Jin X S,Tang Q S,Su Y Q. 2003. Variation analyses of RAPD and mitochondrial 16S rRNA gene sequence of Trichiurus lepturus and Eupleurogrammus muticus in the Yellow Sea[J]. Progress in Natural Science,13(11):1170-1176.] doi:10.3321/j.issn:1002-008X.2003.11.009.
王可玲,张培军,刘兰英,尤锋,徐成,王建飞. 1994. 中国近海带鱼种群生化遗传结构及其鉴别的研究[J]. 海洋学报,16(1):93-104. [Wang K L,Zhang P J,Liu L Y,You F,Xu C,Wang J F. 1994. Study on population biochemical genetic structure and identification of hairtail (Trichiurus haumela Forskal) in the coastal waters of China[J]. Acta Oceanologica Sinica,16(1):93-104.]FF6A4387-7446-4F61-9047-3F7726652A6C
吳仁协,张浩冉,牛素芳,苗奔奔,翟云. 2019. 东海近岸带鱼(Trichiurus japonicus)线粒体控制区序列的群体遗传变异研究[J]. 海洋与湖沼,50(6):1318-1327. [Wu R X,Zhang H R,Niu S F,Miao B B,Zhai Y. 2019. Study on population genetic variation of Trichiurus japonicus in nearshore of the East China Sea in mitochondrial control region sequences[J]. Oceanologia et Limnologia Sinica,50(6):1318-1327.] doi:10.11693/hyhz20190400077.
肖韵铮,韩世明,秦昭,李春奇. 2020. 滇黄精转录组测序及类黄酮合成相关基因的分析[J]. 河南农业大学学报,54(6):931-940. [Xiao Y Z,Han S M,Qin Z,Li C Q. 2020. Analysis of transcriptome sequencing and related genes of flavonoids biosynthesis from Polygonatum kingianum[J]. Journal of Henan Agricultural University,54(6):931-940.] doi:10.16445/j.cnki.1000-2340.2020.06.004.
徐汉祥,刘子藩,许源剑. 1997. 带鱼资源动态综述及管理现状分析[J]. 浙江水产学院学报,16(3):53-59. [Xu H X,Liu Z F,Xu Y J. 1997. Comment on hairtail stock dynamic and discuss on management situation[J]. Journal of Zhejiang College of Fisheries,16(3):53-59.]
徐汉祥,刘子藩,周永东. 2003. 东海带鱼生殖和补充特征的变动[J]. 水产学报,27(4):322-327. [Xu H X,Liu Z F,Zhou Y D. 2003. Variation of Trichiurus haumela productivity and recruitment in the East China Sea[J]. Journal of Fisheries of China,27(4):322-327.] doi:10.3321/j.issn:1000-0615.2003.04.006.
严利平,胡芬,李建生,刘勇,程家骅. 2005. 东海带鱼年龄与生长的研究[J]. 海洋渔业,27(2):139-142. [Yan L P,Hu F,Li J S,Liu Y,Cheng J H. 2005. Age and growth of Trichiurus haumela in the East China Sea[J]. Marine Fisheries,27(2):139-142.] doi:10.3969/j.issn.1004-2490. 2005.02.009.
杨利艳,张玉荣,杨雅舒,王美霞,陈保国,赵丽,张丽光,王创云. 2020. 基于RNA-seq数据分析玉米抗虫响应基因的可变剪接事件[J]. 河南农业大学学报,54(2):181-188. [Yang L Y,Zhang Y R,Yang Y S,Wang M X,Chen B G,Zhao L,Zhang L G,Wang C Y. 2020. Analysis of alternative splicing events of insect-resistant response genes based on RNA-seq data in Zea mays[J]. Journal of Henan Agricultural University,54(2):181-188.] doi:10.16445/j.cnki.1000-2340.20200403.009.
杨天燕,高天翔. 2007. 黄海和东海带鱼群体同工酶分析[J]. 海洋水产研究,28(3):44-49. [Yang T Y,Gao T X. 2007. Isozyme analyses of Trichiurus haumela in the Yellow Sea and East China Sea[J]. Marine Fisheries Research,28(3):44-49.] doi:10.3969/j.issn.1000-7075.2007.03.007.
虞杭,张得芳,樊光辉,王占林. 2018. 枸杞转录组SSR分布特征分析及其与基因组SSR分布特征的比较[J]. 江苏农业科学,46(14):24-27. [Yu H,Zhang D F,Fan G H,Wang Z L. 2018. Characteristic analysis of transcriptome SSR distribution of Lycium barbarum and its comparison with genomic SSR distribution[J]. Jiangsu Agricultural Scien-ces,46(14):24-27.] doi:10.15889/j.issn.1002-1302.2018. 14.006.
张浩冉,梁镇邦,吴仁协,牛素芳,柯宗宇,宿帅,韦欢,王权,孙贝. 2019. 利用SLAF-seq技术开发带鱼(Trichiurus japonicus)微卫星标记以及跨物种扩增检测[J]. 基因组学与应用生物学,38(2):574-585. [Zhang H R,Liang Z B,Wu R X,Niu S F,Ke Z Y,Su S,Wei H,Wang Q,Sun B. 2019. Development of microsatellite loci for hairtail (Trichiurus japonicus) by using SLAF-seq technology and cross-species amplification tests[J]. Genomics and Applied Biology,38(2):574-585.] doi:10.13417/j.gab.038. 000574.FF6A4387-7446-4F61-9047-3F7726652A6C
张继民,刘霜,赵建民,马兆党,张洪亮. 2009. 带鱼(Trichiurus haumela)鱼卵DNA的提取及其18S rDNA初步分析[J]. 海洋通报,28(6):62-65. [Zhang J M,Liu S,Zhao J M,Ma Z D,Zhang H L. 2009. Preliminary study on DNA extraction and 18S rDNA of the Trichiurus haumela fish egg[J]. Marine Science Bulletin,28(6):62-65.] doi:10.3969/j.issn.1001-6392.2009.06.011.
章霞,柳敏海,李凌刚,徐志进,李伟业,殷小龙,傅荣兵. 2019. 东海带鱼(Trichiurus japanicus)肝脏转录组SSR和SNP特征分析[J]. 渔业研究,41(4):269-277. [Zhang X,Liu M H,Li L G,Xu Z J,Li W Y,Yin X L,Fu R B. 2019. SSR and SNP analysis based on Trichiurus japanicus transcriptome[J]. Journal of Fisheries Research,41(4):269-277.] doi:10.14012/j.cnki.fjsc.2019.04.001.
赵彦花,区又君,温久福,李加儿,周慧. 2019. 基于转录组测序技术的黄唇鱼SSR分子标记筛选[J]. 南方农业学报,50(9):2078-2087. [Zhao Y H,Ou Y J,Wen J F,Li J E,Zhou H. 2019. Development of SSR markers in Bahaba flavolabiata by transcriptome sequencing[J]. Journal of Southern Agriculture,50(9):2078-2087.] doi:10.3969/j.issn.2095-1191.2019.09.26.
郑文娟,杜一超,林洁,蒋晨华,丁沃娜,朱世华. 2015. 基于线粒体DNA D-loop区部分序列分析舟山海域带鱼种群遗传结构[J]. 水生生物学报,39(2):408-413. [Zheng W J,Du Y C,Lin J,Jiang C H,Ding W N,Zhu S H. 2015. Genetic diversity analysis of Trichiurus lepturus in Zhoushan based on mitochondrial DNA D-loop region partial sequences[J]. Acta Hydrobiologica Sinica,39(2):408-413.] doi:10.7541/2015.53.
周康奇,潘贤辉,黄姻,覃俊奇,徐俊龙,杜雪松,文露婷,陈忠,潘志忠,邓潜,林勇. 2020. 基于RNA-seq技术的奥利亚罗非鱼转录组SSR位点信息分析[J]. 河南农业科学,49(11):147-152. [Zhou K Q,Pan X H,Huang Y,Qin J Q,Xu J L,Du X S,Wen L T,Chen Z,Pan Z Z,Deng Q,Lin Y. 2020. Analysis of SSR site information in transcriptome of Oreochromis aurea based on RNA-seq[J]. Journal of Henan Agricultural Sciences,49(11):147-152.] doi:10.15933/j.cnki.1004-3268.2020.11.019.
周永东,徐汉祥,刘子藩,薛利建. 2002. 东海带鱼群体结构变动的研究[J]. 浙江海洋学院学报(自然科学版),21(4):314-320. [Zhou Y D,Xu H X,Liu Z F,Xue L J. 2002. A study on variation of stock structure of hairtall,Trichiurus haumela in the East China Sea[J]. Journal of Zhejiang Ocean University(Natural Science),21(4):314-320.] doi:10.3969/j.issn.1008-830X.2002.04.002.
朱濱,常剑波. 1999. 微卫星DNA及其在鱼类中的应用[J]. 水生生物学报,23(6):721-728. [Zhu B,Chang J B. 1999. Micosatellite DNA and its application in fishes[J]. Acta Hydrobiologica Sinica,23(6):721-728.]
An H S,Lee J H,Noh J K,Kim H C,Park C J,Min B H,Myeong J I. 2010. Ten new microsatellite markers in cutlassfish Trichiurus lepturus derived from an enriched geno-mic library[J]. Animal Cells and Systems,14(3):169-174. doi:10.1080/19768354. 2010.504347.
Beier S,Thiel T,Münch T,Scholz U,Mascher M. 2017. MISA-web:A web server for microsatellite prediction[J]. Bioinformatics,33(16):2583-2585. doi:10.1093/bioinformatics/btx198.FF6A4387-7446-4F61-9047-3F7726652A6C
Bolger A M,Lohse M,Usadel B. 2014. Trimmomatic:A flexible trimmer for Illumina sequence data[J]. Bioinforma-tics,30(15):2114-2120. doi:10.1093/bioinformatics/btu 170.
Fang D A,Zhou Y F,Duan J R,Zhang M Y,Xu D P,Liu K,Xu P,Wei Q. 2015. Screening potential SSR markers of the anadromous fish Coilia nasus by de novo transcriptome analysis using Illumina sequencing[J]. Genetics and Molecular Research,14(4):14181-14188. doi:10.4238/2015.November.13.1.
Haas B J,Papanicolaou A,Yassour M,Grabherr M,Blood P D,Bowden J,Couger M B,Eccles D,Li B,Lieber M,MacManes M D,Ott M,Orvis J,Pochet N,Strozzi F,Weeks N,Westerman R,William T,Dewey C N,Henschel R,LeDuc R D,Friedman N,Regev A. 2013. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis[J]. Nature Protocols,8(8):1494-1512. doi:10.1038/nprot. 2013.084.
Harr B,Schl?tterer C. 2000. Long microsatellite alleles in Drosophila melanogaster have a downward mutation bias and short persistence times,which cause their genome-wide under representation[J]. Genetics,155(3):1213-1220. doi:10.1093/genetics/155.3.1213.
Hu Y H,Zhao P,Zhang Q,Wang Y,Gao X X,Zhang T,Zhou H J,Dang M,Woeste K E. 2015. De novo assembly and characterization of transcriptome using Illumina sequen-cing and development of twenty five microsatellite mar-kers for an endemic tree Juglans hopeiensis Hu in China[J]. Biochemical Systematics and Ecology,63:201-211. doi:10.1016/j.bse. 2015.10.011.
Kelkar Y D,Tyekucheva S,Chiaromonte F,Makova K D. 2008. The genome-wide determinants of human and chimpanzee microsatellite evolution[J]. Genome Research,18:30-38. doi:10.1101/gr.7113408.
Mikheyev A S,Vo T,Wee B,Singer M C,Parmesan C. 2010. Rapid microsatellite isolation from a butterfly by de novo transcriptome sequencing:Performance and a comparison with AFLP-derived distances[J]. PLoS One,5(6):e11212. doi:10.1371/journal.pone.0011212.
Temnykh S,DeClerck G,Lukashova A,Lipovich L,Cartinhour S,McCouch S. 2001. Computational and experimental analysis of microsatellites in rice(Oryza sativa L.):Frequency,length variation,transposon associations,and genetic marker potential[J]. Genome Research,11(8):1441-1452. doi:10.1101/gr.184001.
Wang H,Iwai Jr T,Zhao B P,Lee C S,Yang J Z. 2010. Identification of microsatellite DNA markers for Pacific threadfin parentage assignment[J]. Journal of the World Aquaculture Society,41(1):640-647. doi:10.1111/j.1749-7345.2010.00405.x.
Ward R D. 2000. Genetics in fisheries management[J]. Hydrobiologia,420(1):191-201. doi:10.1023/A:100392832 7503.
Yang W T,Feng F,Yue G H. 2007. Isolation and characterization of microsatellites from a marine foodfish species ribbonfish Trichiurus haumela[J]. Molecular Ecology Resources,7(5):781-783. doi:10.1111/j.1471-8286.2007. 01700.x.
Yuan S Q,Xia Y,Zheng Y C,Zeng X M. 2014. Development of microsatellite markers for the spiny-bellied frog Quasipaa boulengeri(Anura:Dicroglossidae) through transcriptome sequencing[J]. Conservation Genetics Resources,7(1):229-231. doi:10.1007/s12686-014-0344-z.
Zhao Y Y,Zhu X C,Li Z,Xu W B,Dong J,Wei H,Li Y D,Li X D. 2019. Genetic diversity and structure of Chinese grass shrimp,Palaemonetes sinensis,inferred from transcriptome-derived microsatellite markers[J]. BMC Gene-tics,20(1):75. doi:10.1186/s12863-019-0779-z.
(責任编辑 兰宗宝)FF6A4387-7446-4F61-9047-3F7726652A6C
