基于宏转录组学技术解析浓香型酒醅活性微生物群落结构及功能变化特征
2022-06-02胡晓龙田瑞杰李保坤何培新朱文优
胡晓龙,田瑞杰,李保坤,张 勇,迟 雷,何培新,魏 涛,朱文优
(1.宜宾学院农林与食品工程学部,四川 宜宾 644000;2郑州轻工业大学食品与生物工程学院,食品生产与安全河南省协同创新中心,河南 郑州 450000)
浓香型白酒作为我国优势传统发酵食品的典型代表,其以窖香浓郁、香味协调、尾净余长等为特征的风格深受消费者喜爱。其生产主要采用复杂且独特的半开放、固态发酵模式,包括原辅料处理、续醅拌和、配入曲药、多微共酵及精心勾调等上百道工序。浓香型白酒酿造微生物主要来源于源酿造环境、器具、发酵剂、原料及窖泥,主要包括细菌、酵母、霉菌及古细菌,其在窖池内长期进行的物质和能量交换、转化是形成浓香型白酒呈香物质及独特风格的主要驱动力。
酒醅作为微生物发酵的主要载体和白酒呈香物质的直接来源,其微生物群落多样性及其演替性一直是研究的热点。近年来,免培养技术的广泛应用(变性梯度凝胶电泳、克隆文库、二代测序等),使得浓香型酒醅微生物群落多样性、结构及其演替规律逐渐清晰。例如,在酒醅中观察到的细菌和真菌属数量分别高达496和155。其中,、、、和等是优势细菌属(相对丰度≥1%);、、和等是优势真菌属。整体上,发酵前期(前7 d左右)酒醅中细菌群落多样性较高,随后细菌群落多样性急剧下降,且逐渐成为酒醅中绝对优势属。
尽管以上通过16S rRNA或ITS基因测序(DNA水平)方法得到的研究结果极大地丰富了人们对酒醅微生物群落物种多样性(多样性)及组成动态变化(多样性)的认知,但基于DNA的方法可以检测到死亡或休眠状态细胞的DNA,却不能准确反映浓香型白酒发酵过程中微生物的生理或转录活性(简称活性)及其功能。目前,基于RNA水平的宏转录组测序技术能够准确揭示目标环境样品中活性微生物群落结构。诸多研究表明,分别采用基于DNA和RNA方法分析的环境微生物群落结果存在较大差异。例如,中国台湾乌龙茶中常见的细菌在DNA水平上为Bacteroidaceae(21.7%)、Microbacteriaceae(22%)和Fusobacteriaceae(12.3%),而在RNA水平上为Bacillaceae(42%)和Enterobacteriaceae(36%)。Duan Shan等利用宏转录组测序在虾酱细菌群落中检测到了、、和,而利用16S rRNA基因测序未检测到。同时,宏转录组测序技术能更为准确地揭示环境微生物功能及代谢途径,如酱香型白酒发酵过程中涉及乙醇和乳酸合成的主要途径分别为糖酵解和丙酮酸代谢。
目前关于浓香型白酒酒醅活性微生物群落及其功能变化的研究鲜有报道。因此,本研究以浓香型白酒固态发酵主发酵期不同发酵节点(3、7 d和25 d)的酒醅样品为研究对象,运用宏转录组学技术对浓香型白酒固态发酵过程中活性微生物群落结构进行解析并确定其优势活性微生物组成,同时对发酵过程中功能基因的差异表达情况进行探究,以期为对浓香型白酒固态发酵机理解析及发酵过程控制提供理论基础。
1 材料与方法
1.1 材料与试剂
白酒发酵过程中的酒醅样品来自河南省某知名白酒企业(E111°33’~112°01’,N34°36’~35°05’)。
十二烷基硫酸钠(sodium dodecyl sulfate,SDS) 上海百赛生物技术股份有限公司;-巯基乙醇、异戊醇、聚乙二醇、焦碳酸二乙酯 上海麦克林生化科技有限公司;水饱和酚(pH 4.5) 上海源叶生物科技有限公司;无水乙醇 天津市富宇精细化工有限公司;Tris、聚乙烯吡咯烷酮、RNase-Free Water、2×Master Mix、2000 Marker 北京索莱宝科技有限公司;氯化镁、氯化钠、氯化钙、草酸钠、磷酸二氢钠、磷酸氢二钠、EDTANa北京北化精细化学品有限责任公司;Ribo-ZeroMagnetic Gold Kit(Yeast)试剂盒 北京中北林格科技发展有限公司;分离用有机溶剂均为国产分析纯。
1.2 仪器与设备
MP200A精密电子天平 上海良丰仪器仪表有限公司;TGL-20M高速离心机 上海卢湘仪离心机仪器有限公司;DYY-8C电泳仪 北京市六一仪器厂;Mixer4K微型涡旋混合仪 生工生物工程(上海)股份有限公司;85-2恒温磁力搅拌器 江苏金坛市中大仪器厂;IMS-40全自动雪花制冰机 常熟市雪科电器有限公司;LDZX-50KBS立式压力蒸汽灭菌器 上海中安医疗器械厂;SW-CJ-1超净工作台 苏州净化设备有限公司;WD-9403C紫外分析仪 北京六一生物科技有限公司;NanoDrop2000微量分光光度计 赛默飞世尔科技公司;2100生物分析仪 北京爱立斯生物科技有限公司。
1.3 方法
1.3.1 样品采集
分别对发酵第3、7、25天的窖池进行酒醅样品采集,每个发酵节点随机选取3 个平行窖池,对每个窖池中层位置的酒醅进行取样并混合作为该发酵节点的代表样,将代表样放入无菌袋中并置于盛有干冰的泡沫箱中,然后立即带回实验室进行总RNA提取。
1.3.2 酒醅总RNA的提取
按照胡晓龙等SDS-苯酚法对酒醅进行总RNA提取,然后采用1%琼脂糖凝胶电泳、NanoDrop2000微量分光光度计和2100生物分析仪评估总RNA的纯度和完整性,最后选择高质量的RNA样本构建Illumina测序文库。
1.3.3 宏转录组测序文库准备和Illumina测序
RNA样本检测合格后,使用Ribo-ZerorRNA Removal Kits(Bacteria)和Ribo-ZeroMagnetic Gold Kit(Yeast)试剂盒分别去除细菌和真菌核糖体RNA。文库的构建和测序在北京奥维森基因科技有限公司Illumina HiSeq 2500(Illumina,San Diego,CA,United States)平台上完成。
1.4 数据分析
测序下机获得原始数据,通过过滤带有测序接头、未知碱基含量大于1%和低质量碱基(≤20)含量大于50%的reads得到clean reads,利用diamond将其进行NR数据库比对分析,并利用megan进行微生物分类学解析。以酒醅活性细菌属和真菌属水平数据为基础计算酒醅中活性微生物群落的Shannon指数,以评估酒醅活性微生物物种多样性。采用Trinity(v0.27)对clean reads进行拼接,然后使用prodigal软件对拼接组装得到的contig序列进行开放阅读框预测,再通过CD-HIT软件对预测的基因序列以0.95相似度去除冗余基因,从而得到非冗余基因集。通过FPKM(每百万片段中来自某一基因每千碱基长度的片段数目)值对所有基因进行表达丰度水平计算,再使用统计软件R中edgeR包和DESeq2包,分析不同样本间表达丰度具有差异的基因(differentially expressed gene,DEG),并使用火山图对DEG的整体分布情况进行分析,阈值设定为|logfold change|>1且value<0.05。最后对DEG进行基因本体论(Gene Ontology,GO)富集分析,通过把所有DEG向GO数据库(http://www.geneontology.org/)的各个term映射,计算每个term的基因数目,然后找出与整个基因组背景相比,在DEG中显著富集的term,且1 个DEG可以分布在多个条目中。选取每个比较组中对应Corrected_value值由小到大排序的前30 个功能条目进行绘柱状图,其中Corrected_value<0.05说明这些功能条目在DEG中显著富集。
2 结果与分析
2.1 序列预处理结果
由表1可知,经高通量双端测序及序列质量控制,每个样品获得的序列的和碱基分别高于97%和92%,表明序列数据可靠,可以用于下一步分析。不同发酵时间节点酒醅宏转录组序列中GC含量差异较大(41%~48%),表明不同发酵时间酒醅中活性生物群落组成或转录的基因序列组成存在较大的差异。
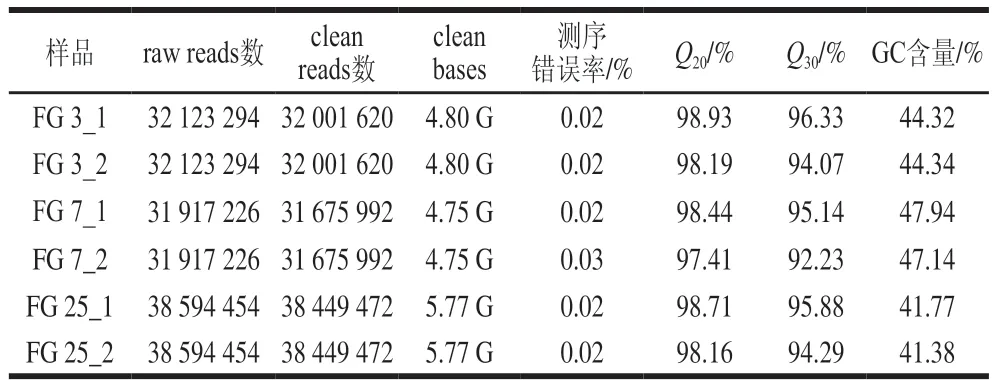
表1 样品宏转录测序信息Table 1 Metatranscriptomic sequencing information of each sample
2.2 酒醅中活性物种注释

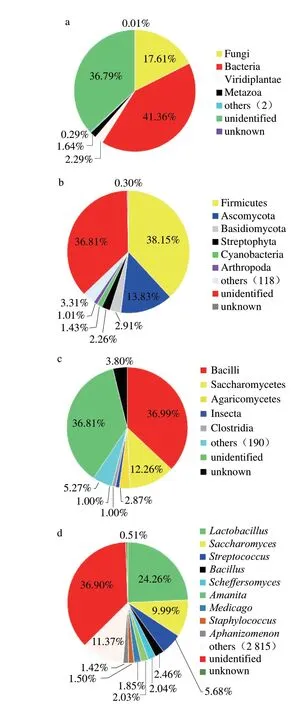
图1 浓香型酒醅中不同分类水平的优势生物(含微生物)群落组成Fig.1 Composition of dominant microbial communities at different taxonomic levels in strong-flavor baijiu fermented grains
2.3 活性微生物群落α多样性及β多样性
在长期发酵过程中,酒醅微生物群落不断演替,期间伴随着物质和能量代谢,因此从RNA水平揭示不同发酵节点酒醅活性微生物群落多样性(组内多样性)及多样性(组间多样性)将更有助于进一步揭示酒醅微生物的功能、代谢途径及浓香型白酒发酵机制。
2.3.1 酒醅活性微生物群落多样性
在酒醅主发酵期,细菌群落多样性呈现先升后降趋势,发酵7 d,细菌多样性最高,发酵25 d细菌多样性最低(图2a);活性真菌群落物种多样性呈上升趋势,且主要集中在3~7 d(图2b),这些结果与之前大多数报道的结果相似。
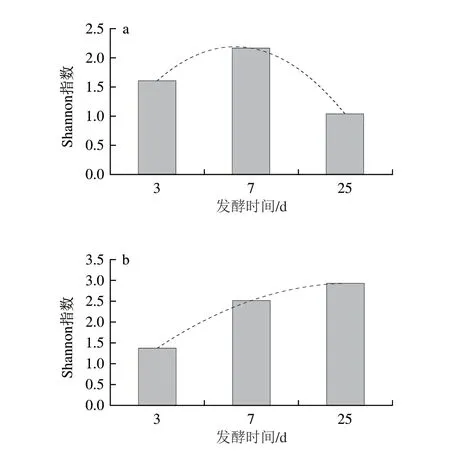
图2 不同发酵节点酒醅活性细菌(a)和真菌(b)群落Shannon指数Fig.2 Shannon index of active bacterial (a) and fungal (b) communities in fermented grains at different fermentation times
2.3.2 酒醅活性微生物群落多样性
由图1可知,真菌界和细菌界总含量占可鉴定界含量的93%以上,表明优势活性生物群落的动态变化主要由微生物群落变化驱动。在酒醅发酵过程中,不同发酵节点活性优势生物群落的变化存在明显的规律性(图3)。

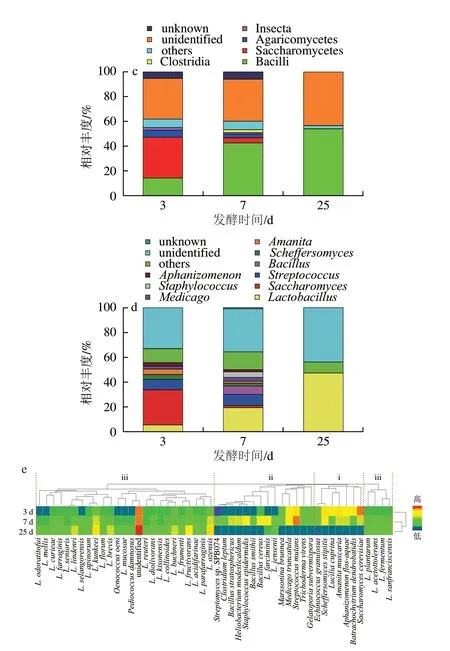
图3 界(a)、门(b)、纲(c)、属(d)、种(e)分类水平下酒醅微生物动态变化规律Fig.3 Dynamic changes of microbial communities in fermented grains at kingdom (a),phylum (b),class (c),genus (d) and species (e) levels
在界水平(图3a),优势微生物为细菌界和真菌界,随着发酵时间延长(≤25 d),细菌界含量呈增加趋势,由17.7%增至56.3%,发酵7 d后成为第一优势界;而真菌界含量呈下降趋势,由42.8%降至0.01%,表明真菌是酒醅发酵前3 d的主要活性微生物,而随发酵延长细菌界成为优势活性微生物。这主要是由于大多数真菌为好氧性微生物,随着发酵时间延长,氧气含量不断减少且养分不断消耗,导致真菌含量减少,这也与之前相关可培养结果一致;而一些厌氧或兼性厌氧、耐酸的微生物在发酵过程中相对含量不断增加,且主要为乳酸菌。
在门水平(图3b),优势微生物门主要为Firmicutes(3 个节点酒醅中平均相对含量38.2%)和Ascomycota(13.8%),还有少量的担子菌门(Basidiomycota,2.9%)和蓝细菌门(Cyanobacteria,1.4%)。在发酵过程中,Firmicutes和Ascomycota含量的变化趋势分别与细菌界和真菌界的变化趋势一致,且其分别占细菌界和真菌界含量的90.2%和71.2%,表明这两个门是驱动酒醅活性微生物群落变化的主要微生物。
在纲水平(图3 c),酒醅发酵3 d 优势微生物Saccharomycetes含量最高(32.8%),其次为Bacilli(14.6%)、Agaricomycetes(5.5%)和Clostridia(0.01%)。随着发酵时间延长,Saccharomycetes相对含量明显下降,由32.8%降至0.04%,且含量下降主要发生在前7 d,由32.8%降至3.9%。而Bacilli含量在发酵过程中不断增加,在发酵第7天,已成为酒醅第一优势纲(42.7%),随后又增加至53.7%。Agaricomycetes在发酵过程中呈现逐渐下降趋势,由5.5%降至0.03%。Clostridia含量呈现先升后降的趋势,在发酵7 d 达到最大值(2.5%)。在本实验前期研究中,发现Clostridia是窖泥中的含量高且具有重要潜在功能微生物类群,有助于稳定窖泥微生态并能合成浓香型白酒重要呈香化合物及其前体(尤其是多种脂肪酸)。此外,采用基于DNA水平分子生物学方法也发现Clostridia是酒醅中的优势微生物,并检测到多种梭菌纲中的属,如、和等。
在属水平(图3d),随着发酵时间延长,酒醅优势活性微生物、、和逐渐减少,其中含量下降主要发生在前7 d,由28.4%降至1.6%。、和含量呈先升后降趋势,尤其是后两个属,其含量分别由第3天的0.1%和0.04%增加至第7天的6.8%和4.4%。在发酵过程中不断上升,在发酵第25天成为唯一优势活性微生物属,而上述其他微生物在发酵25 d含量降至0.01%~0.46%之间。
图3e展示了酒醅中含量前50的种(含unidentified)随时间变化的特征,其中发酵7 d和发酵25 d聚为一簇,而3 d酒醅单独聚为一簇。这表明在酒醅主发酵期,随着发酵时间的延长前50活性物种组成逐渐趋于一致,并与发酵初期的组成存在明显的差异。此外,根据其在不同发酵节点的酒醅含量差异可以分为3 类,其中第i类(共7 种)是随发酵时间延长含量逐渐减少的物种,尤其是酿酒酵母()相对含量由27.8%逐渐下降至1.6%(7 d)和0.04%(25 d),以及树干毕赤酵母()相对含量由4.0%下降至2.1%(7 d)和0.02%(25 d);第ii类是发酵7 d含量最高的物种(共14种),主要包括非属中的物种,如变形链球菌()、蜡样芽孢杆菌()和柔嫩梭菌()等,尤其是相对含量在7 d高达8.4%;第iii类生物(28 种和unidentified)主要是随发酵时间延长而增加的物种,其主要为中的物种,且大多数物种是第一次检测到,如和等。综上,在主发酵期,随着发酵时间延长,等真菌物种含量逐渐减少;大多数非属(如及等)中的物种的含量先升后降,在发酵7 d达到最高;而大多数中的物种含量一直在增加,从而在发酵过程中得到了富集。
2.4 DEG分析
2.4.1 DEG筛选结果
图4展示了不同发酵节点酒醅中显著性DEG数量及变化情况,从3 d vs 7 d、7 d vs 25 d、3 d vs 25 d的酒醅样品中测得的DEG数量分别为6 895、2 640 个和6 124 个。这说明3 d酒醅与7 d和与25 d酒醅均有较多的DEG数量(>6 100 个),而7 d酒醅与25 d酒醅的DEG数量相对较少,表明酒醅中活性微生物群落功能明显变化主要发生在发酵前7 d。
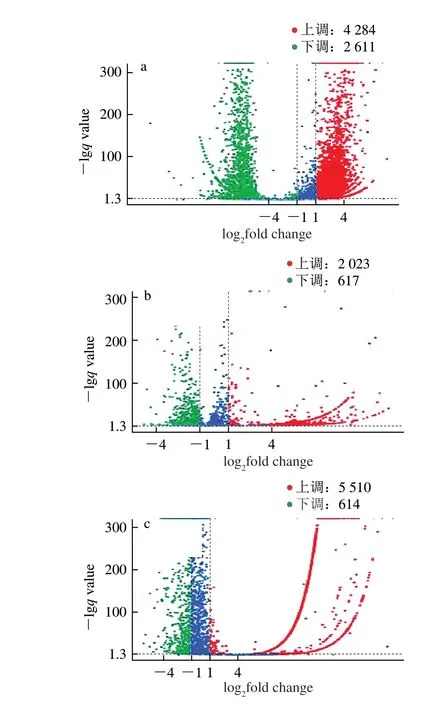
图4 3 d vs 7 d(a)、7 d vs 25 d(b)、3 d vs 25 d(c)酒醅之间的DEG火山图Fig.4 Volcano plot of differentially expressed genes between 3-vs 7-day (a),7-vs 25-day (b),and 3-vs 25-days (c) fermented grains
此外,与7 d酒醅样品相比,3 d酒醅样品显著下调4 284 个(占该组DEG数量的62.1%)和上调2 611 个(37.9%)DEG。同理,25 d酒醅样品相比7 d酒醅样品显著下调2 023 个(占该组DEG数量的76.6%)和上调617 个(23.4%)DEG。综上,随着发酵时间延长,整个主发酵过程中下调的DEG数量明显多于上调数量,表明酒醅微生物群落功能多样性及代谢活性可能逐渐减小,同时也有少部分功能得到增强。
2.4.2 DEG的GO富集分析
由图5a、b可知,发酵3 d酒醅较7 d酒醅下调和上调DEG富集的前30 个功能条目均具有显著性,表明发酵前期微生物的上述代谢功能均存在显著变化。其中7 d较3 d酒醅(图5a)显著上调DEG显著富集的前30功能条目中与生物过程相关的条目20 个,DEG数量共为1 340 个,其中每个条目中DEG数量为9~103 个;与细胞组成相关的条目1 个,含14 个DEG;与分子功能相关的条目9 个,DEG数量共为1 194 个,其中每个条目中基因的数量为5~988 个unigenes。7 d较3 d酒醅显著下调的DEG显著富集的前30功能条目中与生物过程相关的条目6 个,DEG数量共为6 997 个;与细胞组分相关的条目24 个,DEG数量共为47 749 个(图5b)。25 d较7 d酒醅(图5c)中显著上调DEG富集的前30功能条目中仅有催化活性功能条目显著性富集,而显著下调DEG(图5d)富集的前30功能条目均呈显著性富集。其中25 d较7 d酒醅(图5c)中显著上调DEG富集的前30功能条目中与生物过程相关的26 个条目,DEG数量共为1 194 个,与分子功能相关的条目4 个,DEG数量共为239 个;显著下调DEG富集的前30功能条目中,与生物过程相关的条目13 个,DEG数量共为3 426 个;与分子功能相关的条目3 个,DEG数量共为333 个;与细胞组分相关的条目14 个,DEG数量共为6 629 个。综上,不同发酵节点酒醅中显著上调或上调DEG富集的前30功能条目随着发酵时间延长存在一定的动态变化规律:1)发酵7 d相对3 d上调DEG富集的每个功能条目相关基因总数量(共2 548 个)远高于25 d相对7 d上调DEG(共706 个),且富集的条目均集中在与生物过程和分子功能相关的条目,尤其是生物过程;2)发酵7 d相对3 d下调DEG富集的每个功能条目相关基因总数量(共54 746 个)远高于25 d相对7 d下调DEG(共10 388 个),其中富集的条目均集中在与细胞组分和生物过程相关的条目,尤其是细胞组分。
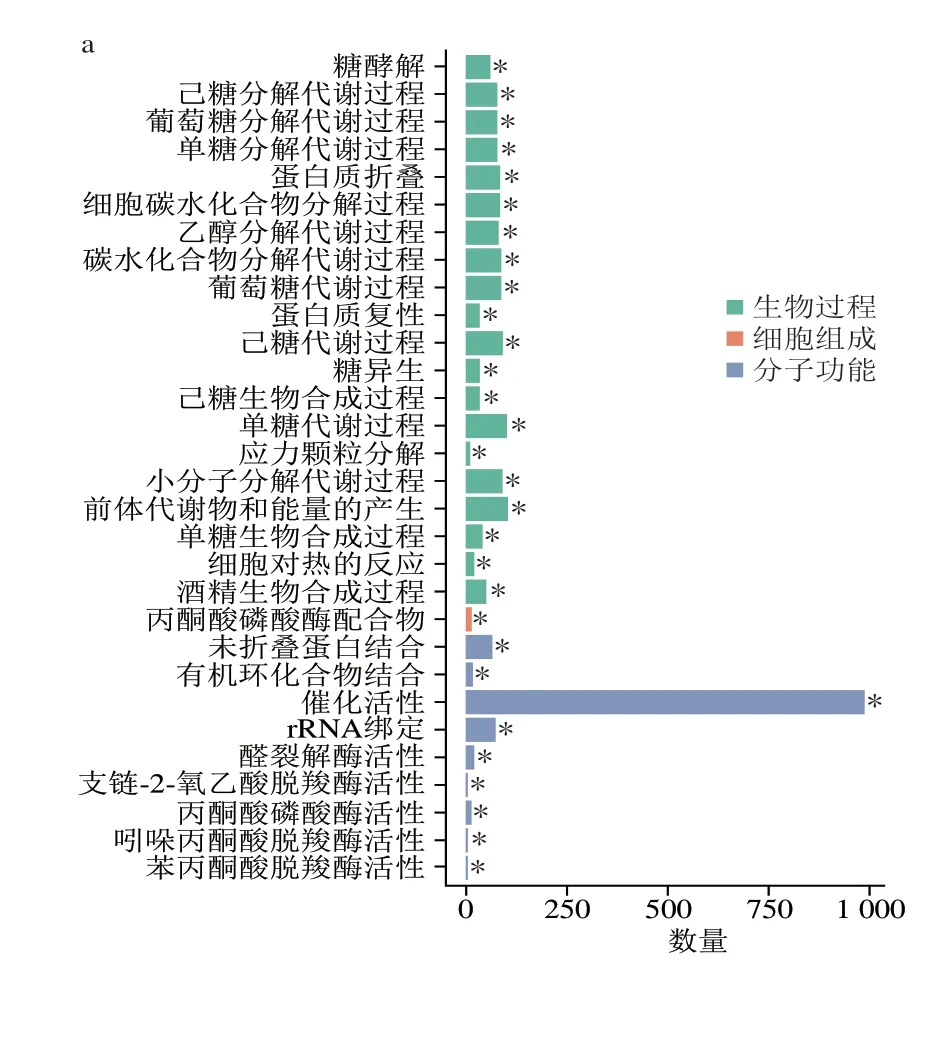
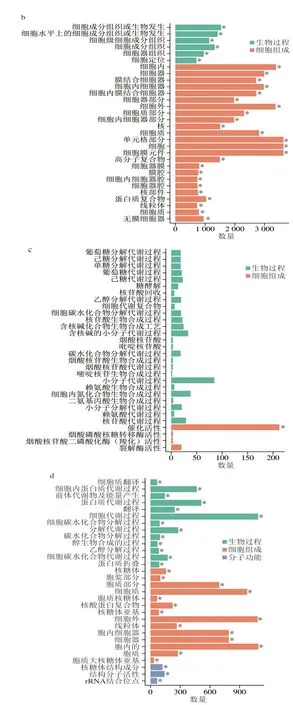
图5 3 d vs 7 d酒醅下调(a)和上调(b)基因、7 d vs 25 d酒醅之间下调(c)和上调(d)基因的GO富集柱状图Fig.5 Significantly enriched GO terms of down-regulated (a) and up-regulated (b) genes between 3 d vs 7 d fermented grains,and downregulated (c) and up-regulated (d) genes between 7 d vs 25 d fermented grains
由图5可知,随着发酵时间延长,酒醅生物上调和下调DEG富集的前30功能条目均存在一定的差异。发酵到7 d,上调DEG显著富集条目中包括糖酵解过程、葡萄糖分解代谢过程、醇类物质合成过程、细胞对热反应等生物过程,以及催化活性和丙酮酸磷酸酶活性等分子功能等30 个条目均得到了显著性富集,其中与糖和醇直接相关的生物过程有14 个条目共含996 个基因(图5a),占所有生物过程条目中基因总数量的74.3%(996/1 340)。从发酵第7天到第25天,上调DEG在富集的前30 个条目中仅有催化活性功能条目具有显著性,其中与糖和醇直接相关的生物过程有9 个条目共含172 个基因,与核酸及氨基酸代谢直接相关的生物过程有15 个条目共含188 个基因(图5c)。发酵7 d,下调DEG富集的30 个条目均具有显著性,且几乎均与细胞组分或与其相关的生物过程相关。从发酵第7天到第25天,下调DEG富集的30 个条目均具有显著性,且主要细胞组分生物过程,含14 个条目6 629 个基因,同时也发现与蛋白质代谢过程及醇类合成过程的条目得到了显著富集。这些结果表明:1)发酵7 d后,酒醅微生物菌群参与糖和醇类化合物代谢的功能可能会显著增强,这也与诸多文献报道结果相似,即发酵1 周后还原糖含量明显下降,而乙醇含量明显上升;2)从发酵第3天至发酵第25天,酒醅微生物丰度可能会出现显著降低,这也Wang Xueshan等研究结果一致,酒醅原核微生物丰度在发酵3 d后呈显著下降趋势。
3 结论
采用宏转录组技术从RNA水平对浓香型不同发酵节点酒醅中活性生物群落及其功能变化规律进行了解析,主要结论包括:酒醅主发酵期浓香型酒醅中可鉴定的活性生物群落主要由细菌和真菌组成,共检测到2 034 个可鉴定的细菌和真菌属,在很大程度上丰富了浓香型酒醅微生物群落多样性内涵;揭示了主发酵期浓香型酒醅中可鉴定的不同分类水平下活性生物群落组成及其变化规律,如Firmicutes和Ascomycota是酒醅中主要优势菌门,其中前者呈上升趋势,而后者呈下降趋势;主发酵期浓香型酒醅微生物群落功能多样性及代谢活性可能逐渐减小,同时也有少部分功能得到增强,且其明显变化主要发生在发酵前7 d;发酵7 d后,酒醅微生物菌群参与糖和醇类化合物代谢的功能可能会显著增强;发酵3~25 d,酒醅活性微生物丰度可能会出现持续显著降低。本研究从活性微生物群落结构和功能角度有助于进一步揭示浓香型白酒固态酿造机制,以期为浓香型白酒生产控制及定向群落组装技术开发提供一定的理论支持。
