细菌RND外排泵的结构与作用机制研究进展
2022-05-30杜静怡王铖郭君慧谢浩
杜静怡?王铖?郭君慧?谢浩


摘要:细菌RND(resistance-nodulation-cell division)外排泵是一类由内膜转运蛋白、周质融合蛋白、外膜外排蛋白组成的蛋白复合体,能够将抗菌药物和多种小分子化合物如毒素、染料、洗涤剂、脂质、群感信号分子等排出细菌细胞外。通过影响RND外排泵的组装和功能,可逆转多重耐药性的发生与防止耐药性的发展。细菌具有多种外排泵,本文以RND外排泵为代表,介绍与其结构以及外排药物的转运和调控机制的最新进展。
关键词:RND外排泵;细菌耐药性;蛋白复合体结构;调控机制
中图分类号:Q735 文献标志码:A
Research progress in the structure and function of RND efflux pump
Du Jing-yi, Wang Chen, Guo Jun-hui, and Xie Hao
(School of Chemistry, Chemical Engineering, and Life Science, Wuhan 430070)
Abstract RND (resistance-nodulation-cell division) efflux pump consists of inner membrane protein, periplasmic membrane fusion protein, and outer membrane protein. It pumps out a wide spectrum of antibiotics and leads to development of multi-drug resistance of host cells. Substrates of RND efflux pump are not limited to antibiotics, but also small molecules such as toxins, dyes, lipids, and quorum sensing signals. Due to the importance of RND efflux pump in multi-drug resistance, one can affect and inhibit the assembly and function of RND efflux pump to prevent and/or reserve multidrug resistance. In this article, the important and latest research progress of the structure as well as the transportation and regulation mechanism of representative RND efflux pumps is reviewed.
Key words RND efflux pump; Antimicrobial resistance; Protein structure; Regulation mechanism
在过去的20年中,多重耐药细菌的种类、数量在快速增加,危害性也日益增強,例如每年美国有高达6.3万人因感染“超级耐药细菌”而死亡,远超过感染艾滋病毒的死亡人数[1]。世界卫生组织的全球监测报告指出,如果不能有效遏制“超级细菌”在全球范围的扩散,由此造成的死亡人数每年超过10万人[1]。但是新的抗菌药物的研发缓慢,远远跟不上耐药细菌出现的速度。因此,迫切需要了解细菌耐药机制,寻找新的治疗方法来对抗耐药细菌。
目前的研究表明,细菌主要通过5种机制(表1)产生耐药性[2-10],分别为:①产生抗菌药物灭活酶,②干扰抗菌药物的作用靶位,③降低细胞膜对抗菌药物的通透性,④主动外排抗菌药物,⑤形成生物被膜。
药物外排泵是位于细胞膜上的蛋白复合体,可以帮助细菌将抗菌药物排出细胞,降低其在细胞内的浓度,在临床中导致病原体对抗菌药物表现出耐药性[11]。在细菌中,主要有5类外排泵[12-13],分别是MFS家族(major facilitator superfamily)[14]、ABC家族(ATP-binding cassette superfamily)[15]、MATE家族(multidrug and toxic compound extrusion superfamily)[16]、RND家族(resistance-nodulation-cell division superfamily)[17]和SMR家族(small multidrug resistance superfamily)[18]。其中,RND外排泵使革兰阴性菌具有内源耐药性,相关的感染更加难以治疗[19-20]。本文将介绍与细菌RND外排泵相关的研究进展,主要包括RND家族成员分布、结构特点、转运机制、相关及潜在应用,从而为基于RND外排泵的新型抗耐药小分子药物研发提供指导。
1 RND家族成员分布
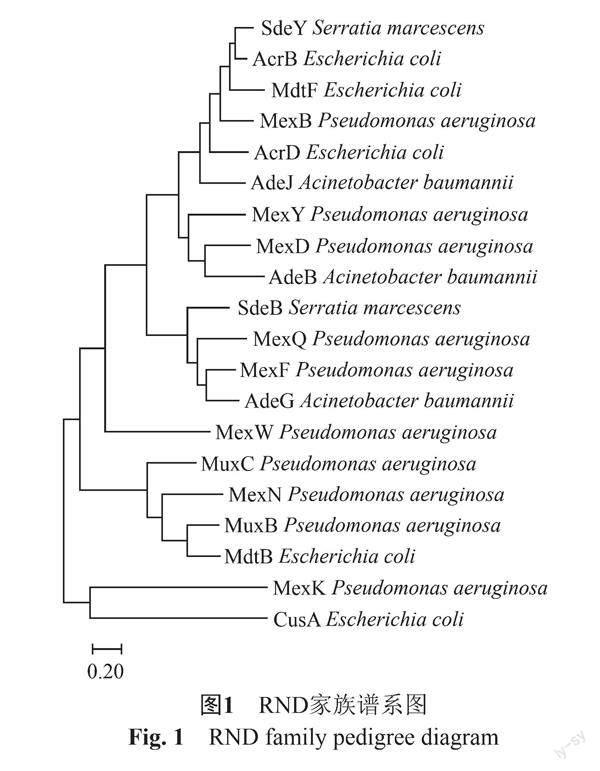
RND蛋白家族广泛存在于细菌、古生菌和真核生物(图1),RND蛋白所组成的外排泵系统主要包括内膜蛋白、周质膜融合蛋白、外膜蛋白3个部分,能够介导多种小分子如抗菌药物、疏水性染料和洗涤剂的跨膜运输[21]。以下是来源于不同菌种的几种代表性的RND外排泵结构及其底物(表2)。
沙雷菌(Serratia marcescens)有8种RND外排泵[22]。其中,SdeCDE系统含有两个内膜蛋白,SdeS系统缺少周质膜融合蛋白。含有周质膜融合蛋白的RND外排泵具有更加广泛的底物范围,在内源性多重耐药机制中具有重要意义[23]。而缺乏周质膜融合蛋白的RND外排泵的底物范围狭窄,例如SdeS系统只外排红霉素、新生霉素、SDS和脱氧胆酸[22]。
大肠埃希菌(Escherichia coli)至少有7种RND外排泵,可以分为疏水/两亲性RND外排泵和重金属RND外排泵两类,分别外排疏水/两亲性抗菌药物和有毒金属离子[24-26]。在疏水/两亲性RND外排泵中,AcrAB-TolC系统与大肠埃希菌天然固有耐药性相关[27]。AcrAB-TolC系统的编码基因为acrAB和tolC,由转录激活因子RamA、SoxS、MarA和Rob调控表达。在环境胁迫下,相关转录激活因子解除抑制,结合到acrAB启动子区域,激活表达[28]。
鲍曼不动杆菌(Acinetobacter baumannii)有7种RND外排泵。其中,最常见的是AdeABC系统、AdeIJK系统和AdeFGH系统[29]。AdeABC系统和AdeFGH系统与获得性耐药相关,过表达时导致多重耐药;AdelJK系统与天然耐药相关。AdeABC系统过表达可造成β-内酰胺类等的外排而导致药物耐受,而对氨基糖苷或氟喹诺酮的影响可以忽略不计[30]。AdeIJK系统中的adeJ表达水平提高可外排哌拉西林、美罗培南、亚胺培南、替加环素、头孢他啶、头孢噻肟和环丙沙星等而导致多重耐药[31]。AdeFGH系统过表达与抗菌药物耐药性以及生物膜形成关系不大[32]。
铜绿假单胞菌(Pseudomonas aeruginosa)有12种RND外排泵,其中MexAB-OprM、MexCD-OprJ、MexEF-OprN、MexXY-OprM在临床耐药菌株中最为常见[33-34]。MexAB-OprM、MexCD-OprJ和MexXY-OprM属于负调节表达,分别受调控基因nalD、nalC、mexR、nfxB和mexZ调控相关操纵子的表达。而MexEF-OprN属于正调节表达,受正调节蛋白MexT调控。MexAB-OprM系统是外排氧氟沙星的主要系统,MexXY-OprA系统是缺乏MexAB的突变体外排氧氟沙星的代用系统,而MexCD-OprJ和MexEF-OprN系统的高水平表达与左氧氟沙星、氟喹诺酮耐药相关[35]。另外,mexB、mexC和mexY基因的过表达导致铜绿假单胞菌的多重耐药性[33]。
肺炎克雷伯菌(Klebsiella pneumoniae)中,目前已报道的RND外排泵有6种,分别是AcrAB、KexD、OqxAB、EefABC、KexEF和KexC。AcrAB外排泵可外排各类抗菌药物,如氟喹诺酮类、大环内酯类、氯霉素和四环素类。其调节因子RamA突变可导致AcrAB过表达,从而降低肺炎克雷伯菌对替加环素的耐药性[36-38]。OqxAB介导肺炎克雷伯菌对喹诺酮类、氯霉素、喹乙醇和替加环素等多种抗菌素的耐药性,受到调节因子RamA和RarA的调控[39-40]。
副溶血性弧菌(Vibrio parahaemolyticus)是一种轻度嗜盐的革兰阴性细菌,可导致食物中毒,主要有12种RND外排泵。其中,VmeAB是导致邻氯青霉素等药物抗性的主要原因,在主动外排胆汁酸过程中也起到重要作用[41]。副溶血性弧菌中的其他RND外排泵与VmeAB类似。其中的VmeCD、VmeEF、VmeYZ分别与VpoC共表达,可以使副溶血性弧菌提高外排底物的种类和范围。
霍乱弧菌(Vibrio cholerae)中已报道的RND外排泵有6种。其中,VexEF外排泵系统具有广泛的底物范围[42],VexCD外排泵的底物范围较窄,与VexAB基本一致。
通过比较不同细菌的RND外排泵,可以发现这些RND外排泵具有以下共同特征。①RND外排泵对广谱的药物外排特性,而含有周质膜融合蛋白的RND周质膜融合蛋白,具有更广泛的外排药物范围。②RND外排泵的底物与来源菌种的生存环境有密切关系,例如大肠埃希菌中的AcrAB-TolC外排泵系统的主要底物是肠道中的多种胆酸盐,副溶血性弧菌的VmeEF-VpoC和VmeYZ-VpoC复合体的主要底物是十二指肠中的胆汁酸。
2 RND外排泵的结构及转运机制
RND外排泵主要是内膜转运蛋白、周质融合蛋白、外膜外排蛋白组成的蛋白复合体(图2A),其结构信息主要来自对大肠埃希菌和铜绿假单胞菌的AcrAB-TolC和MexAB-OprM的研究[28]。这两个复合体是目前理解RND外排泵机制的结构和功能模型,二者的内膜蛋白AcrB与MexB具有86%的相似度,而且在功能上也有一定的相似性。
在AcrAB-TolC中,内膜蛋白AcrB含有跨膜结构域和胞浆结构域,由AcrB亚基组成同源三聚体中存在3种构象,即开放获取(松散的,L)状态,药物结合(紧密的,T)状态,和药物排出(开放的,O)状态,并通过不同构象的循环改变实现药物运输[43]。从AcrB的周质/外膜瓣通过周质域的药物外排途径是许多研究的焦点。AcrB位于胞浆的底物结合位点包含一个浅(近端)结合袋和一个深(远端)结合袋,被残基614-621组成的开关环分隔并影响底物的转运[22]。当开关环中侧链基团较小的甘氨酸残基615突变为侧链基团较大的残基时,会影响大环内酯类抗菌药物(如红霉素)的转运,但不会影响分子量较小化合物(如新生霉素、乙錠、氯霉素)的转运活性[44]。因此,可以利用针对开关环的外排泵抑制剂,影响抗菌药物的外排。Du等[28]构建了外排泵四维结构模型并揭示了TolC、AcrA和AcrBZ复合体的晶体连接结构,通过低温电磁图和热力学角度等研究证明,AcrA分别与AcrB和TolC在周质中独立桥接(图2B):①AcrA的α-发夹结构与周质中圆柱体的6个等效接触面联系整合,该结构与TolC的α-螺旋结构起到桥接作用;②AcrA的膜近端结构域和β-桶状结构域与AcrB子域相互作用且形成封闭的周质通道;③形成的通道结构与AcrB或TolC都没有相互作用。这些发现揭示了自然条件下AcrAB-TolC的结构特点,证实了复合体以3:6:3的AcrB: AcrA: TolC比例组装[45]。
MexAB-OprM系统的整体结构是一个垂直拉长的棒状结构,OprM、MexA和MexB的化学计量比为1:2:1。MexA形成漏斗状六聚体使MexB和OprM结合。在MexA的4个结构域中:α-hairpin结构域与OprM相互作用;β-barrel结构域和MP结构域与MexB相互作用;MexA的脂酰结构域不与OprM、MexB相连,而是与β-barrel结构域形成一个六聚环。Tsutsumi等[46]基于MexAB-OprM的结构研究而构建的底物转运机制模型如下(图3):①6个MexA原聚体与MexB三聚体结合形成一个六聚体管;②封闭的OprM三聚体打开H3-H4螺旋与MexA六聚体相互作用;③H7-H8螺旋向外旋转,被疏水相互作用固定在MexA的一侧;④形成开放状态的MexAB-OprM系统;⑤根据药物浓度的不同,MexB中的静息前聚体会改变其构象,形成结合状态;⑥药物被带入MexB,通过MexA-OprM管,然后根据浓度梯度将药物排出细胞外。
3 RND外排泵的结构和功能调控
3.1 RND外排泵结构的组装及调控
RND型外排泵的内膜蛋白、周质膜融合蛋白和外膜蛋白3个组件,需要正确的装配在一起才能发挥作用。因此,干扰复合体的结构装配过程,可以对功能产生影响。在AcrAB-TolC系统中,AcrB与TolC之间没有直接的作用,而是分别与AcrA发生作用[28]。低温电子断层扫描研究表明,AcrAB复合体通过改变其构象募集TolC[47],AcrB与底物结合时为AcrA六聚体的合成提供了基础,以便于其与TolC的连接过程。这项研究也证实了天然环境下AcrA-AcrB-TolC的存在形式。肽聚糖在AcrAB-TolC的组装过程中起重要作用,AcrA的α-发夹结构域与肽聚糖结合有助于维持AcrAB复合体在细胞膜中的稳定性,因此可以通过干扰AcrA与肽聚糖或AcrB之间的相互作用而阻断AcrAB-TolC的结构组装。
MexAB-OprM是与大肠埃希菌AcrAB-TolC同源的系统。AcrB和MexB的氨基酸序列同源性为70%,AcrA和MexA的同源性为57%,TolC和OprM的同源性为19%[48]。在组装过程中如果将MexB与AcrB互换时,可形成AcrA-MexB-TolC复合体,但与原复合体的形成相比发生率较低[49],因此可能存在通用的机制触发外排泵的组装,形成效率取决于周质膜融合蛋白的结合亲和力。基于这些发现,可通过阻止蛋白质识别或阻碍其结构适应性影响外排泵系统的结构组装,为研究外排泵抑制剂提供了思路。
3.2 RND外排泵基因的表达及调控
外排泵的表达调控涉及众多基因和蛋白,它们以复杂的方式,构成了多层次的网络调控系統。在MexAB-OprM和MexXY系统中,mexR、nalC和nalD基因参与调控MexAB-OprM系统,mexZ、armZ基因参与调控MexXY系统[50]。另外,MexZ通路、PA5471通路、AmgRS和ParRS通路也可诱MexXY表达,其中PA5471通路(图4)研究较为清晰。对于ParRS系统而言[51],ParR调控基因的作用位点,如外排蛋白MexXY (MexXY)编码、多胺生物合成途径PA4773-PA4774-PA4775等,与ParRS一起促进了细菌的耐药性。阐明外排泵基因的调控机制,研发相关抑制剂,阻断调控通路,为逆转耐药性开辟了新前景。
RND外排泵基因在细菌内过表达是细菌耐药性形成的重要原因,而群体感应系统对 RND外排泵基因表达的调控起到重要作用[20]。同时,RND外排泵通过外排群体感应信号分子在群体感应系统中发挥重要作用。两者之间存在互相依赖、互相制约的联系[20]。一方面,群体感应系统能够调控RND外排泵基因的表达,例如以3-oxo-C12-HSL和C4-HSL作为群体感应信号因子的相关试验中,加入的群体感应信号分子显著地诱导外排泵基因表达,提高了菌株对相关抗菌药物的抗性。另一方面,RND外排泵也能影响群体感应系统,例如通过外排泵基因的过量表达而间接影响群体感应系统信号合成基因的表达,从而进一步通过群体感应来降低毒性物质的产量。因此,研究转录调控与群体感应系统的相互关系,可能为解决含有RND蛋白细菌的耐药性提供新思路。
3.3 RND外排泵抑制剂的研发
药物外排泵是细菌耐药性的重要研究靶点,对其抑制剂的发掘尤为重要。对于RND外排泵系统而言,甾体化合物conessine与抗生素竞争和MexAB-OprM的结合位点,抑制外排泵的功能,从而避免细胞内抗生素流失,维持和恢复其抗菌活性[53]。PaβN及其衍生物能够抑制铜绿假单胞菌RND外排泵的功能,提高细菌对氟喹诺酮类、β-内酰胺类和大环内酯类等药物的敏感性[54]。抑制剂虽然能够调控外排泵的活性,但是其临床应用会受到其毒性的限制。目前,外排泵抑制剂的主要研究方向在于植物活性分子,因其具有易于获得以及对人体副作用小等特点。自然产生的利血平、胡椒碱、单萜、儿茶素、花青素和2-(4-丙氧苯基)喹啉衍生物等对金黄色葡萄球菌和肺炎链球菌等菌株有良好抑制作用,甚至可以对其耐药性产生逆转效果[55]。
4 结语
RND外排泵分布于许多致病菌株,是革兰阴性菌产生多重耐药性的主要原因之一。对于AcrAB-TolC、MexAB-OprM、MexXY-OprM等的研究表明,RND外排系统的结构和作用机制主要有以下特点。①外膜蛋白、内膜蛋白、周质膜融合蛋白按一定比例形成跨越内膜、膜间隙、外膜的蛋白复合体,从而在功能上发生关联。②对于大肠埃希菌AcrAB-TolC系统而言,内膜蛋白AcrB存在3种构象(开放获取状态,药物结合状态和药物排出状态),与底物有不同的结合状态,并通过构象循环实现药物运输。其他RND外排泵的内膜蛋白是否也存在与底物结合时发生构象变化的情况,尚有待进一步研究。
基于對RND外排泵的结构和功能研究,为开发新型抗耐药小分子药物提供了思路。首先,可以通过影响RND外排泵结构装配过程以干扰其功能,如通过干预AcrA与肽聚糖或AcrB之间的相互作用而阻断AcrAB-TolC的组装。其次,可以通过阻断RND外排泵的调控通路干扰其外排作用,如阻断MexXY系统中的PA5471通路。深入研究RND外排泵的转录调控以及与群体感应系统的相互关系,也可以为新药研发提供思路。另外,基于RND结构、功能和调控的抑制剂,也是逆转耐药性和未来研究应重点关注的问题。
参 考 文 献
World Health Organization. Antimicrobial resistance: Global report on surveillance 2014[R]. Geneva: WHO, 2014.
Bush K, Jacoby G A, Medeiros A A. A functional classification scheme for beta-lactamases and its correlation with molecular structure[J]. Antimicrob Agents Chemother, 1995, 39(6): 1211-1233.
Khan A A, Manzoor K N, Sultan A, et al. Pulling the brakes on fast and furious multiple drug-resistant (MDR) bacteria[J]. Int J Mol Sci, 2021, 22(2): 859.
Blair J M, Webber M A, Baylay A J, et al. Molecular mechanisms of antibiotic resistance[J]. Nat Rev Microbiol, 2015, 13(1): 42-51.
van Boxtel R,Wattel A A, Arenas J, et al. Acquisition of carbapenem resistance by plasmid-encoded-AmpC-expressing Escherichia coli[J]. Antimicrob Angents Chemother, 2016, 61(1): e01413-16.
Shen T, Chen L, Liu Y, et al. Decanoic acid modification enhances the antibacterial activity of PMAP-23RI-Dec[J]. Eur J Pharm Sci, 2021, 157: 105609.
Poole K. Multidrug resistance in Gram-negative bacteria[J]. Curr Opin Microbiol, 2001, 4 (5): 500-508.
Maslov D A, Shur K V, Vatlin A A, et al. MmpS5-MmpL5 transporters provide mycobacterium smegmatis resistance to imidazo[1,2-b][1,2,4,5]tetrazines[J]. Pathogens, 2020, 9(3): 166.
Cepas V, López Y, Mu?oz E, et al. Relationship between biofilm formation and antimicrobial resistance in Gram-negative bacteria[J]. Microb Drug Resist, 2019, 25(1): 72-79.
Rather M A, Gupta K, Mandal M. Inhibition of biofilm and quorum sensing-regulated virulence factors in Pseudomonas aeruginosa by Cuphea carthagenensis (Jacq.) J. F. Macbr. Leaf extract: An in vitro study[J]. J Ethnopharmacol, 2021, 269: 113699.
Cha H J, Müller R T, Pos K M. Switch-loop flexibility affects transport of large drugs by the promiscuous AcrB multidrug efflux transporter[J]. Antimicrob Agents Chemother, 2014, 58(8): 4767-4772.
AlMatar M, Albarri O, Makky E A, et al. Efflux pump inhibitors: New updates[J]. Pharmacol Rep, 2021, 73(1): 1-16.
Sharma A, Gupta V K, Pathania R. Efflux pump inhibitors for bacterial pathogens: From bench to bedside[J]. Indian J Med Res, 2019, 149(2): 129-145.
Wang S C, Davejan P, Hendargo K J, et al. Expansion of the major facilitator superfamily (MFS) to include novel transporters as well as transmembrane-acting enzymes[J]. Biochimica et Biophysica Acta (BBA)-Biomembranes, 2020, 1862(9): 183277.
Lin Y, Dong Y, Gao Y, et al. Identification of CTL epitopes on efflux pumps of the ATP-binding cassette and the major facilitator superfamily of Mycobacterium tuberculosis[J]. J Immunol Res, 2021, 2021: 8899674.
Kusakizako T, Miyauchi H, Ishitani R, et al. Structural biology of the multidrug and toxic compound extrusion superfamily transporters[J]. Biochim Biophys Acta Biomembr, 2020, 1862(12): 183154.
Klenotic P A, Moseng M A, Morgan C E, et al. Structural and functional diversity of resistance-nodulation-cell division transporters[J]. Chem Rev, 2020. doi: 10.1021/acs.chemrev. 0c00621.
Li X Z, Plésiat P, Nikaido H. The challenge of efflux-mediated antibiotic resistance in Gram-negative bacteria[J]. Clin Microbiol Rev, 2015, 28(2): 337-418.
Butler M S, Cooper M A. Antibiotics in the clinical pipeline in 2011[J]. J Antibiot (Tokyo), 2011, 64(6): 413-425.
梁志彬, 陳豫梅, 陈昱帆, 等. RND外排泵及其与微生物群体感应系统的相互关系[J]. 遗传, 2016, 38(10): 894-901.
Malvacio I, Buonfiglio R, DAtanasio N, et al. Molecular basis for the different interactions of congeneric substrates with the polyspecific transporter AcrB[J]. BiochimBiophys Acta Biomembr, 2019, 1861(7): 1397-1408.
Toba S, Minato Y, Kondo Y, et al. Comprehensive analysis of resistance-nodulation-cell division superfamily (RND) efflux pumps from Serratia marcescens, Db10[J]. Sci Rep, 2019, 9(1): 4854.
Chen J, Kuroda T, Huda M N, et al. An RND-type multidrug efflux pump SdeXY from Serratia marcescens[J]. J Antimicrob Chemother, 2003, 52(2): 176-179.
Nishino K, Yamaguchi A. Analysis of a complete library of putative drug transporter genes in Escherichia coli[J]. J Bacteriol, 2001, 183(20): 5803-5812.
Baranova N, Nikaido H. The baeSR two-component regulatory system activates transcription of the yegMNOB (mdtABCD) transporter gene cluster in Escherichia coli and increases its resistance to novobiocin and deoxycholate[J]. J Bacteriol, 2002, 184(15): 4168-4176.
Bleuel C, Grosse C, Taudte N, et al. TolC is involved in enterobactin efflux across the outer membrane of Escherichia coli[J]. J Bacteriol, 2005, 187(19): 6701-6107.
Du D, Wang Z, James N R, et al. Structure of the AcrAB-TolC multidrug efflux pump[J]. Nature, 2014, 509(7501): 512-515.
Youlden G H, Ricci V, Wang-Kan X, et al. Time dependent asymptotic analysis of the gene regulatory network of the AcrAB-TolC efflux pump system in Gram-negative bacteria[J]. J Math Biol, 2021, 82(4): 31.
Venter H, Mowla R, Ohene-Agyei T, et al. RND-type drug efflux pumps from Gram-negative bacteria: Molecular mechanism and inhibition[J]. Front Microbiol, 2015, 6: 377.
Rajamohan G, Srinivasan V B, Gebreyes W A. Molecular and functional characterization of a novel efflux pump, AmvA, mediating antimicrobial and disinfectant resistance in Acinetobacter baumannii[J]. J Antimicrob Chemother, 2010, 65(9): 1919-1925.
Rosenfeld N, Bouchier C, Courvalin P, et al. Expression of the resistance-nodulation-cell division pump AdeIJK in Acinetobacter baumannii is regulated by AdeN, a TetR-type regulator[J]. Antimicrob Agents Chemother, 2012, 56(5): 2504-2510.
Kim C M, Park G, Ko Y J, et al. Relationships between relative expression of RND efflux pump genes, H33342 efflux activity, biofilm-forming activity, and antimicrobial resistance in Acinetobacter baumannii clinical isolates[J]. Jpn J Infect Dis, 2021, 74(6): 499-506.
Zahedi Bialvaei A, Rahbar M, Hamidi-Farahani R, et al. Expression of RND efflux pumps mediated antibiotic resistance in Pseudomonas aeruginosa clinical strains[J]. Microb Pathog, 2021, 153: 104789.
Fan Z, Pan X, Wang D, et al. Pseudomonas aeruginosa polynucleotide phosphorylase controls tolerance to aminoglycoside antibiotics by regulating the MexXY multidrug efflux pump[J]. Antimicrob Agents Chemother, 2021, 65(2): e01846-20.
Goli H R, Nahaei M R, Rezaee M A, et al. Contribution of mexAB-oprM and mexXY (-oprA) efflux operons in antibiotic resistance of clinical Pseudomonas aeruginosa isolates in Tabriz, Iran[J]. Infect Genet Evol, 2016, 45: 75-82.
Khoshnood S, Heidary M, Hashemi A, et al. Involvement of the AcrAB efflux pump in ciprofloxacin resistance in clinical Klebsiella pneumoniae isolates[J]. Infect Disord Drug Targets, 2020. doi: 10.2174/1871526520999200905121220.
Padilla E, Llobet E, Doménech-Sánchez A, et al. Klebsiella pneumoniae AcrAB efflux pump contributes to antimicrobial resistance and virulence[J]. Antimicrob Agents Chemother, 2010, 54(1): 177-183.
Xu Q, Jiang J, Zhu Z, et al. Efflux pumps AcrAB and OqxAB contribute to nitrofurantoin resistance in an uropathogenic Klebsiella pneumoniae isolate[J]. Int J Antimicrob Agents, 2019, 54(2): 223-227.
Xu Q, Sheng Z, Hao M, et al. RamA upregulates multidrug resistance efflux pumps AcrAB and OqxAB in Klebsiella pneumoniae[J]. Int J Antimicrob Agents, 2021, 57(2): 106251.
Ghanavati R, Kazemian H, Asadollahi P, et al. Characterization of antimicrobial resistance patterns of Klebsiella pneumoniae isolates obtained from wound infections[J]. Infect Disord Drug Targets, 2021, 21(1): 119-124.
Matsuo T, Nakamura K, Kodama T, et al. Characterization of all RND-type multidrug efflux transporters in Vibrio parahaemolyticus[J]. Microbiologyopen, 2013, 2(5): 725-742.
Rahman M M, Matsuo T, Ogawa W, et al. Molecular cloning and characterization of all RND-type efflux transporters in Vibrio cholerae non‐O1[J]. Microbiol Immunol, 2007, 51(11): 1061-1070.
Grimsey E M, Fais C, Marshall R L, et al. Molecular interactions of carbapenem antibiotics with the multidrug efflux transporter AcrB of Escherichia coli[J]. mBio, 2020, 11(3): e00465-20.
Murakami S, Nakashima R, Yamashita E, et al. Crystal structures of a multidrug transporter reveal a functionally rotating mechanism[J]. Nature, 2006, 443(7108): 173-179.
Hobbs E C, Yin X, Paul B J, et al. Conserved small protein associates with the multidrug efflux pump AcrB and differentially affects antibiotic resistance[J]. Proc Natl Acad Sci U S A, 2012, 109(41): 16696-16701.
Tsutsumi K, Yonehara R, Ishizaka-Ikeda E, et al. Structures of the wild-type MexAB-OprM tripartite pump reveal its complex formation and drug efflux mechanism[J]. Nat Commun, 2019, 10(1): 1520.
Shi X, Chen M, Yu Z, et al. In situ structure and assembly of the multidrug efflux pump AcrAB-TolC[J]. Nat Commun, 2019, 10(1): 2635.
Welch A, Awah C U, Jing S, et al. Promiscuous partnering and independent activity of MexB, the multidrug transporter protein from Pseudomonas aeruginosa[J]. Biochem J, 2010, 430(2): 355-364.
Daury L, Orange F, Taveau J C, et al. Tripartite assembly of RND multidrug efflux pumps[J]. Nat Commun, 2016, 7: 10731.
Suresh M, Nithya N, Jayasree P R, et al. Mutational analyses of regulatory genes, mexR, nalC, nalD and mexZ of mexAB-oprM and mexXY operons, in efflux pump hyperexpressing multidrug-resistant clinical isolates of Pseudomonas aeruginosa[J]. World J Microbiol Biotechnol, 2018, 34(6): 83.
Puja H, Bolard A, Noguès A, et al. The efflux pump MexXY/OprM contributes to the tolerance and acquired resistance of Pseudomonas aeruginosa to colistin[J]. Antimicrob Agents Chemother, 2020, 64(4): e02033-19.
鞠曉红, 王月华, 孙艳美. 铜绿假单胞菌MexXY外排泵调控机制研究进展[J]. 中国抗生素杂志, 2019, 44(1): 40-46.
Siriyong T, Srimanote P, Chusri S, et al. Conessine as a novel inhibitor of multidrug efflux pump systems in Pseudomonas aeruginosa[J]. BMC Complement Altern Med, 2017, 17(1): 405.
Askoura M, Mottawea W, Abujamel T, et al. Efflux pump inhibitors (EPIs) as new antimicrobial agents against Pseudomonas aeruginosa[J]. Libyan J Med, 2011. doi: 10.3402/ljm.v6i0.5870.
Sundaramoorthy N S, Mohan H M, Subramaniam S, et al. Ursolic acid inhibits colistin efflux and curtails colistin resistant Enterobacteriaceae[J]. AMB Express, 2019, 9(1): 27.
收稿日期:2021-06-24
基金项目:国家自然科学基金项目(No. 31771032,No. 51911530153);国家级大学生创新创业训练计划资助(No. S202010497055)
作者简介:杜静怡,女,生于2000年,主要研究方向为细菌耐药性研究,E-mail: studentDJY@163.com
*通讯作者,E-mail: h.xie@whut.edu.cn
