铜绿假单胞菌生物被膜基因表达生物信息学研究
2022-05-30廖黎鲁兰杨晨刘昆赵玉婷
廖黎?鲁兰?杨晨?刘昆?赵玉婷

摘要:目的 通過生物信息学探究铜绿假单胞菌生物被膜形成的差异表达基因及可能作用机制。方法 通过GEO数据库筛选获得铜绿假单胞菌生物被膜数据芯片GSE120760, GEO2R工具筛选出铜绿假单胞菌浮游状态和生物被膜状态的差异表达基因;差异表达基因通过String数据库和Cytoscape3.7.1软件中构建靶点PPI网络图, 并在DAVID和KOBAS数据库中对差异表达基因进行GO生物富集和KEGG通路分析。用铜绿假单胞菌体外生物被膜模型测定5种氨基酸(D/L型)的抗生物被膜作用效果。结果 由芯片GSE120760筛选得到556个差异表达基因, 其中上调基因143个, 下调基因413个;从差异表达基因中筛选到62个关键基因, 这些基因主要与30S和50S核糖体蛋白相关;GO功能富集得到40个条目;KEGG 富集到44条通路, 主要涉及代谢途径、次生代谢产物的生物合成、氨基酸生物合成通路、多种氨基酸代谢、群体感应、氨酰tRNA生物合成通路等;体外验证试验中,D-氨基酸对铜绿假单胞菌生物被膜有抑制作用, 而L-氨基酸对其形成无影响。结论 通过生物信息学挖掘出铜绿假单胞菌生物被膜形成的关键基因与靶点, 并与代谢途径、氨基酸合成和代谢、群体感应、氨酰tRNA生物合成通路等相关, 为铜绿假单胞菌生物被膜的靶向抗感染药物的研发提供思路。
关键词:铜绿假单胞菌;生物被膜;生物信息学;差异表达基因;D-氨基酸;L-氨基酸
中图分类号:R9文献标志码:A
Bioinformatics analysis of Pseudomonas aeruginosa biofilm on gene expression
Liao Li, Lu Lan, Yang Chen, Liu Kun, and Zhao Yu-Ting
(Antibiotics Research and Re-evaluation Key Laboratory of Sichuan Province, Sichuan Industrial Institute of Antibiotics, School of Pharmacy, Chengdu University, Chengdu 610106)
Abstract Objective The aim of study is to investigate differentially expressed genes (DEGs) in the biofilm formation of Pseudomonas aeruginosa (PA) and its possible mechanism through bioinformatics analysis. Methods Data chip GSE120760 of biofilm in Pseudomonas aeruginosa was obtained in GEO database. The DEGs in planktonic status and biofilm status of PA were identified by GEO2R. The PPI network map of DEGs was constructed by string database and Cytoscape 3.7.1 software. In David and Kobas database, GO bioaccumulation and KEGG pathway analysis were carried out on DEGs. The anti-biofilm effects of five amino acids (D/L) were determined by the in vitro biofilm model of PA. Results Five hundred and fifty-six DEGs were selected from GSE120760, including 143 up-regulated genes and 413 down-regulated genes. Sixty-two key genes were screened out from DEGs, which were mainly related to 30S and 50S ribosomal proteins. Forty items were enriched in GO function. In KEGG pathway analysis, forty-four pathways were obtained, which were mainly related to metabolic pathway, biosynthesis of secondary metabolites, biosynthesis of amino acid, amino acid metabolism, quorum sensing, aminoacyl-tRNA biosynthesis. In vitro, D-amino acids had inhibitory effects on PA biofilm formation, while L-amino acids had no effect on PA biofilm formation. Conclusion Key genes in PA biofilm formation were identified by bioinformatics analysis, which was related to metabolic pathway, biosynthesis and metabolism of amino acid, aminoacyl-tRNA biosynthesis. Our findings would provide ideas for the development of anti-infective drugs targeting biofilm in PA.
Key wordsPseudomonas aeruginosa; Biofilm; Bioinformatics; Differentially expressed genes; D-amino acids; L-amino acids
铜绿假单胞菌(Pseudomonas aeruginosa, PA)是临床常见条件致病菌,当人体的组织屏障破坏、进行有创医疗操作或免疫功能低下時,可引起人体组织局部化脓性炎症及全身感染,如呼吸道感染、肺炎、败血症等[1]。人体感染铜绿假单胞菌后,导致的慢性感染和反复感染很难完全治愈,这与其形成生物被膜(biofilm, BF)有关[2]。临床数据显示,铜绿假单胞菌在肺囊性纤维化(cystic fibrosis, CF)中形成对耐药生物被膜而阻碍抗生素疗效[3]。另外,伤口慢性感染、慢性鼻窦炎、慢性中耳炎等也与铜绿假单胞菌生物被膜生成有关[4]。生物被膜是附着在惰性或活性表面,由细菌及其分泌胞外多糖等物质形成结构化膜状细菌群体[5]。研究发现,高达80%的人类细菌感染与其生物被膜形成有关[6],此外成熟生物被膜细菌耐药性比浮游菌高500~5000倍[7]。这可能与生物被膜阻止药物进入、限制细菌营养供给、减缓细菌生长、降低其对抗生素的敏感性、逃逸宿主免疫系统作用等相关[8]。
本研究通过生物信息学技术,筛选与铜绿假单胞菌生物被膜形成有关的功能基因,并与浮游菌比较获得差异表达基因(DEGs)[9],再对这些差异表达基因进行富集功能及通路分析,探讨铜绿假单胞菌形成生物被膜机制,为实验与临床抑制铜绿假单胞菌生物被膜提供理论依据和新思路。
1 材料与方法
1.1 公共数据收集
美国国立生物技术信息中心(NCBI)基因表达综合数据库GEO (http://www.ncbi.nlm.gov/geo/)是高通量基因表达,芯片和微阵列数据的功能基因组学数据库[10]。从数据库筛选出铜绿假单胞菌在慢性创面渗出物中的表达分析数据芯片GSE120760(芯片信息:Affymetrix Pseudomonas aeruginosa array,GPL84)。该芯片以3株浮游铜绿假单胞菌及3株生物被膜铜绿假单胞菌为研究对象。
1.2 数据筛选处理
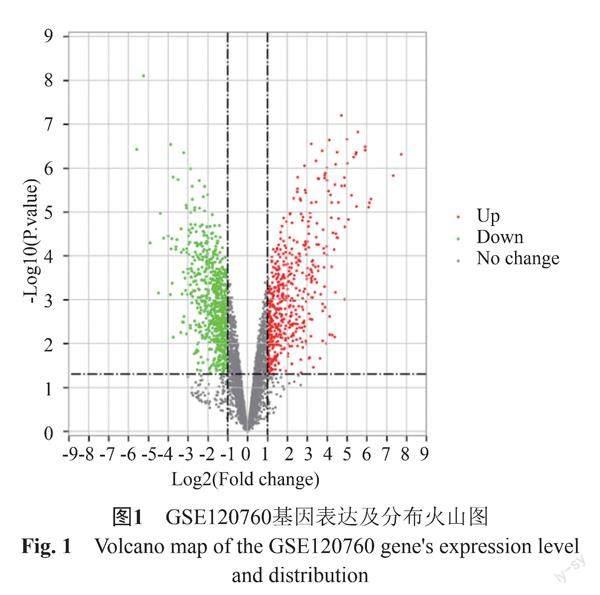
GEO数据库自带在线分析工具GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)是一个交互式网页工具,它可以比较GEO系列中的两个或多个数据集,确定实验条件下的差异表达基因[11],用其筛选浮游铜绿假单胞菌和生物被膜铜绿假单胞菌中的差异表达基因,筛选标准为P<0.05,log FC >1或log FC <-1,log FC >1的基因为上调差异表达基因,log FC<-1的基因为下调差异表达基因。绘制差异表达基因火山图并进行后续分析。
1.3 差异表达基因相互作用分析和核心基因筛选
将差异表达基因导入String数据库(http://string-db.org/),设置种属铜绿假单胞菌,去除无连接靶点,minimum required interaction score设置为0.9,预测蛋白相互作用。再将分析结果导入Cytosacpe3.7.1软件构建蛋白交互网络图(protein-protein interaction,PPI)。使用CytoHubba插件对其网络进行关联度分析[12],根据节点度大小筛选核心基因。
1.4 基因本体与通路富集分析
将差异表达基因分别在DAVID数据库(http://david.ncifcrf.gov/)中进行基因本体(gene ontology,GO)生物富集分析,以P<0.05作为筛选标准,用生物学过程(biological process,BP)、细胞组分(cell component,CC)、分子功能(molecular function,MF),绘制GO注释图;在KOBAS数据库(http://kobas.cbi.pku.edu.cn/)中进行京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路注释分析,以P<0.05作为标准筛选,绘制KEGG分析图。
1.5 D/L-氨基酸体外对铜绿假单胞菌生物被膜作用试验
1.5.1 材料和仪器
D-色氨酸(BTSJ20200906)、D-丝氨酸(BTSJ20200701)、D-缬氨酸(BTSJ20201021)、D-丙氨酸(BTSJ20201004)、D-酪氨酸(BT8J19XBQ)、L-色氨酸(BTSJ20200714)、L-丝氨酸(BTSJ20200114)、L-缬氨酸(BTSJ20200902)、L-丙氨酸(BTSJ20200516)、L-酪氨酸(BTSJ20200309),均购于成都润泽本土化工有限公司;近期收集的临床分离致病铜绿假单胞菌10株,来源于四川抗菌素工业研究所;胰酪大豆琼脂培养基 TSA(批号:8063172),美国BD公司;营养肉汤 NB(批号:20200512),北京奥博星生物技术有限责任公司;生理盐水(批号:M0080204C),四川科伦药业股份有限公司;结晶紫(批号:9301041),北京索莱宝科技有限公司;无水乙醇(批号:2019121801),成都市科龙化工试剂厂;无菌96孔板(批号:010617A005), NEST公司;麦氏比浊管(批号:075870),广东环凯微生物科技有限公司;Multiskan FC型酶标仪,赛默飞世尔上海仪器有限公司;GNP-9080隔水式恒温培养箱,上海精宏实验设备有限公司;Sartorius BS124S型电子天平,德国Sartorius公司。
1.5.2 方法
(1)铜绿假单胞菌产生物被膜菌株筛选
取铜绿假单胞菌10株,于TSA平板复苏过夜,挑取单个菌落于无菌生理盐水,调至麦氏比浊度1.0,灭菌营养肉汤稀释20倍备用。取96孔板于第2~11列,每列第2~7行6个复孔中分别加入稀释好的10株菌液200 μL,其余孔加入无菌生理盐水200 μL,置37℃恒温培养72 h。
取出96孔板,倒掉剩余培养液,用无菌生理盐水小心洗涤2次,去除浮游菌,加入2%结晶紫溶液250 μL染色10 min,再用无菌生理盐水缓慢洗去多余结晶紫,加入250 μL无水乙醇溶解,最后用酶标仪测定96孔板各孔在 570 nm处A值[14]。
(2)D/L-氨基酸体外对铜绿假单胞菌生物被膜形成抑制作用试验
取筛选出产膜能力强的铜绿假单胞菌1株于TSA平板复苏过夜,挑取单个菌落于无菌生理盐水,调至麦氏比浊度1.0,灭菌营养肉汤稀释10倍备用。取96孔板,在第1~2列加入无菌营养肉汤100 μL,作为菌对照;第3~12列中分别加入D-酪氨酸、L-酪氨酸、D-色氨酸、L-色氨酸、D-丙氨酸、L-丙氨酸、D-丝氨酸、L-丝氨酸、D-缬氨酸、L-缬氨酸各200 mmol/L的营养肉汤100 μL[13],每列6个复孔。再在第1~12列的6个复孔中加入稀释好的菌液100 μL,其余孔加入无菌生理盐水200 μL,置37℃恒温培养72 h。按“1.5.2(2)”项下的结晶紫染色方法处理,并测定其在570 nm 处A值。
1.5.3 统计学分析
结果用平均数±标准差(x±s)表示。使用Prism-graph 8.0对铜绿假单胞菌试验数据进行分析和统计图绘制。各组间的比较进行单因素方差分析,并进行统计学分析的Tukey's测试。P<0.05被认为具有统计学意义。
2 结果
2.1 铜绿假单胞菌中差异表达基因分析
GSE120760芯片中的3株浮游铜绿假单胞菌设为control组,3株生物被膜铜绿假单胞菌设为biofilm组,利用GEO2R工具共筛选出556个差异表达基因,其中143个为上调基因,413 个为下调基因,见图1,红色代表上调基因,绿色代表下调基因,灰色代表无显著差异表达基因。
2.2 蛋白交互PPI网络图构建及核心基因分析
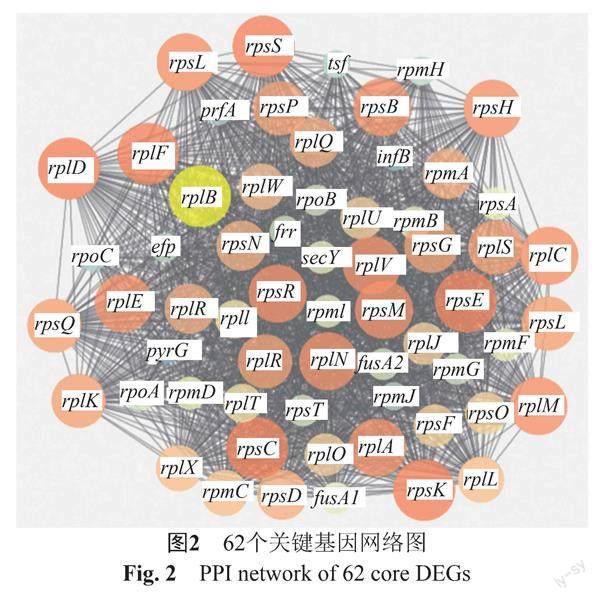
通过String数据库和Cytosacpe3.7.1构建556个差异表达基因间的蛋白交互网络。去除无交集蛋白,得到428个点,2537条边。通过CytoHubba对网络进行分析,根据节点degree值大小排名,其中degree值越大,表明其在网络图中相互作用程度越高,取degree值大于30的基因,得到62个关键基因,部分信息见表1,多数基因为30S核糖体蛋白和50S核糖体蛋白。将这62个关键基因用String数据库和Cytosacpe3.7.1软件构建蛋白交互網络图(PPI),见图2。根据每个节点degree值大小确定其大小颜色,得到边线数1668条,聚类系数为0.924,平均节点degree值为53.806,表明基因连通性好。
2.3 差异表达基因GO生物富集和KEGG通路分析结果
利用DAVID数据库对差异表达基因进行GO生物富集分析,得到生物学过程(BP)、细胞组分(CC)、分子功能(MF)条目共40个,见图3,其中BP主要富集于翻译、TCA循环、IMP生物合成过程、ATP合成与质子电子传输耦合、吩嗪生物合成过程、次生代谢产物生物合成过程、吡哆醇生物合成过程、苏氨酸、组氨酸、精氨酸生物合成过程、糖酵解、有氧呼吸等;CC主要富集于胞浆大小核糖体亚单位、细胞质、核糖体等;MF主要富集于核糖体结构组成、RNA结合、镁离子结合、ATP结合、氧化还原酶活性、细胞色素c氧化酶活性等。
通过KOBAS数据库对差异表达基因进行KEGG通路分析,得到44条通路,见图4,差异表达基因通路主要富集于代谢途径、抗生素生物合成、次生代谢产物的生物合成、氨基酸生物合成、不同环境中的微生物代谢、群体感应、甘氨酸、丝氨酸及丙氨酸等多种氨基酸代谢、赖氨酸、酪氨酸及缬氨酸等多种氨基酸生物合成、TCA循环、糖酵解、铜绿假单胞菌生物被膜形成、氨酰tRNA生物合成、DNA复制、D-丙氨酸代谢、阳离子抗菌肽(CAMP)耐药性等。
2.4 D/L-氨基酸对铜绿假单胞菌生物被膜形成影响
KEGG通路中,L型氨基酸色氨酸、丝氨酸、缬氨酸、丙氨酸、酪氨酸参与代谢途径,氨基酸生物合成,氨酰tRNA生物合成,为了验证这些D型氨基酸与L型氨基酸竞争参与上述通路而对铜绿假单胞菌生物被膜形成起到抑制作用,通过D/L氨基酸体外对铜绿假单胞菌生物被膜形成抑制作用试验。首先采用结晶紫染色法检测近期收集的10株临床分离铜绿假单胞菌产生生物被膜的水平,见图5,由结果筛选出铜绿假单胞菌1809产膜能力强,用于后续试验。
D/L-氨基酸对铜绿假单胞菌1809生物被膜形成影响作用如表2与图6所示。各氨基酸在96孔板终浓度为100 mmol/L时,D-酪氨酸、D-色氨酸、D-丙氨酸、D-丝氨酸、D-缬氨酸与对照组比较,各组吸光度均显著降低(P<0. 05或P<0. 01),表明其抑制铜绿假单胞菌生物被膜形成。L-酪氨酸、L-色氨酸、L-丙氨酸、L-丝氨酸、L-缬氨酸与对照组比较,各组吸光度无显著变化,表明其对铜绿假单胞菌生物被膜形成无明显影响。
3 讨论
随着抗生素大量应用,临床耐药铜绿假单胞菌分离率逐年增高,其耐药形势严峻,已成为临床治疗其感染难题[15]。铜绿假单胞菌耐药机制复杂多变,生物被膜形成是其产生耐药的重要途径,也是导致院内长期和反复感染的原因之一[16]。针对临床铜绿假单胞菌的生长及代谢特点,研发新的抗感染药物及建立新的治疗模式非常迫切。生物信息学以基因芯片研究为基础,从基因芯片中得到大量、复杂生物信息数据,运用序列比对、统计分析、生物分子网络、通路分析及可视化作图等方法,对生物信息数据进行挖掘[17],从而全面对铜绿假单胞菌生物被膜进行系统研究。本研究通过分析GEO数据库中GSE120760芯片筛选生物被膜铜绿假单胞菌和浮游铜绿假单胞菌间有显著差异表达基因556个,其中上调基因143个,下调基因413个。
通过String数据库和Cytosacpe3.7.1构建差异表达基因间的PPI网络图,利用CytoHubba插件得到62個关键基因,包括50S核糖体蛋白(rplB、rplD、rplM、rplC和rplE等)、30S核糖体蛋白(rpsE、rpsK、rpsS、rpsC和rpsM等)及其他(rpoB、secY和frr等),而核糖体是蛋白质合成主要场所,核糖体、氨基酸、蛋白质与铜绿假单胞菌生物被膜形成、发展密切相关。GO富集分析表明,差异表达基因富集在翻译、TCA循环、ATP合成与质子电子传输耦合、苏氨酸、组氨酸、精氨酸生物合成过程、糖酵解、有氧呼吸等生物过程。KEGG通路分析表明,差异表达基因富集于与代谢途径、次生代谢产物生物合成、氨基酸生物合成、群体感应、甘氨酸、丝氨酸及丙氨酸等多种氨基酸代谢、TCA循环、铜绿假单胞菌生物被膜形成、氨酰tRNA生物合成、D-丙氨酸代谢等。生物体通常选择L-氨基酸作为合成多肽基本单元和关键代谢中间体,参与蛋白质合成[18] ,蛋白质合成系统也依靠 L-立体专一性氨基酰-tRNA合成酶区分L/D型氨基酸,并将L-氨基酸掺入到合成的蛋白质链中,如酪胺酰-tRNA合成酶(TyrRS)无法区分L-酪氨酸和D-酪氨酸,从而造成D-酪氨酸对细菌潜在毒性[19]。
此外,D-氨基酸还可通过改变生物被膜细胞肽聚糖层结构,干扰蛋白质合成等造成细胞外基质构象疏松,使生物被膜整体结构塌陷[5]。文献报道,D型氨基酸能抑制细菌生物被膜形成,如D-缬氨酸(75 mmol/L)能抑制牙龈卟啉单胞菌生物被膜的形成并分散成熟的生物被膜,而L-缬氨酸能促进生物被膜的形成[20]。D-亮氨酸、D-蛋氨酸、D-色氨酸和D-酪氨酸在毫摩尔甚至微摩尔数量级下能分散枯草芽孢杆菌形成的生物被膜,从而破坏其生物被膜,而相应的L-型氨基酸对其生物被膜无影响[21]。D-酪氨酸能显著抑制铜绿假单胞菌生物被膜,联合丁胺卡那霉素可提高其抗铜绿假单胞菌生物被膜效果,增强其抑制铜绿假单胞菌生物被膜菌群的活性[22]。这些发现均表明一些D-氨基酸在抑制生物被膜形成和分散成熟生物被膜中具有的潜力[23-24]。
KEGG通路中,L型氨基酸色氨酸、丝氨酸、缬氨酸、丙氨酸、酪氨酸等都参与代谢途径,氨基酸生物合成,氨酰tRNA生物合成。D型氨基酸与L型氨基酸空间构型的不同,是否D型氨基酸能与L型氨基酸在生物合成反应中存在竞争关系,从而阻断这些通路来抑制铜绿假单胞菌生物被膜形成,值得我们探索。因此,本研究选用上述D/L-氨基酸进行体外抗铜绿假单胞菌生物被膜作用比较试验。结果得到这些D-氨基酸在浓度为100 mmol/L时,能显著抑制铜绿假单胞菌生物被膜形成,而L-氨基酸对其生物被膜形成无明显影响,从而推测D型氨基酸可能通过与L型氨基酸竞争参与代谢途径,氨基酸生物合成,氨酰tRNA生物合成和氨基酸代谢通路对铜绿假单胞菌生物被膜形成起到抑制作用。
综上所述,本研究通过生物信息学挖掘铜绿假单胞菌生物被膜相关基因芯片数据,并分析其差异表达基因、关键靶点、通路,通过这些信息我们将进一步认识铜绿假单胞菌形成生物被膜过程及可能作用机制,并通过体外试验验证D型氨基酸对铜绿假单胞菌生物被膜形成的抑制作用及可能作用机制,为以铜绿假单胞菌生物被膜为靶点的靶向抗感染药物的研发提供思路。
参 考 文 献
刘传玲, 王佳贺. 中药抗耐药铜绿假单胞菌机制的研究进展[J]. 中国医药导报, 2019, 16(3): 40-43.
Girard G, Bloemberg G V. Central role of quorum sensing in regulating the production of pathogenicity factors in Pseudomonas aeruginosa[J]. Future Microbiol, 2008, 3(1): 97-106.
Moreau-Marquis S, Coutermarsh B, Stanton B A. Combination of hypothiocyanite and lactoferrin (ALX-109) enhances the ability of tobramycin and aztreonam to eliminate Pseudomonas aeruginosa biofilms growing on cystic fibrosis airway epithelial cells[J]. J Antimicrob Chemother, 2015, 70(1): 160-166.
Sanchez Z, Tani A, Kimbara K. Extensive reduction of cell viability and enhanced matrix production in Pseudomonas aeruginosa PAO1 flow biofilms treated with a D-amino acid mixture[J]. Appl Environ Microbiol, 2013, 79(4): 1396-1399.
齐华, 王鹤龄, 李保胜, 等. D-氨基酸对生物膜抑制作用的研究进展[J]. 现代口腔医学杂志, 2017, 31(4): 242-244.
Wood T K, Hong S H, Ma Q. Engineering biofilm formation and dispersal[J]. Trends Biotechnol, 2011, 29(2): 87-94.
何年安, 王文平. 超声在细菌生物膜相关感染治疗中的应用[J]. 中华超声影像学杂志, 2012(9): 815-817.
Peeters E, Nelis H J, Coenye T. Comparison of multiple methods for quantification of microbial biofilms grown in microtiter plates[J]. J Microbiol Methods, 2008, 72(2): 157-165.
邱洁萍, 孙梦雨, 左瑞东, 等. 基于GEO数据库的胃癌差异表达基因的生物信息学分析[J]. 中国免疫学杂志, 2020, 36(19): 2394-2399.
Ma C, Xu T, Sun X, et al. Network pharmacology and bioinformatics approach reveals the therapeutic mechanism of action of baicalein in hepatocellular carcinoma[J]. Evid Based Complement Alternat Med, 2019(2): 224-238.
Li Z, Liu J, Wang W, et al. Investigation of hub genes involved in diabetic nephropathy using biological informatics methods[J]. Ann Transl Med, 2020, 8(17): 1087-1098.
Chin C H, Chen S H, Wu H H, et al. CytoHubba: Identifying hub objects and sub-networks from complex interactome[J]. BMC Systems Biol, 2014, 8(S4): S11.
KolodkinGal I, Romero D, Shugeng C, et al. D-Amino acids trigger biofilm disassembly[J]. Science, 2010, 328(5978): 627-629.
程強, 曾铭, 鲁兰, 等. 四季青水煎液对铜绿假单胞菌生物被膜生成、毒力因子分泌的抑制作用及机制研究[J]. 中药药理与临床, 2019, 35(4): 111-116.
胡付品, 郭燕, 朱德妹, 等. 2018年CHINET中国细菌耐药性监测[J]. 中国感染与化疗杂志, 2020, 20(1): 1-10.
郭孝兰, 李永伟. 铜绿假单胞菌耐药机制与生物被膜相关性研究进展[J]. 新发传染病电子杂志, 2017, 2(3): 188-191.
郁乐, 张轶雯, 辛文秀, 等. 基于GEO芯片数据的肝癌特征基因生物信息学及预后关联性分析[J]. 中国现代应用药学, 2019, 36(17): 2177-2182.
薛二淑, 吴昊, 宋倩倩, 等. 细菌中D-氨基酸生物合成及调控作用研究进展[J]. 中国生物工程杂志, 2019, 39(4): 106-113.
纪芳, 陈博磊, 梁勇. D-氨基酸在细菌生理中的结构性能和调节功能的研究进展[J]. 微生物学报, 2018, 58(12): 2078-2086.
张慧彦, 李保胜, 张震阳, 等. D/L-缬氨酸对牙龈卟啉单胞菌及其生物膜的作用[J]. 口腔医学研究, 2020, 36(7): 648-653.
Romero D, Vlamakis H, Losick R, et al. An accessory protein required for anchoring and assembly of amyloid fibres in B. subtilis biofilms[J]. Mol Microbiol, 2011, 80(5): 1158-1168.
She P, Chen L, Liu H,, et al. The effects of D-Tyrosine combined with amikacin on the biofilms of Pseudomonas aeruginosa[J]. Microb Pathog, 2015, 86(6): 38-44.
Cava F, Lam H, Miguel A, et al. Emerging knowledge of regulatory roles of D-amino acids in bacteria[J]. Cell Mol Life Sci, 2011, 68(5): 817-831.
佘鹏飞, 陈丽华, 许欢, 等. D-氨基酸在生物膜分散中的作用研究进展[J]. 中国病原生物学杂志, 2015, 10(4): 377-380.
收稿日期:2021-03-29
基金项目:国家自然科学基金(No. 81803812)
作者简介:廖黎,女,生于1987年,在读硕士研究生,研究方向为中药药理及机理,E-mail: 493993621@qq.com
*通讯作者,E-mail: 345747278@qq.com
