MRI 不同影像组学模型预测胶质瘤MGMT启动子甲基化状态的研究
2022-05-28陈思璇许悦叶梅萍李扬于芷轩青钊王正阁张冰张鑫
陈思璇,许悦,叶梅萍,李扬,于芷轩,青钊,2,王正阁,张冰,2,张鑫*
作者单位:1.南京大学医学院附属鼓楼医院医学影像科,南京210093;2.南京大学脑科学研究院,南京210093;3.南京大学健康医疗大数据国家研究院,南京210093
胶质瘤是由不同分化阶段的神经上皮组织衍生的胶质细胞生成的肿瘤的总称,它是中枢神经系统最常见的肿瘤[1]。O6-甲基鸟嘌呤-DNA 甲基转移酶(O6-methylguanine-DNA methyltransferase,MGMT)启动子甲基化是评估脑胶质瘤患者对烷化剂药物替莫唑胺敏感性的重要分子依据,并且是评价脑胶质瘤患者预后情况的重要分子指标,在指导临床制订治疗方案以延长患者生存期方面具有重要价值[2-3]。检测MGMT启动子甲基化对诊断及鉴别诊断脑胶质瘤具有重要意义。而目前检测主要依赖于手术获取组织进行基因分析[4]。
分子诊断、人工智能算法的迅速发展使得利用影像组学研究肿瘤分子标志物成为热门研究课题之一。影像组学是指高通量地提取大量影像特征,应用自动化数据特征化算法将影像数据转化为具有高分辨率、可深度挖掘的特征空间数据并进行建模分析,对疾病诊断、治疗及预后等进行客观定量分析,其可以反映潜在的组织和病变特征,如肿瘤的异质性[5,6]。MGMT 启动子甲基化与胶质瘤MRI 研究不充分,预测胶质瘤MGMT 启动子甲基化仍然是临床上面临的挑战,需要进一步的研究[7]。选择不同影像组学模型及影像图像建模会影响模型的诊断性能[7-8]。
目前在运用影像组学进行胶质瘤MGMT启动子甲基化的预测研究中,研究主要局限在胶质母细胞瘤以及单一的影像组学方法研究,缺乏对不同影像组学模型进行对比研究,因此,本研究基于MRI 平扫T1WI、T2WI、ADC和增强T1WI的影像组学特征,采用不同的影像组学模型构建模型进行研究,探讨胶质瘤MGMT启动子甲基化状态的预测效能。
1 材料与方法
1.1 研究对象
回顾性分析2017 年1 月至2019 年12 月南京鼓楼医院符合以下标准的患者病例,纳入标准:(1)经手术切除,病理证实WHO 分级2~4 级(WHO 2021 版中枢神经系统肿瘤分类);(2)术前行MRI 平扫及增强检查(T1WI、T2WI、ADC、T1WI 增强序列);(3)术前均未进行肿瘤治疗。排除标准:(1)图像质量不佳;(2)图像序列缺损。共114例符合上述标准的患者纳入研究,其中MGMT 启动子甲基化阳性58 例,阴性56 例。数据按8∶2 的比例分割为训练组与验证组,训练组91 例,验证组23例。本回顾性研究经南京大学医学院附属鼓楼医院医学伦理委员会批准(编号:2019-065-01),免除受试者知情同意。
1.2 MRI扫描
采用Philips Achieva 3.0 T MRI扫描仪和联影uMR770 3.0 T MRI 扫描仪。MR 增强扫描:轴位T1WI增强扫描,对比剂为钆喷替酸葡甲胺(Gd-DPTA),用量为0.2 mmol/kg。扫描参数如表1所示。

表1 MRI扫描参数Tab.1 MRI scanning parameters
1.3 MRI特征提取
使 用ITK-SNAP 软 件( 版 本3.8.0,https://itksnap.org/)对未进行预处理T2WI 及T1WI 增强DICOM 原始图像进行三维分割。在T2WI 及T1WI 增强图像上,由2 名影像科医生(具有3 年工作经验)分别独立勾画肿瘤加水肿区域及肿瘤核心区域2 种感兴趣区域(region of interest,ROI) (肿瘤加水肿区域参考T2WI 高信号区域勾画,肿瘤核心区通过参照增强T1WI强化区域勾画),ROI包含每层病灶的图像。如果两位影像科医生勾画的ROI 之间存在的差异低于5%,将两个ROI 融合。如果两个ROI 之间存在的差异高于5%,由1名影像科医生(具有10年工作经验)来确定最终勾画。
为了能够高通量地从MRI图像中提取肿瘤部分的影像组学特征,我们采用开源软件PyRadiomic(https://pyradiomics.readthedocs.io/en/latest/index.html)从T1WI、T2WI、增强T1WI、ADC 这四个序列的肿瘤加水肿区和肿瘤核心区中分别提取影像组学特征,将各个序列配准至相同的物理空间,从而使得同一个患者的ROI在各个序列上对应相同的病灶区域。每个患者在四种序列中的两种ROI 区域,即共计8 种区域中进行影像组学特征提取。在每个区域中,我们提取了86 个特征:包含18 个一阶特征,68 个纹理特征(包括14个灰度依赖矩阵特征、16个灰度尺寸区域矩阵特征、16个灰度游程长度矩阵特征、22个灰度共生矩阵特征),基于以上特征可以量化肿瘤不同维度的特性。对于每个患者能够提取8 个区域的86 个特征,总计688个特征。
1.4 特征选择和预测模型的建立
1.4.1 数据归一化
由于不同特征计算出的数值差别较大,需将不同特征直接的数值缩放到同一数量级,利用Normalize to unit 对特征矩阵进行归一化处理,计算公式如公式1,其中表示第n个特征,m表示样本数[9]。

1.4.2 数据降维
采用主成分分析(principal component analysis,PCA)方式进行数据降维,将潜在相关的高维特征转变为线性独立的低维特征。
1.4.3 特征选择
利用多变量方差分析(analysis of variance,ANOVA)对多变量进行统计分析,计算每个降维特征对label 的F值权重,从大到小排序后,筛选最相关的特征[9]。
1.4.4 模型建立及评估
分别采用逻辑回归(Logistic regression,LR)算法、Lasso 的逻辑回归算法(Logistic regression via Lasso,LR-Lasso)、支持向量机(support vector machine,SVM)、贝叶斯分类器(native Bayes,NB)构建诊断模型。应用5 折交叉验证方式进行验证。绘制ROC 曲线用于评估不同模型的预测性能,并且计算模型的曲线下的面积(area under curve,AUC)、准确率、特异度、敏感度,根据模型在测试集上的AUC来筛选最优模型,实验建模流程图如图1所示。
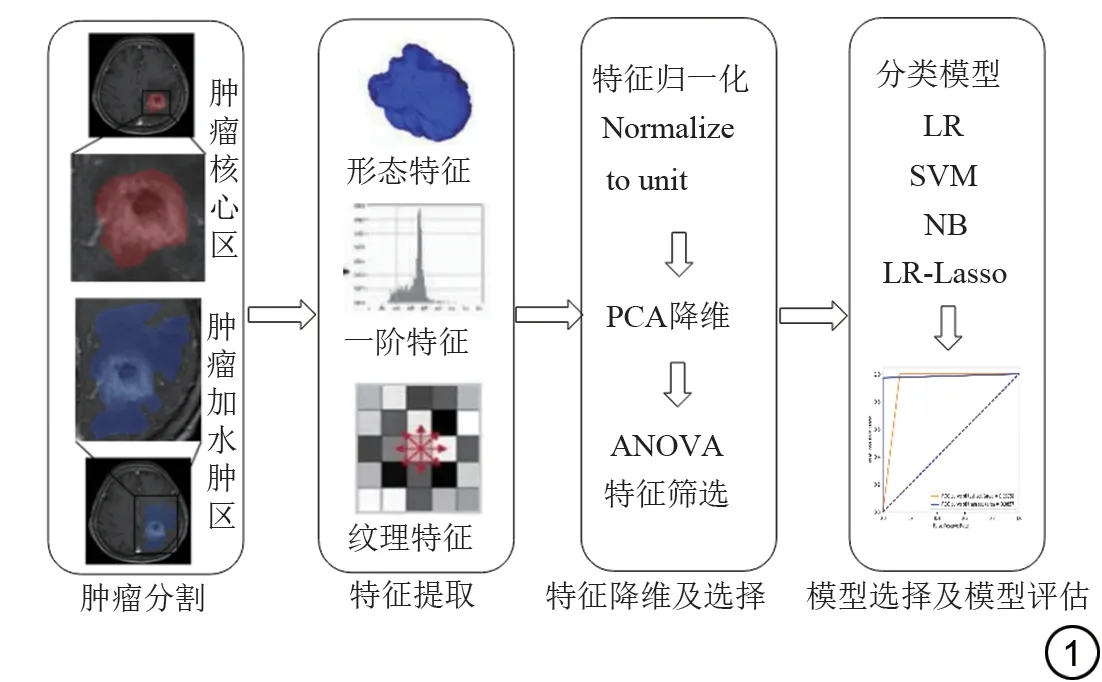
图1 实验流程图。Fig.1 Experimental flow chart.
1.5 统计分析
应用SPSS 软件(22.0 版)对临床基本资料进行统计分析,组间性别、肿瘤分级差异采用Pearson Chi-squared 检验进行评估,年龄分布的差异采用独立样本t检验进行评估。P<0.05 认为差异具有统计学意义。本实验使用的特征选择和分类方法、模型建立采用Python 中的scikit-learn包(scikit-learn 版 本0.22,Python 版 本3.7)以及FeAture Explorer 软件(https://github.com/salan668/FAE)。
2 结果
2.1 临床资料结果
共有MGMT 启动子甲基化结果大脑胶质瘤患者117 例,排除图像缺损、质量不佳的病例3 例,最终共纳入114例,分为训练组(91例)和验证组(23例),包括58 例胶质瘤MGMT 启动子甲基化阳性(平均年龄52岁;男/女=27/31)和阴性56例(平均年龄56岁;男/女=36/20)。本试验的临床特征如表2 所示,两组在性别和年龄上差异无统计学意义。
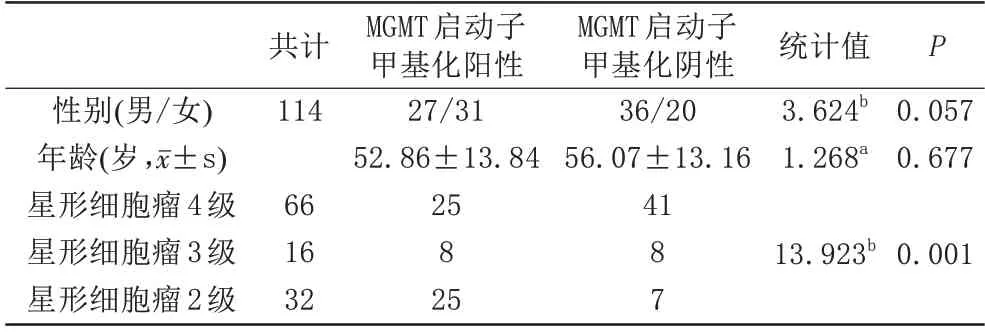
表2 两组间一般资料的比较Tab.2 Comparison of general data between the two groups
2.2 影像组学结果
本研究共计提取688个特征,经标准化处理并经PDA 降维,得到4 个模型预测效能如下:LR 模型的AUC值、准确率为0.90 和91% (表3,图2);SVM 模型的AUC值、准确率为0.89 和87% (表4,图3);LR-Lasso 模型的AUC 值、准确率为0.80 和74% (表5,图4),NB 模型的AUC 值、准确率为0.69 和74% (表6,图5)。基于LR模型预测效能最高。

图2 LR 模型在交叉验证集(CV Validation)、训练集(Train)、测试集(Test)中的ROC 曲线。 图3 SVM 模型在交叉验证集(CV Validation)、训练集(Train)、测试集(Test)的ROC 曲线。 图4 LR-Lasso 模型在交叉验证集(CV Validation)、训练集(Train)、测试集(Test)中的ROC 曲线。 图5 NB 模型在交叉验证集(CV Validation)、训练集(Train)、测试集(Test)中的ROC曲线。Fig. 2 ROC curves of LR model in cross validation set, training set and validation set. Fig. 3 ROC curves of SVM model in cross validation set, training set and validation set. Fig. 4 ROC curves of LR-Lasso model in cross validation set, training set and validation set. Fig. 5 ROC curves of NB model in cross validation set,training set and validation set.

表3 LR模型预测胶质瘤MGMT启动子甲基化状态的性能Tab.3 The performance of LR model in predicting the status of MGMT promoter methylation in glioma

表4 SVM模型预测胶质瘤MGMT启动子甲基化状态的性能Tab.4 The performance of SVM model in predicting the status of MGMT promoter methylation in glioma

表5 LR-Lasso模型预测胶质瘤MGMT启动子甲基化状态的性能Tab.5 The performance of LR-Lasso model in predicting the status of MGMT promoter methylation in glioma

表6 NB模型预测胶质瘤MGMT启动子甲基化状态的性能Tab.6 The performance of NB model in predicting the status of MGMT promoter methylation in glioma
3 讨论
本研究基于T1WI、T2WI、ADC 和增强T1WI 的影像组学特征,采用LR、LR-Lasso、SVM、NB 影像组学算法构建模型对胶质瘤MGMT启动子甲基化的预测效能进行研究,结果发现影像组学模型对术前预测脑胶质瘤MGMT 启动子甲基化的状态具有一定应用价值,LR模型预测效能最佳(AUC 值、准确率分别为0.90 和91%),SVM 模型的诊断效能较好(AUC 值、准确率分别为0.89和87%),LR-Lasso模型预测效能尚可(AUC值、准确率分别为0.80 和74%),NB 模型预测效能欠佳(AUC 值、准确率分别为0.69 和74%)。本研究创新性地对不同影像组学模型的效能进行了评估与对比,筛选最适合预测胶质瘤MGMT启动子甲基化状态的影像组学模型,为无创评估胶质瘤MGMT 启动子甲基化状态提供新途径,对实现脑胶质瘤精准个体化治疗具有一定的临床指导意义。
MGMT 启动子甲基化是近年研究较多的脑胶质瘤分子标志物,是评估脑胶质瘤患者对烷化剂药物替莫唑胺敏感性的重要分子依据,并且是评价脑胶质瘤患者预后情况的重要分子指标,在指导临床制订治疗方案以提高疗效及改善预后方面具有重要价值[2-3,10]。目前组织及分子病理学分析是检测MGMT启动子甲基化金标准,但获取标本不仅是有创操作、过程复杂、可重复性差,而且由于标本取材的限制,有时不能反映肿瘤组织的整体情况,使得无创的影像组学方法成为脑肿瘤分子标志物的研究热点[4,11-12]。
3.1 不同影像组学模型对胶质瘤MGMT启动子甲基化的预测效能的比较
本研究基于T1WI、T2WI、ADC 和增强T1WI 的MRI影像组学特征,采用LR、LR-Lasso、SVM、NB 影像组学算法构建模型对胶质瘤MGMT启动子甲基化的预测效能进行研究,发现LR 模型预测效能最高,SVM 模型的诊断效能较高,LR-Lasso模型预测效能尚可,NB模型预测效能欠佳。LR 模型在时间和内存需求上非常高效,它可以用较少的资源处理大型数据,其对于数据中小噪声的鲁棒性较好,在部分临床预测模型中,其他种类机器学习模型的预测性能并没有优于LR[13]。SVM 在相对小样本,高维数特征、非线性问题具有一定优势[14]。LR和SVM模型均属于分类判别模型,常用于二分类。两种模型均属于线性分类算法,因此两种算法结果比较相近。本实验中基于LR模型预测效能最高,LR 模型与SVM 模型诊断效能差别不大,均具有较好的预测效能,这与上述两种模型的特点及之前的研究相符。Sasaki 等[15]利用Lasso 算法预测胶质母细胞瘤MGMT甲基化状态的准确率为67%,而本研究LR-Lasso 预测胶质瘤MGMT 启动子甲基化准确率为74%,与之前的研究结果相符。NB是一种基于条件概率和计数的非常简单的算法,由于算法非常简单,NB 算法建模性能常常被其他经过适当训练的分类算法超越,这可能是本研究中NB 模型预测效能不佳的原因。
3.2 影像组学方法在预测胶质瘤MGMT 启动子甲基化状态的临床价值
本研究基于T1WI、T2WI、ADC 和增强T1WI 的MRI影像组学特征,采用不同影像组学模型构建预测模型,验证了影像组学预测MGMT 启动子甲基化状态方面具有较高应用价值,这与之前的研究相符。Xi等[16]对98 例胶质母细胞瘤用支持向量机建模,发现联合T1WI、增强T1WI 和T2WI 模型的预测准确率最高(0.86)。Wei 等[17]在T1WI、ADC 和FLAIR 图像上从肿瘤和瘤周水肿提取影像组学特征,应用LR 建模预测MGMT启动子甲基化,训练集AUC为0.92,验证集AUC为0.90,预测效能优于临床因素和ADC参数。Jiang等[18]发现MRI 影像组学有助于预测低级别胶质瘤患者MGMT 启动子甲基化状态,而不同序列的影像组学特征的融合可能会提高预测效能。有学者基于EfficientNet-B3卷积神经网络构建独立的基于T2WI、T1WI 增 强、T2WI 联 合T1WI 增 强 的 预 测 模 型(T2-net、T1C-net、TS-net),TS-net 模型的准确度为81.8%,AUC为0.78[7]。上述的研究主要局限在单一的影像组学或深度学习方法研究,缺乏对不同影像组学模型进行对比研究。然而每种分类算法具有各自的优点及局限性,因此有必要尝试多种分类算法,并且评估不同算法的性能[19-20]。
3.3 本研究的优越性及局限性
本研究的创新点在于采用多种(包括LR、LR-Lasso、SVM、NB)而非一种分类算法,比较了不同算法构建模型预测的效能,这与之前的一些仅用单一分类算法的研究不同。本研究使用多种算法建模是因为单个分类算法的成功可能会受到机会因素影响,并且可能存在数据稳定的问题。本研究发现诊断效能较高的分类算法并不局限于单一的算法,LR及SVM 算法构建模型均取得了较高的预测准确度。本研究的ROI 及序列选择与之前的一些研究有所不同,之前的研究大多提取单序列、1 种ROI 特征,而本研究的肿瘤分割区域包括2种ROI (既包括肿瘤加水肿区域的ROI,又包括肿瘤核心区域的ROI),并匹配到包括T1WI、T2WI、增强T1WI、ADC 的4 个序列中,从而提取的肿瘤影像组学特征更全面。此外,本研究的特征降维方法与之前的一些研究有所不同,之前一些研究使用Pearson相关进行单因素分析降维,而本研究中影像组学特征降维采用PCA方法,通过线性变换将潜在相关的高维特征转变为线性独立的低维特征,优点是去除特征之间的互相影响的因子,并且不受数据集以外的因素干扰;计算方法简单,主要运算是特征值分解,易于实现。
本研究的局限性在于采用回顾性研究,样本量相对较少,样本可能存在偏倚;另外,研究中的ROI分割是人工勾画,操作不方便,效率较低,并且具有一定主观性;此外,采用同一家医院的数据,扫描参数相同,模型具有局限性;采用PCA 降维得到的综合特征是经过计算得到的各个特征的含义具有一定的模糊性,算法降维得到的不是直接从原始图像中提取的原始特征,而是综合计算的特征,而主成分与原始特征间的关系以直线方程表示,不能直接用于阐释和说明[20-22]。本研究应用的影像组学算法种类较少,也具有一定局限性。下一步还需与其他研究中心合作开展多中心研究,进一步增加病例数、更换降维方法及影像组学算法,增加模型的稳定性。
综上所述,MRI 影像组学模型对术前预测脑胶质瘤MGMT 启动子甲基化的状态具有一定应用价值,为评估胶质瘤预后、鉴别复发与假进展提供更多信息,为实现脑胶质瘤精准治疗提供新思路。
作者利益冲突声明:全部作者均声明无利益冲突。
