基于纤毛鹅观草特异的卫星重复序列开发寡核苷酸探针
2022-05-20程梦豪MiroslavaKarafitov孙昊杰KateinaHoluovJaroslavDoleel宋新颖王海燕王秀娥
程梦豪,Miroslava Karafitov,孙昊杰,Kateina Holušov, Jaroslav Doležel,宋新颖,王海燕,王秀娥*
(1.南京农业大学作物遗传与种质创新国家重点实验室/细胞遗传研究所/ 现代作物生产省部共建协同创新中心,江苏 南京 210095; 2.捷克科学院实验植物学研究所/哈纳地区生物技术和农业研究中心,捷克共和国 奥洛穆茨 77900)
纤毛鹅观草(Roegneriaciliaris,2n=4x=28,StStYY或ScScYcYc)是鹅观草属多年生异源四倍体物种,在我国广泛分布。纤毛鹅观草既是优良的牧草植物,也是栽培小麦的三级基因资源库。研究表明,纤毛鹅观草具有抗赤霉病等小麦病害及抗非生物逆境等优良性状[1]。为利用其优异基因,前人将纤毛鹅观草与普通小麦杂交,先后获得远缘杂种和双二倍体,并创制鉴定出普通小麦-纤毛鹅观草二体异附加系等异染色体系种质资源[1-3]。
基因组原位杂交(genomicinsituhybridization,GISH)和荧光原位杂交(fluorescenceinsituhybridization,FISH)是鉴定特定染色体组来源或追踪外源染色体片段的有效手段。纤毛鹅观草含有St(Sc)和Y(Yc)2个基因组。大多数学者认为,纤毛鹅观草St基因组的供体来源于拟鹅观草属二倍体物种,但具体来源于哪个种有争议[4]。Y基因组的来源未知,目前主要有2种推测:一是根据ITS序列分析和细胞学鉴定,认为Y起源于St基因组;二是根据核基因序列分析,认为Y与St基因组来源不同,与Xp基因组有一定的同源性,可能起源于独立的二倍体[4]。还有研究利用GISH和FISH识别鹅观草属染色体,并推测其基因组归属,如:利用基于DNA重复序列和质粒探针pCbTaq4.14、pPITaq2.5和pEt2识别纤毛鹅观草染色体,发现pEt2能在1StL、3StL和5StL上产生FISH信号[3];利用pCbTaq4.14对8个普通小麦-纤毛鹅观草异附加系进行FISH,发现在1St、2St、3St、7St染色体上可检测到FISH信号[2];利用探针pAs1和pHvG39进行顺序双色FISH/GISH,成功区分出St、P和Y染色体组的所有染色体[5];开发1个新的FISH探针St2-80,可以在St染色体组产生丰富信号,但Y和E基因组上也能检测到杂交信号[6];利用4个重复序列探针pTa794、pTa71、RcAfa和(GAA)10,构建了纤毛鹅观草的FISH核型,可以成功识别并区分纤毛鹅观草的 14条染色体[1]。
上述研究中,识别纤毛鹅观草品系各条染色体的探针,均来源于小麦族物种,由于缺乏纤毛鹅观草基因组序列,迄今尚未开发出纤毛鹅观草特异、特别是可以区分这2个基因组特异的探针,对各染色体基因组归属还是推测,缺乏直接证据,2个基因组之间的关系、特别是Y染色体组的来源尚不明了。此外,鹅观草属有多个物种,自然界也存在纤毛鹅观草来源不同的品系,这些物种、品系之间的系统进化关系也尚不明了,自然品系中是否存在丰富的染色体水平变异也不清楚,这些都限制了鹅观草属、特别是纤毛鹅观草的有效利用。
近年来发展起来的基于寡核苷酸探针的FISH(oligo-FISH)技术[7]和基于单拷贝探针库的染色体涂染(oligo-painting)技术[8]已经成为染色体研究的重要手段。染色体涂染技术主要针对已测序物种的基因组序列,开发单拷贝的oligo探针库,可精准识别染色体或染色体片段,发现结构重排。由于小麦族物种的基因组庞大,重复序列占比高,单拷贝oligo探针库的覆盖密度较低且合成成本较高,目前在小麦族物种中应用较少[8-9]。oligo-FISH技术首先基于重复序列开发oligo探针,单个探针就能产生丰富的FISH信号,1个或几个探针组合可产生更丰富的信号,可用于准确识别染色体或染色体片段。oligo-FISH与染色体涂染相比,虽然在识别染色体或染色体片段和检测结构重排的准确度上有所欠缺,但是更加经济、简便、快捷,是目前小麦族物种染色体研究最常用的方法。目前在纤毛鹅观草染色体鉴定研究中,利用的重复序列DNA探针来源于其他物种,对纤毛鹅观草染色体无特异性,且常需要探针标记制备过程,程序繁琐,染色体鉴定效率较低。迫切需要基于纤毛鹅观草基因组信息,开发纤毛鹅观草特异的oligo探针,进一步完善鉴定体系,提高纤毛鹅观草染色质的鉴定精度、特异性和效率。
小麦族物种基因组中富含散布的移动元件家族(逆转录因子和DNA转座子)和串联分布的卫星DNA(SatDNA)等重复序列类型[10]。根据重复单元的长短,SatDNA又分为微卫星(2~7 bp)、小卫星(几十bp)和卫星(几百bp)重复序列[11]。SatDNA主要位于染色体着丝粒和端部区域,是真核生物基因组中最活跃的成分之一,其高进化速率导致相当大的序列多样性,因此大多数SatDNA家族具有物种特异性或属特异性[12]。在植物染色体研究中,基于SatDNA基序开发的oligo探针是最常用的FISH探针资源之一[13]。
TRF(tandem repeats finder)和TAREAN(tandem repeats analyzer)等是重复序列鉴定的主要生物信息学工具[11,14]。TRF软件需要长度超过一个重复单元的序列作为输入序列,尽管这种长度的序列通常可以从组装基因组数据中获得,但组装数据中常缺乏卫星重复序列[15],因此TRF预测的重复序列中可能漏掉一些长的卫星序列;TAREAN软件利用基于图形的聚类算法从未组装的测序Reads中识别和表征卫星DNA序列,仅需要基因组覆盖度0.01~0.50倍的Reads数据作为输入序列,有利于鉴定出重复单元长的卫星重复序列[11]。
本研究基于前期选育的普通小麦-纤毛鹅观草异附加系DA6Sc和DA2ScL,利用流式细胞仪分拣纤毛鹅观草6Sc染色体和2ScL染色体臂,通过二代测序获得染色体基因组序列,利用TAREAN鉴定卫星重复序列,基于其基序开发oligo探针,利用oligo-FISH筛选纤毛鹅观草特异oligo探针。通过研究,进一步完善纤毛鹅观草染色体鉴定体系,服务于纤毛鹅观草基因组和优异基因的转移和利用研究。
1 材料与方法
1.1 植物材料
纤毛鹅观草(Roegneriaciliaris,2n=4x=28,基因组SSYY)品系2003I-37(引种号W614249)引自美国西部地区植物引种站(Western Regional Plant Introduction Station,USA)。小麦野生近缘物种拟斯卑尔脱山羊草(Aegilopsspeltoides,2n=2x=14,基因组SS,Accession No.:PI542238)、黑麦(Secalecereal,2n=2x=14,基因组RR,Accession No.:PI542176)、大麦(Hordeumvulgare,2n=2x=14,基因组HH,Accession No.:PI542176)引自National Plant Germplasm System,USA。普通小麦(Triticumaestivum,2n=6x=42,基因组AABBDD)品种‘中国春’(Chinese Spring,CS)、CS-百萨偃麦草(Thinopyrumbessarabicum,2n=2x=14,基因组JJ)双二倍体、一整套普通小麦-纤毛鹅观草二体异附加系(DA1Sc-DA7Sc和DA1Yc-DA7Yc)和端二体异附加系DA2ScL均由南京农业大学细胞遗传研究所创制或保存。
1.2 染色体分拣与测序
为了减少由于DNA扩增引起的偏差,制备了2种不同类型的DNA测序文库。第1种类型的DNA测序文库,染色体DNA根据imkov等[18]的方法纯化,用Nextera DNA Library Prep试剂盒,通过5个扩增循环将50 ng纯化的DNA制备成DNA测序文库。第2种类型的DNA测序文库,用TruSeq DNA PCR-Free试剂盒扩增染色体DNA制备而成。所有文库均在Illumina HiSeq2500系统上使用HiSeq Rapid SBS Kit v2进行测序,每条染色体的测序深度大于50倍,产生2×250 bp的双端Reads测序数据,利用Trimmomatic[20]剔除原始数据中低质量的碱基对,最后利用Meraculous v2.0.5[21](k-mers=111 bp)组装成scaffolds(剔除短于1 kb的scaffolds)。
1.3 重复序列鉴定与oligo探针开发
从2ScL和6Sc的测序数据中,分别随机选择250 000对双端Reads,经质检和筛选,利用TAREAN[11]进行聚类分析,从中鉴定卫星重复序列。基于鉴定出的重复序列基序,首先利用Oligo7软件直接搜索长度为50~59 bp的序列作为oligo探针设计的候选序列,对未能搜索到候选序列的,则将此卫星重复序列与2ScL或6Sc的组装数据进行本地BLAST比对,在比对率最高的一段序列中,随机截取50~59 bp作为oligo探针设计的候选序列(随机截取的序列要保证合适的Tm值)。每个卫星重复序列开发1个oligo探针,oligo探针由南京擎科生物有限公司合成,探针的5′端由羟基荧光素(carboxy fluorescein,FAM)或羧基四甲基罗丹明(carboxytetramethylrhodamine,TAMRA)修饰,用于oligo-FISH。经oligo-FISH验证,选择能在纤毛鹅观草染色体上产生稳定FISH信号的探针进行后续研究。
卫星重复序列和探针命名:重复序列命名为CL+染色体(臂)+序列聚类簇的编号,例如来自2ScL基因组序列的cluster 24的重复序列命名为CL 2ScL-24;基于该重复序列开发的oligo探针相应命名为oligo-2ScL-24。
1.4 有丝分裂中期染色体制片
根尖细胞中期染色体制片参照Lei等[7]的方法。将固定好的根用ddH2O洗净,切下1~2 mm的根尖分生组织,放入盛有20 μL酶液[4%纤维素酶Onozuka R-10(Yakult,Tokyo,Japan)、1%果胶酶Y23(Karlan,Osaka,Japan)和KCl缓冲液(75 mmol·L-1KCl,7.5 mmol·L-1EDTA,pH4.0)]的离心管中,37 ℃酶解50~60 min后,加入1×TE溶液反应5 min,随后吸干TE溶液,用70%乙醇漂洗2次,风干后加入35 μL悬浮液(醋酸与甲醇体积比为5∶1),用解剖针将根尖搅碎并混匀,悬浮根尖细胞,然后将干净的载玻片放在湿盒里,吸取3~5 μL悬浮液滴在载玻片上,风干,置于紫外交联仪交联3 min,用于后续的原位杂交试验。
1.5 基因组原位杂交(GISH)和荧光原位杂交(FISH)
GISH参照Zhang等[22]的程序,全基因组DNA探针标记采用缺刻平移法。FISH在Lei等[7]的基础上稍作修改。制片首先在含0.15 mol·L-1NaOH的70%乙醇溶液中变性5 min,随后依次在70%、70%和100%的乙醇中脱水各5 min,气干备用。将15 μL杂交液滴加在制片上,杂交液组成:7.5 μL甲酰胺(Formamide,FA),1.5 μL缓冲液(20×SSC),0.5 μL鲑鱼精DNA(Salmon sperm DNA,ssDNA),10 pmol oligo探针,然后用ddH2O补足至15 μL。盖上盖玻片,放进湿盒,置37 ℃避光杂交不少于6 h;杂交后的制片在2 × SSC和ddH2O中各清洗5 min,气干,滴加6.5 μL含DAPI的H1200(VECTA)防荧光淬灭剂,盖上盖玻片。置Olympus BX51型荧光显微镜下观察杂交信号,使用OLYMPUS DP72型CCD(Cooled Color Digital,彩色数字相机)相机拍摄图像,使用Adobe Photoshop软件处理图像。
2 结果与分析
2.1 纤毛鹅观草2ScL和6Sc染色体分拣测序及其特异重复序列筛选
将分拣的2ScL和6Sc悬浮液用(GAA)7-FITC和端粒DNA作为探针进行FISHIS,分拣的目标染色体纯度分别为86%和88%,符合进一步的测序要求。对分拣的染色体DNA进行扩增,分别获得2ScL的DNA 6.88 μg、6Sc的DNA 8.08 μg,用于Illumina测序分别获得目标染色体基因组序列,经过序列拼接,2ScL和6Sc染色体的基因组组装长度分别为338.8 Mb和302.6 Mb。
利用Repeatexplorer2[11]分析重复序列构成,发现2ScL和6Sc基因组中的重复序列分别占63.57%和63.91%,低于其他已测序的麦类作物基因组中的重复序列含量,推测由于Repeatexplorer2这类基于Reads预测重复序列的软件在预测转座元件时敏感度较低,导致这类重复序列预测的含量可能低于实际含量[23]。2ScL的重复序列类型主要包括LTR反转座子(占45.56%)、DNA转座子(2.16%)和卫星重复序列(1.15%),LTR反转座子中Ty3_gypsy和Ty1_copia超家族分别占31.84%和9.97%;6Sc的重复序列类型和占比与2ScL基因组相似,LTR反转座子、DNA转座子和卫星重复序列分别占44.91%、2.24%和0.23%,LTR反转座子中Ty3_gypsy和Ty1_copia超家族分别占30.25%和11.14%(表1)。
2条染色体基因组序列中共鉴定出11种卫星重复序列,其中8种来自2ScL,3种来自6Sc。基序最长的是2ScL的CL24(2 812 bp),命名为CL 2ScL-24,在2ScL基因组中占比最高(0.670%);基序最短的2条序列是CL 2ScL-246和CL 2ScL-248,长度分别为177和188 bp,在2ScL基因组中的比例较低,分别占0.012%和0.011%;其余8种序列的基序长度为380~662 bp,在基因组中的占比为0.014%~0.190%。分别将11种重复序列基序在NCBI数据库BLASTN[24](Percent Identity≥80%,Query Coverage≥80%)中进行序列比对,发现5条序列可以比对到已知同源序列,其中2条(CL 2ScL-100和CL 2ScL-161)的同源序列不是典型的串联重复序列,二者分别与小麦醇溶蛋白相关序列和转录因子STP6序列同源。3条与已知重复序列相似,CL 2ScL-119为Tail家族串联重复序列,与大赖草重复序列pLrTail-4相似度最高,为90.54%;CL 2ScL-240为pAW161家族重复序列,与冰草重复序列ACRI_CL20[25]相似度最高,为98.68%;CL 2ScL-163与野生黑麦的卫星重复序列Lt1-6相似度为91.60%。其余的6条序列(CL 2ScL-24,CL 2ScL-246,CL 2ScL-248,CL 6Sc-111,CL 6Sc-200,CL 6Sc-232)未比对到已知同源序列,推测可能是纤毛鹅观草基因组特异重复序列。
2.2 oligo探针开发及其基因组特异性分析
基于上述11种重复序列,利用Oligo7软件开发了11个oligo探针,其基序为50~59 bp。oligo-FISH发现,6个探针(oligo-2ScL-119、oligo-2ScL-161、oligo-2ScL-163、oligo-2ScL-248、oligo-6Sc-111和oligo-6Sc-200)仅在纤毛鹅观草染色体上可观察到明显FISH信号,而在‘中国春’染色体上未观察到明显信号,表明它们可以用作纤毛鹅观草基因组特异探针,特异识别小麦背景中的纤毛鹅观草染色质(图1,表2)。其中3个探针oligo-2ScL-248、oligo-6Sc-111和oligo-6Sc-200来源于先前推测为纤毛鹅观草特异的3个重复序列 CL 2ScL-248、CL 6Sc-111和CL 6Sc-200,其余3个探针oligo-2ScL-119、oligo-2ScL-161和oligo-2ScL-163分别来源于先前推测为非特异于纤毛鹅观草的3个重复序列CL 2ScL-119、CL 2ScL-161和CL 2ScL-163(图1,表2)。

图1 纤毛鹅观草和普通小麦‘中国春’根尖有丝分裂中期染色体的oligo-FISHFig.1 Oligo-FISH on root tip metaphase chromosomes of Roegneria ciliaris and Triticum aestivum ‘Chinese Spring’ A1—A2. 探针oligo-2ScL-119;B1—B2. 探针oligo-2ScL-161;C1—C2. 探针oligo-2ScL-163;D1—D2. 探针oligo-2ScL-248;E1-E2. 探针 oligo-6Sc-111;F1—F2. 探针oligo-6Sc-200。染色体用DAPI套染(蓝色);6个寡聚核苷酸探针用5′ TAMRA修饰(红色)。A1-A2. Probe oligo-2ScL-119;B1-B2. Probe oligo-2ScL-161;C1-C2. Probe oligo-2ScL-163;D1-D2. Probe oligo-2ScL-248;E1-E2. Probe oligo-6Sc-111;F1-F2. Probe oligo-6Sc-200. Chromosomes were counterstained with DAPI(blue);six oligo probes were modified with 5′ TAMRA(red).

表2 开发的6个oligo探针的序列信息Table 2 Sequence information of six oligonucleotide probes developed in this study
为验证这6个oligo探针对于纤毛鹅观草的特异性,利用B2DSC[26](pident=85,qcovhsp=80)搜索上述探针序列在‘中国春’参考基因组中的拷贝数和物理位置,oligo-2ScL-163未能搜索到同源序列,其余5个探针序列可以比对到‘中国春’参考基因组中的同源序列,但数量和位置分布不同。oligo-2ScL-161、oligo-6Sc-111和oligo-6Sc-200在‘中国春’基因组的不同区域预测的拷贝数很少,且同一位置(1 Mb区间内)低于6个拷贝。其中oligo-2ScL-161来源于先前推测为非特异的重复序列CL 2ScL-161,其在小麦中比对到的同源序列为转录因子STP6,不属于重复序列(表2);oligo-2ScL-119来源于Tail家族重复序列CL 2ScL-119,其同源序列存在于除3A、4A、2D和4D之外的其他小麦染色体上,同一位置(1 Mb区间)最高拷贝数为128(5D染色体的189~190 Mb区间),尽管拷贝数较高,但2个物种中该重复序列同源性较低(低于85%),这也可能是oligo-2ScL-119未在小麦染色体上产生信号的主要原因;oligo-2ScL-248来源于先前推测为纤毛鹅观草特异的重复序列CL 2ScL-248,但其同源序列在3D和7D上分别有6个和116个拷贝,我们未能在这2条染色体上观察到FISH信号,推测其原因是拷贝数较低或者与二者序列差异较大。
进一步利用oligo-FISH分析6个探针在其他5个小麦族物种的基因组特异性,结果发现,6个探针均不能在大麦染色体上观察到明显的FISH信号(图2-A1—F1);oligo-2ScL-119和oligo-6Sc-200可以在黑麦一对染色体端部产生FISH信号(图2-A2、F2),探针oligo-pSc119.2-1进行FISH分析证实该染色体为4R[27],表明oligo-2ScL-119和oligo-6Sc-200可用于特异识别4R短臂;oligo-2ScL-119和oligo-2ScL-163在拟斯卑尔脱山羊草Ss染色体上仅观察到较弱的FISH信号,而oligo-2ScL-248在Ss染色体组上可产生丰富的FISH信号,表明oligo-2ScL-248可以用于识别拟斯卑尔脱山羊草Ss染色体组(图2-A3、C3、D3)。GISH/oligo-FISH发现,oligo-2ScL-119和oligo-2ScL-248分别在百萨偃麦草Eb基因组2对和4对染色体端部产生FISH信号(图2-A4,E4);GISH/oligo-FISH发现,6个探针均可在鹅观草的St或Y基因组的部分染色体上检测到信号,oligo-2ScL-163在12对染色体上有FISH信号,oligo-6Sc-111在9对染色体上有FISH信号,oligo-2ScL-119在7对染色体上有FISH信号,oligo-2ScL-161和oligo-6S-200在5对染色体上有FISH信号,oligo-2ScL-248在2对染色体上有FISH信号(图2-A5—F5)。
2.3 6个oligo探针在纤毛鹅观草不同染色体上的信号分布
将纤毛鹅观草基因组DNA探针分别与6个oligo探针组合,在一整套普通小麦-纤毛鹅观草二体异附加系上进行GISH/oligo-FISH分析。CL 2ScL-163和CL 6Sc-111的基序具有89.47%的相似性,GISH/oligo-FISH分析表明,oligo-2ScL-163和oligo-6Sc-111信号分布也相似,仅在个别染色体上的信号强度略有差异,FISH信号均位于Sc组的所有染色体端部、近端部和近着丝粒区域(图3-C、E),而Yc组染色体上未见信号,可以作为Sc基因组特异探针;二者在2Sc和7Sc短臂近端部和端部的信号强度存在差异,oligo-2ScL-163信号强于oligo-6Sc-111(图3-C、E)。
oligo-2ScL-119的信号主要分布在11对染色体的端部和近端部,包括6对Sc组(1Sc、2Sc、4Sc、 5Sc、6Sc、7Sc)和5对Yc组(1Yc、2Yc、5Yc、6Yc、7Yc)染色体;oligo-2ScL-161的信号主要分布在12对染色体的端部和近端部,包括6对Sc组(1Sc、2Sc、4Sc、5Sc、6Sc、7Sc)和6对Yc组(1Yc、2Yc、4Yc、5Yc、6Yc、7Yc)染色体;oligo-2ScL-248的信号位于染色体1Sc和2Sc的长臂顶端;oligo-6Sc-200的信号主要分布在12对染色体的端部和近端部,包括所有Sc组和5对Yc组(1Yc、2Yc、5Yc、6Yc、7Yc)染色体(图3)。
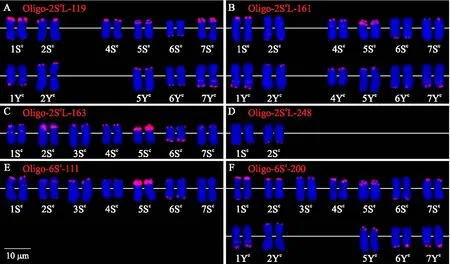
图3 一整套小麦-纤毛鹅观草二体异附加系根尖中期染色体的oligo-FISHFig.3 Oligo-FISH on root tip metaphase chromosomes of a complete set of T.aestivum-R.ciliaris DALs A. 探针oligo-2ScL-119;B. 探针oligo-2ScL-161;C. 探针oligo-2ScL-163;D. 探针oligo-2ScL-248:E. 探针oligo-6Sc-111;F. 探针oligo-6Sc-200。染色体用DAPI套染(蓝色),6个寡聚核苷酸探针用5′ TAMRA修饰(红色)。A. Probe oligo-2ScL-119;B. Probe oligo-2ScL-161;C. Probe oligo-2ScL-163;D. Probe oligo-2ScL-248;E. Probe oligo-6Sc-111;F. Probe oligo-6Sc-200. Chromosomes were counterstained with DAPI(blue);six oligonucleotide probes were modified with 5′ TAMRA(red).
根据上述oligo探针的信号分布特征,将oligo-2ScL-161和oligo-2ScL-163组合,对一整套普通小麦-纤毛鹅观草二体异附加系进行双色oligo-FISH,构建了异附加系中纤毛鹅观草染色体的oligo-FISH核型(图4),该探针组合可以准确识别Sc组的各条染色体,但由于信号少且位于端部,对Yc组染色体的识别要结合FISH信号分布、染色体长度和臂比等核型特征。此外,异附加系中DA1Yc和DA7Yc中外源染色体信号分布与染色体形态高度相似,这与Kong等[1]的结果一致。

图4 一整套异附加系中纤毛鹅观草染色体的双色oligo-FISH核型Fig.4 Dual-color oligo-FISH-based karyotype of R.ciliaris chromosomes in a complete set of T.aestivum-R.ciliaris DALs 探针oligo-2ScL-161用5′ TAMRA 修饰(红色),oligo-2ScL-163用5′ FAM修饰(绿色)。染色体用DAPI套染(蓝色)。Probe oligo-2ScL-161 was modified with 5′ TAMRA(red),oligo-2ScL-163 was modified with 5′ FAM(green). Chromosomes were counterstained with DAPI(blue).
3 讨论与结论
纤毛鹅观草是异源四倍体,基因组序列尚未发布,单条染色体的分拣能起到将大基因组化整为零的作用,克服基因组过大的分析障碍,提高特定染色体片段和特定基因分离和研究的效率,从而大大加速基因组分析的进程。本研究基于前期选育的普通小麦-纤毛鹅观草异附加系2ScL和6Sc,利用流式细胞仪分拣外源染色体2ScL和6Sc,经测序和组装获得2条染色体的基因组序列。该策略适用于没有参考基因组、基因组较为庞大的小麦近缘物种。纤毛鹅观草2ScL和6Sc基因组序列的获得,为进一步开发纤毛鹅观草染色体的特异分子标记、定位与克隆外源优异基因、构建比较基因组物理图谱、解析物种间共线性与进化特征等提供了宝贵数据。
以往用于鉴定纤毛鹅观草染色体的FISH探针均来自其他物种的串联重复序列,由多个探针组合而成,结合GISH和FISH核型,可以识别小麦背景中的纤毛鹅观草染色体,但由于这些FISH探针在小麦染色体上也产生较强的信号,给染色体或染色体片段的识别带来了困难[1,3,5-6]。开发基因组特异探针是快速准确识别染色体的重要手段,早期在缺乏物种全基因组序列的情况下,研究者主要利用全基因组DNA重复序列的复性动力学特征,或利用BAC文库杂交等,鉴定高度重复序列,筛选物种特异重复序列,然后开发质粒探针进行FISH,验证其物种或基因组特异性[28]。伴随着基因组测序技术的发展和越来越多物种全基因组序列发布,利用生物信息学分析筛选物种特异重复序列、开发物种或基因组特异FISH探针成为可能。本研究利用TAREAN从2ScL和6Sc染色体的测序数据中鉴定出卫星重复序列并开发成oligo探针,通过与小麦参考基因组的序列比对结合oligo-FISH,成功开发了纤毛鹅观草特异探针,并通过探针组合构建了纤毛鹅观草FISH核型,结合GISH和其他核型特征,不但可以识别小麦背景中的外源染色体,还可以区分纤毛鹅观草各条染色体。特异探针开发效率高,可预见性强,可以为其他无参考基因组物种探针开发提供借鉴。
本研究开发的11个oligo探针中,3个探针(oligo-2ScL-24、oligo-2ScL-246和oligo-6Sc-232)在纤毛鹅观草染色体上无法产生FISH信号,2个探针(oligo-2ScL-100和oligo-2ScL-240)在纤毛鹅观草染色体和普通小麦染色体上均能产生FISH信号。oligo-2ScL-24来源于CL 2ScL-24,它在2Sc基因组序列中的占比最高(0.670%),但其基序长达2 812 bp,这可能是oligo-2ScL-24无法在纤毛鹅观草染色体上产生FISH信号的原因;oligo-2ScL-246和oligo-6Sc-232来源于CL 2ScL-246和CL 6Sc-232,它们在各自基因组的占比较低,分别占0.014%和0.045%,这可能是oligo-2ScL-246和oligo-6Sc-232无法在纤毛鹅观草染色体上产生FISH信号的原因;oligo-2ScL-100和oligo-2ScL-240来自CL 2ScL-100和CL 2ScL-240,它们在小麦族各物种中广泛分布,所以探针oligo-2ScL-100和oligo-2ScL-240无法作为纤毛鹅观草特异探针。
Kong等[1]利用4个探针[pTa71、pTa794、RcAfa和(GAA)10]首次构建了纤毛鹅观草的核型,可识别纤毛鹅观草14条染色体。然而,除了探针(GAA)10以外,其余3个探针均不是oligo探针,需要较繁杂的探针制备过程,且这4个探针均不是纤毛鹅观草特异探针,在对复杂材料鉴定时不易排除背景中的小麦或其他近缘物种染色质的干扰。本研究基于特异oligo探针oligo-2ScL-161与oligo-2ScL-163构建的纤毛鹅观草染色体核型,不但可以识别纤毛鹅观草14条染色体,还可以清晰分辨出纤毛鹅观草Sc组的各条染色体。但6个oligo探针都来自Sc基因组染色体,只有oligo-2ScL-119、oligo-2ScL-161和oligo-6Sc-200能在Yc组的染色体上产生信号,且在Yc组上的信号分布十分相似,仅在4Yc上的信号有差别,因此仅凭其中1个探针很难识别所有Yc组染色体。
GISH是检测小麦中外源染色质渐渗片段的传统有效手段。本研究开发的6个oligo探针均能在纤毛鹅观草染色体上产生明显信号而在普通小麦染色体上无信号,说明这6个oligo探针可以应用于快速从小麦背景中检测纤毛鹅观草染色质的渐渗。美中不足的是这6个oligo探针的信号均主要分布于染色体端部和近端部,可能无法检测涉及纤毛鹅观草染色体着丝粒区域的渐渗片段。为更准确鉴定出小麦中纤毛鹅观草染色质的渐渗片段,建议将GISH与新开发的oligo探针结合使用,增加FISH信号在不同区域的丰富度。
纤毛鹅观草为异源四倍体,目前认为Pseudoroegneria是Sc染色体组的供体,而Yc染色体组来源未知。Wang等[6]开发的探针St2-80可以将St基因组与Y和E基因组区分开来,但St2-80也能在Y和E的部分染色体上检测到信号,并不是St基因组特异探针。本研究开发的6个oligo探针中,oligo-2ScL-163、oligo-6Sc-111和oligo-2ScL-248仅在Sc染色体组上产生信号,而oligo-2ScL-119、oligo-2ScL-161和oligo-6Sc-200在Sc和Yc染色体组上均产生较丰富的信号。这些结果进一步验证前人提出的Sc和Yc染色体组亲缘关系较近的推论,也为区分Sc和Yc染色体组提供了新的oligo探针资源。探针oligo-2ScL-163和oligo-6Sc-111在纤毛鹅观草1Sc—7Sc染色体上均能产生信号而在1Yc—7Yc染色体上无法产生信号,且在大部分不含St染色体组的物种上不能产生明显信号,表明这2个探针能快捷且清楚将St染色体组与小麦族大部分其他染色体组区分开来,是未来研究Sc和Yc染色体组起源的重要探针资源。
