海南鳽贵州雷公山小种群遗传多样性分析
2022-05-16李斌强詹昊峰何小涛吕小艳杨宗才王野影
李斌强 詹昊峰 何小涛 吕小艳 杨宗才 王野影*
(1.贵州师范大学生命科学学院,西南喀斯特山地生物多样性保护国家林业和草原局重点实验室,贵阳,550025;2.贵州工程职业学院药学院,铜仁,565200;3.贵州省雷公山国家级自然保护区管理局,雷山,557199)
海南鳽(Gorsachiusmagnificus)又称海南虎斑鳽,隶属于鸟纲(Avea)鹈形目(Pelecaniformes)鹭科(Ardeidae)夜鳽属[1],被世界自然保护联盟(IUCN)评定为濒危物种(EN)[2],是我国一级重点保护野生动物。根据2016年的调查数据,海南鳽的全球数量为250~999只,由于其数量稀少,曾被列为全世界30种最濒危鸟类之一[3]。在我国主要分布在广西和江西两省,零星分布在海南、广东、湖北、湖南、贵州、四川、云南、安徽、福建和内蒙古10个省份[1,4],但分布地覆盖到保护区的数量与面积极低[5],受人类活动、栖息地破坏和环境变化的影响,该物种的生存繁殖正受到严重威胁[6]。国外主要在越南[7]、柬埔寨[8]和印度[9]等国发现了海南鳽的活动踪迹。
1899年,海南鳽首次由Whitehead在海南地区的五指山收集到标本,并由Grant将其命名为海南鳽[10],后在国内逐步展开系统分类地位、繁殖生态、行为、分布和日活动节律的研究。Lei等[11]通过组织相容性复合体(MHC)基因进行了包括海南鳽在内的9种鹭科鸟类系统发育分析,发现海南鳽系统发育较为独立。进一步对海南鳽线粒体基因进行测序,同鹭科其他鸟类综合分析之后,海南鳽与白鹭(Egrettagarzetta)的亲缘关系较近,支持海南鳽从夜鳽属(Gorsachius)划分出来,独立成属(Oroanassa)[12]。
濒危物种种群数量较少,种群缺乏遗传多样性,在进化过程中种群基因流动减少和适应性下降,容易导致物种灭绝[13]。脊椎动物线粒体基因组(mitochondrial DNA,mtDNA)具有严格的母系遗传、进化速率快、突变率高、无重组和细胞内稳定存在等特性,能够作为物种遗传多样性研究的有效分子标记[14]。贵州省于2007年首次在雷公山保护区发现海南鳽[15];直到2018年再次发现[16];2019年,在保护区内的方祥、干脑和大塘镇3个地点均发现海南鳽活动[16-17]。本研究在筛选分子标记的基础上,利用从贵州省野生动物救助中心获取的5只海南鳽,开展雷公山小种群的遗传多样性分析,以期了解该种群的生存发展能力,为其保护政策的制定提供科学依据和基础研究资料。
1 材料与方法
1.1 基因序列及样本来源
从NCBI数据库中下载海南鳽、黑冠鳽(Gorsachiusmelanolophus)和栗头鳽(G.goisagi)[12]线粒体全基因组序列,试验中提取的海南鳽组织样本来自贵州省野生动物救助中心在2018—2021年于雷公山国家级自然保护区内收集的不明原因死亡的5只海南鳽,样品编号为HBJ1~HBJ5(表1)。样品均在腿部组织采样,采样后在无菌离心管中加入无水乙醇,放置-20 ℃冷冻保存以备提取DNA。

表1 扩增产物与夜鳽属线粒体全基因组登录号信息
1.2 研究方法
1.2.1 最优分子标记筛选
使用DnaSP 6[18]分析软件计算海南鳽、黑冠鳽和栗头鳽3条线粒体基因组各片段变异位点(variable sites)数量和变异比例。选取位点变异比例高、序列片段长度中等的线粒体基因片段作为物种鉴定的最优分子标记。
1.2.2 样品DNA提取扩增及测序
称取0.5 g海南鳽肌肉组织进行DNA提取,提取方法参考陈晓芳等[19]的研究。提取后加入50~100 μL灭菌ddH2O溶解,使用核酸分析仪测浓度。用1.2%琼脂糖凝胶电泳检测提取DNA的完整度,置于冰箱4 ℃保存待用。扩增引物使用Zhou等[20]设计的引物Cytb3F(5′-TGAGTAGGCAGCCAACCAGTAGA-3′),CR2R(5′-CTGACCGAGGAACCAGAGG-3′)扩增线粒体Cytb-D-loop区基因片段序列。委托生工生物工程(上海)股份有限公司(以下简称上海生工)合成引物。PCR反应采用25.0 μL扩增体系:加入1.0 μL(100 ng/μL)模板DNA,各1.0 μL(10 μmol/L)正向和反向引物;12.5 μLTaqPCR Master Mix(2×,blue dye);ddH2O补足到25.0 μL。反应程序:94 ℃预变性4 min,进入循环;94 ℃变性30 s,55 ℃复性45 s,72 ℃延伸2 min,循环30次;72 ℃终延伸7 min,4 ℃结束。扩增完成后,用3 μL扩增产物在1.2%琼脂糖凝胶中电泳,观察扩增产物在1 400 bp左右是否存在条带,将出现扩增条带对应的PCR产物送上海生工双向测序。
1.2.3 序列分析
PCR产物测序得到的序列峰图文件,使用BioEdit 7.0.5(https://www.softpedia.com/get/Science-CAD/BioEdit.shtml#download)打开,将得到的单峰序列在NCBI数据库中使用BLAST比对,然后将正反向引物测序得到的2条单峰序列使用Dnastar 7.1软件包中的SeqMan程序进行正反向序列拼接,排序对齐后的序列截取NADH6-tRNAGlu-D-loop部分序列。
用DnaSP 6[18]软件统计序列的保守位点(conserved sites)、简约信息位点(parsimony informative sites)、单一多态位点(singleton variable sites)、A+T含量、G+C含量、变异位点、单倍型数(number of haplotypes)、单倍型多样性(haplotype diversity)、核苷酸多样性(Nucleotide diversity)、多态位点数(number of polymorphic sites)和平均核苷酸差异(mean number of pairwise differences)等遗传参数。用Tajima’sD和Fu’sFs检验检测种群进化是否严格遵循物种的中性理论,分析动态扩张的错配分布(mismatch distributions)[21-22]。使用Network 4.6.1.2软件中Median-joining模型构建种群单倍型网络,网络构建时均选择软件默认参数。
2 结果与分析
2.1 线粒体基因组
海南鳽线粒体全基因组(KT364529)长度为19 212 bp,黑冠鳽线粒体全基因组(KT364531)长度为18 470 bp,栗头鳽线粒体全基因组(KT364530)长度为18 562 bp(表2)。物种线粒体全基因组序列根据每段序列的功能不同分类注释:由于夜鳽属3个物种线粒体全基因组序列存在misc feature基因片段,导致各个功能基因的数量不同。海南鳽蛋白编码序列(CDS)片段有14个,黑冠鳽和栗头鳽各有13个,海南鳽比黑冠鳽和栗头鳽重复表达1段NADH6序列;海南鳽tRNA基因编码片段序列分类注释后为25个,黑冠鳽和栗头鳽为22个,海南鳽比其他2个种多的编码片段是重复表达的tRNAThr、tRNAPro、tRNAGlu;3个种的rRNA基因序列均为2个。海南鳽存在3个misc feature基因片段,2个非编码控制区(D-loop),1段Cytb推测剩余序列,而黑冠鳽和栗头鳽存在5个misc feature基因片段,2个非编码控制区,1段Cytb推测剩余序列,1段tRNAThr推测剩余序列,1段tRNAPro-NADH6-tRNAGlu推测剩余序列。
通过夜鳽属3种鸟类线粒体基因组各片段变异位点数及变异比例与线粒体全基因组得到平均变异比例相比,夜鳽属线粒体各片段中变异位点及比例较高的前5个片段依次是:D-loop2(0.230 764)、NADH6(0.229 885)、D-loop1(0.215 026)、ATP8(0.184 524)和tRNAGlu(0.175 676),线粒体基因其他片段具体差异见表2。tRNAPhe、tRNAVal、16SrRNA、tRNALeu、tRNATrp、tRNATyr、tRNAThr、D-loop1和D-loop29个基因在3个种之间碱基长度不等,在各自物种的线粒体基因功能进行表达时,引起物种遗传的多态性,造成群体性状之间的变异。16SrRNA序列片段较长,但是变异比例(0.116 541)比夜鳽属线粒体全基因组变异(0.155 003)平均值低,不采用该片段作为分子标记;6个tRNA序列片段长度较短,在基因编码过程中表达的信息量相对较少,同样不适宜用来做分子标记;而NADH6序列作为蛋白编码基因,3个种NADH6序列片段变异比例高于线粒体全基因组平均变异比例,长度适中,适合做夜鳽属物种的遗传分子标记;在线粒体全基因组序列中,D-loop序列片段是非编码区域,发生突变的概率比其他片段都高,发生变异并被保留下来的概率比其他区域高,所以线粒体D-loop也适合作为物种鉴定的有效分子标记;tRNAGlu编码片段位于D-loop区与NADH6区之间,而且该片段的变异位点比例与线粒体全基因组序列变异比例高;最终选用NADH6-tRNAGlu-D-loop片段作为海南鳽物种遗传鉴定的优先分子标记,并进行接下来的遗传多样性分析研究。
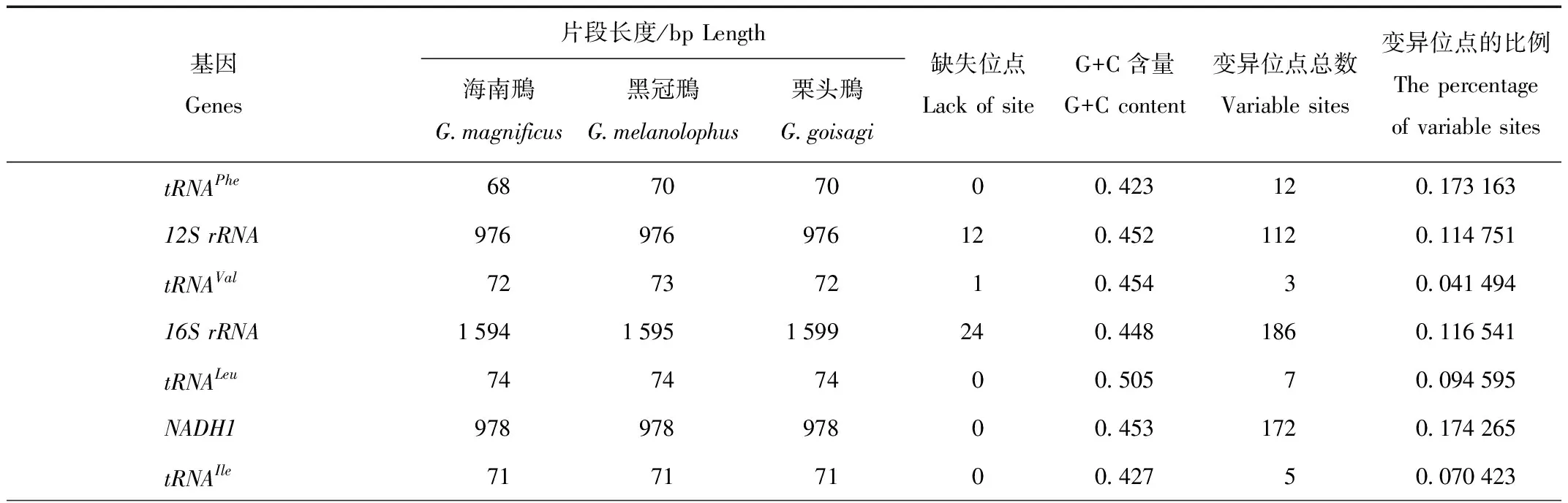
表2 海南鳽、黑冠鳽和栗头鳽线粒体基因组比较
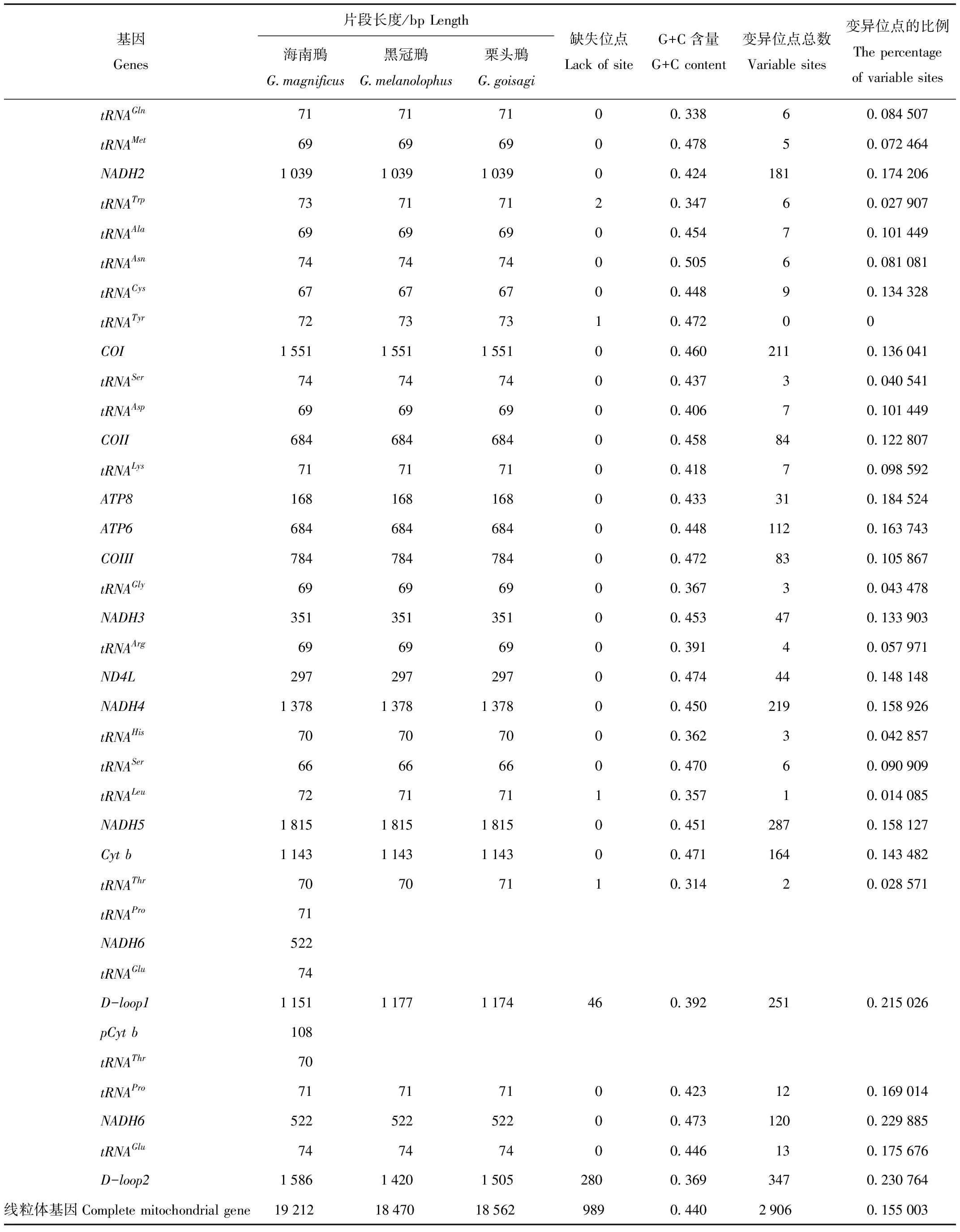
续表2
2.2 样品DNA扩增
样品DNA配制25 μL反应体系经PCR仪扩增完成后,取3 μL扩增产物在1.2%琼脂糖凝胶中电泳,提取的DNA全部成功扩增出目的基因,目的条带大小为1 400 bp(图1)。
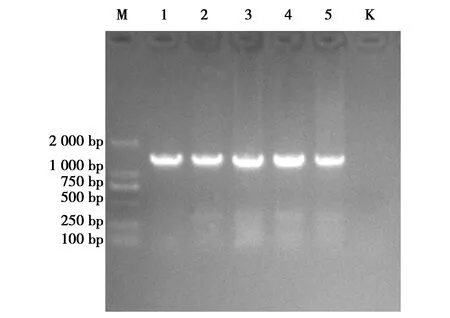
图1 海南鳽目的片段PCR扩增产物凝胶电泳Fig.1 Gel electrophoresis of Gorsachius magnificus target fragment PCR amplification 注:M.D2000 DNA Marker;1~5.扩增产物;K.空白对照 Note:M,D2000 DNA Marker.1-5,PCR product.K,Blank control
2.3 碱基组成
通过测序共获得5条海南鳽Cytb-D-loop片段序列,测序得到的序列在NCBI数据库中进行BLAST比对,比对结果与数据库已经公布的海南鳽线粒体基因序列的相似性最高,5条序列的相似性均在99%以上,可以确定PCR扩增得到的序列即为海南鳽线粒体基因序列。将得到的序列NADH6-tRNAGlu-D-loop部分串联拼接,去掉两端多余序列,使用BioEdit软件将这段序列手动比对并补齐,补齐后的NADH6-tRNAGlu-D-loop基因序列片段总长度为1 032 bp。序列已经上传NCBI数据库,序列登录号为(MZ381449~MZ381453)(表1)。用MEGA 7.0计算5条海南鳽NADH6-tRNAGlu-D-loop基因序列的碱基平均数量,其中A、T、C、G平均含量分别是31.3%、20.0%、31.0%、17.7%;5段序列中A+T平均含量为51.3%,C+G平均含量48.7%;NADH6-tRNAGlu-D-loop共检测到1 029个保守位点、8个变异位点,其中单一多态位点5个,简约信息位点3个;Ts为3,Tv为6,转换/颠换(Ts/Tv)平均值为0.6;鹭科鸟类线粒体碱基序列含量都存在A+T碱基偏好性特点,NADH6-tRNAGlu-D-loop片段碱基偏好性整体符合鹭科鸟类线粒体的特点。
2.4 遗传多样性与历史动态
利用DnaSP 6软件分析海南鳽线粒体NADH6-tRNAGlu-D-loop串联基因序列的变异位点、单倍型数量、单倍型多样性和核苷酸多样性,并进行中性检验。5条海南鳽NADH6-tRNAGlu-D-loop串联序列变异位点数为8;单倍型数量为4;单倍型多样性较高,为0.900;核苷酸多样性为0.003 69;多态位点数为8;平均核苷酸差异值为3.800 00。本研究获得的5条串联序列分属于4种单倍型,样品HBJ1为Hap1,HBJ 2和HBJ3为Hap2,HBJ4为Hap3,HBJ5属于Hap4,种群单倍多样性较高,其中Hap2为海南鳽种群的优势单倍型。江西省海南鳽线粒体全基因序列中该片段定义为1种单倍型Hap5。中性检验Tajima’sD值为-0.073 39(P>0.10),Fu’sFs值为0.051(P>0.10),均未达到显著差异水平。错配分布分析结果显示,曲线呈多峰分布(图2)。以上结果说明该种群在历史上未经历过明显的扩张。
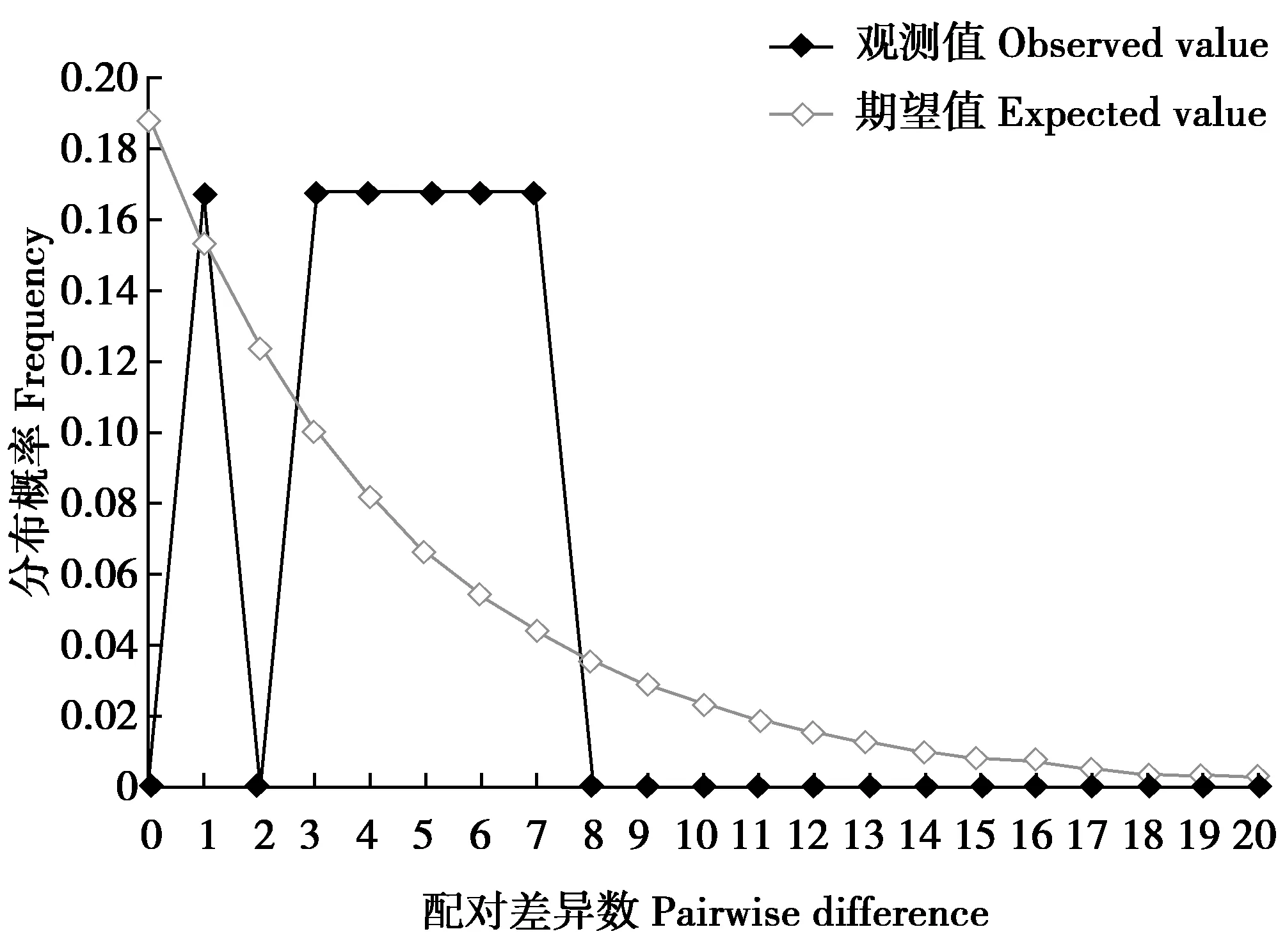
图2 海南鳽线粒体NADH6-tRNAGlu-D-loop错配分布Fig.2 Mismatch map of mitochondrial NADH6-tRNAGlu-D-loop of Gorsachius magnificus
基于4条贵州省(GZ)雷公山海南鳽单倍型序列和1条江西省(JX)海南鳽单倍型序列NADH6-tRNAGlu-D-loop片段构建单倍型网络,共呈现出5种单倍型,3个支系,各单倍型之间以缺失的祖先单倍型相连接,呈3个分支,雷公山小种群含有4种单倍型(Hap1~Hap4),其中Hap2为共有单倍型,江西海南鳽为单独的1种单倍型(Hap5)(图3)。单倍型Hap4是由Hap3经一步突变而来的,Hap5是由Hap2经一步突变而来的,说明贵州省海南鳽单倍型分布较为分散,推测该5个个体至少来自3个巢穴。江西省海南鳽可能是由贵州海南鳽扩散过去的。
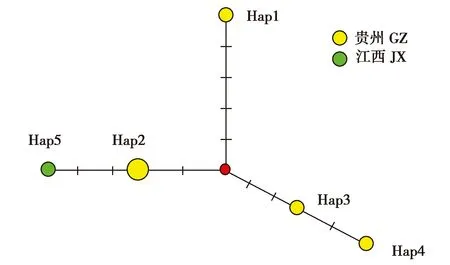
图3 基于贵州、江西省海南鳽NADH6-tRNAGlu-D-loop的单倍型网络Fig.3 Network diagram of NADH6-tRNAGlu-D-loop haplotypes of Gorsachius magnificus in Guizhou and Jiangxi 注:黄颜色圆圈代表贵州省单倍型,绿色圆圈代表江西省单倍型,红色圆圈代表缺失单倍型 Note:Yellow circles represent Guizhou haplotypes,green circles represent Jiangxi haplotypes,and red circles represent missing haplotypes
3 讨论
3.1 线粒体基因组片段分析及分子标记筛选
本研究通过分析海南鳽、黑冠鳽和栗头鳽3个物种线粒体全基因序列片段,发现D-loop区、NADH6和tRNAGlu3个基因片段的变异比例较高。D-loop区是线粒体基因组中进化速率最快、遗传突变较高的一段序列,序列发生变异后容易保留下来,是目前分子鉴定常用的分子标记[23],适用于亲缘关系较近的种间差异研究[24]。该片段作为分子标记被广泛应用于白鹭(Egrettaeulophotes)[23,25]、红原鸡(Gallusgallus)[26]、马(Cleveland Bay hourse)[27]、羊(Ovisaries)[28]、驴(Equusasinus)[29]、藏猪(Tibetan pig)[30]和驯鹿(Rangifertarandus)[31]等动物遗传多样性和系统发育研究中。NADH6基因序列长度、进化速率适中,基因突变频率高,被认为是研究脊椎动物间遗传多样性和系统发育分析的较适合的分子标记[32-33]。该片段被成功应用于解析金丝猴(Rhinopithecusspp.)性状与环境间的关系[34],揭示了家马(Equuscaballus)和藏马(Tibetan horse)基因对不同海拔的适应性机制[35-36]。综上所述,D-loop区和NADH6基因是一个被广泛应用于各种动物遗传多样性研究中的分子标记。然而Ibrahim等[28]建议使用长基因片段进行线粒体遗传多样性研究。所以,在节约测序成本的情况下,NADH6-tRNAGlu-D-loop片段是一个代替线粒体全基因组的分析海南鳽遗传多样性的最优分子标记。该分子标记A+T碱基含量为51.3%,与其他鹭科鸟类的研究结果相似,均表现出一定的A和T的偏向性[37],转换与颠换比值为0.6,说明序列变异多来自颠换,而颠换率高于转换率的基因被认为更适合构建系统发育树[38],再次证实该联合基因片段是最优分子标记。
3.2 基于NADH6-tRNAGlu-D-loop联合基因的遗传多样性分析
遗传多样性除了反映个体间性状的差异外,还反映核苷酸的微小差异[33]。mtDNA的核苷酸多样性(Pi)和单倍型多样性(Hd)是评价群体多态性和遗传分化的2个重要指标,Pi为0.001~0.010,可划分为3个类别,即高类别(0.008~0.010)、中类别(0.005~0.007)和低类别(0.001~0.004)[39],Hd值在0~0.5属于低等类别,0.5~1.0被列入高类别[40]。本研究中海南鳽NADH6-tRNAGlu-D-loop基因序列Hd值为0.900,Pi值为0.003 69,呈现出与白鹭研究结果较一致的低核苷酸多样性和高单倍型多样性[23],符合Grant等[41]提出的进化模式,推测雷公山海南鳽小种群曾经历过瓶颈效应,随后出现了迅速地种群扩张与变异的积累,但还没有足够的时间积累核苷酸变异[42]。而Tajima’sD值为负数,Fu’sFs值为正值,差异表达均不显著,核苷酸错配分布图为多峰,证实了该种群在历史上未经历过扩张[43-44],但不排除现阶段是扩张状态。通过单倍型网络的分析,推测江西海南鳽可能来自雷公山海南鳽小种群的分化,提示贵州可能是海南鳽分化的中心之一,应给予更高的保护重视,而目前海南鳽的分子数据极其匮乏,暂时还无法做这个濒危物种的起源与进化方面的研究。
4 结论与展望
合适的线粒体分子标记对濒危物种的鉴定与遗传多样性分析起关键性作用。本研究对海南鳽、黑冠鳽和栗头鳽3种鸟类线粒体全基因组序列进行分析,确定NADH6-tRNAGlu-D-loop序列可以作为海南鳽及其近缘物种遗传多样性研究的最优分子标记;通过对雷公山海南鳽NADH6-tRNAGlu-D-loop序列的遗传多样性、中性检验、错配分布和单倍型网络分析,推测雷公山海南鳽小种群处于刚经历了瓶颈效应后的扩张状态,雷公山可能为海南鳽的分化中心之一,建议对贵州境内的海南鳽种群分布和数量进行全面调查,加强国内和国际合作,深入全面地了解海南鳽栖息状态及遗传多样性水平,从而能够更好地制定保护计划。
