不同海拔喜马拉雅旱獭线粒体基因组比较分析
2022-05-16南新营李耀东
南新营 李 优 李耀东
(青海大学,西宁,810016)
喜马拉雅旱獭(Marmotahimalayana)属哺乳纲(Mammalia)啮齿目(Rodentia)松鼠科(Sciuridae)旱獭属,体形肥大,是松鼠科中体型最大的一种,主要栖息于我国青藏高原及其毗邻的印度、尼泊尔及巴基斯坦等地的各类高寒草甸草原[1]。作为世居高原的一个特有物种,喜马拉雅旱獭经过长期的自然选择和自身适应,在生理生化上获得了适应高原地区低温低氧的稳定遗传学特性,是研究高原低氧适应的天然理想动物模型[2]。
早在20世纪80年代末,陈钦铭等[3]首次用喜马拉雅旱獭作为动物模型来研究其适应高原低氧的机理。随后,贾荣莉[4]从心脏、血液和内分泌等角度分析,发现喜马拉雅旱獭的红细胞数及心肌细胞的形态结构、生理功能与生化酶均未受到高原低氧的不良影响,表明对高原低氧有很好的适应性。随着二代测序技术的迅猛发展,其高通量、快速、低成本的特点成为越来越多的学者解决生物学问题的首选。线粒体基因组中编码蛋白质的所有基因都位于电子转移呼吸链上,在氧的利用和能量代谢中起着关键作用。Xu等[5]认为线粒体基因的进化可能反映了高原适应的一些过程;Chao等[6]通过对喜马拉雅旱獭线粒体基因组测序,已获得喜马拉雅旱獭线粒体基因组全序列。线粒体能量代谢在生物进化和物种形成过程中起着重要作用,本研究以不同海拔喜马拉雅旱獭线粒体为供试材料,利用二代测序技术组装线粒体基因组,通过线粒体差异基因的比较分析,从mtDNA一级结构上筛选喜马拉雅旱獭低氧适应相关的基因,为进一步在分子水平上阐明高原低氧适应机理提供理论依据。
1 材料与方法
1.1 样品采集
在青海省海东市乐都区引胜乡(海拔2 279 m,3例)、黄南州同仁县瓜什则乡(海拔3 273 m,3例)和玉树州曲麻莱县麻多乡(海拔4 452 m,2例)3个地区采集喜马拉雅旱獭肝脏组织共8例,于液氮中保存并带回,冰箱内-80 ℃储存。
1.2 喜马拉雅旱獭基因组DNA提取
用痕量样品基因组DNA提取试剂盒(Qiagen公司)提取基因组DNA。
1.3 喜马拉雅旱獭线粒体基因组测序
采用Illumina HiSeqX10[7]测序技术对8个样品的DNA进行paired-end测序,用fastQC软件(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)观测测序质量,然后使用NGSQC[8]软件对测序数据质控。测序由百迈客生物科技有限公司完成。
1.4 喜马拉雅旱獭线粒体基因组组装
使用SPAdes[9]对质控过的clean data初步拼接,经过同源比对和蛋白序列比对后,利用PRICE(Paired-Read Iterative Contig Extension)[10]进行迭代延伸、gap补洞及重拼接,直至获得完整的线粒体基因组序列。
1.5 数据分析
获得完整的线粒体基因组序列后,使用MEGA X软件[11]分析不同海拔喜马拉雅旱獭线粒体基因组大小和碱基组成。采用mafft软件[12]进行序列比对,用Mega X软件采用最大似然法(Maximum likelihood,ML)[13]构建系统发育树。用SPSS 19.0对3个海拔喜马拉雅旱獭线粒体基因组中13个蛋白编码基因的突变位点个数进行统计学分析。使用MEGA X做基于密码子的比对。用KaKs_Calculator 2.0[14](https://sourceforge.net/projects/kakscalculator2/)计算Ka/Ks,计算方法选择γ-YN,若Ka/Ks>1,则认为有正选择效应;若Ka/Ks=1,则认为存在中性选择作用;若Ka/Ks<1,则认为是纯化选择作用。密码子偏好性分析使用 CodonW 软件,同义密码子相对使用度(relative synonymous codon usage,RSCU)等于1,则密码子使用没有偏好;若大于1则表明其使用频率相对较高,反之则该密码子使用频率相对较低。
2 结果与分析
2.1 喜马拉雅旱獭线粒体基因组测序
2.1.1 测序数据统计
通过Illumina HiSeq X10高通量测序平台,采用2×150 bp双末端策略,对8个样品的文库进行线粒体基因组测序。根据原始数据过滤后结果,Q20值均在97%以上,Q30值均在93%以上(表1)。表明测序数据整体质量较好,可用于后续线粒体基因组的组装。
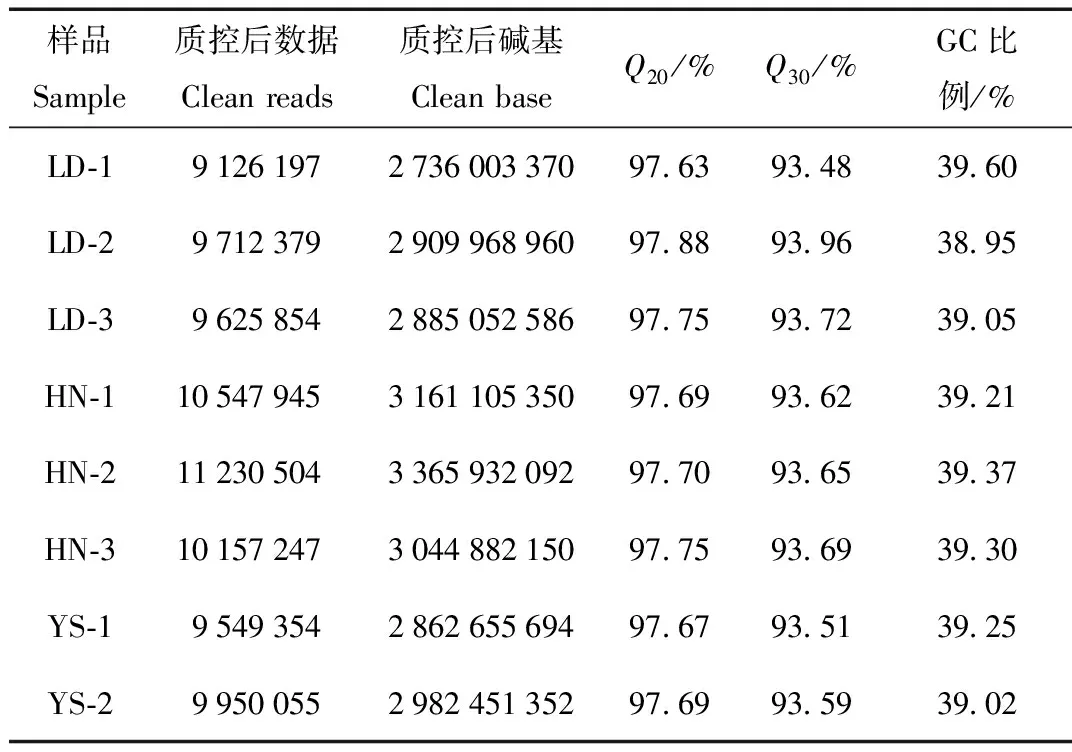
表1 喜马拉雅旱獭样本过滤后的测序数据
2.1.2 线粒体基因组序列
测序获得了3个海拔共8个样品的喜马拉雅旱獭线粒体基因组(表2),发现不同海拔喜马拉雅旱獭线粒体序列长度之间并无差异,均为16 442 bp,其核苷酸组成也无差异,A、T、G、C平均含量为32.1%、31.4%、12.8%、23.7%,有明显的AT偏倚。每个样品由37个片段拼接而成,最大片段为1 827 bp,最小片段为59 bp,GC含量为36.5%,8个样品之间没有差异。
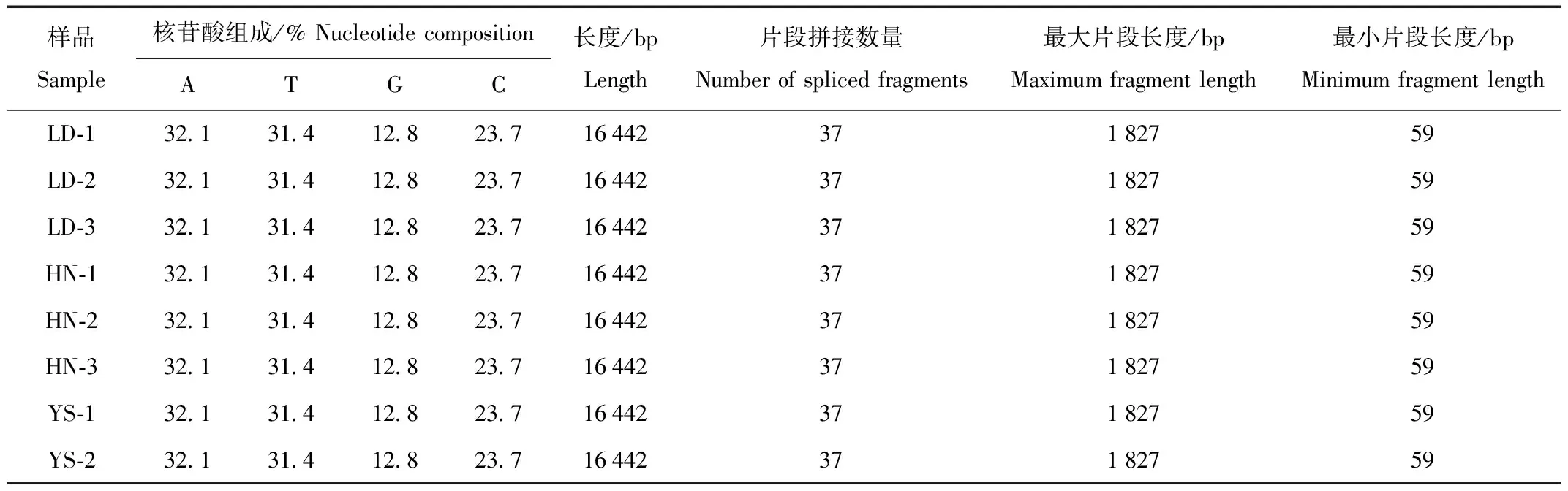
表2 喜马拉雅旱獭线粒体DNA序列信息
2.2 系统发育分析
黄娅琳等[15]研究发现基于单一基因位点构建系统发育关系容易造成偏差,使用线粒体基因组全序列构建的系统发育树更为精确可靠。基于线粒体基因组组装好的序列,以温哥华旱獭(Marmotavancouverensis)[16](序列号:MK859 897.1)为外群,用MEGA X软件采用最大似然法构建系统发育树(图1),从系统树可看出,黄南的2、3号聚为一支,再与乐都的3号聚为一支,乐都的1、2号聚为一支后又同黄南1号聚为一支,最后与玉树的1、2号聚为一支,表明与玉树相比,乐都和黄南的喜马拉雅旱獭亲缘关系相对较近。
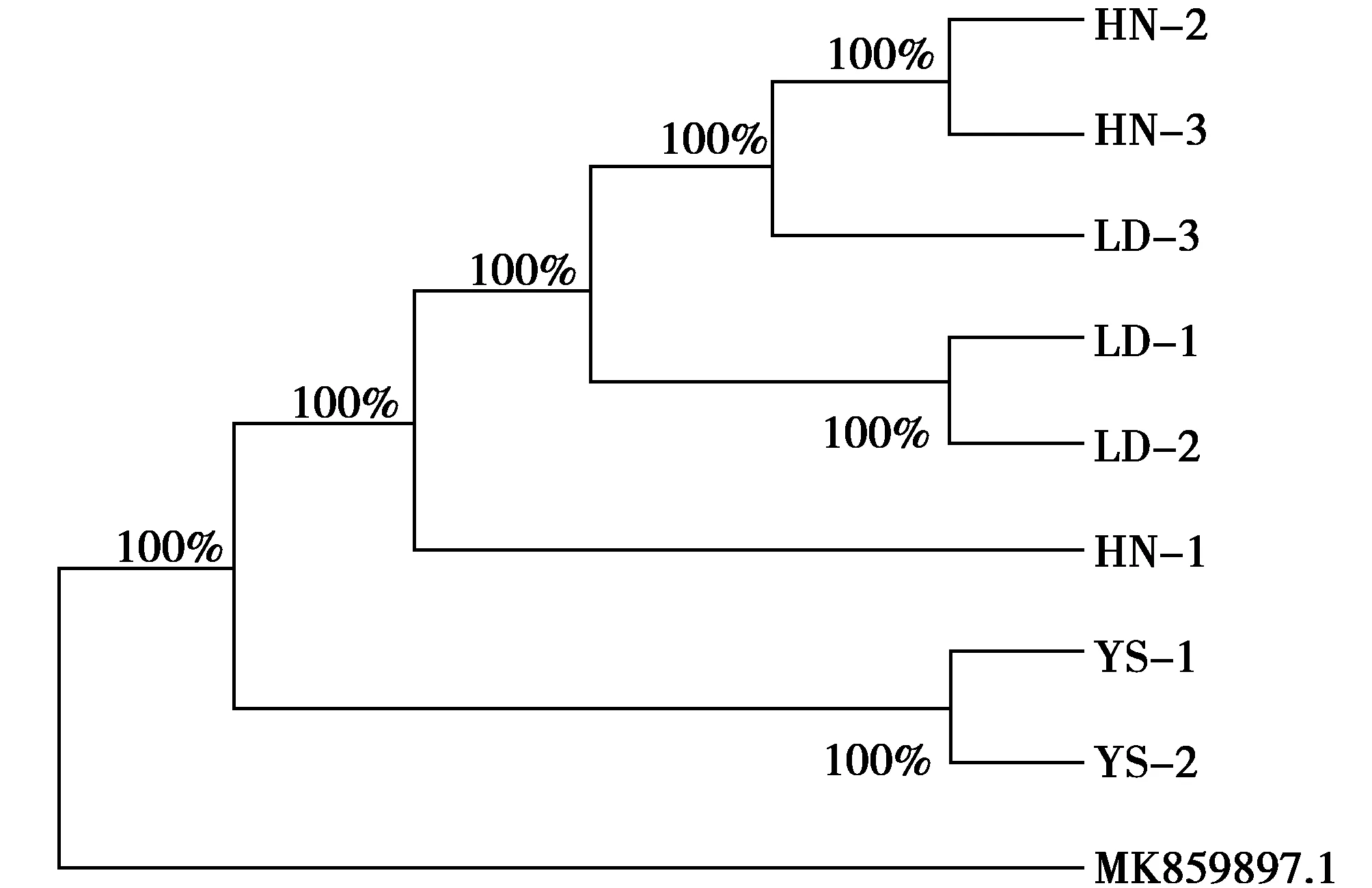
图1 ML法构建的不同海拔喜马拉雅旱獭发育树Fig.1 Phylogenetic relationship of eight Marmota himalayana constructed by ML approach
2.3 喜马拉雅旱獭线粒体DNA蛋白质编码基因差异
2.3.1 13个蛋白编码基因突变位点个数
对不同海拔喜马拉雅旱獭线粒体基因组中13个蛋白编码基因的突变位点个数进行统计,发现共有65个突变位点,其中COX3基因最为保守,没有发生变异,而ND5基因有18个突变位点,比例最高(27.7%)。玉树的突变位点个数极显著高于乐都和黄南(P<0.01),即随着海拔的增加,突变位点个数显著增多(表3)。
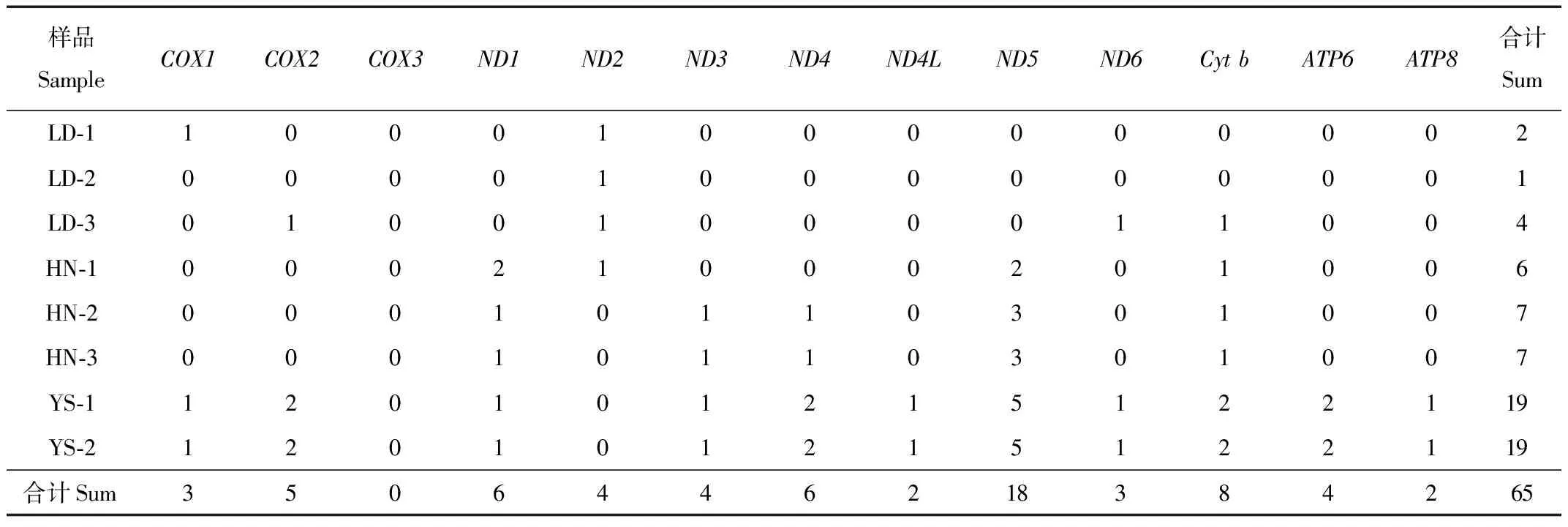
表3 不同海拔喜马拉雅旱獭13个蛋白编码基因突变位点个数
2.3.2 功能基因蛋白编码序列差异
分别比较了13个编码蛋白的核苷酸和氨基酸序列,发现除了COX3基因没有突变,COX1、COX2、ND4L、ATP6和ATP8虽然在基因水平上发生了突变,但并没有发生相应的氨基酸改变,即同义突变(same sense mutation),共有41个基因位点发生了同义突变,如玉树-1、玉树-2的COX1基因第1 380位G→A,COX2基因第648位T→A,ND4L基因的第513位T→C,ATP6基因第324位A→G(图2,图3),ATP8基因第6位C→T。而ND1、ND2、ND3、ND4、ND5、ND6和Cytb这些基因某些位点的改变导致了氨基酸的改变,即非同义突变(non-synonymous mutation),这7个基因里共有24个非同义突变(表4)。
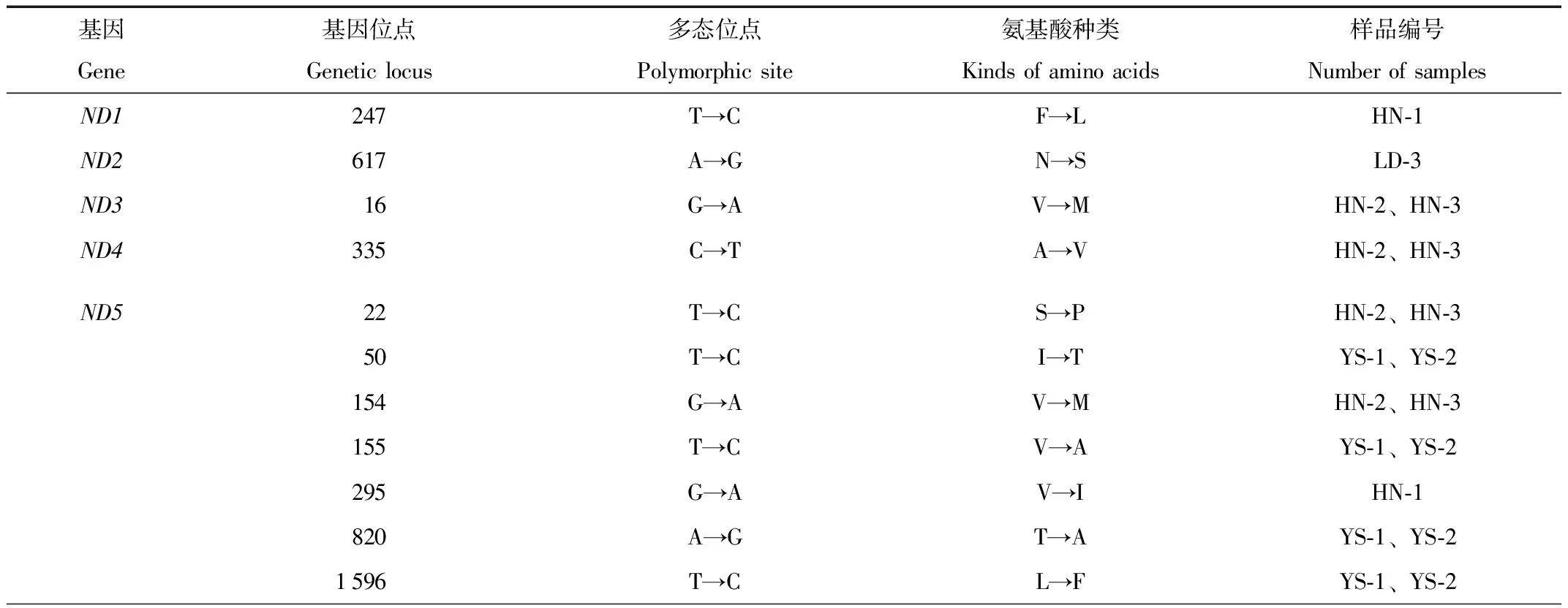
表4 蛋白编码基因非同义突变位点

续表4
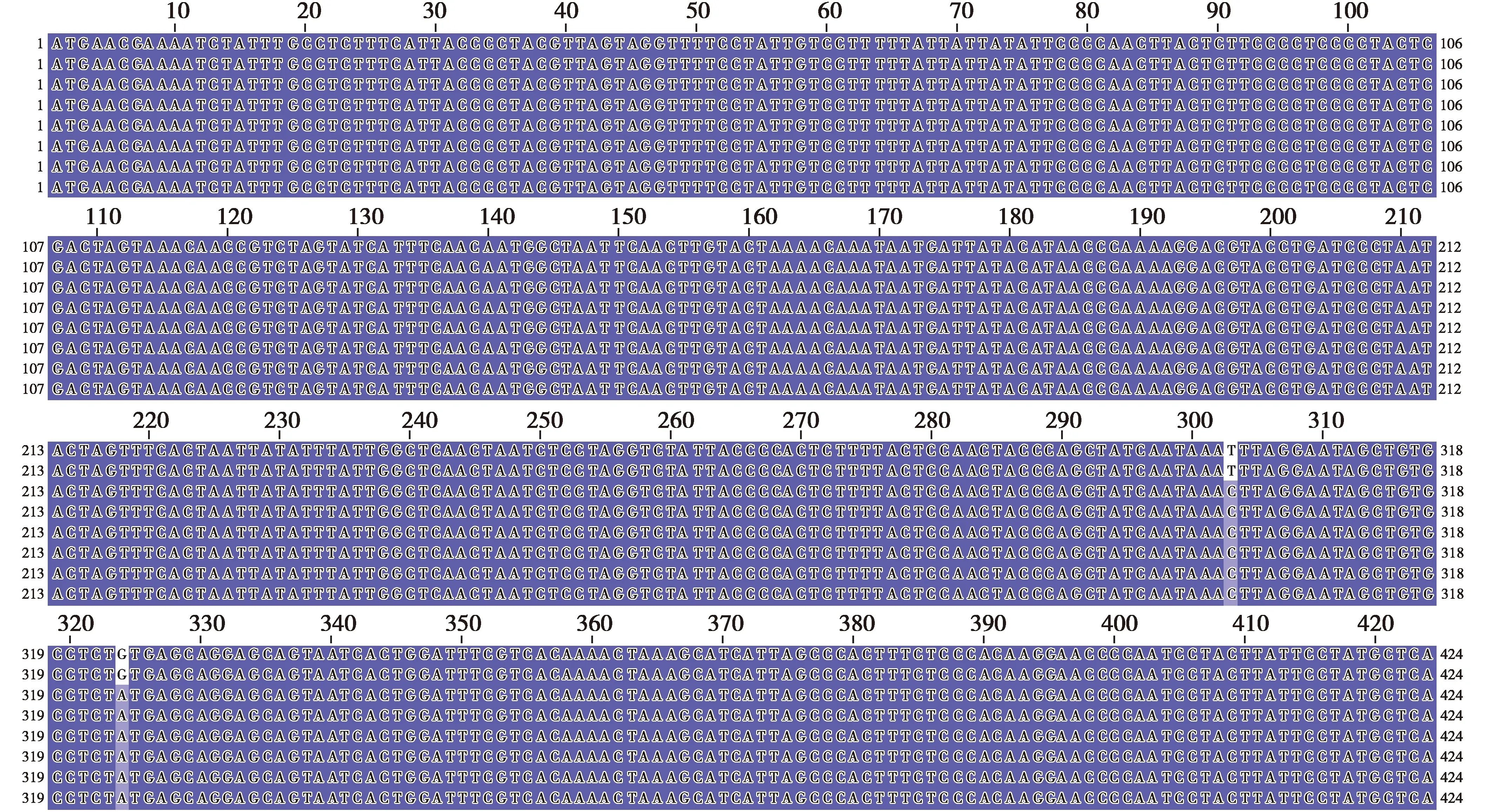

图2 ATP6的核苷酸序列比较Fig.2 Comparison of nucleotide sequences of ATP6 注:从上到下依次为YS-1、YS-2,LD-1、LD-2、LD-3、HN-1、HN-2、HN-3,下同 Note:From top to bottom are YS-1,YS-2,LD-1,LD-2,LD-3,HN-1,HN-2 and HN-3,the same as below

图3 ATP6的氨基酸序列比较Fig.3 Comparison of amino acid sequences of ATP6
由图4和图5可见,黄南的非同义突变位点个数最多,有13个,如HN-2、HN-3的ND4基因第335位C→T,导致ND4氨基酸序列的第112位A→V(丙氨酸→缬氨酸),ND5基因第22位T→C,导致ND5氨基酸序列的第8位S→P(丝氨酸→脯氨酸);玉树的次之,有8个,YS-1、YS-2的ND5基因第50位T→C,导致ND5氨基酸序列的第17位I→T(异亮氨酸→苏氨酸);乐都的最少,只有3个,LD-3的Cytb基因第514位T→C,导致Cytb氨基酸序列的第172位Y→H(酪氨酸→组氨酸),ND2基因第617位A→G,导致ND2氨基酸序列的第206位N→S(天冬酰胺→丝氨酸),ND6基因第361位T→A,导致ND6氨基酸序列的第121位L→M(亮氨酸→蛋氨酸)。ND5基因的非同义突变位数最多,有7个。

图4 ND6的核苷酸序列Fig.4 Comparison of nucleotide sequences of ND6

图5 ND6的氨基酸序列Fig.5 Comparison of amino acid sequences of ND6
2.3.3 13个蛋白编码基因的Ka/Ks
通过13个蛋白编码基因,计算不同海拔喜马拉雅旱獭碱基的Ka、Ks、Ka/Ks,统计结果表明(图6),Ka中ND5的值最高,ND3次之,COX1、COX2、COX3、ND4L、ATP6、ATP8的Ka值为0;Ks中ATP8最高,COX2次之,COX3最小为0,ND5的Ka/Ks>1,表明ND5基因存在正选择效应,可作为适应高海拔低氧环境的候选基因,其余基因均小于1,表明这些基因存在纯化选择作用。
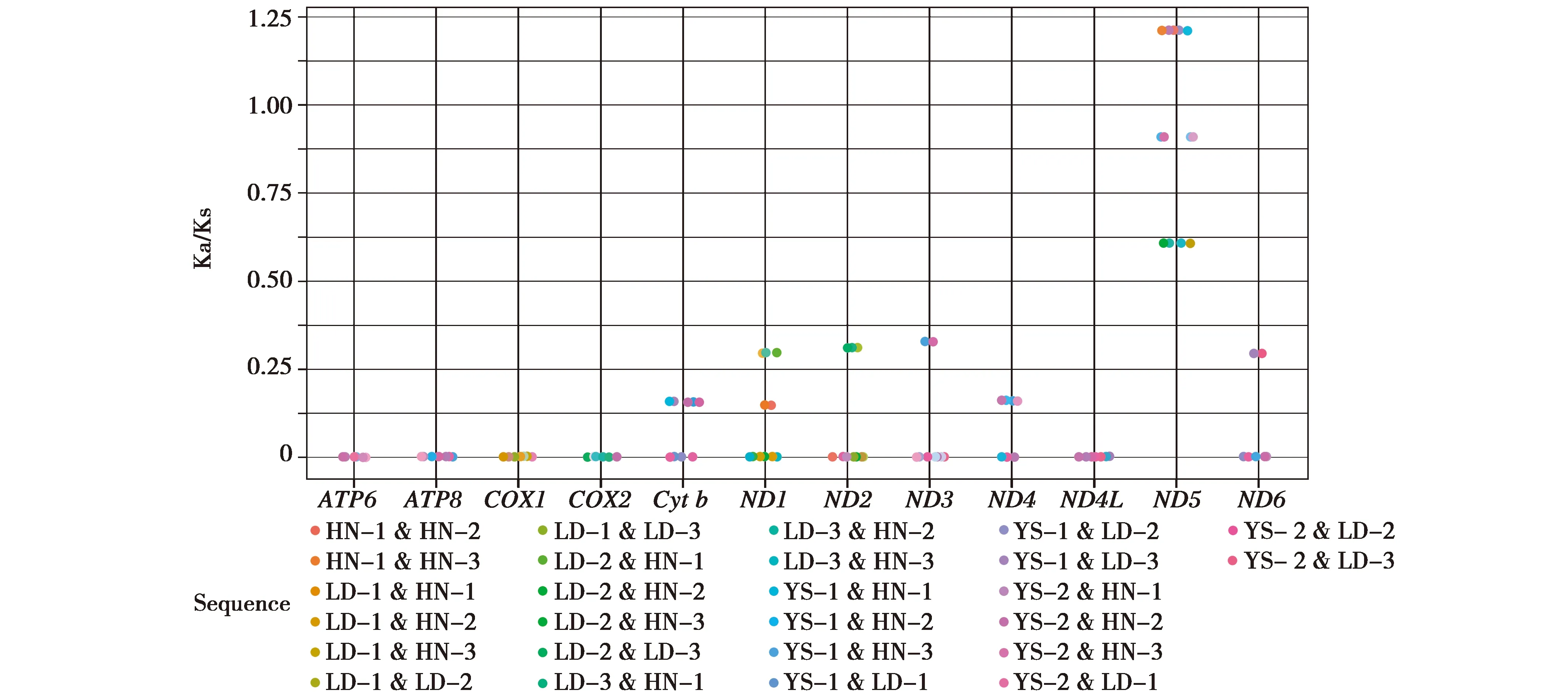
图6 喜马拉雅旱獭蛋白编码基因Ka/Ks值Fig.6 Ka/Ks values of protein-coding genes of Marmota himalayana
2.3.4 喜马拉雅旱獭线粒体密码子偏好性
喜马拉雅旱獭13个蛋白质编码基因的核苷酸序列共编码氨基酸3 812个,去掉起始密码子和终止密码子后共编码氨基酸3 802个,在20种氨基酸中,Leu的使用率最高,达16.18%,Gys的使用率最低,仅有0.71%。在喜马拉雅旱獭中RSCU值>1的密码子有32种,RSCU值<1的密码子有32种,其中AGG的RSCU为0,即在编码过程中没有使用该密码子(图7)。

图7 喜马拉雅旱獭线粒体密码子偏好Fig.7 Mitochondrial codon bias analysis of Marmota himalayana
3 讨论
线粒体作为动物的“能量工厂”,通过氧化磷酸化产生能量,在生物进化和物种形成过程中起着重要作用,因此成为研究生物适应高原低氧环境的分子基础[17]。本研究通过对不同海拔8个样品的喜马拉雅旱獭线粒体基因组测序,发现不同海拔地区的喜马拉雅旱獭在线粒体基因组长度和核苷酸组成上并无差异,可能在长期的进化过程中,喜马拉雅旱獭已适应了高原地区低温低氧气候,获得了稳定的遗传特性。对其13个蛋白编码基因进行统计分析,发现随着海拔的增高,突变位点个数也随之增加,其中ND5基因的突变位点个数和非同义突变位点个数都为最多,且ND5基因Ka/Ks>1,认为有正选择效应,表明ND5基因在适应低氧中发挥着重要作用。跟玉树相比,乐都和黄南的喜马拉雅旱獭亲缘关系相对较近,且每个海拔梯度之间的亲缘关系较近,表明地理隔离是影响喜马拉雅旱獭种群交流的重要因素,这与马英等[18]的研究结果一致。本研究筛选出ND5这一低氧适应相关基因,为进一步研究喜马拉雅旱獭高原低氧适应机制提供了新的方向和思路。
