基于线粒体蛋白编码基因探讨飞虱科Delphacidae13属系统发育关系
2022-05-12张争光苏田娟
张争光,赵 芳,苏田娟,蒋 凯
基于线粒体蛋白编码基因探讨飞虱科Delphacidae13属系统发育关系
*张争光,赵 芳,苏田娟,蒋 凯
(井冈山大学生命科学学院,江西,吉安 343009)
飞虱科昆虫隶属于半翅目蜡蝉总科,是世界上最重要的农业害虫之一。昆虫的线粒体基因是昆虫分子与进化研究中常用且有效的分子标记。本研究基于线粒体蛋白编码基因探讨了飞虱科13属18种的系统发育关系,分别采用距离法、最大似然法和贝叶斯法构建的系统发育树得到的结论是一致的,即在飞虱科13属中,Ugyops属较为原始,处于系统树的基部,其余12个属聚为2支,Bambusiphaga属、Epeurysa属、Tropidocephala属和Saccharosydne属聚为一支,Peregrinus属、Perkinsiella属、Changeondelphax属、Chloriona属、Ishiharodelphax属、Sogatella属、Laodelphax属和Nilaparvata属聚为另一支。18种的进化地位和分类归属与传统分类结果基本一致。
线粒体基因组;系统发育;飞虱科
飞虱科Delphacidae隶属于半翅目Hemiptera头喙亚目Auchenorrhyncha蜡蝉总科Fulgoroidea,是蜡蝉总科中最大的一个类群,广泛分布于世界各地,物种多样性十分丰富,目前全世界已报到427属2226种[1]。该科昆虫许多种类是重要的农林业害虫,主要通过直接取食和作为病原物的传播载体对农作物造成危害[3]。其以刺吸式口器吮吸植物的汁液来夺取植物的营养,使植物营养不良或至枯萎或在吮吸部位出现黄色或黄褐色病斑,有的则因涎液的刺激使植物细胞反常增殖,造成畸形臃肿的虫瘿,作为植物病毒传播的潜在媒介,是玉米、甘蔗、小麦、水稻、大麦等主要粮食作物上的重要的农业害虫之一[2]。由于具有很高的繁殖潜力、扩散能力和对植物病毒疾病的传播能力,一些飞虱种类已经对谷物生产造成了巨大损害,并在一些亚洲国家被确定为造成稻米饥荒的重要原因之一[4]。
飞虱分类始于18世纪欧洲。1815年英国伦敦大英博物馆馆长Leach以Fabricius 为模式属建立了飞虱科(Delphacidae),为飞虱类昆虫和系统分类奠定了基础[5]。我国飞虱科研究起步较晚,早期的飞虱研究始于国外学者,在1941年胡经甫编写《中国昆虫名录》中仅记录飞虱9属14种。此后,Metcalf在《世界飞虱名录》中记载了我国飞虱种类23属55种,其中多数是分布于台湾的种类[6]。上世纪七十年代,由黄其林和葛钟麟主持的飞虱科分类研究开启了我国大陆飞虱科昆虫分类研究工作。经历了70年代中后期创始阶段,进入80年代后我国飞虱分类研究进展较快,相关研究进展详见丁锦华教授的我国飞虱分类研究概况一文[7]。进入新世纪以后,我国飞虱科的研究得到了进一步快速发展,主要研究内容包括飞虱若虫分类、飞虱新种记述和属级阶元厘定、飞虱区系分类和地理分布以及飞虱分子鉴定等。例如陈祥盛等对中国飞虱科若虫分类开展了系列研究[8-11]。郭两珍等对叉飞虱属[12],陈祥盛等对长突飞虱亚科和偏角飞虱属、竹飞虱属[13-14],秦道正等对隆脊飞虱属、帕飞虱属、细突飞虱属、等胸飞虱属、扁角飞虱属开展了系统分类研究,发表了大量新种[15-18];李红荣,陈祥盛开展了飞虱生物地理学研究,分析了贵州飞虱科昆虫物种多样性及地理分布格局[19]。特别是南京农业大学植保学院丁锦华教授编著的《中国动物志—昆虫纲飞虱科》,它的出版是对我国飞虱科昆虫分类研究工作的系统总结,共记述我国飞虱152属335种,其中包括31新属43新种和15新组合种,并核定新异名32种,极大地促进了我国飞虱科分类工作的发展[20]。
近年来,随着分子生物学技术的快速发展,飞虱科的分类研究也从传统单一的形态分类向形态、分子相结合的综合分类和系统进化方向发展。例如侯晓辉,陈祥盛利用16S rDNA基因序列对飞虱科5属10种15个地理种群进行了分子鉴定[21]。Zhu Junjie等报到了灰飞虱的纹状体基因组组装体,并与白背飞虱()和褐飞虱()两种飞虱进行了比较[22]。在系统发育研究方面,一些研究者基于单个基因或联合少数几个基因,如16S rDNA、28S rDNA、COI和Cytb基因研究了Delphacini和Tropidocephalini的系统发育关系[23]。Urban等则基于4个核基因(18S rDNA、28S rDNA、Wingless和COI)和132个形态学编码特征研究了飞虱科高级阶元间系统发育关系,并根据寄主—植物组合的进化模式发现飞虱科多样性的快速分化与寄主转移到禾本科植物有关,特别是从C3植物向C4植物的转移有关[24]。
物种的准确鉴定和物种间进化关系的确定对于指导害虫防治策略的制定非常关键,由于种类繁多和形态近似,飞虱科物种传统的形态鉴定和进化关系研究一直面临着较大挑战。以往的研究大多基于单个基因(如16SrDNA、COI)或少数几个基因联合(如16S rDNA、28S rDNA、COI和Cytb),造成系统发育的信息较少,由于多基因联合能够解决单基因系统发育信息较少的问题,结果更接近物种进化的真实情况,因此在系统发育研究中受到越来越多的关注。随着分子生物学技术和测序技术的发展,线粒体基因组序列的获得变得越来越便捷,线粒体基因在昆虫进化研究中的应用也越来越广泛。比如Song等学者利用49个线粒体基因序列(蛋白编码基因和核糖体RNA基因)探讨了半翅目的目级阶元的亲缘关系,研究结果支持蜡蝉亚目Fulgoromorpha与胸喙亚目Sternorrhyncha为姐妹群,各亚目间的系统发育关系为(((Fulgoromorpha, Sternorrhyncha), Cicadomorpha),Heteroptera),该结论解决了蜡蝉总科的分类地位问题,这与半翅目传统的分类系统相一致[25]。测序技术的快速发展导致GenBank数据库中线粒体基因组数据呈爆发式增长。截止目前,GenBank数据库中已收录飞虱科昆虫mtDNA基因组序列共计18种,分属于13个属,详细信息见表1。本文联合线粒体13个蛋白编码基因,探讨迄今已测定线粒体基因组全序列的飞虱科13属18种的系统发育关系,以验证线粒体蛋白编码基因在飞虱科系统发育研究中的应用价值。
1 材料与方法
1.1 数据来源
18个飞虱科昆虫物种的线粒体基因组序列数据均下载自GenBank,具体情况见表1。
表1 飞虱科线粒体基因及外群的GenBank信息
Table1 GenBank information of mitochondrial genes of Delphacidae and Outgroup
属种GenBank登录号参考文献 UgyopsU. sp.MH352481.1Fang Y. & A.P. Liang, 2018[26] BambusiphagaB. furcaMH293453.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] BambusiphagaB. taibaishanaMH293456.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] EpeurysaE. nawaiiMH293459.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] TropidocephaiaT. brunnipennisMH293471.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] SaccharosydneS. procerusMG515237.1Huang Y.X. & D.Z. Qin, 2018[28] PeregrinusP. maidisMG049917.1Huang Y.X. & D.Z. Qin, 2017[29] PerkinsiellaP. saccharicidaMH293466.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] NilaparvataN. bakeri|KC333655.1Lu L., X.X. Peng & S.L. Jing et al. 2015[30] NilaparvataN. muiriJN563998.1Lu L., X.X. Peng & S.L. Jing et al. 2015[30] NilaparvataN. lugensJN563995.1Lu L., X.X. Peng & S.L. Jing et al. 2015[30] ChangeondelphaxC. velitchkoskyiMG049916.1Huang Y.X. & D.Z. Qin, 2018[31] ChlorionaC. tateyamanaMH293458.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] IshiharodelphaxI. matsuyamensisMH293461.1Huang Y.X., F.J. Ren & C.R. Bartlett et al. 2020[27] Sogatella S. furciferaKC512914.1Zhang K.J., W.C. Zhu & Rong X. et al. 2014[32] SogatellaS. kolophonMW009064.1Yu F. & Z.S. Song 2021[33] SogatellaS. vibixMG515238.1Huang Y.X. & D.Z. Qin, 2018[28] LaodelphaxL. striatellusFJ360695.1Song N. & A.P. Liang, 2009[34] CicadellaC. viridisMK335936.1Zhong L.K., H.X. Li & X.F. Yu, et al. 2019[35] CicadettaC. absconditaMW123088.1unpublished
1.2 分析方法
多序列比对与序列联合:13个线粒体蛋白编码序列分别通过MegaX(Molecular Evolutionary Genetics Analysis across computing platforms)软件进行多序列比对[36],经过人工修剪后保存为fasta格式。然后使用MegaX软件对比对后的蛋白编码基因进行多序列联合,形成单一的联合序列。
碱基序列分析:在MegaX中分别查看各基因碱基长度,计算基因的碱基组成,统计各基因的不变位点(C)、可变位点(V)和信息位点(PI)的碱基数,并计算13蛋白编码基因联合序列的碱基配对频率,包括相同碱基对数(ii)、转换碱基对数(si)、颠换碱基对数(sv)以及转换/颠换比(R)。
距离法构建系统发育树:在MegaX中采用NJ法(Neighbor-joining Method)构建系统发育树,系统发育树的检验(Test of Phylogeny)采用Bootstrap method,重复次数(No. of Bootstrap Replications)为1000次,其余选项采用默认设置。
最大似然法构建系统发育树:利用IQTREE 1.6.12软件进行进化模型的选择[37],然后基于BIC标准选择最佳模型,利用最大似然法构建ML树。
贝叶斯法构建系统发育树:利用MrModeltest 2.0软件分别计算13个编码基因的碱基替换模型[38],然后利用MrBayes3.27构建贝叶斯进化树[39]。
2 结果与分析
2.1 碱基序列分析
通过NCBI网站下载了飞虱科18个物种的线粒体全基因组和2个外群的线粒体基因组,通过线粒体基因组信息,分别提取出每个物种的13个蛋白编码基因。13个线粒体蛋白编码序列分别通过MegaX(Molecular Evolutionary Genetics Analysis across computing platforms)软件进行多序列比对,经过人工修剪后保存为fasta格式。然后使用MegaX软件将比对好的13个蛋白编码基因进行多序列联合,形成单一的联合序列。13个蛋白编码基因的长度、位点信息及碱基多样性信息见表2。其中NAD5基因长度最长(1728 bp),ATP8基因长度最短(129 bp),C/V值最高的是COX1(0.96),最低的是NAD4L(0.21),信息位点(PI)最高的是NAD5,最低的是ATP8。13蛋白编码基因联合序列的碱基组成见表3,其中平均A+T含量为76.33%,远高于G+C含量。13蛋白编码基因联合序列的碱基配对频率见表4,平均相同碱基对(ii = Identical Pairs)7579bp,转换碱基对(si = Transitional Pairs)982bp,颠换碱基对(sv = Transversional Pairs)1856bp,转换/颠换比(R = si/sv)为0.50。
表2 13蛋白编码基因联合序列位点信息
Table 2 Site information of 13 protein coding genes
GeneLengthSitesCVC/VPI NAD210111-10111847850.23646 COX115331012-25447527810.96644 COX26722545-32162544180.61331 ATP81293217-334525800.3165 ATP66663346-40111774770.37386 COX37804012-47912675130.52425 NAD33544792-5145642870.22252 NAD517285146-687344012550.35934 NAD413206874-81933249900.33796 NAD4L2888194-8481492240.21176 NAD65078482-8988934020.23315 CYTB11258989-101134416840.64569 NAD191510114-110282856300.45493 Total110281-11028335575260.456029
注:C代表保守位点;V代表可变位点;Pi代表信息位点;
表3 13蛋白编码基因联合序列碱基组成
Table 3 Nucleotide composition of the combined sequence of the 13 protein coding gene
SpeciesT(U)%C%A%G%Total ength Bambusiphaga furca42.4012.5834.7510.279147 Bambusiphaga taibaishana43.0412.0135.459.499147 Changeondelphax velitchkovskyi42.8614.0631.6111.4710805 Chloriona tateyamana41.8115.0330.8812.2810806 Cicadella viridis44.7911.2332.0211.9510799 Cicadetta abscondita44.4110.2933.8011.4910875 Epeurysa nawaii43.4512.9732.6710.9110824 Ishiharodelphax matsuyamensis44.2312.5532.2910.9310809 Laodelphax striatellus43.4113.2532.3111.0310752 Nilaparvata bakeri44.0612.7232.5410.6910677 Nilaparvata lugens43.9912.8431.9511.2210677 Nilaparvata muiri43.3913.3332.0111.2610680 Peregrinus maidis43.6013.1032.1411.1610809 Perkinsiella saccharicida44.3012.4032.5810.7210809 Saccharosydne procerus45.3011.1033.869.7410806 Sogatella furcifera43.5613.6430.8811.9110803 Sogatella kolophon43.7712.8732.2111.1510809 Sogatella vibix43.8213.2031.5411.4410809 Tropidocephala brunnipennis44.7811.4434.119.6710815 Ugyops sp42.1212.5534.2811.0610827 Avg.43.6712.6632.6611.0110624.25
表4 13蛋白编码基因联合序列碱基配对频率
Table 4 Nucleotide pair frequencies of the combined sequence of the 13 protein coding gene
iisisvRTTTCTATGCCCACGAAAGGGTotal Avg757998218560.503486601127422484727683244338180210417.2
注:ii = Identical Pairs,代表相同碱基对;si = Transitional Pairs,代表转换碱基对;sv = Transversional Pairs,代表颠换碱基对;R =si/sv,代表转换/颠换比
2.2 系统发育分析
基于线粒体13蛋白编码基因联合序列分别采用距离法、最大似然法和贝叶斯推断法构建了飞虱科13属18种的系统发育树(见图1-3)。三种方法得到的系统发育树结构几乎完全一致,均能把相同属的物种或近缘种聚在一起,仅在部分分支的支持率上有所差别。研究结果表明在飞虱科13属中,属较为原始,处于系统树的基部,其余12个属分别聚为2支,属、属、属和属聚为一支,属、属、属、属、属、属、属和属聚为另一支。
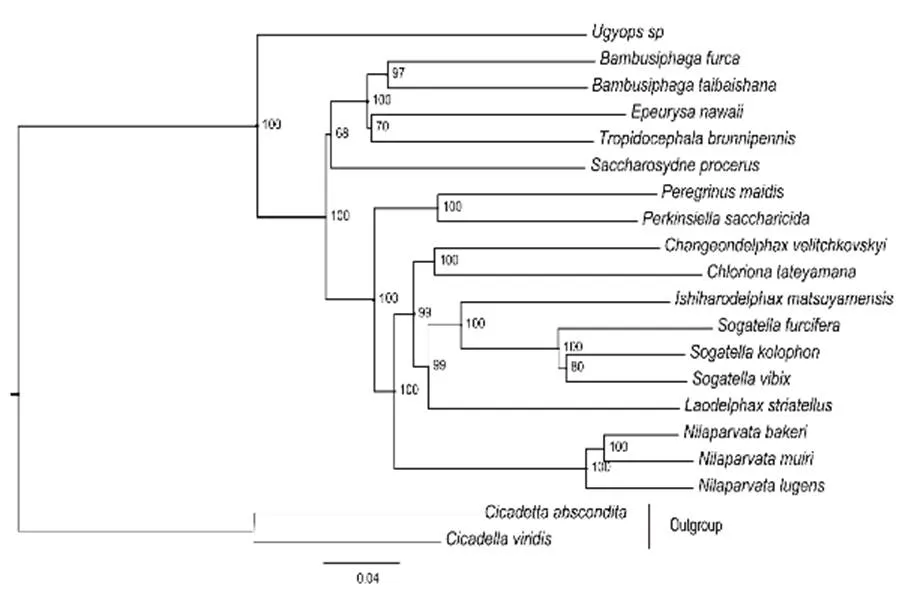
图1 基于13蛋白编码基因构建的邻接法系统发育树
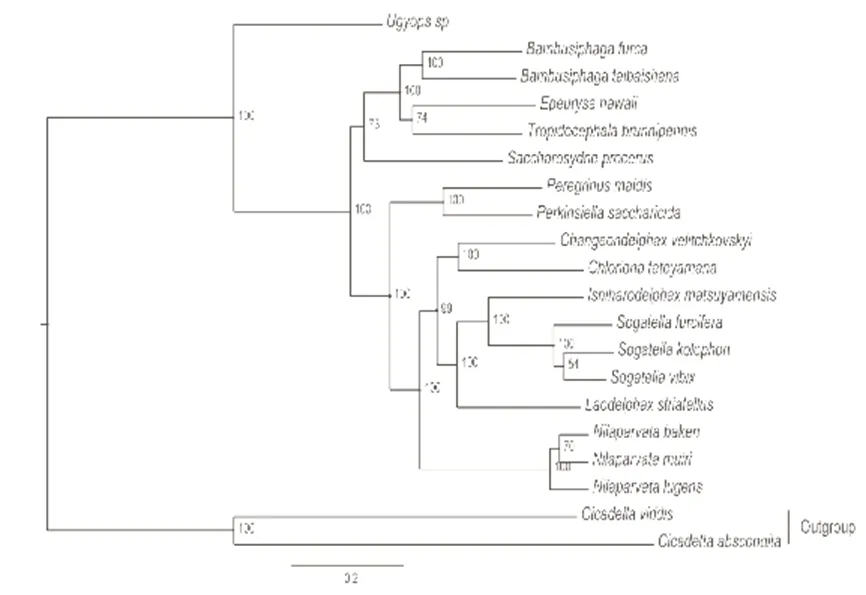
图2 基于13蛋白编码基因构建的最大似然法系统发育树
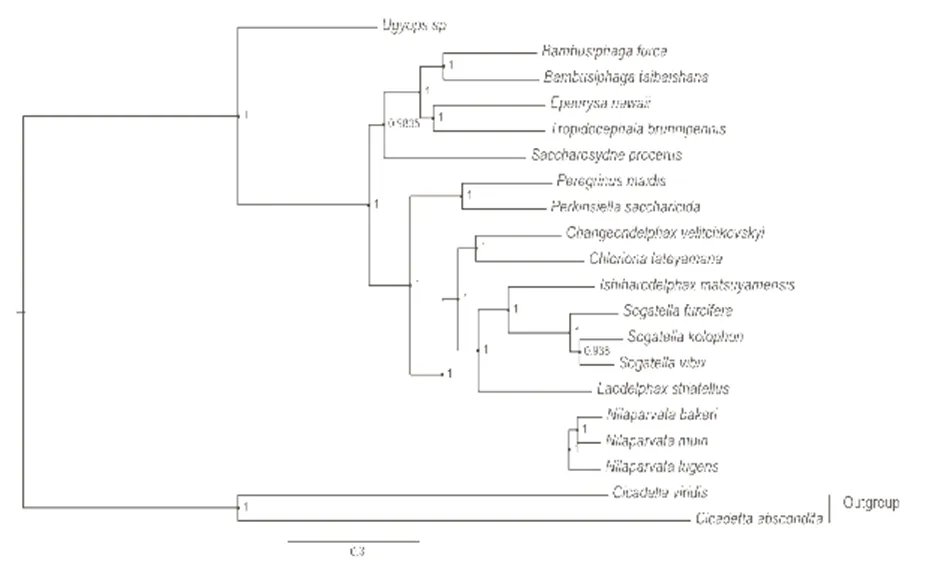
图3 基于13蛋白编码基因构建的贝叶斯系统发育树
3 讨论
通过对昆虫遗传物质的研究,可以探讨不同分类水平上昆虫类群之间的系统发育关系、进化机制和种群遗传变异及分化。目前用于昆虫系统学研究的主要方法有核酸序列分析(DNA sequence analysis)、限制性片段长度多态性分析(restriction fragment length polymorphism, RFLP)、分子杂交技术(molecular hybridization)、随机扩增DNA多态性分析(random amplified polymorphic DNA, RAPD)、单链构象多态性(single-strand conformational polymorphism, SSCP)和双链多态性(double-strand conformational polymorphism, DSCP)分析等。近年来,线粒体基因已成为动物分子系统学研究中应用最为广泛的遗传物质之一。线粒体DNA具有进化速率较核DNA快,遗传过程不发生基因重组、倒位、易位等突变特点,并且遵守严格的母系遗传方式等特点[40]。昆虫的线粒体基因长为15-20 Kb的双链环状DNA分子,包含2个核糖体RNA(rRNA)基因,22个转运RNA(tRNA)基因和13个蛋白编码基因[41]。基于线粒体编码基因开展物种系统发育关系的研究已在节肢动物直翅目[42]、双翅目[43]、鳞翅目[25]44等昆虫以及甲壳类[45]、鱼类[46]和脊椎动物中开展了大量研究,结果表明基于线粒体多基因的联合可以更好的解析物种间系统发育关系。
本研究基于线粒体蛋白编码基因序列构建的飞虱科13属18种的系统发育结果支持属在飞虱科中较为原始,处于系统树的基部,这与Dijkstra, Slotman and Post的结果一致。三种方法获得的系统发育树结构几乎完全一致,均能把相同属的物种或近缘种聚在一起,其中飞虱属先与属、属聚为一支,然后再与属聚为一支,属与属姐妹群,属与属聚为一支,属先与属相聚,再与属聚为一支,这与Park, Xi H, Park J et al.的研究结果一致。基于线粒体编码基因得到的飞虱科种属间系统发育关系符合飞虱科传统分类结果,进一步验证了线粒体编码基因在飞虱科系统发育研究中的应用价值。然而,由于飞虱科物种多样性极其丰富,本研究仅包含了少量飞虱科物种,验证线粒体蛋白编码基因用于飞虱系统发育研究的可行性,若要解决整个飞虱科的系统进化关系则需要更多飞虱物种分子数据的支持。
[1] Bourgoin T. FLOW (Fulgromorpha Lists on The Web): a world knowledge base dedicated to Fulgoromorpha. Version 8, updated [EB/OL]. https://hemiptera-databases.org/flow/,2021.
[2] Wilson S W, L B O’Brien. A survey of planthopper pests of economically important plants (Homoptera: Fulgoroidea)[A]. M R, Wilson, L R Nault (eds.). Proceedings of the 2nd international workshop on leafhoppers and planthoppers of economic importance[M]: Utah: Brigham Young University, Provo, 1986.
[3] Braithwaite K S, Croft B J, Magarey R C. Progress in identifying the cause of Ramu stunt disease of sugarcane[J]. Proc. Aust. Soc. Sugar Cane Technol, 2007, 29:235-241.
[4] Akira Otuka. Migration of rice planthoppers and their vectored remerging and novel rice viruses in East Asia[J]. Frontiers in Microbiology, 2013, 4(309): 1-11.
[5] Leach W E. Entomology[C]. The Edinburgh Encyclopedia. 1815, 9: 57-172.
[6] Metcalf Z P. General catalogue of the hemiptera. Fascicle IV, Fulgoroidea, Part 3, Araeopidae (Delphacidae)[C]. Smith College, Northhampton, Massachusetts, 1943.
[7] 丁锦华. 我国飞虱分类研究概况[C]. 纪念六足学会创建八十周年,江苏省昆虫学会四十周年论文集粹, 2000: 150-153.
[8] 陈祥盛,李子忠,蒋书楠. 中国飞虱科若虫分类研究Ⅰ高原飞虱属、带背飞虱属和黄脊飞虱属(同翅目:蜡蝉总科)[J]. 山地农业生物学报, 2000(1): 21-24.
[9] 陈祥盛,李子忠,蒋书楠. 中国飞虱科若虫分类研究Ⅱ白背飞虱属(同翅目:蜡蝉总科)[J]. 山地农业生物学报, 2000(2): 108-112.
[10] 陈祥盛,陈仕荣,李子忠. 中国飞虱科若虫分类研究Ⅲ.梯顶飞虱属、匙顶飞虱属及簇角飞虱属[J]. 山地农业生物学报, 2000(4): 252-257.
[11] 郑维斌,陈祥盛. 竹飞虱属(Bambusiphaga)若虫分类研究(Ⅱ)[J]. 贵州农业科学, 2013, 41(3): 50-53.
[12] 郭良珍,梁爱萍,丁锦华,等. 叉飞虱属(同翅目,飞虱科)分类研究[J]. 动物分类学报, 2004(4): 736-741.
[13] 陈祥盛,梁爱萍. 偏角飞虱属分类研究(同翅目,蜡蝉总科,飞虱科)[J]. 动物分类学报, 2005(2):374-378.
[14] Hou X H, Chen X S. Oriental bamboo planthoppers: Two new species of the genus bambusiphaga (Hemiptera:Fulgoroidea:delphacidae)from hainan island,China[J].The Florida Entomologist,2010,93:391-397.
[15] 秦道正.中国帕飞虱属分类研究(半翅目,蜡蝉总科,飞虱科)(英文)[J].动物分类学报, 2005(4):857-858.
[16] 秦道正,张雅林. 隆脊飞虱属分类并记一新种(同翅目,蜡蝉总科,飞虱科)(英文)[J].动物分类学报,2005(2):390-392.
[17] 秦道正,张雅林,丁锦华. 中国竹飞虱属分类研究(半翅目,蜡蝉总科,飞虱科)(英文)[J]. 动物分类学报,2006(1): 148-151.
[18] 秦道正.中国飞虱科二属分类概要及三新种记述(半翅目,蜡蝉总科,飞虱科)(英文)[J].动物分类学报, 2006(2): 392-397.
[19] 李红荣,陈祥盛. 贵州飞虱科昆虫物种多样性及地理分布格局(半翅目,蜡蝉总科,飞虱科)[J]. 山地农业生物学报, 2009, 28(6): 485-491.
[20] 丁锦华.中国动物志昆虫纲(第四十五卷同翅目飞虱科)[M]. 北京:科学出版社, 2006:824.
[21] 侯晓晖,陈祥盛. 利用16S rDNA基因序列进行害竹飞虱(半翅目:飞虱科)的分子鉴定[J]. 林业科学研究, 2013, 26(1): 65-69.
[22] Zhu J J, Jiang F, Wang X H, et al. Genome sequence of the small, brown planthopper, laodelphax striatellus[J]. GigaScience, 2017, 6: 1-12.
[23] Huang Y X, Zheng LF, Bartlett CR, et alResolving phylogenetic relationships of Delphacini and Tropidocephalini (Hemiptera: Delphacidae: Delphacinae) as inferred from four genetic loci[J]. Scientific Reports, 2017, 7: 3319.
[24] Urban J M, Bartlett C R, Cryan J R. Evolution of Delphacidae (Hemiptera: Fulogoridea): combined-evidence phylogenetics reveals importance of grass host shifts[J]. Systematic Entomology, 2010, 35: 678-691.
[25] Song, Liang Bu. A molecular phylogeny of Hemiptera inferred from mitochondrial genome sequences[J].Plos One, 2012, 7(11): 1-13.
[26] Fang Y, Liang A P. The complete Mitochondrial Genome of(Hemiptera: Delphacidae)[J]. Journal of Insect Science, 2018, 18(3): 25; 1-13.
[27] Huang Y X, Ren F J, Bartlett C R, et al. Contribution to the mitogenome diversity in Delphacinae: Phylogenetic and ecological implications[J]. Genomics, 2020, 112(2): 1363-1370.
[28] Huang Y X, Qin D Z. First mitogenome for the tribe Saccharosydnini (Hemiptera: Delphacidae: Delphacinae) and the phylogeny of three predominant rice planthoppers[J]. European Journal of Entomology, 2018, 115: 242-248.
[29] Huang Y, Qin D Z. The complete mitochondrial genome sequence of the corn planthopper,(Hemiptera: Fulgoroidea)[J]. Mitochondrial DNA Part B, 2017, 2(2): 783-784.
[30] Lu L, Peng X X, Jing S L, et alIntraspecific and Interspecific Variations in the Mitochondrial Genomes of Nilaparvata (Hemiptera: Delphacidae)[J]. Journal of Economic Entomology, 2015, 108(4): 2021-2029.
[31] Huang Y X, Qin D Z. Sequencing and analysis of the complete mitochondrial genome of(Hemiptera: Fulgoroidea)[J]. Mitochondrial DNA Part B, 2018, 3(1): 90-91.
[32] Zhang K J, Zhu W C, Rong X, et al. The complete mitochondrial genome sequence of(Horváth) and a comparative mitogenomic analysis of three predominant rice planthoppers[J]. Gene, 2014, 533(1):100-9.
[33] Yu F, Song Z S. Characterization of the complete mitochondrial genome of(Hemiptera: Delphacidae)[J]. Mitochondrail DNA Part B, 2021, 6:2, 391-392.
[34] Song N, Liang A P. Complete mitochondrial genome of the small brown planthopper,(Delphacidae: Hemiptera), with a novel gene order[J]. Zoological Science, 2009, 26(12): 851-860.
[35] Zhong L K, Li H X, Yu X F, et al. Complete mitochondrial genome sequence of(Hemiptera: Cicadellidae: Cicadellinae)[J]. Mitochondrail DNA Part B, 2019, 4(1): 1297-1288.
[36] Kumar S, Stecher G, Michael L, et al. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms[J]. Molecular Biology and Evolution,2019, 35: 1547-1549.
[37] Nguyen L T, Schmid H A, Von Haeseler A, et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Mol Biol Evol, 2015, 32(1):268–274.
[38] Nylander J A A. MrModeltest v2. Program distributed by the author[M]. Evolutionary Uppsala University:Biology Centre, 2004.
[39] Ronquist F, Teslenko M, P van der Mark, et al. Huelsenbeck. MRBAYES 3.2: Efficient Bayesian phylogenetic inference and model selection across a large model space[J]. Syst. Biol, 2012, 61:539-542.
[40] 王备新,杨莲芳. 线粒体DNA序列特点与昆虫系统学研究[J]. 昆虫知识, 2002, 39(2):88-92.
[41] Boore J L. Animal mitochondrial genomes[J]. Nucleic Acids Research, 1999, 27(8): 1767-1780.
[42] Zhang H L, Huang Y, Lin L L, et al. The phylogeny of the Orthoptera (Insecta) as deduced from mitogenomic gene sequences[J]. Zoological Studies, 2013, 52: 37.
[43] Foster PG, TMP de Oliveira, Bergo E S, et alPhylogeny of Anophelinae using mitochondrial protein coding genes[J]. Royal Society open Science, 2017, 4: 170758.
[44] Han X, He H, Shen H, et al. Comparative mitochondrial genome analysis of(Lepidoptera: Lasiocampidae) and phylogenetic relationship among Lasiocampidae species[J].Plos One,2020,15(5):0232527.
[45] Carapelli A, Liò P, Nardi F, et al. Phylogenetic analysis of mitochondrial protein coding genes confirms the reciprocal paraphyly of Hexapoda and Crustacea[J]. BMC Evolutionary Biology, 2007, 7, (Suppl 2):S8 .
[46] Akhtar T, Ali G, Shafi N, et al. Sequencing and Characterization of mitochondrial protein-coding genes for(Cypriniformes: cyprinidae) with Phylogenetic Consideration[J].BioMed Research International, 2020(2020):1-13.
[47] Zardoya R, Meyer A. Phylogenetic performance of mitochondrial protein-coding genes in resolving relationships among vertebrates[J]. Molecular Biology and Evolution, 1996, 13(7): 933-42.
PHYLOGENTIC ANALYSIS WITHIN 13 GENERA OF DELPHACIDAE BASED ON THE CONCATENATED PROTEIN CODING GENES OF MITOCHONDRIAL GENOMES
*ZHANG Zheng-guang,ZHAO Fang,SU Tian-juan,JIANG Kai
(School of Life Sciences, Jinggangshan University, Ji’an, Jiangxi 343009, China)
Delphacidae, belonging to Fulgoroidea of Hemiptera, is one of the most important agricultural pests in the world. The mitochondrial genome of insects has been proven to be a useful genetic marker for molecular and evolutionary studies. The objective of this study is to infer phylogenetic relationships among 18 species of 13 genera mitochondrial genomes within Delphacidae. The results of phylogenetic trees constructed by distance method, maximum likelihood method and Bayesian method are consistent. Among the 13 genera of planthopper family, Ugyops is relatively primitive and located at the base of the phylogenetic tree. The other 12 genera cluster into two branches respectively.,,andcluster into one branch,,,,,,,, andconverge into another branch. The evolutionary status and classification of 18 species are consistent with the results of traditional morphological classification.
mitochondrial genomes; phylogenetics; Delphacidae
1674-8085(2022)03-0027-07
S433.3
A
10.3969/j.issn.1674-8085.2022.03.005
2021-06-30;
2022-01-24
国家自然科学基金项目(31960219,31460157);江西省自然科学基金面上项目(20171BAB204010)
*张争光(1979-),男,山东济宁人,副教授,博士,主要从事昆虫系统学和分子生态学研究(Email: zhzhg0537@163.com).
