求和自回归移动平均模型与动态回归模型预测产超广谱β-内酰胺酶肺炎克雷伯菌的检出率
2022-05-07杨金兰陈建华刘如品杜秋争荆自伟
王 升,杨金兰,陈 瑞,陈建华,刘如品,杜秋争,荆自伟
1.信阳市中心医院药学部,信阳 464000;2.郑州大学第一附属医院药学部,郑州 450052
肺炎克雷伯菌是一种条件致病菌,在社区和院内广为流行,可引起泌尿系感染、肺部感染、皮肤感染、软组织感染和血流感染[1]。由于抗菌药物的选择压力和不合理使用,肺炎克雷伯菌的耐药菌株有所增加,产超广谱β-内酰胺酶(ESBLs)是肺炎克雷伯菌耐药的主要机制之一[2]。ESBLs阳性菌株可以对氨基糖苷、喹诺酮和头孢菌素等多类抗菌药物耐药。在肠杆菌属中,有超过200个ESBLs表型已被确认[3],其中CTX-M、TEM 和SHV 最为常见[4]。这些耐药基因通常存在于质粒等移动元件,使耐药基因可以在不同菌株间传播[5],增加了防控难度。细菌耐药性与抗菌药物的使用存在宏观量化关系,本研究将探究我院产ESBLs肺炎克雷伯菌与常用抗菌药物的相关性,运用单纯求和自回归移动平均(ARIMA)模型和纳入以抗菌药物用药频度(DDDs)为外生变量的动态回归模型预测产ESBLs肺炎克雷伯菌的检出率,比较2个模型的拟合和预测效果,旨在为抗菌药物的合理使用和耐药菌传播的科学防控提供依据。
1 资料与方法
1.1 菌株来源与鉴定
菌株来源于2014年第2季度至2020年第4季度送检至微生物检验室的临床标本,包括血液、尿液、痰液、分泌物和胸腔积液,剔除同一患者分离得到的相同菌株。采用VITEK-2型全自动细菌鉴定仪(法国生物梅里埃公司)进行菌株鉴定。质控菌株为大肠埃希菌ATCC25922和肺炎克雷伯菌ATCC700603,由卫生部临床检验中心提供。采用美国临床实验室标准化协会(CLSI)推荐的双纸片法进行确证试验[6],以对头孢曲松和头孢噻肟耐药者作为产ESBLs菌株的初筛标准。使用头孢他啶、头孢噻肟纸片和头孢他啶克拉维酸钾、头孢噻肟克拉维酸钾复合剂纸片对初筛菌株进行确证试验,对初筛菌株贴头孢噻肟和头孢噻肟克拉维酸纸片,比较加克拉维酸和未加克拉维酸的抑菌圈,增加5 mm 以上判定为产ESBLs菌株。
1.2 DDDs
运用PASS临床药学管理系统调取我院2014年第2季度至2020年第4季度常用抗菌药物DDDs,参考世界卫生组织(WHO)推荐的限定日剂量(DDD)、《新编药物学》(第18版)和抗菌药物药品说明书计算DDDs。DDDs=某抗菌药物年使用总量÷该药DDD 值。
1.3 单纯ARIMA 模型
ARIMA 模型是针对时间序列分析常用的建模方法,其原理是提取和模拟时间序列自身蕴含的内在规律,用以预测未来变化趋势。本研究以2014年第2季度至2019年第4季度我院产ESBLs肺炎克雷伯菌检出率的季度数据为样本数据,形成时间序列。建模方法:(1)序列平稳性检验。采用增广迪基-福勒(ADF)法检验序列是否平稳,假设序列存在单位根,P<0.05时拒绝原假设。(2)序列的纯随机性检验。采用杨-博克斯(Ljung-Box)的LB统计量检验序列是否为白噪声序列,P>0.05时,可认为序列为白噪声序列,分析终止。(3)模型识别。通过计算平稳序列的自相关系数(ACF)和偏自相关系数(PACF)对ARIMA(p,d,q)模型进行定阶,其中p为自回归阶数,d为差分次数,q为移动平均阶数。(4)参数估计。使用条件最小二乘法进行参数估计以确定模型口径。(5)模型检验。使用LB统计量检验模型的显著性。其原理为判断残差序列是否为白噪声序列,P>0.05时,拟合的模型显著成立。
1.4 动态回归模型
动态回归模型的原理是多元回归分析和时间序列分析的有机结合。本研究首先考察产ESBLs肺炎克雷伯菌检出率与抗菌药物DDDs的线性相关性,将与前者相关性显著的抗菌药物DDDs形成时间序列作为自变量并纳入模型,建模方法:(1)考察产ESBLs肺炎克雷伯菌检出率与抗菌药物DDDs的线性相关性,以确定输入序列与相应序列间存在的滞后情况。(2)拟合两者的回归模型并得到残差序列。(3)对残差序列进行平稳性检验。残差序列平稳时,说明输入序列与响应序列间存在协整关系。(4)拟合协整动态回归模型。计算残差ACF和PACF系数,对残差序列拟合ARIMA 模型。(5)模型显著性检验,同单纯ARIMA 模型检验方法。
1.5 单纯ARIMA 模型和动态回归模型的比较
本研究拟从2个方面着手比较2种模型的优劣。首先采用最小信息量(AIC)准则评价模型的拟合精度。一个好的模型应综合考虑拟合精度和未知参数个数,而AIC准则是两者的加权函数,AIC值最小的模型通常被认为是最优模型。再者,分别运用2个模型做4期预测,即2020年第1季度至2020年第4季度产ESBLs肺炎克雷伯菌株的检出率,与实际检出率进行比较。并以绝对误差、相对误差和平均相对误差为指标比较2种模型的预测精度。
1.6 统计学方法
用Excel 2013建立样本数据。用线性回归方法分析产ESBLs肺炎克雷伯菌与抗菌药物DDDs的变化趋势。回归系数β>0时,可认为时序数据呈上升趋势。β<0时,可认为呈下降趋势。用Pearson线性相关法分析产ESBLs肺炎克雷伯菌与抗菌药物DDDs的相关性。相关系数r>0时,两者呈正相关。r<0时,两者呈负相关。使用R 3.3.0软件建立ARIMA模型和动态回归模型并预测,所涉及的程序包有tseries、TSA和forecast。检验水准均设定为α=0.05。
2 结果
2.1 产ESBLs肺炎克雷伯菌检出率及与抗菌药物DDDs的相关性
2014年第2季度至2019年第4季度我院ESBLs肺炎克雷伯菌分离率呈整体平稳趋势,2015年第4季度最高,达到42.50%,随后呈快速下降趋势,至2017年第3季度开始反弹,2018年第2 季度又达到高峰值(45.00%),随后快速下降至较低值,随时间整体呈下降趋势(β=-0.91,P=0.01)。分析我院常用抗菌药物DDDs发现,头孢曲松、头孢他啶、头孢唑肟、莫西沙星和头孢哌酮舒巴坦DDDs均随时间呈上升趋势(β>0,P<0.05)。左氧氟沙星DDDs随时间呈下降趋势(β=-102.09,P=0.00)。美罗培南和哌拉西林舒巴坦DDDs随时间变化趋势不显著(P>0.05)。相关性分析发现,产ESBLs肺炎克雷伯菌检出率仅与哌拉西林舒巴坦DDDs呈显著正相关(r=0.75,P<0.05),与其他抗菌药物DDDs未见显著相关性(P>0.05)。哌拉西林舒巴坦DDDs 2015年第3季度最高,随后快速下降,较低值维持在2016年第4季度至2017年第1季度。自2017年第1季度又升至高平台期,至2018年第1季度最高,随后缓慢下降至低平台期,2019年全年消耗量都处于较低水平。产ESBLs肺炎克雷伯菌检出率及常用抗菌药物DDDs变化趋势见表1,产ESBLs肺炎克雷伯菌检出率与哌拉西林舒巴坦DDDs时间序列图见图1。

图1 产ESBLs肺炎克雷伯菌检出率与哌拉西林舒巴坦DDDs的时序Fig.1 Temporal sequence of extended spectrumβ -lactamase producing K lebsiella pneumonia and DDDs of piperacillin-sulbactam

表1 近5年我院产ESBLs肺炎克雷伯菌检出率及常用抗菌药物DDDs的变化趋势Tab.1 Changing trend of detection rate of ESBLs producing K lebsiella pneumonia and DDDs of commonly used antibacterials in our hospital in recent 5 years
2.2 对产ESBLs肺炎克雷伯菌检出率建立单纯ARIMA 模型
使用adf.test程序包对序列进行平稳性检验,结果显示,产ESBLs肺炎克雷伯菌检出率为平稳序列(ADF=-3.61,P<0.05)。纯随机性检验发现,序列为非白噪声序列(χ2=5.97,P<0.05)。分析序列的自相关图和偏自相关图发现,偏自相关图1 阶截尾,自相关图1阶截尾,尝试拟合ARIMA(1,0,1)和ARIMA(1,0,0)模型,通过对2个模型进行参数检验和AIC准则筛选最优模型,结果发现,ARIMA(1,0,0)模型AIC值较小,模型参数均显著,故排除ARIMA(1,0,1)模型。对ARIMA(1,0,0)模型的残差进行白噪声检验,结果发现,残差序列为白噪声序列(χ2=1.63,P>0.05),提示序列信息提取完毕,模型显著。自相关图和偏自相关图见图2,ARIMA(1,0,0)模型诊断信息见表2,ARIMA(1,0,0)的拟合效果见图3A。

图2 产ESBLs肺炎克雷伯菌检出率的自相关图和偏自相关图Fig.2 ACF and PACF diagrams of detection rate of extended spectrumβ -lactamase producing K lebsiella pneumonia
2.3 动态回归模型建立
首先对产ESBLs肺炎克雷伯菌检出率与哌拉西林舒巴坦做线性回归分析,提取回归残差序列。使用adf.test程序包对回归残差序列进行平稳性检验,结果显示,残差序列为平稳序列(ADF=-2.20,P<0.05),提示产ESBLs肺炎克雷伯菌检出率与哌拉西林舒巴坦DDDs存在协整关系,可以建立动态回归模型。对残差序列的纯随机性检验,结果发现,序列为非白噪声序列(χ2=4.53,P<0.05)。绘制残差序列的自相关图和偏自相关图发现,两者均存在一阶截尾现象,通过参数显著性检验和AIC准则,最终为残差序列拟定ARIMA(1,0,1)模型,模型参数均显著(P<0.05)。对动态回归模型的残差序列做白噪声检验,结果发现,残差序列为白噪声序列(χ2=0.65,P>0.05)。回归残差序列的自相关图和偏自相关图见图4,动态回归模型诊断信息见表2,动态回归模型拟合效果见图3B。
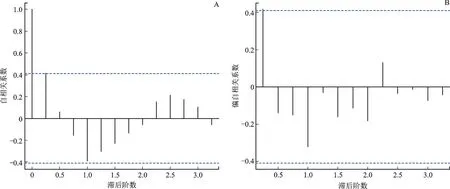
图4 回归残差序列的自相关图和偏自相关图Fig.4 ACF and PACF diagrams of residuals by regression

表2 单纯ARIMA模型与动态回归模型的参数和拟合度比较Tab.2 Comparison of the fitting degree and parameters between unmixed ARIMA model and dynamic regression model
2.4 2种模型预测效果评价
用建立的单纯ARIMA 模型和动态回归模型对我院2020年第1季度至2020年第4季度产ESBLs肺炎克雷伯菌检出率进行预测,将预测结果与同期实际值对比。结果显示,2种模型实际值均在95%可信区间内。2种模型的最大相对误差分别为67.96%、69.40%,均处于预测第4期,动态回归模型的预测相对误差则略小于单纯ARIMA模型。见表3。
3 讨论
耐药菌监测一直都是医院感控工作的重点。全国细菌耐药监测网发布的革兰阴性杆菌检出率的趋势图显示,我国肺炎克雷伯菌的检出率自2010年起跃居第2,仅次于大肠埃希菌。2010~2018年间,肺炎克雷伯菌检出率由14.7%升至20.7%,呈逐年上升趋势。意味着临床需消耗更多的抗菌药物用于治疗肺炎克雷伯菌引起的感染,同时还要应对随之而来的耐药菌株增多等负面效应。我院近5年产ESBLs肺炎克雷伯菌检出率随时间整体呈下降趋势,但并非逐年下降,期间存在反弹现象。同期对比发现,我院产ESBLs肺炎克雷伯菌检出率低于欧阳娜等[7]报道,高于陆彩虹等[8]报道。临床中,碳青霉烯类抗菌药物常用于治疗产ESBLs肠杆菌属引起的严重感染[9]。
近年来,碳青霉烯耐药肺炎克雷伯菌(CRKP)的分离率整体呈快速上升趋势,有研究发现,61.1%的CRKP中携带CTX-M 型ESBLs[10]。据报道,碳青霉烯类抗菌药物的使用与CRKP检出率存在线性相关性[11-12],国家卫健委最新发布的《碳青霉烯类抗菌药物临床应用专家共识》也明确指出,多重耐药菌感染的重症患者才有使用碳青霉烯类抗菌药物的指征,从侧面反映了产ESBLs肺炎克雷伯菌可选药物十分有限的困境,提示应加强对我院产ESBLs肺炎克雷伯菌的监测和预警。
ARIMA 模型是时间序列数据中常用的建模方法,最初应用在经济领域,随着时间的推移,在生物医学领域亦被广泛使用,如流行病的发病率、卫生部门人力资源规划及卫生经费预算等的预测[13]。我院产ESBLs肺炎克雷伯菌分离率形成的时间序列数据符合ARIMA 模型的建模特征和要求,考核样本验证也显示,所建立的ARIMA 模型对产ESBLs肺炎克雷伯菌检出率的4期预测值均在实际值上下小范围浮动,且前3期预测相对误差均小于20%。但所得预测值的平均相对误差要高于储文杰等[14]报道。提示单纯的ARIMA 模型对我院ESBLs肺炎克雷伯菌流行趋势的预测精度仍有提升空间。可能因为影响ESBLs菌株流行的因素较多,仅依靠时间变量很难全面解释ESBLs菌株的变迁。此外,在数据形成的时间段中,如果存在政策性的干预措施通常也会影响时间序列的内在变化规律,使研究对象的惯性趋势改变,从而影响拟合和预测精度,这时则需补充新的时间数据修正模型。
目前,国内外已有多项研究对产ESBLs肺炎克雷伯菌检出率与抗菌药物消耗量间的关系进行探索。徐玉兰等[15]研究表明,产ESBLs肺炎克雷伯菌与抗菌药物使用率和住院抗菌药物使用强度均呈正相关。吕火详等[16]研究显示,含酶抑制剂复合制剂抗菌药物使用量与产ESBLs肺炎克雷伯菌检出率呈正相关。ALDEYAB M A 等[17]研究发现,产ESBLs菌株检出率与阿莫西林克拉维酸钾使用强度呈正相关。说明产ESBLs肺炎克雷伯菌检出率并非仅与某一种或某一类抗菌药物的使用存在相关性。可能因为各医疗机构采购的品种、临床医师的给药方案以及统计学方法的选择不同使得该类研究结果存在差异,提示对于文献报道结论应辩证看待,应结合本医院监测数据来确定产ESBLs肺炎克雷伯菌检出率与抗菌药物间的相关性。本研究以我院常用抗菌药物DDDs为切入点,对两者的相关性进行分析,结果发现,产ESBLs肺炎克雷伯菌检出率与哌拉西林舒巴坦DDDs随时间变化特征基本一致,两者存在显著的线性相关性。随着产酶菌株临床分离率的不断升高,含酶抑制剂的抗菌药物使用更加普遍。哌拉西林舒巴坦是用于治疗产β-内酰胺酶菌株感染的常用药物,应严格根据适应证,仅推荐用于重度感染的治疗[18],警惕此类抗菌药物的使用会带来高产ESBLs菌株的筛选和流行的可能性。
动态回归模型是基于ARIMA 模型演变而来的含有外生变量的模型。国内外多个领域的多项研究运用动态回归模型进行预测,结果大部分研究显示,相较于单纯的ARIMA 模型,动态回归模型能更好地拟合响应序列[19-21]。本研究基于相关性分析结果,筛选出哌拉西林舒巴坦的DDDs数据作为输入序列,对产ESBLs肺炎克雷伯菌检出率建立动态回归模型。结果显示,动态回归模型AIC值低于单纯ARIMA模型,提示动态回归模型的拟合精度优于单纯ARIMA 模型。外推4期预测发现产ESBLs肺炎克雷伯菌检出率实际值均处于动态回归模型95%预测区间内,预测平均相对误差小于单纯ARIMA 模型。此外,通过观察预测值的95%置信区间也可发现,动态回归模型预测值的95%置信区间较单纯ARIMA 模型窄,进一步说明前者预测精度更高。可见引进新的对响应序列具有时间上依存关系的变量作为输入变量,可以提高对响应变量的解释度,优化模型的拟合和预测精度。
综上所述,我院产ESBLs肺炎克雷伯菌的检出率与哌拉西林舒巴坦DDDs存在正相关(P<0.05)。运用单纯ARIMA 模型和动态回归模型对产酶菌株检出率均可进行有效预测。在拟合和预测方面,动态回归模型更具优势。2种模型均存在局限性,不适合做长期预测,在实际运用中应当及时填充样本数据,对模型修正或重新评估。一旦发现实际值超出预测值95%置信区间,应当从手卫生、抗菌药物使用的合理性等方面查找打破模型运行规律的原因,警惕产酶菌株的广泛流行。
