海洋细菌来源β-琼胶酶的生物信息学分析与高效制备
2022-04-22尤钰娴解文嫣班宵逢孔昊存李才明李兆丰
尤钰娴,解文嫣,班宵逢,孔昊存,李才明,李兆丰,
1. 江南大学 食品学院,江苏 无锡 214122
2. 粮食发酵与食品生物制造国家工程研究中心,江苏 无锡 214122
琼胶是由海洋藻类生物合成的一种天然高分子多糖[1-2]。琼胶经过水解后生成聚合度为2~20的琼胶寡糖,不仅溶解性好、生物利用度高,还具有改善水产品储藏性能[3]、抗氧化[4]、抗肿瘤[5]、抗炎症[6]、降血糖[7-8]、改善肠道菌群结构[9-10]以及美白保湿[11]等多项理化和生理功能,在功能性食品、化妆品、药品等多个领域均具有很高的经济价值和广泛的应用前景[12]。
近年我国对琼胶的开发存在规模较小、附加值低、易污染环境等不足,开发功能性琼胶寡糖等高附加值的琼脂衍生物对高效利用海洋资源、增加经济效益、减轻化学污染等意义重大。目前工业上主要利用酸水解[13]和酶解[14]两种方式制备琼胶寡糖,其中利用琼胶酶生物催化水解具有反应条件温和、催化效率高、底物特异性强等优势[15]。琼胶酶根据水解模式和产物的不同可分为α-琼胶酶 (EC 3.2.1.158) 和 β-琼胶酶 (EC 3.2.1.81)[16]。α-琼胶酶可特异性水解α-1,3-糖苷键,生成以3,6-内醚-α-L-半乳糖为还原性末端的琼寡糖,β-琼胶酶可特异性水解β-1,4-糖苷键,生成以β-D-半乳糖为还原性末端的新琼寡糖。β-琼胶酶因来源丰富、稳定性好得以广泛应用,但仍存在菌株产酶性状不稳定、产酶量和催化活性低、产物专一性差等问题,难以满足工业化生产需求。因此,寻找水解活力更高、性状稳定的琼胶酶基因,并在基因工程菌中实现异源表达,成为生产琼胶酶最经济、高效的方法。
目前,已有部分细菌来源的琼胶酶实现了在大肠杆菌 (Escherichia coli)[17-18]、枯草芽孢杆菌(Bacillus subtilis)[19]、短芽孢杆菌 (B. pumilus)[20]、酿酒酵母 (Saccharomyces cerevisiae)[21]和毕赤酵母 (Pichia pastoris)[22]中的表达,其中大肠杆菌因具有生长与表达速度快、易于基因组修饰、表达量高等优势得到最广泛的应用。现有β-琼胶酶在大肠杆菌中的表达量大多高于原始菌株,但绝大多数是胞内分泌表达,不利于后续的分离、纯化和应用,且表达系统的选择多具有随机性,尚未见有结合生物信息学分析等手段指导进行异源表达的报道。本研究利用生物数据库资源挖掘技术和生物信息学分析手段,获得一个来源于海洋细菌——淡黄色噬琼胶菌(Agarivorans gilvus) WH0801的 β-琼胶酶 (β-AGA酶) 基因。通过分子生物学手段和发酵调控策略实现了其在大肠杆菌BL21(DE3)中的高效胞外表达,以期为新琼寡糖资源的高效制备与充分利用提供新途径。
1 材料与方法
1.1 实验材料
大肠杆菌JM109、大肠杆菌BL21(DE3)、质粒pET-20b(+) 保藏于本实验室;pUCm-T simple质粒、限制性内切酶 NcoⅠ和 XhoⅠ、Primer STAR GXL DNA聚合酶、T4 DNA 连接酶、碱性磷酸酶均购自TaKaRa (大连)公司;琼脂糖、质粒小量提取试剂盒、氨苄青霉素等试剂均购自生工生物工程(上海)股份有限公司;分子级酵母粉和胰蛋白胨均购自Oxoid (英国)公司;大豆蛋白胨、酵母提取物 H07014、酵母提取物 H07002均购自上海统园食品技术有限公司;蛋白胨(鱼粉)、牛肉浸膏、酵母浸膏及其他常规培养基均购自国药集团;所用化学试剂均为分析纯[23]。
1.2 实验方法
1.2.1 β-琼胶酶的生物信息学分析
分别以来源于嗜琼胶菌Agarivorans sp. QM38(NCBI登录号:ABM90422.1)[24]、弧菌Vibrio sp.PO-303 (NCBI登录号:BAG71428.1)[25]和火色杆菌Flammeovirga sp. OC4 (NCBI登录号:AJW82061.1)[26]的β-琼胶酶氨基酸序列为探针,利用美国国家生物信息中心(NCBI) 的BLASTp功能 (https://blast.ncbi.nlm.nih.gov/),设置E值为1.0×10-10,长度覆盖率90%以上且同一性大于30%,进行同源性蛋白分子筛选[27],利用MEGA软件中的邻接 (Neighbor-Joining) 法构建系统发生树[28]。
利用在线生物信息学预测工具分别对β-琼胶酶的基本理化性质、分泌特性、结构特征、功能潜力等进行预测分析[29],利用RobeTTAFold在线服务器 (https://robetta.bakerlab.org/) 进行蛋白质三级结构分析和预测。
1.2.2 β-AGA 酶基因的克隆
以淡黄色噬琼胶菌WH0801基因组DNA为模板,根据其中的β-AGA酶序列分别设计含Nco I和Xho I酶切位点的两端引物,通过PCR扩增得到β-aga基因片段[23]。引物设计如下,正向 (P1):5'GCGATGGCCATGGCCACATTTACTAAAAGCAA AATCGCAACCGTTCTT 3' (下划线为Nco I酶切位点));反向 (P2):5'GGTGGTGCTCGAGTTTTT TGTAACGCAGATTATATAGATCACGGTTGAA 3'(下划线为Xho I酶切位点)。
引物由生工生物工程(上海)股份有限公司合成。PCR反应体系及扩增条件参照李兆丰[30]进行。将得到的β-aga基因克隆到质粒pUCm-T simple,获得克隆载体pUCm-T simple/β-aga,并转化大肠杆菌JM109。提取含100 μg·mL-1氨苄青霉素平板筛选所得阳性菌株的质粒进行酶切电泳和测序验证,序列测定由生工生物工程(上海)股份有限公司完成。
1.2.3 表达载体 pET-20b(+)/β-aga的构建和转化
采用限制性内切酶Nco I和Xho I对克隆载体pUCm-T simple/β-aga双酶切,经碱性磷酸酶消化后回收目的片段,再用T4 DNA连接酶与经同样双酶切处理的pET-20b(+)质粒片段连接后转化大肠杆菌JM109,提取质粒并测序鉴定,获得含正确序列的表达载体pET-20b(+)/β-aga,将其转化表达宿主大肠杆菌BL21(DE3),获得基因工程菌E. coli BL21(DE3)(pET-20b(+)/β-aga)。
1.2.4 重组 β-AGA 酶的生产
种子培养和发酵培养参照李兆丰[30]的方法进行,发酵结束后收集上清液即为重组β-AGA酶粗酶液。
1.2.5 发酵条件优化
以体积分数为4%的接种量将种子培养液接至50 mL的发酵培养基中,在不同的发酵初始培养基、诱导剂添加量及添加时间、发酵温度、发酵时间、发酵pH、碳源和氮源条件下进行发酵,测定发酵上清液中重组β-AGA酶活力,以确定最佳的发酵条件,每次优化的结果用于之后的实验。
1.2.6 正交试验
在单因素试验结果的基础上,选择3个影响最大的因素进行正交试验,测定发酵液中重组β-AGA酶活力,获得发酵产酶的最佳条件。
1.2.7 β-AGA 酶水解活力的测定
以质量分数为0.25%的琼脂糖溶液为底物,β-AGA酶的水解活力参照Liu等[31]和Dong等[32]采用3,5-二硝基水杨酸 (DNS) 法测定。
1.2.8 数据处理
2 结果
2.1 β-琼胶酶的生物信息学分析
2.1.1 β-琼胶酶系统进化树的构建
来源于嗜琼胶菌Agarivorans sp. QM38、弧菌Vibrio sp. PO-303和火色杆菌Flammeovirga sp.OC4的β-琼胶酶已被报道具有优良的环境适应力和水解活力,因此以这3个氨基酸序列为探针,利用NCBI数据库中的BLASTp功能进行搜索,保留同源性大于30%的序列,共得到1 039条序列。对筛选出的序列构建系统进化树 (图1),可根据其菌属来源分为11大类,考虑到可获得性和潜在工业价值,最终选取其中与模板序列亲缘关系较近的来源于淡黄色噬琼胶菌的β-琼胶酶(β-AGA酶,NCBI登录号:AQT38174.1) 作为后续研究对象。

图1 β-琼胶酶基因系统进化树Fig. 1 Phylogenetic tree of β-agarase
2.1.2 β-AGA 酶理化性质预测
利用ProtParam预测了β-AGA酶的理化性质。β-AGA酶的氨基酸数量为955个,氨基酸组成见表1,其中含量较多的是丙氨酸(82个,8.6%)、天冬氨酸(81个,8.5%);其中带负电荷的氨基酸残基有135个,带正电荷的氨基酸残基有84个;仅含有1个半胱氨酸,推测可能具有较少的二硫键结构。β-AGA 酶的分子式为 C4 767H7 184N1 238O1 484S21,相对分子量为106.254 kD,理论等电点为 4.66,不稳定指数为24.45,总亲水性平均值为-0.428,表明β-AGA酶为酸性、稳定的亲水性蛋白。

表1 β-AGA酶的氨基酸组成Table 1 Amino acid composition of β-AGAase
2.1.3 β-AGA 酶二级结构分析
利用SOPMA预测了β-AGA酶可能包含的二级结构 (图2),其中303个氨基酸参与构成α-螺旋,30个氨基酸参与构成β-转角,170个氨基酸参与构成延伸链,452个氨基酸参与构成无规则卷曲。

图2 β-AGA酶二级结构预测Fig. 2 Secondary structure prediction of β-AGAase
2.1.4 β-AGA 酶三维结构预测
采用RobeTTAFold对β-AGA酶结构进行在线预测,得到5个预测结构 (图3),置信度均为0.8(满分为1),比对各模型的误差估算图,其中模型2具有相对较小的误差,可以作为基础进行进一步结构解析。

图3 β-AGA酶三级结构预测Fig. 3 Tertiary structure prediction of β-AGAase
2.1.5 β-AGA 酶亲水性/疏水性分析
利用Prot Scale分析了β-AGA酶的亲水性/疏水性 (图4)。β-AGA酶蛋白的最低分值(-3.100)峰区位于第537位脯氨酸,代表其亲水性最强;最高分值(2.422)峰区位于第16位苯丙氨酸,代表其疏水性最强。分析可得,β-AGA酶蛋白的亲水区多于疏水区,总亲水平均值为-0.400,是一种亲水性蛋白。

图4 β-AGA酶亲水性/疏水性预测Fig. 4 Hydropathicity/hydrophobicity prediction of β-AGAase
2.1.6 β-AGA 酶亚细胞定位分析
利用PSORTb version 3.0分析了β-AGA酶的亚细胞定位,结果显示无法准确预测其亚细胞定位。同时利用Loc Tree3分析其亚细胞定位 (图5),结果表明β-AGA酶有一定可能定位在细胞周质中,亚细胞定位分数为36,精确度为89%。

图5 β-AGA酶亚细胞定位预测Fig. 5 Subcellular localization prediction of β-AGAase
2.1.7 β-AGA 酶信号肽分析
利用Signal P 5.0 Server分析了β-AGA酶的信号肽 (图6),可知β-AGA酶的S-mean和Y-max的平均值 (D) 小于临界值,代表不具有信号肽序列,推测β-AGA酶为非分泌性蛋白。

图6 β-AGA酶信号肽分析Fig. 6 Signal peptide prediction of β-AGAase
2.1.8 β-AGA 酶功能结构域分析
利用NCBI的保守域数据库 (CDD) 进行功能结构域分析 (图7)。β-AGA酶在N端48—211、240—424位氨基酸处含有2个Agarase_CBM结构域,该CBM样域在结构上与某些CBM家族非常相似,其中的一个Loop参与了催化位点通道顶部的形成,在β-琼胶酶的外切水解模式中起到了识别底物的作用。这些结构域与琼脂糖链的非还原末端特异性结合,并将附加的催化模块引导至受琼胶酶水解的区域,帮助β-AGA酶捕捉底物,促进参与1,4-β-D-半乳糖苷键水解的催化模块活性。

图7 β-AGA酶功能结构域预测Fig. 7 Conserved domain prediction of β-AGAase
2.2 β-AGA酶大肠杆菌表达系统的构建
β-AGA酶在大肠杆菌中表达载体pET-20b(+)/β-aga的构建见图8。通过双酶切法构建获得分泌型表达载体pET-20b(+)/β-aga。测序验证序列正确性后将表达载体转化宿主大肠杆菌BL21(DE3),获得 E.coli BL21(DE3)(pET-20b(+)/β-aga),实现了β-AGA酶在大肠杆菌中的胞外分泌表达。经测定,发酵所得重组β-AGA酶的初始酶活为2.79 U·mL-1,比酶活为 121.30 U·mg-1。

图8 表达载体pET-20b(+)/β-aga的构建Fig. 8 Construction of expression plasmid pET-20b(+)/β-aga
2.3 重组β-AGA酶的发酵优化
2.3.1 种子生长曲线与种龄的确定
重组β-AGA酶的种子生长曲线见图9-a,菌体在培养前3 h处于延滞期,3~10 h处于对数生长期,10 h后进入稳定期。选择种龄4~9 h的β-AGA酶种子按体积分数为4%的接种量接种,以发酵时间24 h、发酵温度25 ℃的条件进行培养,离心后测定上清液中的胞外酶活力。对数中期 (≥5 h) 的种龄对重组β-AGA酶活力影响不显著,综合考虑时间和经济成本,选择种龄5 h的种子培养液进行发酵 (图9-b)。

图9 重组β-AGA酶的种子生长曲线 (a) 和接种时间对重组β-AGA酶发酵的影响 (b)Fig. 9 Seed growth curve of β-AGAase (a) and effect of inoculation time on fermentation (b) of recombinant β-AGAase
2.3.2 发酵初始培养基的选择
发酵初始培养基对酶活的影响结果见图10。TB培养基对重组β-AGA酶的胞外发酵生产效果最好,胞外酶活力最高,达到3.20 U·mL-1。分析其原因主要为TB培养基营养丰富,更适合菌体生长,且酵母粉含量更高,能够促进重组酶释放到大肠杆菌细胞的周质空间并形成高渗透压便于胞外产酶;同时,TB培养基的pH缓冲能力较强,能够有效维护重组菌的生长。

图10 发酵培养基类型对重组β-AGA酶发酵的影响Fig. 10 Effect of culture medium on fermentation of recombinant β-AGAase
2.3.3 诱导剂对重组 β-AGA 酶生产的影响
诱导剂浓度和添加时间也会在一定程度上影响重组β-AGA酶的生产(图11-a)。当IPTG诱导浓度介于0.005~0.050 mmol·L-1时酶活较稳定,IPTG浓度为0.025 mmol·L-1时酶活最高 (2.589 U·mL-1)。诱导剂添加时间对重组β-AGA酶生产的影响见图11-b,添加诱导剂前培养时间超过0.5 h,酶活均较稳定,培养时间为1 h时酶活最高 (3.96 U·mL-1)。

图11 IPTG浓度和添加时间对重组β-AGA酶发酵的影响Fig. 11 Effects of IPTG concentration and additive time on fermentation of recombinant β-AGAase
2.3.4 发酵时间、温度和 pH 的选择
发酵时间对重组β-AGA酶生产的影响见图12-a,发酵32~64 h其胞外酶活均较稳定,发酵第48小时时酶活达到最高。
发酵温度优化结果见图12-b,重组β-AGA酶活力在20~30 ℃处于较高水平,25 ℃下发酵48 h酶活最高,达到4.37 U·mL-1;超过30 ℃时酶活显著下降。
发酵初始培养基的pH会影响微生物的代谢、发酵及分泌,在pH 7~8.5酶活保持稳定 (图12-c),综合考虑环境和经济因素,选择7.0作为重组β-AGA酶发酵初始pH。

图12 时间、温度和pH对重组β-AGA酶发酵的影响Fig. 12 Effects of time, temperature and initial pH on fermentation of recombinant β-AGAase
2.3.5 碳源和氮源对重组 β-AGA 酶生产的影响
发酵出发培养基优化结果表明,在9种常见的培养基中,TB培养基最有利于重组β-AGA酶的分泌。以此为基础,对其碳源组成进行优化以达到降低成本、提高效率的目的。以去除TB培养基中的碳源为空白,选取并添加与该培养基碳含量相等的果糖、麦芽糖、蔗糖等9种不同的碳源,结果表明碳源的种类和添加量对重组β-AGA酶表达分泌存在较大影响 (图13)。除了葡萄糖,其他碳源在重组β-AGA酶发酵中均能取得比甘油更好的效果,其中果糖作为碳源酶活达到最高 (6.28 U·mL-1)。进一步优化果糖的添加量,当果糖添加量为6 g·L-1时,胞外产酶活力最高 (7.45 U·mL-1)。

图13 碳源种类和添加量对重组β-AGA酶发酵的影响Fig. 13 Effect of carbon sources and their additive amount on fermentation of recombinant β-AGAase
氮源是微生物合成核酸、蛋白质等的重要原料。氮源分为无机氮和有机氮,有机氮更适合大肠杆菌重组菌的发酵生产。以去除TB培养基中的氮源为空白,选取并添加与TB发酵培养基氮含量相等的不同种类的蛋白胨、酵母提取物,发现氮源的种类和添加量对重组β-AGA酶发酵产酶的影响存在明显差异 (表2)。酵母提取物作为氮源时的表达量整体明显优于蛋白胨。其中,使β-AGA酶发酵量达到最高的氮源种类和浓度为添加量30 g·L-1的酵母提取物H07014,此时胞外产酶活力达到15.33 U·mL-1。

表2 有机氮源对重组β-AGA酶发酵的影响Table 2 Effect of organic nitrogen source on fermentation of recombinant β-AGAaseU·mL-1
2.3.6 正交试验
单因素试验结果表明发酵温度、碳源添加量、IPTG添加时间3个因素对重组菌株产酶影响最大,选择这3个因素做三因素三水平正交试验,正交试验设计见表3,试验结果见表4。最佳组合为A2B2C3,即碳源添加量为6 g·L-1,发酵温度为25 ℃,IPTG添加时间为2.0 h,在此条件下发酵48 h后所得粗酶液酶活为16.72 U·mL-1。

表3 正交试验因素和水平表Table 3 Factors and levels in orthogonal array design
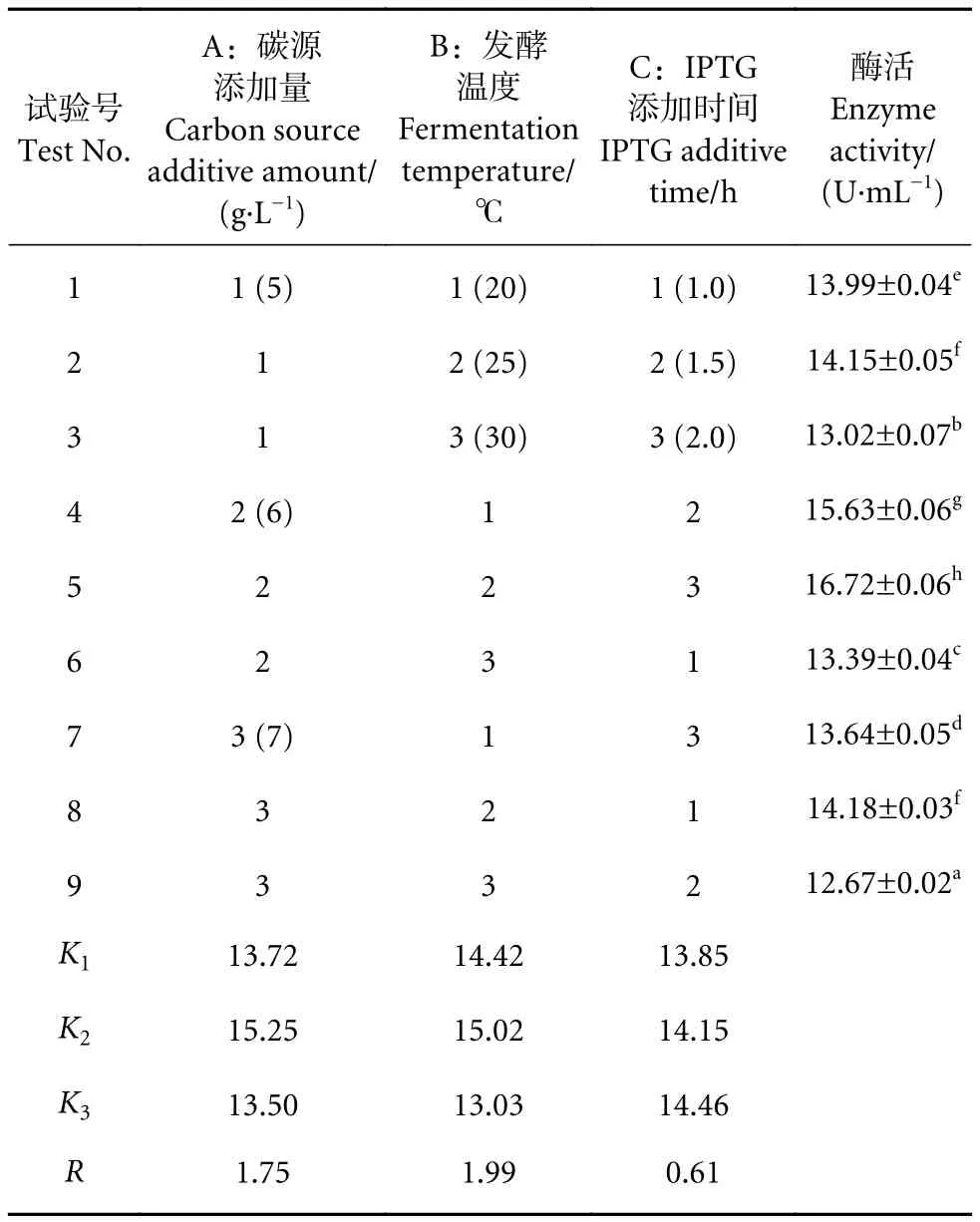
表4 发酵条件优化正交试验结果Table 4 Orthogonal array design layout and experimental results
3 讨论
对琼胶酶进行异源表达可以突破天然酶的分泌限制,利用基因工程菌大幅提高其产量,可实现优良酶制剂的高效制备。目前国内外对琼胶酶在大肠杆菌中的克隆表达主要定位于细胞质或周质空间,存在酶产量有限、制备工艺复杂、分离纯化困难等瓶颈[33],限制了其工业化应用。本研究通过生物信息学分析,发现来源于淡黄色噬琼胶菌的β-AGA酶氨基酸序列中不包含信号肽,因此引入pET-20b(+)质粒中的信号肽进行表达载体的构建,成功实现了重组β-AGA酶的胞外分泌表达。
发酵过程中菌体的生长状态和产酶量可通过多种因素进行系统调控。菌体在生长的不同阶段具备不同的生理特征,处于对数生长期的种子因其活性高、繁殖快、适应性强,更适合用于接种发酵。由于宿主菌E.coliBL21(DE3)中存在由lac I基因调控的T7 RNA聚合酶基因激活机制[34],因此在发酵过程中通常需要添加诱导剂IPTG诱导目的基因的转录。本实验结果表明,IPTG浓度为0.025 mmol·L-1时重组β-AGA酶的水解活力最高,随着诱导剂浓度进一步升高,酶活逐渐降低。这可能是由于IPTG具有一定的细胞毒性,浓度过高时会抑制菌体生长[35],且经高浓度IPTG诱导后重组β-AGA酶的前体蛋白合成速率会大大提高[30,36],容易造成细胞质中无活性包涵体的产生,从而导致正常折叠的蛋白质分泌量显著降低。同时,诱导剂的添加时间也会对重组β-AGA酶的表达量和酶活造成影响,先培养一段时间再添加诱导剂,有利于使菌体增殖到适当的浓度,从而更好地抵御IPTG的毒性,最大限度地诱导细胞产酶。另外,随着发酵时间的延长,菌体在利用营养物质生长繁殖的同时又将更多的代谢产物排出至培养基中,当代谢产物积累到一定量时,会反向阻遏菌体的生产及产酶,因此需要对发酵时间进行严格把控。发酵体系的pH也是影响微生物生长的一个重要因素,适中的pH有利于重组菌的生长、代谢和重组β-AGA酶在发酵期间活力的稳定。除pH外,发酵温度也是调控重组β-AGA酶胞外分泌表达的一项重要指标。本研究结果表明,重组β-AGA酶在25 ℃下发酵活性最高,可能是由于该温度更接近野生型海洋来源β-AGA酶所处的环境温度,此温度下该酶本身稳定性较高,且相对较低的温度也更适合宿主大肠杆菌的生长繁殖和分泌产酶。相反,当发酵温度上升至37 ℃时,宿主菌和重组酶的稳定性均有所下降,在拷贝过程中质粒可能存在较严重的丢失现象,因此胞外酶活显著降低。当诱导温度太低(20 ℃)时,宿主菌的增殖速率和重组酶的转运速率均会降低,对重组β-AGA酶的胞外生产起到一定的限制作用。碳源和氮源的种类和浓度也是影响微生物生长代谢的重要因素,浓度过低时不足以提供充足的营养,过高则会反向抑制微生物的生长和代谢[37],不同微生物对碳、氮源物质的选择偏好性存在明显差异。
综合上述各因素对重组β-AGA酶进行发酵条件优化后的最终条件为:选取37 ℃下培养5 h的种子液用于发酵产酶,以TB作为发酵出发培养基,设置初始pH为7.0,以6 g·L-1的果糖代替其中的碳源,以30 g·L-1的酵母提取液Ⅱ代替其中的氮源,于25 ℃培养2 h后加入终浓度为0.025 mmol·L-1的IPTG,继续诱导48 h,此条件下发酵所得酶活达到16.72 U·mL-1,约为初始酶活的6倍,在目前已报道的β-琼胶酶中处于领先水平[38-40]。优良的水解活力和分泌稳定性使得重组β-AGA酶具有广阔的应用空间,有望成为定向水解琼脂糖,高效生产特异性新琼寡糖的新型酶解工具。
4 结论
本研究利用基因挖掘技术从海洋微生物基因库中筛选得到一个具有优良催化潜力的β-琼胶酶基因,采用分子生物学手段实现了其在大肠杆菌系统中的异源表达,并通过发酵调控策略的优化使其酶活提高了约5倍。该酶具有优良的水解活力、稳定的分泌水平和较高的产物特异性,应用前景广阔。研究结果可为开发新型琼胶水解酶和高效制备不同聚合度的新琼寡糖提供参考。
