CD133+与CD133-人原代胃癌细胞的差异表达基因及核心基因的筛选*
2022-03-31贾岑岑程薇李雷蕾曾晓燕廖永慧谢渊周建奖赵艳
贾岑岑, 程薇, 李雷蕾, 曾晓燕, 廖永慧, 谢渊, 周建奖, 赵艳
(贵州医科大学 地方病与少数民族疾病教育部重点实验室 & 贵州省医学分子生物学重点实验室, 贵州 贵阳 550004)
胃癌是全球第5大常见癌症,亦是死亡率第4位的恶性肿瘤[1]。目前,胃癌具体的发病机制尚未完全明确,其发生发展是多种因素综合的结果,包括幽门螺杆菌感染、遗传因素、饮食习惯、饮酒以及吸烟等[2-4]。随着对肿瘤发病机制的认识和研究不断深入,肿瘤干细胞(cancer stem cells,CSCs)学说对探索新的肿瘤治疗方式具有重要意义[5]。目前已知的大多数 CSC 表面标记都来自已知的胚胎或成人干细胞表面标记,以此来识别癌症组织中CSC[6]。CD133是具有5次跨膜结构的糖蛋白,也是近年来研究较多的肿瘤干细胞标志物。有研究显示,CD133在胃癌中的过度表达与淋巴结转移、化学耐受性以及血管浸润相关,且患者的预后较差[7-11]。CD133在胰腺癌、肺癌、肝癌、脑肿瘤等多种肿瘤组织中均有表达[12-15],且与消化系统恶性肿瘤的发生、发展密切相关。为了研究CD133阳性胃癌干细胞中的关键基因与通路,本研究利用前期分选出的CD133+与CD133-原代胃癌细胞转录组学数据,应用生物信息学分析两者之间的差异表达基因(differentially expressed genes, DEGs),结合Cytoscape 3.8.0、GEPIA、ULCAN和Metascape构建基因的蛋白网络图,筛选关键靶点基因和相关通路,旨在探索参与胃癌发生发展相关的潜在核心基因,为CD133+胃癌干细胞与胃癌的发生发展、诊断、治疗及预后提供新的研究线索。
1 材料与方法
1.1 材料、主要试剂及仪器
CD133+与CD133-人原代胃癌细胞由课题组前期分选鉴定,保存于液氮罐中;胎牛血清、DMEM培养基、双抗、胰蛋白酶购于美国Gibco公司,倒置显微镜购自日本Olympus公司,细胞培养箱购自美国Thermo Scientific公司,超净工作台购于苏州市金净净化设备科技有限公司。
1.2 研究方法
1.2.1数据收集及DEGs筛选 课题组前期通过华大基因科技有限公司对CD133+与CD133-人原代胃癌细胞进行转录组测序,本次研究通过R语言(4.0.4版本)Affy程序包对转录组原始数据进行过滤和标准化处理,再运用limma安装包筛选得到DEGs,以|log2FC|≥1和P<0.05作为筛选标准,对DEGs绘制火山图和聚类热图,直观地展示基因表达情况。
1.2.2基因本体论(gene ontology, GO)和京都基因和基因组数据库 (Kyoto encyclopedia of genes and genomes,KEGG)信号通路富集分析 利用生物信息注释数据库Metascape (http://metascape.org/),以P<0.05为标准,筛选DEGs的GO富集结果和KEGG信号通路分析结果。GO功能富集包含生物过程(biological process, BP)、细胞组成(cellular component, CC)和分子功能(molecular function, MF)3个方面。
1.2.3蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络构建与核心基因的筛选 应用在线数据库STRING(http//string-db.org/),以medium confidence>0. 4为条件来构建CD133+与CD133-人原代胃癌细胞DEGs的蛋白互作网络,并通过Cytoscape3.8.2软件(http://www.cytoscape.org/)将PPI网络可视化,利用CytoHubba插件计算各节点间的数值,并使用MCODE插件从PPI网络中选取最重要的模块进行分析,筛选出核心基因。
1.2.4核心基因与临床患者总生存率的关系分析 利用肿瘤基因组图谱(the cancer genome atlas,TCGA) 数据库,通过在线网站Kaplan-Meier plotter(http://kmplot.com/analysis/)分析核心基因与临床胃癌患者总生存率的关系,筛选条件:(1)癌症,胃癌; (2)基因,见表1;(3)生存期,总生存期;(4)阈值,All; (5)患者,自动选择最佳。评估核心基因在预测胃癌患者预后中的价值。
2 结果
2.1 DEGs的筛选
经R语言limma安装包分析,按照|log2FC|≥1,P<0.05为筛选阈值,在CD133+和CD133-人原代胃癌细胞中共筛选出305个DEGs,其中在CD133+细胞中下调的DEGs 227个,上调的DEGs 78个。见图1。
2.2 GO富集与KEGG通路富集
GO富集以P<0.01,以最小重叠基因=3和最小富集因子>1.5作为阈值,分别对78个上调DEGs和227个下调DEGs进行分析,图2结果显示:上调的DEGs主要参与信号转导受体活性调节、细胞黏附、rho蛋白信号转导调节的生物过程、钙离子通道调节以及钙离子跨膜转运蛋白活性调节等分子功能;下调的DEGs主要参与DNA复制、DNA代谢、ncRNA代谢的生物过程、甲基转移酶活性、单链DNA螺旋酶活性以及乙酰转移酶活性等分子功能。KEGG通路富集发现DEGs主要富集于IL-17信号通路、破骨细胞的分化、RNA运输以及MAPK信号通路等。见图3。
2.3 PPI构建与核心基因的筛选
PPI图中共有187个节点,连接线362个,见图4。应用CytoHubba插件计算各节点间的数值,MCODE插件分析PPI网络中连接最紧密的基因模块,从中选取分数排名前3的模块,见图5;根据度值评分筛选出前20的基因即核心基因,见表1。
2.4 核心基因与胃癌患者总生存率分析
分析20个核心基因与临床胃癌患者总生存率的关系,发现趋化因子8(C-X-Cmotifchemokine8,CXCL8)(HR=0.65)、TCP1伴侣蛋白亚基2(chaperonincontainingTCP1,subunitβ,CCT2) (HR=0.76)、核糖体产物因子 2(ribosomeproductionfactor2,RPF2)(HR=0.77)、蛋白酶体 20S 亚基α4(proteasome20Ssubunitalpha4,PSMA4)(HR=0.56)、胞质伴侣素6A(chaperonincontainingTCP1subunit6A,CCT6A)(HR=0.74)、蛋白酶体 26S 亚基(proteasome26Ssubunit,non-ATPase12,PSMD12)(HR=0.63)以及凝聚素Ⅰ复合物亚基G(non-SMCcondensinIcomplexsubunitG,NCAPG)(HR=0.63)基因表达与总生存率负相关(P<0.05);RUNX 家族转录因子2(RUNXfamilytranscriptionfactor2,RUNX2)(HR=1.51)与总生存率正相关(P<0.05)。图6。
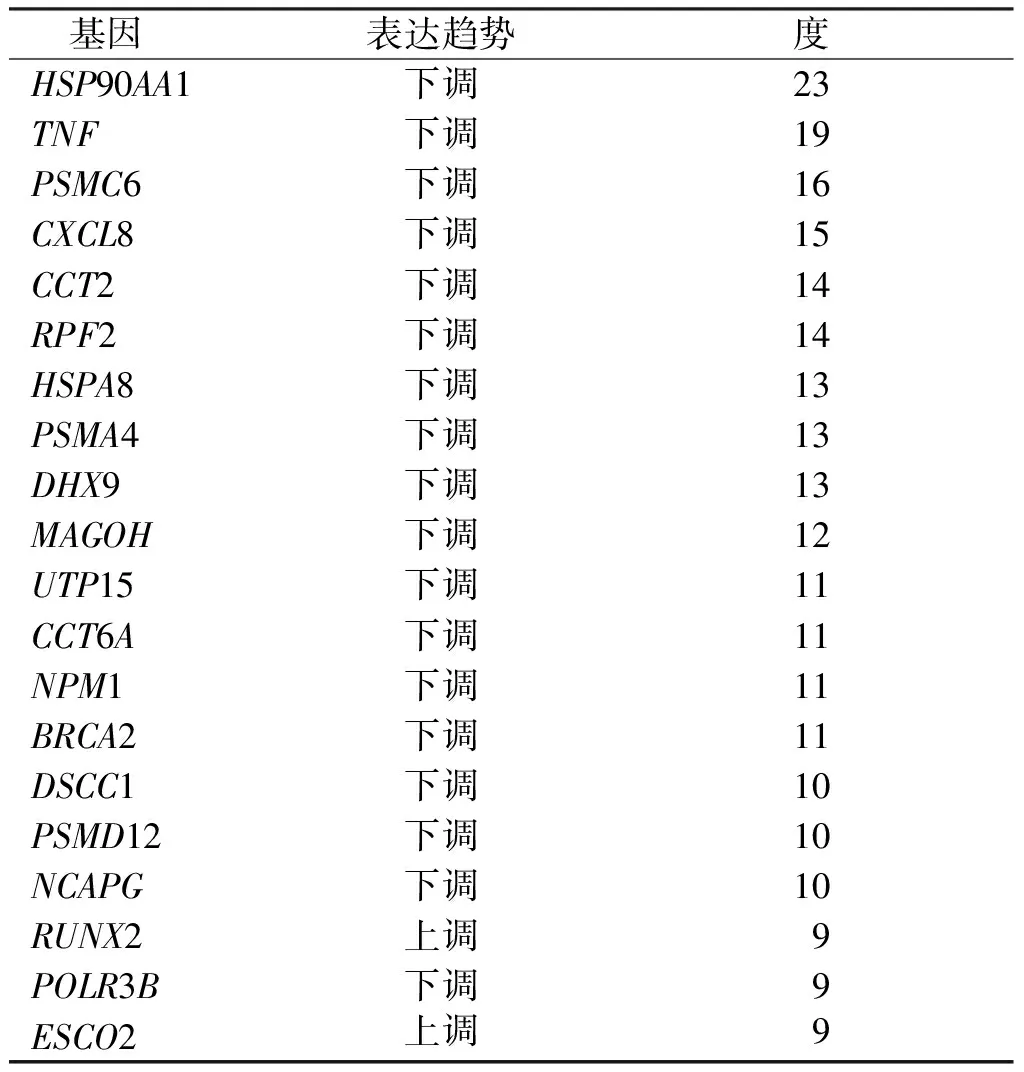
表1 PPI网络中前20的核心基因Tab.1 Top 20 core genes in the PPI network
3 讨论
胃癌仍然是全球癌症死亡的重要原因,死亡率很高。CSC是具有干细胞特征的癌细胞,占整个肿瘤的极少部分,它们具有自我更新和分化能力,被认为是肿瘤复发和转移的起源。研究表明,CD133作为CSC标志物可以在多种实体瘤中调节细胞自我更新和肿瘤发生。因此,进一步基于CD133寻找针对胃癌新的分子标记物,了解胃癌的发展进程和分子机制对于胃癌患者的治疗和预后至关重要。
本研究基于课题组前期分选出的CD133+与CD133-人原代胃癌细胞转录组学数据,筛选出CD133+与CD133-原代胃癌细胞之间的DEGs 305个,其中表达下调的DEGs 227个,上调的DEGs 78个。GO功能富集分析显示DEGs主要参与钙离子通道调节、DNA的复制以及DNA的代谢过程等;KEGG通路富集提示DEGs主要富集于IL-17信号通路、RNA运输以及MAPK信号通路;基于Metascape的GO分析工具,上调的DEGs与信号传导受体活性的调节、细胞粘附以及rho蛋白信号转导的调节、钙离子通道调节及钙离子跨膜转运蛋白活性有关。下调DEGs主要在DNA复制、DNA的代谢过程、ncRNA代谢、甲基转移酶活性、单链DNA螺旋酶活性和乙酰转移酶活性以及细胞内的蛋白络合物富集。通过构建DEGs的PPI网络,并筛选出20个核心基因,其中CCT2、CCT6A、RPF2、PSMA4、PSMD12、NCAPG和RUNX2与胃癌患者的预后相关,以上8个关键基因中除RUNX2在CD133+中上调与总生存率正相关,其余七个基因在CD133+中皆表达下调,且表达与总生存率负相关,提示以上基因与胃癌患者的预后密切相关。
CCT参与细胞中的蛋白质折叠,调节许多肿瘤相关蛋白质的表达和细胞周期,每个环有8个不同的亚基构成,其中 CCT2是CCT的β亚基。既往研究发现CCT2在乳腺癌、胆囊癌、肠癌及肝癌中高表达,且CCT2的表达水平与肿瘤的发生发展、转移以及患者预后密切相关[20-21],CCT2在帮助折叠作用素、管蛋白和其他细胞溶性蛋白方面起关键作用[22-23]。CCT6A则是CCT的第六个亚基的a亚型,CCT6A被发现在调节几种癌症的进展中起重要的作用[24]。 CCT6A能促进非小细胞癌细胞生存和转移,调节肝细胞癌细胞和乳腺癌细胞的细胞周期[25-26]。目前CCT2、CCT6A与胃癌之间的研究较少。RNA结合蛋白在rRNA处理和防止错误折叠rRNA的形成方面起重要作用。RPF2是一个RNA结合蛋白,它能与核糖体合成调控因子1相互作用后影响60S核糖体亚单位的生物合成[27]。目前尚未见RPF2在肿瘤中的研究。PSMA4 是20S蛋白酶核心的α亚基,控制蛋白酶体的活性。已有研究表明PSMA4的缺失会降低蛋白酶体的活性,导致蛋白质的积累,影响转录、细胞周期进展以及凋亡中涉及的蛋白质的降解。因此,若PSMA4缺失则会导致蛋白酶体功能障碍对细胞造成压力[28]。肺癌中PSMA4在癌细胞增殖起着重要的作用,下调会诱发癌细胞凋亡[29],但其在胃癌和其他肿瘤中的作用尚未涉及。PSMD12则是26S蛋白酶复合物的19S调节因子的非ATPase亚基,可以去除错误折叠或受损的蛋白质来调节细胞周期、DNA损伤修复和凋亡。PSMD12在多种肿瘤中高表达并能够促进肿瘤的发生发展,其在乳腺癌中能够促进细胞的生长和迁移并抑制凋亡[30];在胶质瘤中被认为是肿瘤发展和进展的关键调节器,可作为治疗胶质瘤的潜在生物标志物或治疗靶点[31]。目前PSMD12在胃癌中的研究尚未见报道。Runt相关转录因子 (RUNX) 蛋白属于转录因子家族,被称为重要胚胎发育程序的主要调节因子,参与细胞的增殖、分化和细胞凋亡[32]。在哺乳动物中RUNX家族有3个成员,分别是RUNX1、RUNX2和RUNX3,其中RUNX2对成骨至关重要[33]。既往研究发现RUNX2在乳腺癌[34]、肺癌[35]、神经胶质瘤[36]、结直肠癌[37]等多种肿瘤中过度表达并提示预后不良。越来越多的证据表明 RUNX2在肿瘤的发生、进展、侵袭和转移中起着至关重要的作用。RUNX2参与了胃癌的侵袭和转移,与胃癌临床病理参数和患者的预后密切相关[38]。
综上所述, 本研究利用生物信息学筛选出CD133+与CD133-人原代胃癌细胞之间的305个DEGs,成功构建DEGs的PPI网络,筛选出可能参与胃癌发生的20个关键基因,其中CCT2、RPF2、PSMA4、CCT6A、PSMD12、NCAPG和RUNX2与胃癌的预后相关。本研究将为胃癌干细胞的分子机制及胃癌治疗提供新的靶点。
