基于生物信息学分析肝细胞癌潜在的关键基因及miRNA
2022-03-17余春波李大玉李长福
余春波,李大玉,范 芳,李长福
(遵义医科大学 生物化学教研室,贵州 遵义 563099)
肝细胞癌(Hepatocellular carcinoma,HCC)是消化系统最常见的恶性肿瘤之一[1],其发病率和死亡率占全球癌症的第6位和第4位[2],病毒感染、酒精、肥胖等都可能引发HCC的发生,尽管在手术治疗、化疗方面取得一些进展,但肝癌发生的分子机制仍然难以捉摸[3]。由于缺乏有效的治疗靶点,一直以来都是临床治疗阶段的一个难关,随着癌症的基因突变和全基因组研究的开展,许多基因在肿瘤的致癌过程中起作用,这可能对开发有效的肝癌预防和治疗方案有用,因此,筛选及鉴定肝癌发生发展过程中的关键基因和通路,将有助于认识肝癌潜在的发病机制,以期为临床寻找更多的诊断方法、治疗靶点和预后提供一定的参考。
目前,癌症研究已进入基因组学数据测序时代,将更好地了解癌症基因变化,确定潜在的基因突变,进一步发现新的治疗靶点[4],单张数据芯片分析的结果,假阳性率相对较高。本研究从GEO数据库中筛选下载五个数据集,通过对正常肝组织和HCC组织基因芯片中的差异基因进行分析,筛选得到关键基因和信号通路;进一步验证关键基因在表达水平、病理分期关系、患者生存分析和免疫组化分析,这样筛选出来的基因可能更具有临床意义,或将为HCC的诊断和治疗提供新的思路。
1 材料与方法
1.1 芯片来源 在GEO数据库(http://www.ncbi.nlm.nih.gov/)中搜索关键词“Hepatocellular carcinoma”,选择“Homo sapiens、tissue、Expession profiling by array”,在数据集中挑选正常肝组织为对照组,人HCC组织为实验组。3张基因芯片,GSE14520,GSE84598,GSE84402分别有正常肝组织样本19、22、14例,人HCC组织样本有22、22、14例,平台分别为GPL3921、GPL10558、GPL570;2张miRNA数据芯片,GSE98269、GSE57555分别有正常肝组织样本3、5例,人HCC组织样本3、5例,平台分别为GPL20712、GPL18044。
1.2 筛选差异表达基因 利用GEO2R在线分析工具对实验组和对照组数据进行分析,其中以|log2FC(fold change)|≥1.5和P<0.05作为截取标准用于DEGs的筛选,Venn在线制图工具对3组差异表达基因取交集基因,获得3组差异表达基因共同上调、下调的DEGs。
1.3 DEGs的PPI网络构建及关键基因的筛选 将所获得的3组差异表达基因共同上调、下调的DEGs输入STRING数据库(https://string-db.org)构建PPI网络图,随后导入Cytoscape3.7.1软件,利用软件自带MCODE插件进行模块分析,模板筛选标准[5]:自由度截止值=10,Haircut on,Node Score Cutoff =0.2,K-Core=2,及Max.Depth=100,进而用Cytohubba插件对差异基因用常规3种算法(Degree、Closeness、Betweeness)进行筛选,每一种算法所得的前10个基因为关键基因。
1.4 关键基因GO分析及KEGG pathway分析 利用在线DAVID网站(https://david.ncifcrf.gov)对Cytohubba插件筛选出来的关键基因进行GO和KEGG pathway分析。
1.5 关键基因的验证与预后 用GEPIA数据库(http://gepia.cancer-pku.cn)进行在线分析,以|log2FC|>1、P<0.05为标准对筛选得到的关键基因进行表达水平、病理分期关系、患者生存分析等,并用人类蛋白质图谱(http://www.proteinatlas.org)分析关键基因的在HCC组织和正常组织中的免疫组化结果,下载具有代表性的免疫染色图片。
1.6 关键基因的肿瘤免疫细胞浸润分析 利用TIMER在线网站(https://cistrome.shinyapps.io/timer/)检测关键基因在HCC组织中免疫细胞的浸润情况。
1.7 关键基因的miRNA验证 将关键基因输入动物microRNA靶标预测和功能注释数据库(http://mirdb.org/)预测其miRNA,并与2张miRNA数据芯片取DEMs,验证关键基因的miRNA,构建Hub Gene—miRNA网络。
2 结果
2.1 HCC差异基因及miRNA的筛选 3张基因芯片GSE14520、GSE84598、GSE84402分别筛选出DEGs559个(163个上调基因,396个下调基因)、1 021个(450个上调基因,571个下调基因)、857个(403个上调基因,454个下调基因),将这3个芯片数据集取交集,共同表达上调的DEGs为37个(见图1A),共同表达下调的DEGs为127个(见图1B)。
2.2 HCC差异基因的PPI网络构建及关键基因的筛选 将筛选的164个DEGs导入String数据库构建PPI网络(见图2A),再用Cytoscape3.7.1软件的MCODE插件获得2个重要模板(见图2B、C);Hubba插件筛选得到19个Hub基因(见图3)。
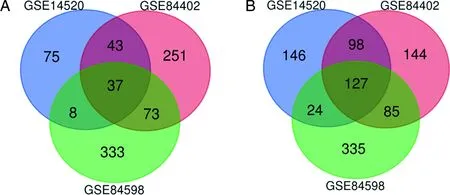
A:上调基因;B:下调基因。图1 HCC组织芯片差异表达基因筛选
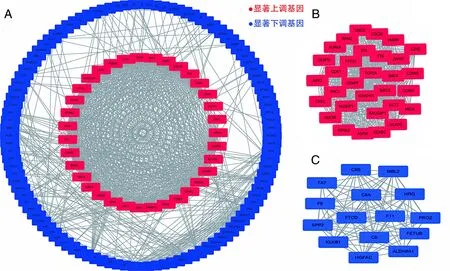
图2 DEGs之间的PPI网络和显著模板分析

A:Degree; B:Closeness; D:Betweennes。图3 3种算法结果排名前10的基因在网络中的相互关系
2.3 HCC关键基因的GO分析和KEGG通路富集结果 通过把19个Hub基因上传到DAVID数据库进行GO分析和KEGG通路富集,差异基因生物学过程集中在细胞周期、有丝分裂过程、视黄酸受体信号通路的调控、对类固醇激素及内源性刺激的反应等(见图4A);分子功能有酶结合功能和蛋白去乙酰化酶结合功能,细胞组成分布在微管、中心体、纺锤体等,信号通路有p53信号通路、卵母细胞减数分裂和细胞周期等(见图4B)。

图4 HCC组织关键基因GO分析和KEGG通路富集
2.4 HCC组织和正常肝组织关键基因表达的验证 通过GEPIA验证Hub基因在HCC组织和正常肝组织的表达结果显示:与正常肝组织比较,有3个Hub基因在HCC组织中低表达,7个Hub基因在HCC组织中高表达(P<0.05,见图5A),且与HPA数据库中免疫组织化学结果相一致,而NDC80和TAT未被HPA数据库收录(见图5B);同时验证Hub基因与HCC患者OS和DFS的关系,结果表明,7个Hub基因高表达组患者的OS情况差于低表达组患者,CYP3A4、ESR1和TAT低表达组患者的OS情况差于高表达组患者(P<0.05,见图6A);7个Hub基因的高表达组患者的DFS情况差低于表达组患者,CYP3A4、ESR1和TAT高表达组患者的DFS情况高于低表达组患者(P<0.05,见图6B);除此之外,这10个Hub基因的表达与HCC临床分期差异显著(P<0.05,见图6C)。
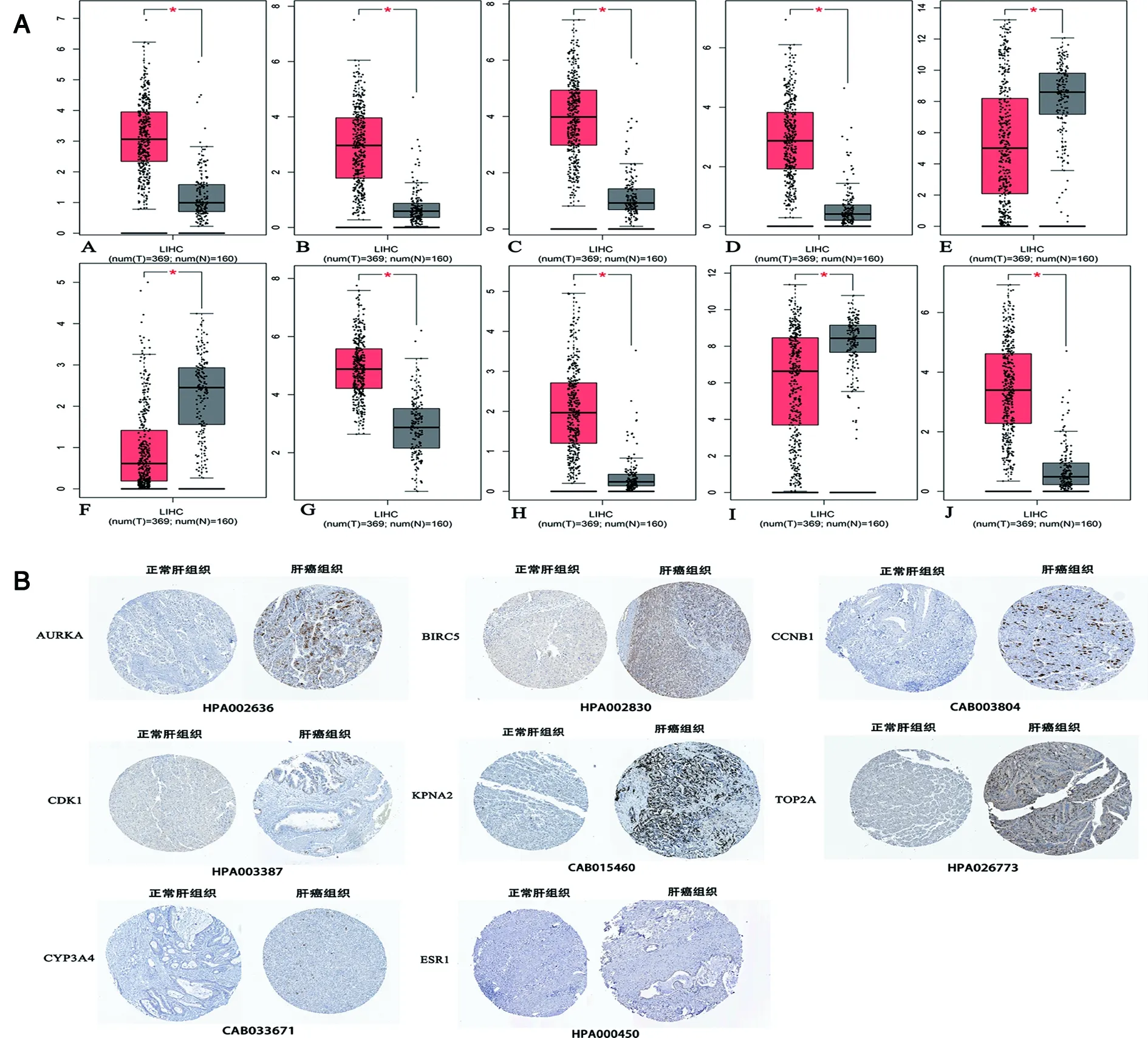
A:AURKA;B:BIRC5;C:CCNB1;D:CDK1;E:CYP3A4;F:ESR1;G:KPNA2;H:NDC80;I:TAT;J:TOP2A。图5 关键基因在HCC组织与正常肝组织中的表达
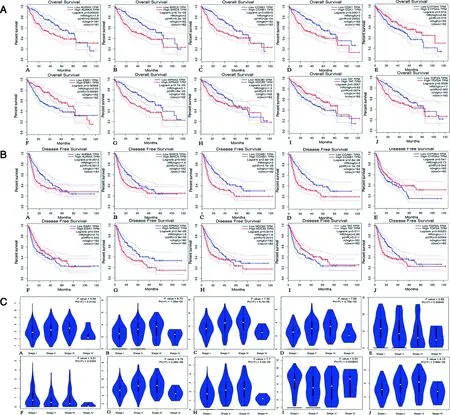
A:AURKA;B:BIRC5;C:CCNB1;D:CDK1;E:CYP3A4;F:ESR1;G:KPNA2;H:NDC80;I:TAT;J:TOP2A。图6 关键基因在HCC组织中表达与患者预后分析
2.5 关键基因的肿瘤免疫细胞浸润分析 通过TIMER数据库对差异表达Hub基因进行检测发现,AURKA、KPNA2、CYP3A4和TAT mRNA在肝癌免疫微环境中与B细胞、中性粒细胞、CD8+T细胞、CD4+T细胞、树突状细胞和巨噬细胞在肝癌组织的免疫微环境中的表达明显相关(P<0.05);而与肿瘤纯度在肝癌组织免疫环境中的表达无确切关系(P>0.05);AURKA、KPNA2 mRNA表达水平与免疫细胞呈正相关,表达量增加;而CYP3A4、TAT mRNA表达水平与免疫细胞呈负相关,表达量下降(见图7)。
BIRC5、CCNB1、CDK1、NDC80和TOP2A mRNA在肝癌免疫微环境中与肿瘤纯度、B细胞、中性粒细胞、CD8+T细胞、CD4+T细胞、树突状细胞和巨噬细胞在肝癌组织的免疫微环境中的表达明显相关(P<0.05);且都与免疫细胞呈正相关,表达量增加(见图7)。
ESR1 mRNA在肝癌免疫微环境中与肿瘤纯度、B细胞和巨噬细胞在肝癌组织的免疫微环境中的表达明显相关(P<0.05);而与中性粒细胞、CD8+T细胞、CD4+T细胞和树突状细胞在肝癌组织免疫环境中的表达无相关性(P>0.05);且都与免疫细胞呈负相关,表达量减少(见图7)。

图7 关键基因与肿瘤免疫细胞间的相关性
2.6 关键基因预测miRNA 将关键基因CDK1、CCNB1、NDC80、TOP2A、KPNA2、BIRC5、AURKA、CYP3A4、TAT、ESR1用miRDB数据库进行在线预测miRNA,发现132个miRNA,与2个miRNA芯片GSE98269、GSE57555取交集,得到共有的hsa-miR-224-5p、hsa-miR-144-3p、hsa-miR-148a-3p、hsa-miR-130a-3p、hsa-miR-22-3p这5个miRNA,并构建 Hub Gene—miRNA网络(见图8)。
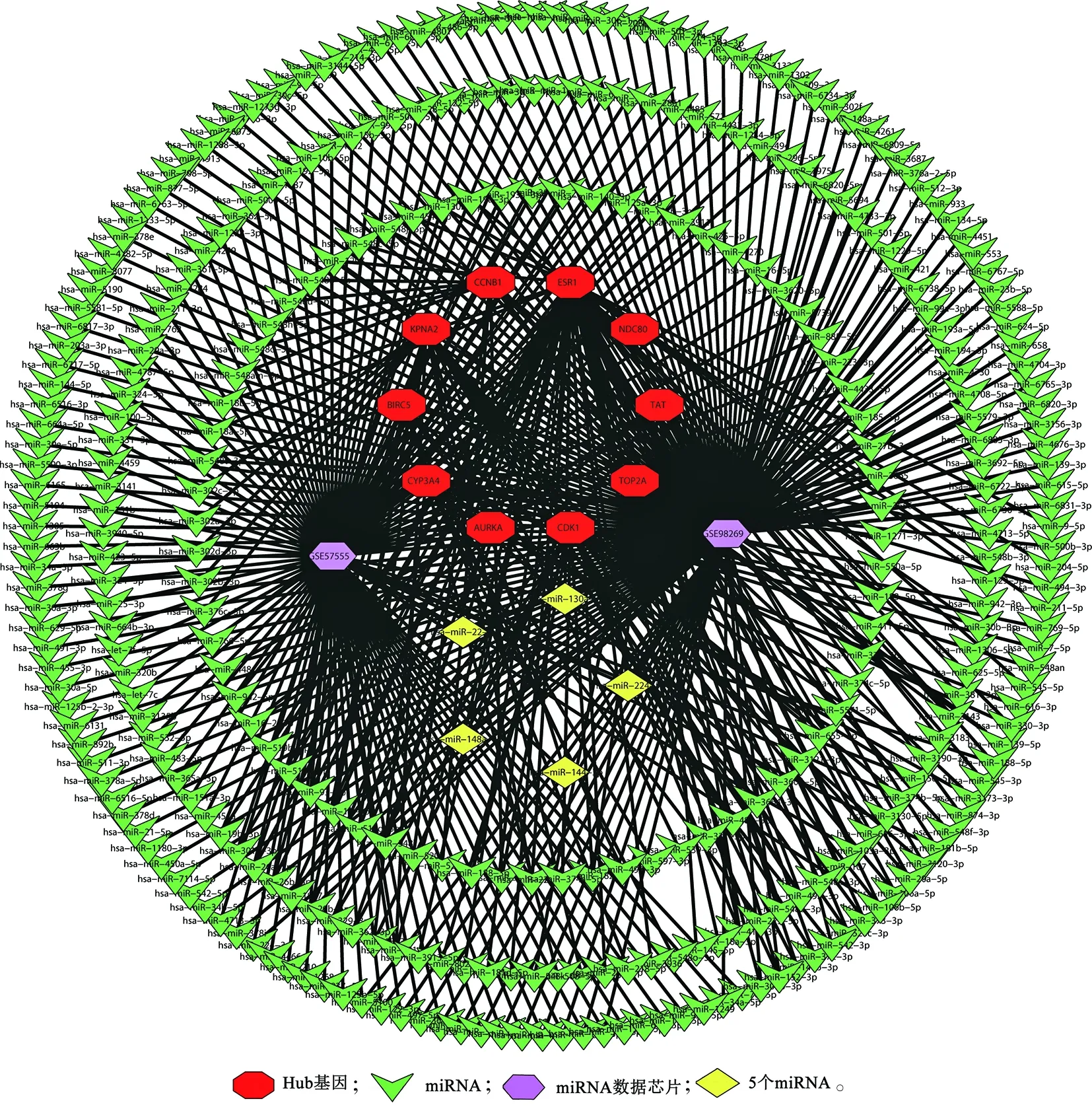
图8 Hub Gene—miRNA网络
3 讨论
肝癌是全球常见癌症之一,其发生发展与转移被认为是一个非常复杂的过程,且HCC的5年生存率仅为18%[6-8],为深入了解其中的分子机制,寻找与HCC密切相关的基因,笔者在GEO数据库检索筛选得到3张基因表达芯片数据集GSE14520、GSE84598和GSE84402,筛选得到164个DEGs并构建PPI网络,Cytoscape3.7.1软件筛选排名前十的Hub基因,并进行GO功能和KEGG通路分析,验证发现7个上调Hub基因在HCC组织中的表达水平明显高与正常肝组织,有3个下调Hub基因在HCC组织中的表达水平低于正常肝组织,与免疫组化结果相一致,进而分析Hub基因与HCC患者OS的和DFS相关,且这10个Hub基因的表达与HCC临床分期相关,提示这些Hub基因具有HCC病理诊断的临床价值,有助于HCC患者的预后评估。进一步探讨Hub基因与肿瘤微环境免疫细胞浸润之间的关系,发现Hub基因在HCC组织的免疫微环境中的表达明显相关;与免疫细胞呈现出正相关或负相关,表达量有增加或减少,提示这些Hub基因与HCC肿瘤组织中的免疫细胞的浸润存在相关性。随后将10个Hub基因导入miRDB数据库预测得到miRNA并于2张miRNA数据芯片GSE98269、GSE57555取交集,得到5个miRNA。
由此笔者推测,这10个关键基因CDK1、CCNB1、NDC80、TOP2A、KPNA2、BIRC5、AURKA、CYP3A4、TAT和ESR1可能是肝癌潜在治疗的靶点及预后标志物。目前已证实AURKA是肝癌的一个有效药物靶点,且取得了初步成功,其发挥的原癌基因作用已探究明确[9-10],抑制AURKA可促进黑色素瘤免疫的积累[11],激活p53协同AURKA拮抗可促进免疫介导的肿瘤清除[5]。TOP2A在肝癌中参与p53途径、癌症途径和细胞凋亡信号传导途径等几种与癌症相关的关键途径,发挥原癌基因作用,是肝癌靶向治疗中的一个重要靶点[12],且介导的DNA分裂与肿瘤炎症微环境相关,TOP2A是HCC发生发展中重要的基因[13]。CDK1与HCC中的免疫细胞浸润有关[14],抑制CDK1可防止免疫介导的远端肿瘤破坏[15]。而KEGG分析发现Hub基因富集在p53信号通路,p53信号通路异常能促进肝癌恶性增殖,并参与HCC的发生,p53信号通路关键基因p53在正常细胞和癌细胞的代谢中起到重要作用[16],并与肿瘤免疫、炎症微环境相关,由此笔者推测AURKA、TOP2A和CDK1和p53信号通路可能与肝细胞再生及肿瘤免疫、炎症微环境相关。
NDC80是染色体正常分离所必需的基因,有丝分裂过程中有重要的作用,异常表达会造成染色体的异常分离,致使染色体不稳定,最终导致肿瘤的发生[17]。ESR1是HCC的关键基因、诊断和治疗的标志物[18]。CCNB1与HCC免疫细胞侵润相关[14]。KPNA2基因可能是通过影响G2/M期、p53信号通路进而发挥抑制肿瘤细胞增殖、诱导细胞凋亡的作用[19]。KPNA2参与机体免疫应答,在多种肿瘤的发生、发展和预后中发挥重要作用,其高表达能够显著性增加肿瘤的恶性增殖,与肿瘤的恶性程度密切相关[20-22]。BIRC5是肝癌免疫相关的预后基因[23]。CYP450是体内药物转化关键的代谢酶[24]。TAT表达异常让酪氨酸不能有效降解,致使血液及尿液中酪氨酸及其代谢产物明显增加,这种疾病是酪氨酸血症Ⅱ型常染色体隐性遗传病。以上均反映了这个10个关键基因作为肝癌诊断、治疗靶点的潜在可能性。
通过2张miRNA数据芯片GSE98269、GSE 57555筛选得到的差异miRNA,与10个Hub基因导入miRDB数据库预测得到miRNA取交集,得到hsa-miR-224-5p、hsa-miR-144-3p、hsa-miR-148a-3p、hsa-miR-130a-3p、hsa-miR-22-3p这5个miRNA,有研究发现,hsa-miR-224-5p被认为是肝细胞癌的最佳生物标志物[25],在对乙酰氨基酚诱导的人肝毒性中起到自我保护的作用[26]。hsa-miR-144-3p在肝细胞癌患者血清中细胞外囊泡明显升高[27]。酒精性肝炎疾病中hsa-miR-148a-3p介导CYP2B6下调[28],hsa-miR-148a-3p通过AGO1依赖性方式在对照和乙醇暴露的肝细胞中促进ADH4表达[29]。miR-130a-3p控制多种癌细胞中的细胞生长,迁移和侵袭[30],miR-130a-3p通过抑制吉西他滨代谢肝癌细胞中的Smad4调节细胞迁移和侵袭[30]。小檗碱抑制HepG2细胞生长并上调miR-22-3p表达[31]。梓醇通过调节miR-22-3p/MTA3信号抑制HCC的细胞增殖、侵袭和迁移[32]。以上均反映了这个5个miRNA在肝癌的诊断、治疗靶点的潜在可能性。
综上所述,对GEO数据库中筛选的3个基因芯片和2个miRNA数据芯片进行生物信息学分析发现,HCC发生的关键基因可能是CCNB1、NDC80、CDK1、TOP2A、KPNA2、BIRC5、AURKA、CYP3A4、TAT和ESR1,重要的信号通路可能是卵母细胞减数分裂和p53信号通路,hsa-miR-224-5p、hsa-miR-144-3p、hsa-miR-148a-3p、hsa-miR-130a-3p、hsa-miR-22-3p这5个miRNA可能是HCC关键的miRNA,为HCC的分子机制研究和预后判断提供新靶点,对肝癌的早期诊断和个体化治疗,为后续的实验研究提供指导。
