斑地锦RT-qPCR内参基因的筛选
2022-03-16宋美玲黄胜和陈祖杰邹嘉轩刘欢胜全文军
宋美玲 黄胜和 陈祖杰 邹嘉轩 刘欢胜 全文军



摘 要: 實时荧光定量PCR(RT-qPCR)的前提条件之一是具有合适的内参基因。为筛选斑地锦(Euphorbia maculata)合适的RT-qPCR内参基因,该文利用同源克隆法克隆斑地锦GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP等基因片段,RT-qPCR检测7个候选内参基因在斑地锦不同生长期根、茎、叶和果实中的表达情况,并用geNorm、NormFinder和BestKeeper等生物学软件对各候选基因表达稳定性进行评价。结果表明:(1)克隆的GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP基因片段为729、808、753、422、233、656、313 bp,分别编码242、269、250、140、77、218、103个氨基酸,与其他植物相应氨基酸序列的最高同源性均在85%以上。(2)综合3个分析软件分析内参基因表达稳定性得出,表达稳定性排名为UBQ>EF-1α>TUB-α>eIF-4A>GAPDH>CYP>act。因此,可以选取UBQ作为斑地锦RT-qPCR分析的内参基因,用于不同生长期基因组织特异性表达研究。
关键词: 斑地锦, 基因克隆, 内参基因, RT-qPCR
中图分类号: Q943; R286.12
文献标识码: A
文章编号: 1000-3142(2022)02-0340-09
Screening of reference genes for RT-qPCR
in Euphorbia maculata
SONG Meiling1, HUANG Shenghe1*, CHEN Zujie2,
ZOU Jiaxuan3, LIU Huansheng3, QUAN Wenjun3
( 1. Department of Basic Medicine, Jiangxi College of Traditional Chinese Medicine, Fuzhou 344000, Jiangxi, China; 2. Jiangxi Medical College,
Nanchang University, Nanchang 330031, China; 3. Fuzhou Medical College, Nanchang University, Fuzhou 344000, Jiangxi, China )
Abstract: The suitable reference genes is a prerequisite for real-time quantitative PCR (RT-qPCR). In order to find a suitable reference gene for gene expression analysis using RT-qPCR in Euphorbia maculata, GAPDH, EF-1α, act, UBQ, TUB-α, eIF-4A, and CYP gene fragments from roots, stems, leaves and fruits at different growth stages were cloned with the method of homologous cloning. Subsequently, the expression patterns of the seven candidate reference genes were obtained by RT-qPCR in E. maculata, and the expression stability was assessed by geNorm, NormFinder, and BestKeeper. The results were as follows: (1) The fragment sequences of GAPDH, EF-1α, act, UBQ, TUB-α, eIF-4A and CYP contained 729 bp (encoding 242 amino acids), 808 bp (encoding 269 amino acids), 753 bp (encoding 250 amino acids), 422 bp (encoding 140 amino acids), 233 bp (encoding 77 amino acids), 656 bp (encoding 218 amino acids), and 313 bp (encoding 103 amino acids), respectively. And the seven amino acid sequences shared over 85% identity with other GAPDH, EF-1α, act, UBQ, TUB-α, eIF-4A, CYP by BlAST in GenBank. (2) The order of expression stability was UBQ>EF-1α>TUB-α>eIF-4A>GAPDH>CYP>act by geNorm, NormFinder, and BestKeeper. Therefore, UBQ can be selected as a reference gene for RT-qPCR in E. maculata using for gene expression analysis in different plant tissues at different growth stages.
Key words: Euphorbia maculata, gene cloning, reference genes, RT-qPCR
地锦草药材为大戟科大戟属一年生草本植物地锦(Euphorbia humifusa)或斑地锦(E. maculata)的干燥全草,又名鬼见愁、血筋草、奶汁草等,分布于中国江苏、江西、浙江、湖北、河南、河北、新疆和内蒙古等地(中国科学院中国植物志编辑委员会,1997),是中医、维医、蒙医常用药材,具有清热解毒、凉血止血、利湿退黄等功效,主治痢疾、泄泻、咯血、尿血、便血、崩漏、瘡疖痈肿、湿热黄疸等(国家药典委员会,2015),目前已开发有肠炎宁片、三七止血片、泻痢宁片等中成药。斑地锦主要含有黄酮类、萜类、酚酸类和生物碱类等成分,具有抗氧化、抗炎、抗菌、止血、免疫调节等作用(柳润辉等,2001;安惠霞等,2008)。目前,斑地锦研究多聚焦在质量标准控制、药理学作用、化学成分的提取及鉴定等方面(胡建新等,2018;Tian et al., 2019),分子生物学相关研究报道较少。
实时荧光定量PCR是在普通PCR的基础上引入荧光基团,通过相应的荧光信号积聚实时监测整个反应的进程,为未知序列进行定量分析的方法,其特异性强、灵敏度高、重复性好、定量准确(张玉芳等,2014;Wang et al., 2019),已经成为分子生物学中研究基因表达的重要工具之一,而使用合适的内参基因是获得可信定量结果的前提(Shakeel et al., 2018)。在实际应用中,RNA提取、cDNA合成及PCR扩增效率等因素会直接影响实验结果,因此常用表达稳定的内参基因进行校正和标准化(Bustin, 2002),以减少样品之间和样品内部的误差。最近研究结果表明,内参基因不存在绝对通用性,若不经筛选而以一种基因作为任何条件下的内参,得到的结果精确度大幅度降低,甚至错误(Zhu et al., 2019)。另外,单一传统的内参基因有时会对结果精确性产生影响,新内参基因将逐渐取代某些表达稳定性差的传统管家基因(Nguyen et al., 2018),或应用内参基因组合有效地减少基因表达误差(袁伟等,2012),以获得更准确的分析结果。同时,基于基因芯片技术和基因表达数据库的筛选(Liang et al., 2018),也将有助于获得更可靠的内参基因。因此,在分析目标基因表达之前,有必要对候选内参基因进行筛选与评估,而评价内参基因表达稳定性的软件较多,其中geNorm、NormFinder和BestKeeper应用较为广泛(Kiarash et al., 2018;Zhong et al., 2019)。目前,常用的内参基因有act(actin)、GAPDH(glyceraldehyde-3 phosphate dehydrogenase)、EF-1α(elongation factor-1 alpha)、UBQ(ubiquitin)、TUB-α(tubulin alpha)、eIF-4A(eukaryotic translation initiation factor 4A)、CYP(cyclophilin)、18S rRNA(18S ribosomal RNA)等(Haller et al., 2004;Kozera & Rapacz, 2013)。为筛选斑地锦合适的RT-qPCR内参基因,本研究同源克隆斑地锦GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP等7个候选内参基因片段,RT-qPCR检测在斑地锦不同生长期(苗期、花期、果期)根、茎、叶和果实中的表达情况,利用geNorm、NormFinder和BestKeeper等软件对各候选基因表达稳定性进行评价,为斑地锦不同生长期基因组织特异性表达研究奠定基础。
1 材料与方法
1.1 实验材料
斑地锦种子由江西天施康生态中药种植有限公司惠赠,种植于南昌大学抚州医学院实验田,经江西中医药高等专科学校药学系中药教研室鉴定为斑地锦。分别采集种植70 d后的苗期(未开花)、花期(开3朵以上的花且未结果)的根、茎、叶,以及果期(结3个以上的果)的根、茎、叶、果,每个样品皆为3株以上的混合样品,采集后立即置于-80 ℃冰箱备用。
1.2 主要试剂与仪器
植物总RNA提取试剂盒(Spin Column Plant Total RNA Purification Kit)、胶回收试剂盒(SanPrep Column DNA Gel Extraction Kit)、引物合成、测序等,生工生物工程(上海)股份有限公司;反转录试剂盒(PrimeScriptTM RT Reagent Kit with gDNA Eraser)、荧光定量PCR试剂(PrimeScriptTM RT Master Mix)等,购于宝生物工程(大连)有限公司;Pfu酶(TransStart FastPfu DNA Polymerase)、Taq酶、T-载体(pEASY-Blunt Simple Cloning Kit),购于北京全式金生物技术有限公司。
普通PCR仪(T100TM Thermal Cycler)、荧光定量PCR仪(CFX96 Real-Time)等,美国Bio-Rad公司;超微量分光光度计(NanoDrop One),美国Thermo Scientific公司。
1.3 实验方法
1.3.1 总RNA的提取和cDNA的合成 斑地锦不同生长期根、茎、叶、果实总RNA的提取和cDNA的合成皆参照试剂盒说明书操作。总RNA提取后通过琼脂糖凝胶电泳检测RNA的质量,并用超微量分光光度计测定RNA浓度,以保证后续实验的进行。
1.3.2 引物设计 在GenBank中查询相关序列,尤其是同科植物蓖麻(Ricinus communis)、麻疯树(Jatropha curcas)和橡胶树(Hevea brasiliensis)等的GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP的基因序列,比对找出高度保守区段,分别设计克隆斑地锦相关基因的简并引物。后续根据测序结果,利用Primer Premier 5.0 软件分别设计RT-qPCR引物。本实验所用引物见表1。
1.3.3 候选内参基因片段的克隆与分析 PCR反应体系(25 μL):5×GC Buffer 5 μL,dNTPs(各2.5 mmol·L-1)2.5 μL,简并引物F(10 μmol·L-1)、R(10 μmol·L-1)各1 μL,cDNA模板1 μL,Pfu酶(5 U·μL-1)0.3 μL,灭菌ddH2O 14.2 μL。扩增条件:94 ℃ 5 min;94 ℃ 30 s,相应退火温度30 s,72 ℃ 60 s,30个循环;72 ℃ 5 min。PCR产物用1.5%琼脂糖凝胶电泳进行检测,目的片段的回收純化按照试剂盒操作说明进行。将回收的DNA片段连接T-载体后转化大肠杆菌DH5α,菌液PCR鉴定阳性克隆,送测序。所得序列在GenBank中进行BLAST比对。
1.3.4 RT-qPCR引物检测与熔解曲线分析 为检验引物特异性,排除引物二聚体及非特异性扩增产物对结果的影响,用相应引物qF、qR进行普通PCR,然后进行RT-qPCR,再加热RT-qPCR产物从65 ℃到95 ℃,每隔0.5 ℃停留5 s检测1次荧光强度以获取熔解曲线。RT-qPCR反应体系(20 μL):2×SYBR Green qPCR Master Mix 10 μL,引物qF(10 μmol·L-1)、qR(10 μmol·L-1)各0.4 μL,cDNA模板2 μL,灭菌ddH2O 7.2 μL;扩增条件:95 ℃ 5 min;95 ℃ 30 s,相应退火温度30 s,72 ℃ 30 s,40个循环。
1.3.5 候选内参基因的筛选 分别将样品总RNA的反转录产物cDNA作为模板,进行RT-qPCR扩增,geNorm、NormFinder和BestKeeper软件对3个候选内参基因表达稳定性进行评估。将Ct值转化为相对表达量(吴建阳等,2017),按照公式Q=2(Ct min- Ct sample)进行转化,用于geNorm和NormFinder软件分析,于BestKeeper软件中直接输入Ct值进行分析,最后进行综合排名,得出最适合斑地锦不同生长期基因组织特异性表达研究的内参基因。
2 结果与分析
2.1 候选内参基因片段的克隆与分析
以总RNA反转录所得到的cDNA为模板,相应简并引物F、R为引物进行PCR扩增,扩增产物大小符合预期(图1)。对其测序后显示,克隆的GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP基因片段分别为729、808、753、422、233、656、313 bp,分别编码242、269、250、140、77、218、103个氨基酸。将氨基酸序列在GenBank中进行BLAST比对后,GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP分别与莴苣GAPDH(XP023733724)、蓖麻EF-1α(XP002518073)、荔枝act(ADV17460)、山梨猕猴桃UBQ(GFZ13847.1)、大麦TUB-α(CAB76917.1)、玉米eIF-4A(U17979.1)、油桐CYP(ARV78452.1)的同源性为95%、99%、100%、99%、99%、87%、86%。将相应核苷酸序列登录到GenBank,获得登录号EmGAPDH(MT044466)、EmEF-1α(MT044465)、Emactin(MT044464)、EmUBQ(MW815120)、EmeIF-4A(MW815119)、EmTUB-α(MW815118)、EmCYP(MW815117)。
2.2 RT-qPCR引物检测与熔解曲线分析
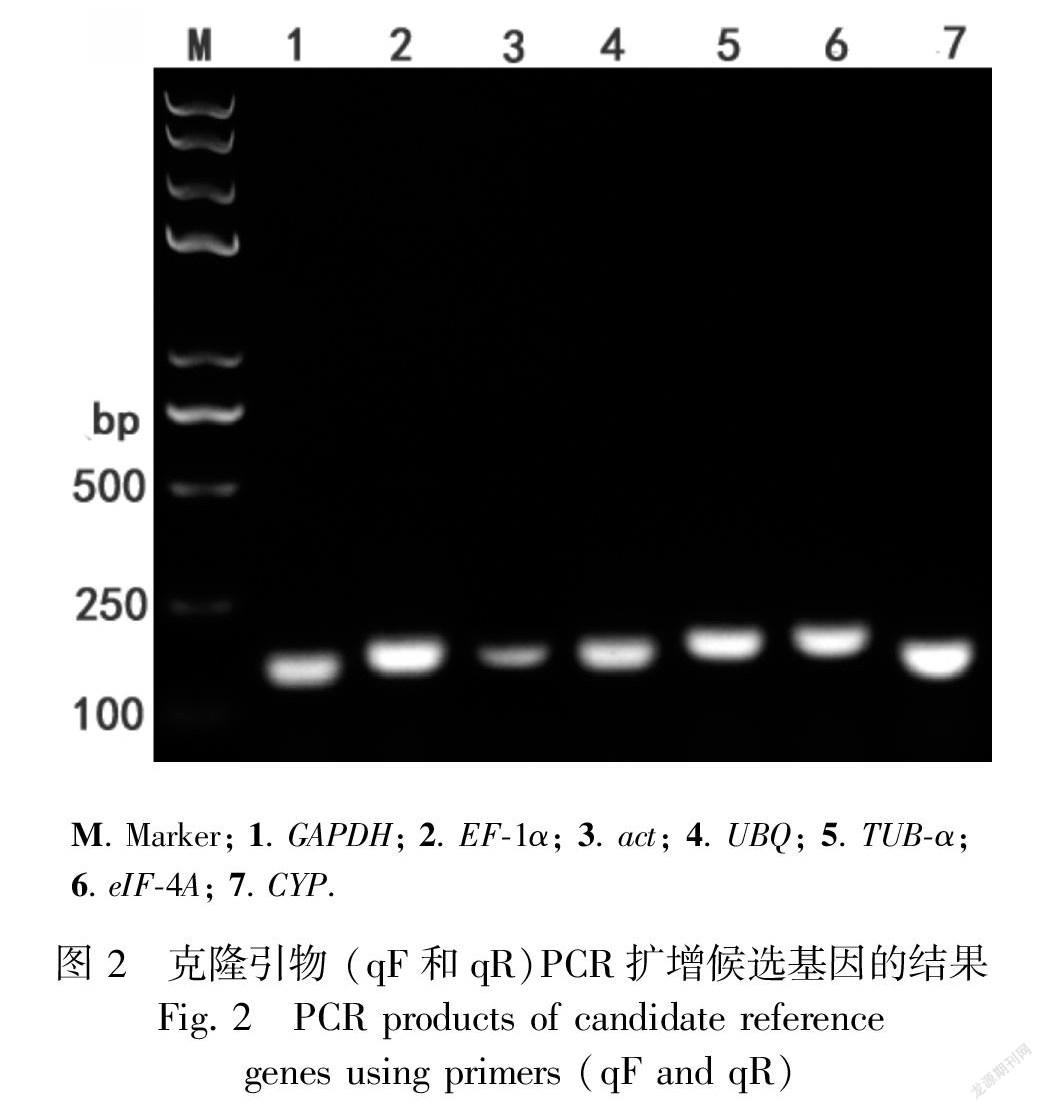
用相应引物qF、qR进行普通PCR扩增,产物长度与预期一致(图2)。将荧光定量RT-PCR 产物进行熔解曲线分析,各基因引物的熔解曲线均显示为单峰(图3)。以上结果表明,本实验所设计定量引物无引物二聚体及非特异性扩增,可用于后续的RT-qPCR分析。
2.3 候选内参基因的Ct值分析
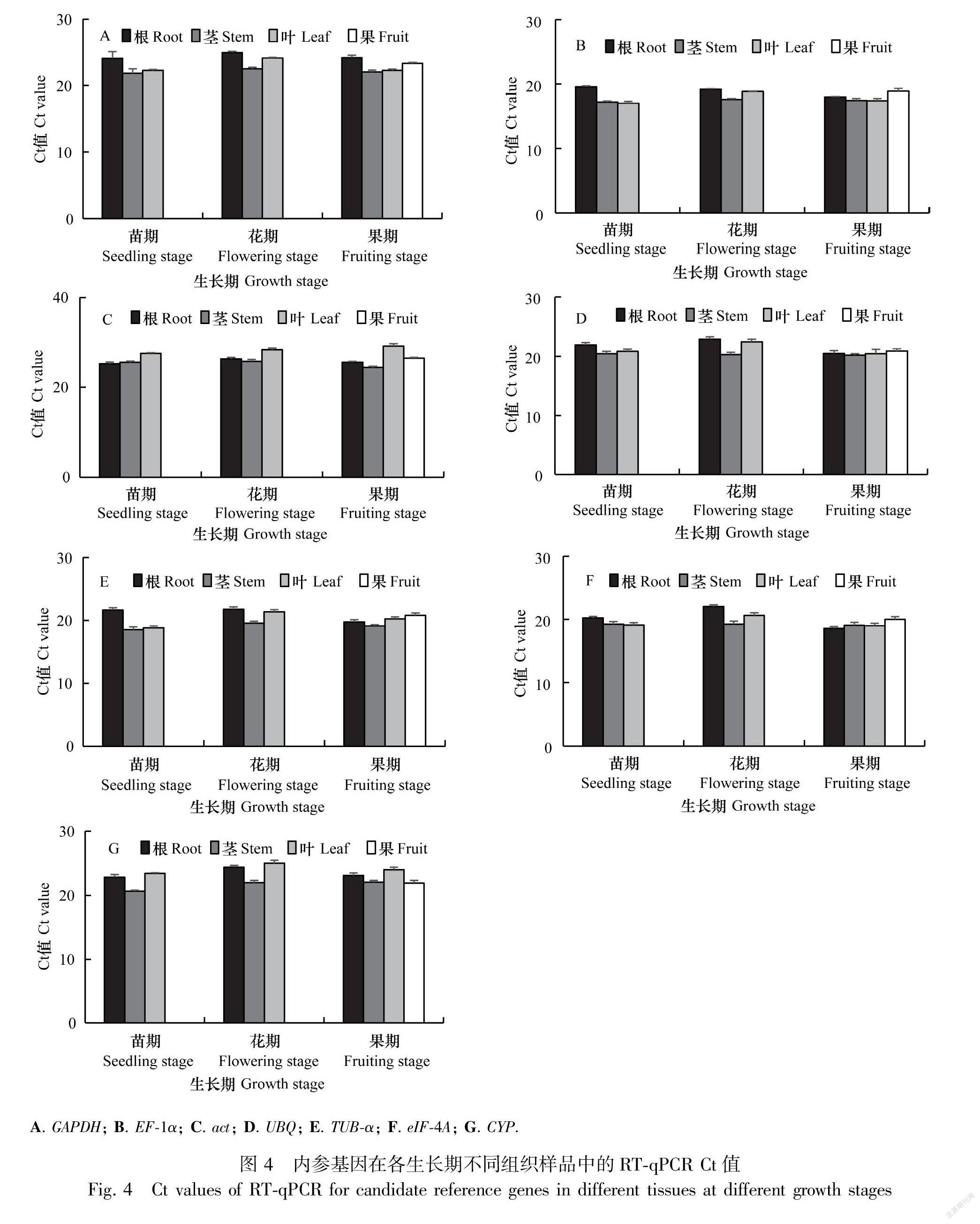
对斑地锦不同生长期各组织(根、茎、叶和果)的cDNA样品进行RT-qPCR扩增,运用Ct值评估各内参基因的表达量,Ct值与其表达量成反比,即Ct值越小,表达量越高。内参基因的表达值排序为EF-1α>TUB-α>eIF-4A>UBQ>CYP>GAPDH>act,Ct值分别为17.04~19.55、18.52~21.81、18.58~22.04、20.19~22.90、20.63~24.99、21.85~24.95、24.44~29.26(图4)。3次重复之间各内参基因的表达量变动较小,且表达量都有所变化。
2.4 候选内参基因表达稳定性分析
2.4.1 geNorm软件分析 geNorm软件通过计算稳定系数M来分析基因表达稳定性,M值越小,稳定性越好。除act外,6个候选内参基因在不同组织中表达的M值都小于1.5,稳定性排名为UBQ>TUB-α>EF-1α>eIF-4A>GAPDH>CYP>act(表2)。
2.4.2 NormFinder软件分析 与geNorm类似,NormFinder软件也是通过各候选内参基因的M值评估其表达稳定性。经NormFinder软件分析,7个内参基因在各组织中的表达稳定程度存在差异,按M 值大小排序为UBQ>TUB-α>EF-1α>eIF-4A>GAPDH>CYP>act(表3)。UBQ的M值最低,总体稳定性最好。
2.4.3 BestKeeper软件分析 BestKeeper直接根据各基因的Ct值,计算标准偏差(standard deviation,SD)和变异系数(coefficient of variation,CV)来评估各内参基因的稳定性。一般来说,稳定的内参基因具有较小的SD值和CV值。BestKeeper分析的基因表达稳定性排名为EF-1α>UBQ>eIF-4A>GAPDH>TUB-α>CYP>act(表4)。其中,EF-1α、UBQ、eIF-4A的SD值和CV值比较相近,表达都相对稳定。
2.4.4 综合分析 由于各内参基因在3个软件中的排序略有差异,故运用几何平均值算法进行各候选内参基因的综合排名,内参基因几何平均值越低,则其稳定性越好。各内参基因在斑地锦不同生长期的不同组织中表达稳定性综合排名为UBQ>EF-1α>TUB-α>eIF-4A>GAPDH>CYP>act(表5)。
3 讨论与结论
地锦草是中医、维医、蒙医常用药材,主要含有黄酮类、萜类、酚酸类和生物碱类等,其中槲皮素含量作为其质量标准,目前,斑地锦的分子生物学相关报道较少。本研究首次克隆了斑地锦GAPDH、EF-1α、act、UBQ、TUB-α、eIF-4A、CYP等常用的传统内参基因片段,并作为候选内参基因进行RT-qPCR,分别用geNorm、NormFinder和BestKeeper评价在各生长期(苗期、花期、果期)根、茎、叶和果实中的表达稳定性。本实验中,各候选内参基因在不同生长期各组织中的表达丰度除act外都较高,Ct值皆在25以下,符合要求。由于3个评估软件采用不同统计学算法,分析结果通常存在差异,需要综合分析得到最适合的内参基因(Kiarash et al., 2018;Zhong et al., 2019),其中geNorm和NormFinder的分析结果较为一致,UBQ、TUB-α、EF-1α、eIF-4A、GAPDH的M值都小于1,比较稳定,最佳内参基因为UBQ,而BestKeeper评价结果与前二者略有差异,EF-1α、UBQ、eIF-4A、GAPDH的SD值小于1,该软件筛选的最优内参基因为EF-1α,而UBQ和eIF-4A表达也比较稳定,与EF-1α并无明显差异。UBQ是泛素蛋白,与蛋白质调节系统有关,参与细胞代谢过程,UBQ是常用的内参基因,在许多植物中得到应用,例如分析芍药花瓣不同发育时期和不同组织的基因表达时可选用UBQ作为内参基因(李健,2017)。EF-1α是转录延伸因子α亚基基因, 在真核生物中参与转录控制、凋亡以及信号转导等一系列重要的生命活动过程,也是较为常用的内参基因,在朱顶红不同组织中表达较稳定(刘晓婷等,2018)。GAPDH、act、TUB-α、eIF-4A、CYP等不是斑地锦各生长期不同组织RT-qPCR的合适内参基因,但在其他一些植物中可能稳定表达,这也证明在不同植物或不同实验条件下进行RT-qPCR有必要对内参基因进行筛选与评估(Kozera & Rapacz, 2013)。因此,若研究斑地锦不同生长期基因组织特异性表达时,可以选取UBQ作为内参基因,如果选择内参基因组合,则UBQ和EF-1α较为合适。此结果为后续斑地锦分子生物学相关研究提供了便利条件。当然,随着斑地锦基因发掘和表达研究的深入,不排除会出現更稳定的内参基因。
参考文献:
AN HX, LI ZJ, GULINA D, et al., 2008. Research advance of the uighur medicine Dijincao [J]. Lishizhen Med Mat Med Res, 19(12): 2866-2868. [安惠霞, 李治建, 古丽娜·达吾提, 等, 2008. 地锦草的研究进展 [J]. 时珍国医国药, 19(12): 2866-2868.]
BUSTIN S, 2002. Quantification of mRNA using real-time reverse transcription PCR RT-PCR: trends and problems [J]. J Mol Endocrinol, 29(1): 23-29.
Chinese Pharmacopoeia Commission, 2015. The Pharmacopoeia of the People’s Republic of China (Vol. I) [M]. Beijing: China Medical Science Press:127. [国家药典委员会, 2015. 中华人民共和国药典(一部) [M]. 北京: 中国医药科技出版社: 127.]
Delectis Florae Reipublicae Popularis Sinicae Agendae Academiae Sinicae Edita, 1997. Flora Reipublicae Popularis Sinicae (Vol. 3) [M]. Beijing: Science Press, 44: 49-50. [中国科学院中国植物志编辑委员会, 1997. 中国植物志(第三分册) [M]. 北京: 科学出版社, 44: 49-50.]
HALLER F, KULLE B, SCHWAGER S, et al., 2004. Equivalence test in quantitative reverse transcription polymerase suitable for normalization chain reaction: Confirmation of reference genes [J]. Anal Biochem, 335(1): 1-9.
HU JX, XI XT, WANG XM, et al., 2018. Mechanism of Humifuse euphorbia in regulating tumor angiogenesis by NF-κB/VEGF signal pathway [J]. Chin J Exp Trad Med Form, 24(20): 167-172. [胡建新, 席晓甜, 王晓敏, 等, 2018. 地锦草调控NF-κB/VEGF信号通路抑制肿瘤血管生成的机制 [J]. 中国实验方剂学杂志, 24(20): 167-172.]
KIARASH JG, WILDE HD, AMIRMAHANI F, et al., 2018. Selection and validation of reference genes for normalization of RT-qPCR gene expression in wheat (Triticum durum L.) under drought and salt stresses [J]. J Genet, 97(5): 1433-1444.
KOZERA B, RAPACZ M, 2013. Reference genes in real-time PCR [J]. J Appl Genet, 54(4): 391-406.
LI J, 2017. Selection and validation of reference genes for quantitative real-time PCR in herbaceous peony [J]. Mol Plant Breed, 15(7): 2544-2549. [李健, 2017. 芍药实时定量PCR内参基因的筛选和验证 [J]. 分子植物育种, 15(7): 2544-2549.]
LIANG WX, ZOU XX, CARBALLAR-LEJARAZU R, et al., 2018. Selection and evaluation of reference genes for RT-qPCR analysis in Euscaphiskonishii Hayata based on transcriptome data [J]. Plant Methods, 14(1): 42.
LIU RH, WANG HB, KONG LY, 2001. Studies on chemical constituents of Euphorbia humifusa [J]. Chin Trad Herb Drugs, 32(2): 107-108. [柳润辉, 王汉波, 孔令义, 2001. 地锦草化学成分的研究 [J]. 中草药, 32(2): 107-108.]
LIU XT, WANG SL, XUE JQ, et al., 2018. Selection of reference genes for quantitative real-Time PCR in different tissue and organ of barbadoslily [J]. Acta Hortic Sin, 45(5): 919-930. [刘晓婷, 王顺利, 薛璟祺, 等, 2018. 朱顶红实时荧光定量PCR中不同组织器官内参基因的筛选 [J]. 园艺学报, 45(5): 919-930.]
NGUYEN N, SUOKAS M, KARPPINEN K, et al., 2018. Recognition of candidate transcription factors related to bilberry fruit ripening by de novo transcriptome and RT-qPCR analyses [J]. Sci Rep, 8(1): 1-12.
SHAKEEL M, RODRIGUEZ A, TAHIR UB, et al., 2018. Gene expression studies of reference genes for quantitative real-time PCR: an overview in insects [J]. Biotechnol Lett, 40(2): 227-236.
TIAN S, WEN E, MI N, et al., 2019. Determination of three active components in Euphorbia humifusa Will. U dsing high-performance liquid chromatography with diode-array detection and autophagy and apoptosis analysis of normal rat kidney and HeLa cells [J]. Pharmacogn Mag, 15(61): 348.
WANG YZ, DAI MS, CAI DY, et al., 2019. Screening for quantitative real-time PCR reference genes with high stable expression using the mRNA-sequencing data for pear [J]. Tree Genet Genom, 15(4): 54.
WU JY, HE B, DU YJ, et al., 2017. Analysis method of systematically evaluating stability of reference genes using geNorm,NormFinder and BestKeeper [J]. Mod Agric Sci Technol, (5): 278-281. [吴建阳, 何冰, 杜玉洁, 等, 2017. 利用geNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法 [J]. 现代农业科技, (5): 278-281.]
YUAN W, WAN HJ, YANG YJ, 2012. Characterization and selection of reference genes for real-time quantitative RT-PCR of plants [J]. Chin Bull Bot, 47(4): 427-436. [袁伟, 万红建, 杨悦俭, 2012. 植物实时荧光定量PCR内参基因的特点及选择 [J]. 植物学报, 47(4): 427-436.]
ZHANG YF, ZHAO LJ, ZENG YL, 2014. Selection and application of reference genes for gene expression studies [J]. Plant Physiol J, 50(8): 1119-1125. [张玉芳, 赵丽娟, 曾幼玲, 2014. 基因表达研究中内参基因的选择与应用 [J]. 植物生理学报, 50(8): 1119-1125.]
ZHONG SL, ZHOU SY, YANG SJ, et al., 2019. Identification of internal control genes for circular RNAs [J]. Biotechnol Lett, 41(10): 1111-1119.
ZHU LF, YANG CQ, YOU YH, et al., 2019. Validation of reference genes for RT-qPCR analysis in peel and flesh of six apple cultivars (Malus domestica) at diverse stages of fruit development [J]. Sci Hortic, 244: 165-171.
(責任编辑 李 莉)
收稿日期: 2021-04-19
基金项目: 江西省教育厅科学技术研究项目(GJJ191230) [Supported by Scientific and Technological Research Program of Jiangxi Education Department (GJJ191230)]。
第一作者: 宋美玲(1989-),硕士,讲师,主要从事药用植物生物技术研究,(E-mail)sml0703@163.com。
*通信作者: 黄胜和,博士,副教授,主要从事药用植物生物技术研究,(E-mail)hsh712@163.com。
2936501186280
