缺氧相关长链非编码RNA作为肝癌预后预测标志物的潜在价值
2022-03-08程敏张静曹鹏博周钢桥1
程敏,张静,曹鹏博,周钢桥1,,3
研究报告
缺氧相关长链非编码RNA作为肝癌预后预测标志物的潜在价值
程敏1,3,张静4,曹鹏博2,周钢桥1,2,3
1. 南京医科大学公共卫生学院流行病学系,南京 211166 2. 军事科学院军事医学研究院辐射医学研究所,蛋白质组学国家重点实验室,国家蛋白质科学中心,北京 100850 3. 南京医科大学全球健康中心,南京 211166 4. 河北大学生命科学学院,保定 071002
肝细胞癌(hepatocellular carcinoma, 简称肝癌)是一种常见的恶性肿瘤。缺氧是肝癌等实体肿瘤的一个重要特征,同时也是诱导肿瘤恶性进展的重要因素。然而,肝癌缺氧相关的长链非编码RNA(long non-coding RNA,lncRNA)的鉴定及其在临床生存预后等方面的价值仍未得到系统的研究。本研究旨在通过肝癌转录组的整合分析鉴定肝癌缺氧相关的lncRNA,并评估其在肝癌预后中的价值。基于癌症基因组图谱(The Cancer Genome Atlas, TCGA)计划的肝癌转录组数据的整合分析,初步鉴定到233个缺氧相关的候选lncRNA。进一步筛选具有预后价值的候选者,基于其中12个缺氧相关lncRNA (、、、、、、、、、、和)建立了肝癌预后风险模型。Cox比例风险回归分析显示,基于该模型计算的缺氧风险评分作为肝癌患者新的独立预后预测指标,优于传统的临床病理因素。基因集富集分析显示,缺氧风险评分反映了细胞增殖相关通路的活化和脂代谢过程的失活。综上所述,本研究构建了一个基于缺氧相关lncRNA的风险评分模型,可以作为肝癌患者预后预测的候选指标,并初步提示了这些缺氧相关的lncRNA在肝癌防治中的重要作用。
肝细胞癌;缺氧;长链非编码RNA;预后模型
肝癌是全球最常见的恶性肿瘤之一,其在男性和女性中的死亡率分别位居第二和第六位[1]。近年来针对肝癌的早期诊断和临床治疗策略均取得很大进展,但肝癌的长期预后仍然较差。因此,建立肝癌复发和转移风险等相关的预后预测模型有利于指导临床治疗。以往研究显示,肿瘤淋巴结转移分期和血管侵犯等传统临床病理指标有助于预测肝癌患者的预后;然而,由于肝癌巨大的异质性,这些传统指标的预测效果仍远不能令人满意。
实体肿瘤微环境中的氧分子含量水平较低,被称为缺氧(hypoxia)。已有大量研究报道,实体肿瘤微环境的缺氧与包括肝癌在内的多种癌症的进展密切相关[2,3]。因此,针对肝癌微环境缺氧相关的分子机制研究受到关注。lncRNA是一种长度超过200个核苷酸、缺乏开放阅读框(open reading frame, ORF)、蛋白质编码能力较弱的RNA。以往多项研究已表明,lncRNA可参与肿瘤发生发展的多种生物学过程[4,5]。近年来,越来越多的与缺氧应答相关的lncRNA被陆续发现。缺氧可以通过缺氧诱导因子(hypoxia- inducible factor 1-alpha,)转录激活多个参与肿瘤发生发展的lncRNA,例如[6]、[7]、[8]、[9]和[10]等。此外,也有大量研究发现lncRNA可以通过调控的表达或活性促进肿瘤的进展。例如,Dong等[11]发现lncRNA可通过参与调控miR-98-5p-STAT3-HIF-1α正反馈环路而促进卵巢癌的增殖、凋亡和转移。lncRNA可通过激活PKM2-HIF-1α糖酵解级联反应促进肿瘤细胞的代谢适应,以增加细胞在葡萄糖饥饿和缺氧应激下的存活能力[12]。然而,目前关于肝癌缺氧相关lncRNA的鉴定及其在临床预后方面的价值仍未得到系统研究。
基于与缺氧相关的转录组表达特征,本研究对TCGA计划(The Cancer Genome Atlas)中的肝癌组织样本进行了缺氧评分,基于此评分系统筛选出233个与缺氧相关的候选lncRNA,继而基于其中12个lncRNA构建了肝癌预后模型,并系统评价了此模型的预测能力。此模型为肝癌患者的预后风险分层、预后评估及指导临床治疗等提供了新的参考指标,具备潜在的应用价值。
1 材料与方法
1.1 研究对象及组学数据
从TCGA数据库(https://portal.gdc.cancer.gov/)下载获得肝癌患者(包括374个肝癌肿瘤组织和50个邻近非肿瘤组织)的mRNA和lncRNA表达谱数据和临床信息。该表达谱数据包括20530个蛋白编码基因和15093个lncRNA基因。基于这些数据集建立了预后风险评分模型,并对367例随访信息完整的患者进行了后续分析。数据集的详细信息见附表1。从基因表达综合数据库(gene expression omnibus, GEO)获得另一个公开的肝癌lncRNA表达谱数据集(编号:GSE40144;包括59例病例)作为独立的验证队列,用以评估建立的预后风险模型。从TCGA数据库下载肝癌的拷贝数变异数据集,从UCSC Xena获取了患者相应的临床信息。
1.2 肝癌中缺氧相关lncRNA的鉴定
首先,收集了文献报道的与肿瘤缺氧相关的蛋白编码基因集[13, 14]。其次,基于TCGA中肝癌的癌组织基因表达数据集,利用基因集变异分析(gene set variation analysis, GSVA)[15]计算每个肿瘤样本的缺氧评分。最后,依据缺氧评分与lncRNA表达水平的Spearman相关性来筛选缺氧相关的候选lncRNA。以相关系数||>0.35和<10–6为标准,共鉴定到233个肝癌缺氧相关的候选lncRNA。
1.3 基于肝癌缺氧相关的lncRNA构建预后风险评分模型
基于TCGA肝癌队列分两步构建预后风险评分模型。首先,对每个候选lncRNA进行单因素Cox回归分析,以筛选与肝癌总体生存期(overall survival, OS)相关的lncRNA,其中<5×10–4的lncRNA作为候选对象。其次,采用多因素Cox分析,以建立基于Akaike信息标准(Akaike information criterion, AIC;AIC = 1314.509)的最佳预后风险模型。最后,根据下列公式计算每个患者的风险评分:风险评分(risk score) = Coef(lncRNA1) × Expr(lncRNA1) + Coef(lncRNA2) × Expr(lncRNA2) + …… + Coef(lncRNAn) × Expr(lncRNAn)。其中,Expr(lncRNAn)代表某一特定lncRNA的表达水平,Coef(lncRNAn)代表lncRNA多因素Cox分析中的系数。以TCGA肝癌队列中患者的总体风险评分的中位数为界,将患者划分为高风险组和低风险组;采用单因素和多因素Cox回归分析评估该风险模型对患者总体生存风险的预测能力。在验证队列中,同样基于以上模型计算每个患者的风险评分,然后以患者总体风险评分的中位数为界,将队列中的患者划分为高风险组和低风险组,进而采用多因素Cox回归分析评估该风险模型对患者总体生存风险的预测能力。
1.4 预后分层分析和列线图的绘制
为了综合评价预后风险评分模型,将TCGA肝癌患者按照性别、年龄和肿瘤分期进行分层后评估风险评分与患者总体生存期的关系。在生存分析中,首先依据每个临床参数将患者分成两组[16],然后按照风险评分(以中位数分组)将患者进一步分为高风险组和低风险组,比较两组间生存期的差异。此外,采用多因素Cox回归构建列线图(nomogram)来量化患者的生存风险,进而预测患者的临床预后。分析中纳入的临床参数包括性别、年龄、肿瘤分期和基于预测模型计算的风险评分。采用R中的rms包来绘制列线图,同时采用校准曲线(calibration curve)来评价该模型预测患者生存的准确性[17]。此外,还采用时间依赖的受试者工作特征(receiver operating characteristic, ROC)曲线评估该风险模型在预测患者预后方面的能力[18]。
1.5 基因集富集分析
采用基因集富集分析(gene set enrichment analysis, GSEA)[19]对高、低风险组(以中位数分组)进行富集分析,鉴定与缺氧生存风险相关的信号通路。采用的参考基因集为MsigDB(7.2版本)中的GO(Gene Ontology)基因集,样本置换检验1000次,< 0.05作为显著性的评价标准。采用enrichplot[20]包对富集的基因集进行聚类分析。
1.6 细胞系
人正常肝细胞系L02,人肝癌细胞系HepG2、HCCLM3和Huh7均来自本实验室细胞库。细胞培养实验所用培养基均为含10% FBS和1%青、链霉素双抗的DMEM。L02、HepG2、HCCLM3和Huh7细胞培养条件均为37℃,5% CO2,并保持一定的湿度。
1.7 细胞RNA的提取及实时定量PCR实验
取对数生长期细胞分别接种于两组6孔板中,当细胞量生长到60%~70%时,将其中1组6孔板细胞移至氧气浓度为2%的低氧培养箱中继续培养24 h,另一组6孔板细胞继续在二氧化碳培养箱中培养24 h。各组细胞总RNA的提取根据康为世纪生物有限公司的RNA提取试剂盒说明书进行。RNA逆转录反应根据TaKaRa公司的逆转录试剂盒说明书进行。相关基因的实时定量PCR(quantitative reverse transcription-PCR, RT-qPCR)实验根据SYBR Green荧光定量试剂盒说明书进行。所有实验均设置3个重复。以肌动蛋白()基因为对照,使用∆∆Ct法对mRNA定量结果进行归一化处理。对所有PCR实验均采用熔解曲线分析以排除非特异性扩增。PCR引物由华大基因公司合成,引物信息见附表5。
1.8 统计学分析
采用2检验进行分类变量组成的差异比较;采用秩和检验计算TCGA肝癌患者不同分组间风险评分的差异;采用单因素方差分析计算在TCGA肿瘤及癌旁非肿瘤组织中的差异表达。采用Spearman相关分析计算TCGA肝癌组织中基因拷贝数与其表达水平的相关性。采用Kaplan-Meier法进行生存期估计;采用Log-rank检验评估组间的生存期差异;采用单因素Cox比例风险回归分析计算风险比(hazard ratio, HR)和95%置信区间(confidence interval, CI)。使用GEPIA[21]网站进行泛癌肿瘤(TCGA肿瘤样本)与非肿瘤组织(TCGA癌旁样本和基因型–组织表达数据库(genotype-tissue expression,GTEx)样本)的差异表达分析和TCGA泛癌肿瘤样本高低表达组间的生存分析。主成分分析(principal component analysis, PCA)[22]用于肝癌缺氧相关lncRNA表达谱风险模型的有效降维、模式识别和高维数据的可视化。所有统计学分析均采用R软件(版本4.0.2,www.rproject.org)。
2 结果与分析
2.1 肝癌中与缺氧相关的lncRNA的鉴定
为了鉴定肝癌中与缺氧相关的lncRNA,首先从以往报道的研究[13,14]中获得了124个与肿瘤缺氧相关的编码基因(图1A,附表2)。其次,基于TCGA肝癌癌组织的基因表达谱数据集,采用GSVA计算每个样本的缺氧评分。最后,通过缺氧评分与lncRNA表达水平的Spearman相关性分析,共鉴定出233个与肝癌缺氧相关的lncRNA。其中与肝癌缺氧评分呈正相关的lncRNA有49个,呈负相关的有184个(图1:B和C)。
2.2 基于缺氧相关lncRNA建立肝癌的预后风险模型
为了从上述233个lncRNA中进一步筛选与肝癌患者预后相关的lncRNA,采用单变量Cox比例风险分析评估了每个候选lncRNA的预后价值。结果显示,其中20个lncRNA具有显著的预后价值(< 0.0005)。随后,采用多变量Cox分析从中进一步鉴定出12个具有独立预后价值的候选lncRNA,包括、、、、、、、、、、和(图2A,附表3)。这12个与缺氧相关的lncRNA中,包括1个保护型lncRNA(HR<1)和11个风险型lncRNA(HR>1)(图2A)。
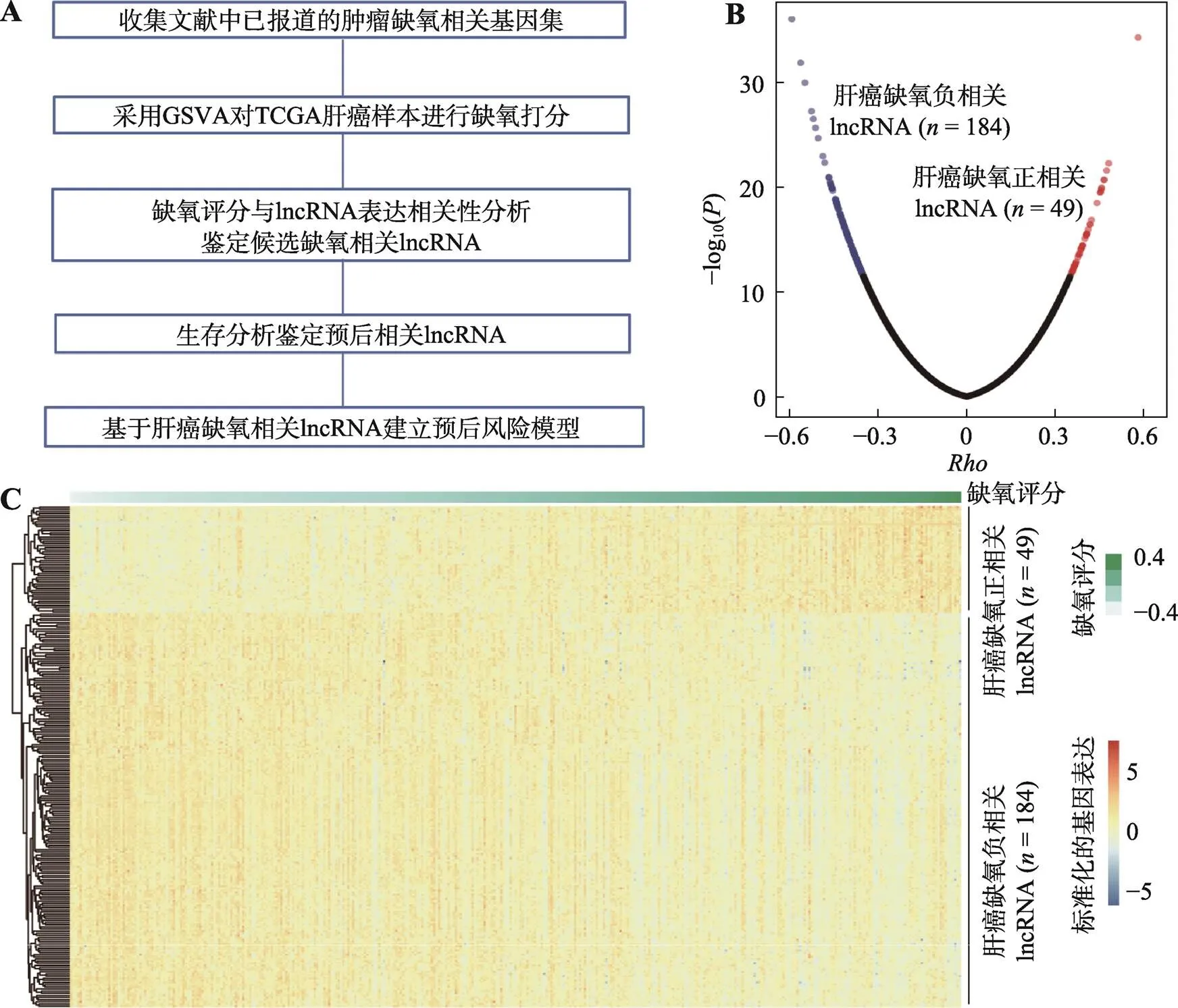
图1 肝癌中缺氧相关lncRNA的鉴定
A:肝癌中缺氧相关lncRNA的鉴定流程。B:火山图显示了TCGA肝癌组织中lncRNA的表达水平与缺氧评分之间的相关性。缺氧评分基于缺氧相关基因集由GSVA计算所得。红色的点代表显著正相关,蓝色的点代表显著负相关。相关系数()和值采用Spearman相关性分析计算所得。C:TCGA肝癌队列中缺氧相关lncRNA表达水平的热图。
随后,基于这12个lncRNA的表达水平及其对应的多变量Cox回归分析系数,在TCGA肝癌患者队列中建立了预后评分模型:风险评分= (0.38976795 × Expr) + (0.405757379 × Expr) + (0.357444336 × Expr) + (0.36846134 × Expr) + (0.473496973 × Expr) + (0.422085606 × Expr) + (0.417047269 × Expr) + (-0.355404128 × Expr) + (0.399819433 × Expr) + (0.391626222 × Expr) + (0.579995375 × Expr) + (0.3673704 × Expr)。进一步根据风险评分的中位数将肝癌患者分为高风险组和低风险组。这些缺氧相关的lncRNA在肝癌组织样本中的表达水平热图显示,高风险组中风险型lncRNA表达上调,而保护型lncRNA在低风险组中高表达(图2B)。主成分分析同样显示,这些缺氧相关的lncRNA可以区分低风险组和高风险组的肝癌样本(图2C)。风险评分和生存状况相关性分析显示,风险评分越高,患者的死亡率则越高(图2:D和E)。Kaplan-Meier生存分析显示,高风险组患者的总体生存率显著低于低风险组患者(HR = 2.55,< 0.0001)(图2F),提示此风险评分具有重要的预后价值。为了验证此风险模型的预测价值,在另一独立的肝癌患者队列(GSE40144)中对此进行了验证[23]。基于缺氧相关lncRNA风险评分的Kaplan-Meier生存分析显示,高风险组患者的死亡率显著高于低风险组患者(HR = 1.93,= 0.015)(图2G)。

图2 肝癌缺氧相关lncRNA预后模型的构建
A:多变量Cox比例风险回归分析。森林图显示了候选lncRNA的HR(95% CI)和值。B:热图显示了高风险组患者和低风险组患者中候选lncRNA的表达水平。C:主成分分析显示基于12个候选lncRNA的表达谱可以显著区分低风险组和高风险组肝癌患者。D:缺氧相关lncRNA预后特征的高、低风险肝癌患者风险评分分布。E:肝癌患者的生存时间与基于缺氧相关lncRNA构建的预后特征的风险评分之间的相关性。F:Kaplan-Meier生存曲线显示,TCGA肝癌队列中基于缺氧相关lncRNA构建的预后特征的高风险评分患者的生存时间显著短于低风险评分患者。G:Kaplan-Meier生存曲线显示,GSE40144队列中基于缺氧相关lncRNA构建的预后特征的高风险评分患者的生存时间显著短于低风险评分患者。H:风险模型评分和临床特征的1、3和5年生存预测能力评价。PC1,主成分1(principal component 1);PC2,主成分2(principal component 2);PC3,主成分3(principal component 3);T,肿瘤大小;N,淋巴结转移;M,远端转移。
为了进一步评价风险评分预测肝癌患者短期和长期生存的能力,估算了风险评分预测不同生存期的受试者工作特征曲线下面积(area under the curve, AUC)。结果显示,1年、3年和5年生存风险评分的AUC分别为0.74、0.70和0.71,显著优于年龄、性别、T(肿瘤大小)分期、N(淋巴结转移)分期及M(远处转移)分期等临床病理因素的预测能力(AUC均小于0.67;图2H),提示该模型能较好地预测肝癌患者的短期和长期生存状况。
2.3 肝癌缺氧相关lncRNA预后模型的评估
为了评估上述由12个缺氧相关lncRNA构建的风险模型是否具备独立的预后预测价值,对TCGA肝癌患者进行了风险评分的单因素和多因素生存分析。单因素Cox回归分析显示,HR和95% CI分别为2.55和1.78~3.70(= 3.29×10–7)(图3A)。校正了其他的临床病理因素后的多因素Cox回归分析显示,HR和95% CI分别为2.77和1.68~4.56(= 6.07×10–5) (图3A)。这些结果表明,缺氧相关lncRNA的风险模型是独立于临床病理因素的最显著的预后因素。
为了进一步验证其临床意义,将TCGA肝癌队列按临床病理特征进行了分层,并比较不同分组间风险评分的差异。患者分别按年龄(≤60岁>60岁)、性别(女性男性)、T(肿瘤大小)分期(T1和T2T3和T4)进行分组。不同年龄患者风险评分的秩和检验显示,年轻和老年患者之间的风险评分在统计学上相似(图3B)。Kaplan-Meier生存分析显示,老年组(HR = 2.24,= 0.00083)和年轻组(HR = 2.95,< 0.0001)高风险患者的总体生存率均显著低于低风险患者(图3B)。不同性别患者风险评分的秩和检验显示,女性患者的风险评分和男性患者的风险评分在统计学上相似(图3C)。该风险模型对男性患者的预后具有预测价值(HR = 3.51,< 0.0001),而对女性患者则没有这种预测价值(HR = 1.34,= 0.30)(图3C)。此外,不同临床分期患者的风险评分秩和检验显示,临床分期较高的肝癌患者其风险评分(T2、T3和T4期)显著高于临床分期较低的肝癌患者(T1期)(图3D)。T1~T2期患者(HR = 2.12,= 0.0012)和T3~T4期患者(HR = 2.27,= 0.0028)的Kaplan- Meier生存分析均显示,高风险患者的总体生存率显著低于低风险患者(图3D)。总之,这些结果表明该预后预测模型是肝癌患者的一个重要的独立预后因素,特别是在男性患者中具有更好的预测能力。
2.4 基于临床特征参数与预后预测评分模型建立列线图
列线图是临床上用于准确预测患者生存时间的方法,可根据列线图中包含的每个预后因素的分值计算总评分。为了进一步建立可应用于预测肝癌患者总体生存率预测的直接定量方法,将预测模型评分和临床特征参数进行了多因素Cox比例风险回归分析,并构建了列线图(图4A)。临床特征参数包括年龄、性别、T、N和M分期。建立的列线图显示,相较于其他临床特征参数,预后预测评分模型贡献了最大的风险值(范围为0~100)(图4A),提示此预测模型在列线图的所有变量中的作用最为重要。校准曲线显示,与参考线相比,实际和预测的1、3及5年生存率均较一致(图4:B~D)。这些结果均提示了建立的肝癌缺氧相关lncRNA风险评分列线图的准确性。
2.5 肝癌中缺氧相关lncRNA特征的生物学意义
为了进一步探究肝癌缺氧相关lncRNA可能参与调控的生物学过程,基于TCGA肝癌高风险组和低风险组样本的基因表达数据进行了GSEA分析。结果显示,高风险组肝癌显著富集于与细胞的分裂增殖相关的信号通路,如姐妹染色单体分离、染色体组织的负调控和核分裂的负调控等(图5A,附表4),提示高风险评分反映了增殖信号的异常活化。而这些通路的异常活化与肿瘤的发生发展密切相关[24]。相反,低风险组肝癌显著富集于代谢相关通路,包括脂肪氧化、脂肪酸分解代谢过程、有机酸分解代谢过程和脂肪酸乙酰氨基甲酸分解代谢过程(图5A,附表4),提示低风险评分的患者保持了相对正常的肝功能。此外,通过enrichplot分析构建的网络图的结果与这些发现一致:与细胞周期、代谢和氧化磷酸化相关的信号通路发生了异常(图5B)。总之,这些结果为未来针对不同风险的肝癌患者的个体化治疗提供了参考。
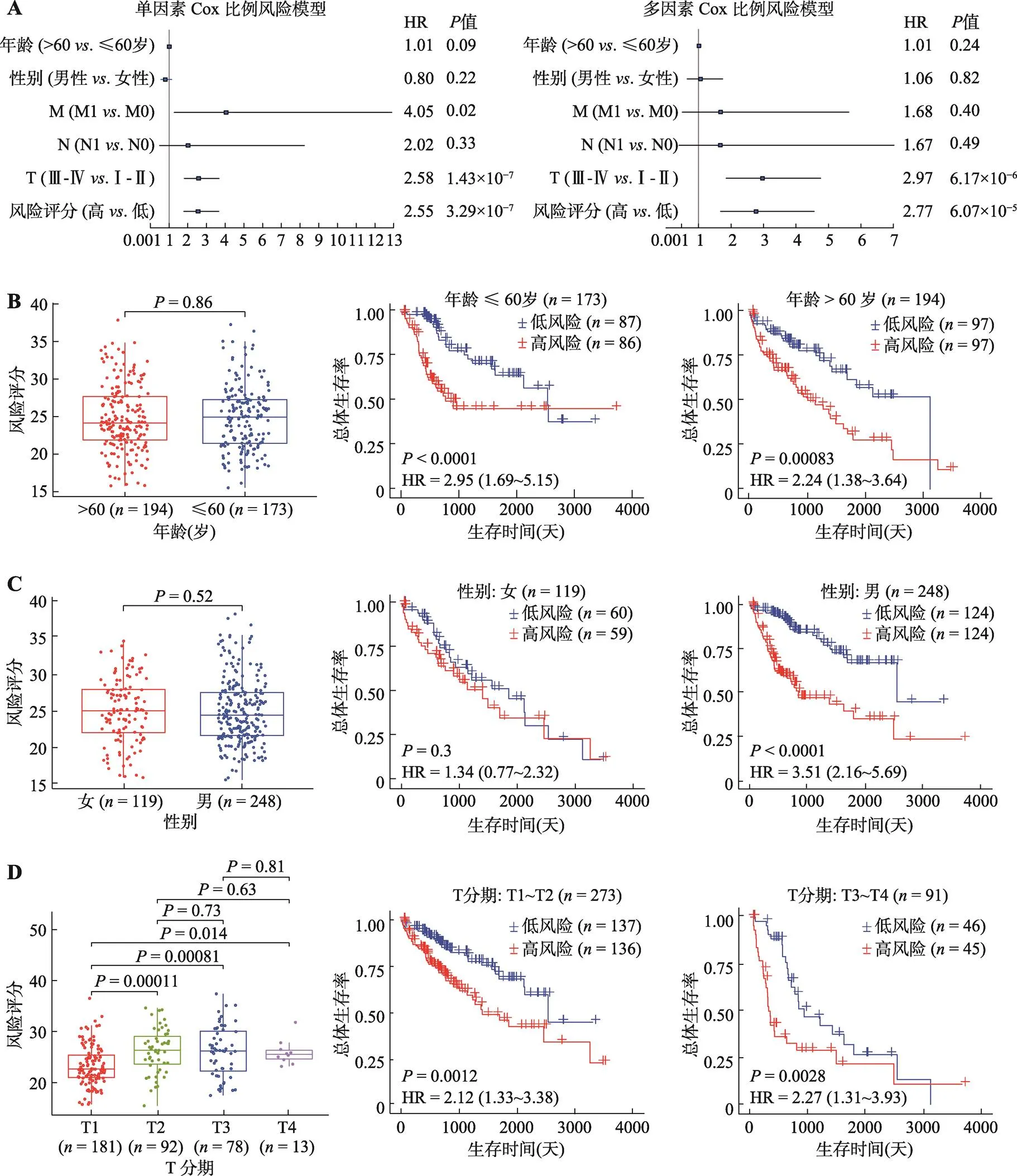
图3 缺氧相关lncRNA预后特征与肝癌患者临床特征的相关性分析及分层生存分析
A:单因素(左)及多因素(右)Cox比例风险回归分析风险模型评分和临床特征与肝癌患者预后的关系。临床特征包括年龄、性别、T(肿瘤大小)、N(淋巴结转移)及M(远处转移)分期。B~D:相关性分析分别按(B)年龄(≤60岁>60岁)、(C)性别(男性女性)和(D)T分期(T1和T2T3和T4)将TCGA肝癌样本分组后比较组间肝癌缺氧相关风险评分的差异,并评估肝癌缺氧相关风险评分在分层后的生存预后价值。
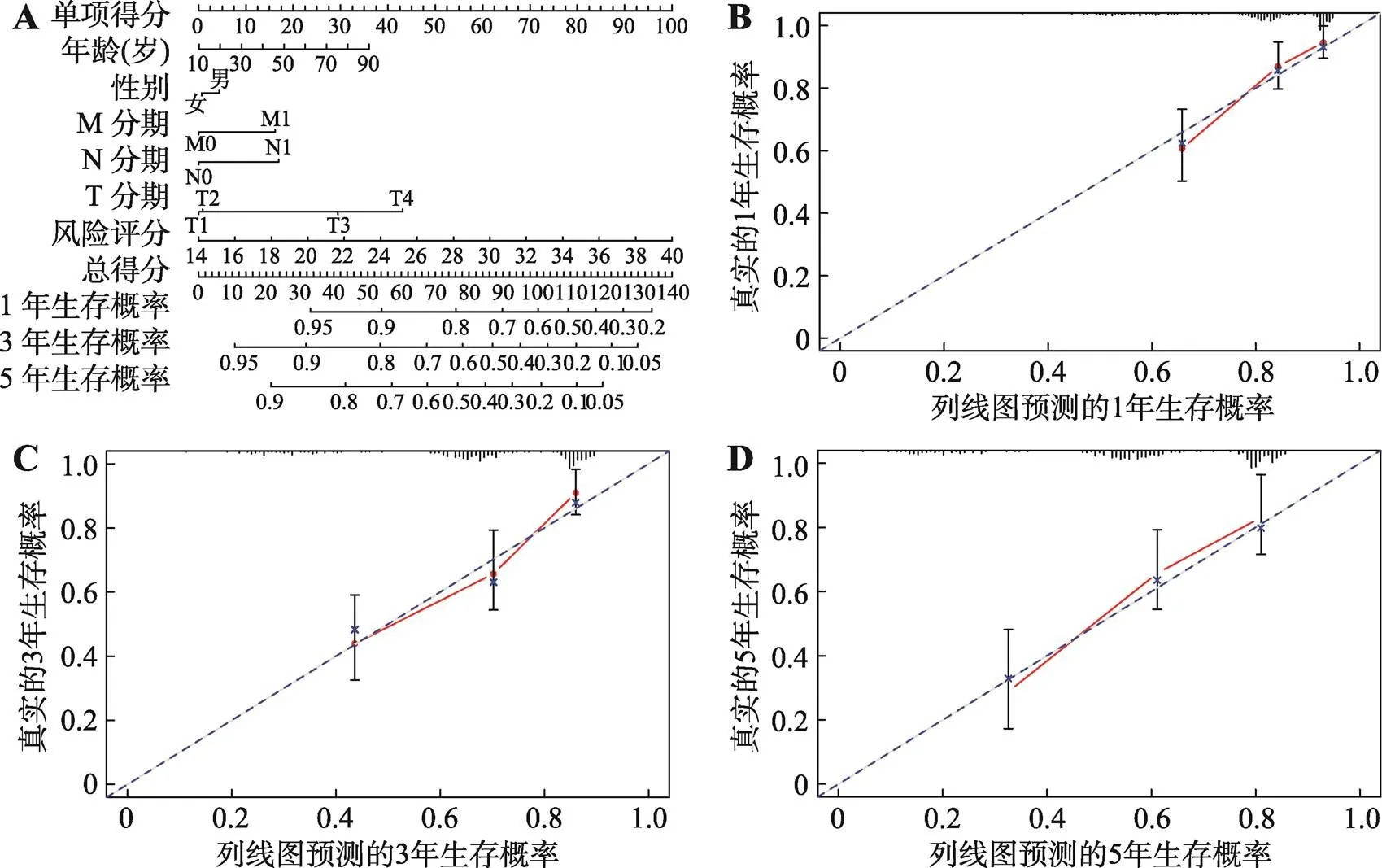
图4 基于缺氧相关lncRNA预后特征构建的列线图
A:包括缺氧相关的lncRNA预后特征风险评分、年龄、性别、T、N及M分期等临床病理参数在内的肝癌患者1、3和5年生存概率预测的列线图。B~D:校正曲线显示了根据偏倚校正后的预后列线图预测的肝癌患者1年生存概率(B)、3年生存概率(C)和5年生存概率(D)与实际生存率的一致性。
2.6 特征性lncRNA MIR210HG在肝癌中表达水平上调且与患者的不良预后显著相关
在上述12个与肝癌预后相关的lncRNA中,(miR-210宿主基因)在多因素Cox分析中显示出与不良预后显著的相关性(HR = 2.13,= 0.0030;图2A)。miR-210是参与缺氧应激的重要miRNA,其在多种肿瘤细胞和肿瘤组织中表达水平均上调[25]。作为实体瘤生物标志物[26],miR-210可以调节肿瘤细胞的增殖[27]、凋亡[28]和转移[29]。阻断miR-210-5p可以逆转缺氧诱导的线粒体自噬核心调节基因(ATPase family AAA-domain containing protein 3A,)表达水平的下调,并通过促进线粒体自噬增加肝癌细胞对索拉非尼的敏感性[30]。基于此,进一步评估了在肝癌中潜在的临床意义。
首先,基于TCGA数据集研究了在肝癌组织中的表达水平变化。结果显示,与癌旁非肿瘤肝脏组织相比,在肝癌组织中显著上调表达(图6A)。泛癌分析显示,在TCGA大多数癌症的癌组织中显著上调表达(图6B),如肾上腺皮质癌(adrenocortical carcinoma, ACC)、肾透明细胞癌(kidney renal clear cell carcinoma, KIRC)和胰腺癌(pancreatic adenocarcinoma, PAAD)等;在少数几种癌种中下调表达,如急性髓细胞样白血病(acute myeloid leukemia, LAML)、食管癌(esophageal carcinoma, ESCA)和皮肤黑色素瘤(skin cutaneous melanoma, SKCM)等。生存分析显示,的高表达与肝癌患者的较短生存期显著相关(HR = 1.98,= 0.00018;图6A)。泛癌分析显示,对肾上腺皮质癌、结肠癌(colon adenocarcinoma, COAD)、肾嫌色细胞癌(kidney chromophobe, KICH)和胰腺癌也具有生存风险的预测能力(图6C)。然后,进一步探究了在肝癌组织中上调表达的潜在机制。分析了TCGA肝癌组织的基因组拷贝数变化(copy number alteration, CNA)与的表达水平的相关性。结果显示,的表达水平与其拷贝数显著正相关(= 0.13,= 0.013;图6D),提示的上调表达部分受其基因组拷贝数扩增的调控。最后,评估了的表达水平和缺氧评分之间的相关性,结果显示两者呈显著的正相关性(= 0.65,< 0.00001;图6E)。

图5 基于缺氧相关lncRNA构建的预后特征的生物学意义
A:GSEA结果显示,高风险肝癌患者的细胞周期相关信号通路显著富集,而低风险肝癌患者的代谢信号通路显著富集。根据肝癌缺氧相关lncRNA特征风险评分将TCGA肝癌队列中的患者分为高风险和低风险两组,并通过GSEA鉴定与风险评分相关的信号通路或生物学过程。NES和值由GSEA分析所得。B:GSEA显著富集的信号通路的网络图。圆圈的大小代表了通路中包含的基因的数量;线的粗细代表了通路之间共有的基因的数量。NES,校正后的富集分数(normalized enrichment score)。
为了进一步探究是否参与缺氧应激,采用qRT-PCR技术检测了和其他几个已被报道参与缺氧应激过程的基因的表达水平。结果显示,与常氧条件相比,缺氧条件下[31][32][33]和[34]的表达均显著增加(图6F)。值得注意的是,对TCGA肝癌组织样本中和的表达水平进行Pearson相关性分析,发现两者呈显著的正相关性(= 0.13,= 0.013)(图6G)。综上所述,作为miR-210的宿主基因,可能参与了肝癌细胞的缺氧应激过程,进而促进肝癌的进展。
3 讨论
近年来越来越多的研究已表明lncRNA参与了肝癌的进展[35,36],为揭示肿瘤的发生发展机理提供了新的视角,也为肝癌的诊治提供了新的候选靶点。TNM分期、血管侵犯和血清甲胎蛋白水平等常规临床参数在一定程度上有助于预测患者的预后[37~39]。然而,由于肝癌的高度异质性,迫切需要鉴定新的预后生物标志物并建立更为准确的预后预测模型。与采用单一的临床病理参数相比,整合不同的生物标志物并建立预后模型是预测肿瘤预后更为有效的方法。近年来,已经有多个基于lncRNA建立的肿瘤预后模型被报道,包括Li等[40]的11基因模型、Li等[41]的12基因模型和Jin等[42]的6基因模型等。缺氧是实体肿瘤的一个重要特征,在包括肝癌在内的多种癌症进展中均发挥重要作用[43~45]。以往研究显示,许多与缺氧响应相关的lncRNA参与了缺氧对肿瘤生物学行为的影响[46]。本研究旨在鉴定与肝癌微环境缺氧相关的lncRNA,并建立基于这些lncRNA的肝癌预后模型。
通过转录组整合分析鉴定到233个缺氧相关的lncRNA,并基于其中12个lncRNA建立了肝癌生存风险预测模型。在这些特征性lncRNA中,有多个lncRNA已被报道参与调控缺氧介导的肿瘤进展。例如,缺氧诱导的主效转录因子可直接与lncRNA启动子结合并激活其转录;同时,通过内源性竞争RNA机制促进的表达,从而上调的表达,在肝癌细胞中形成一个反馈环路[47]。lncRNA高表达是肝癌患者不良预后的独立危险因素;通过海绵作用吸附miR-125b-5,促进的表达,从而抑制肝癌细胞的凋亡[48]。此外,可通过靶向miR-103-RAB10轴促进胃癌的进展[49],并调控miR-378a-5pSERPINE1轴促进结肠癌细胞上皮间质转化和对奥沙利铂的耐药性[50]。本研究显示,在TCGA的大多数癌症中表达上调。例如,已有研究报道lncRNA在肝癌中作为癌基因可促进肝癌细胞的增殖、侵袭和迁移[51]。可通过调节缺氧参与结直肠腺癌的进展[52]。通过HMGA2-TGF-β/Wnt通路抑制miR-337-3p/137,从而促进子宫内膜癌的进展[53]。调节miR-874-STAT3轴促进非小细胞肺癌的进展[54]。调控miR-503-5p-TRAF4轴促进宫颈癌细胞的增殖和侵袭[55]。还可通过miR-125b-5p-HK2PKM2轴促进胰腺癌细胞的糖酵解、细胞增殖和迁移[56]。同时,还被报道可以增强在三阴性乳腺癌中的翻译,促进Warburg效应和肿瘤生长[57]。虽然其他特征性lncRNA(包括、、、和)在癌症中的功能特征尚未有研究报道,但其与肝癌缺氧特征和预后的显著相关性提示这些候选lncRNA值得开展深入的功能和机制研究。

A:在TCGA肝癌癌组织及癌旁组织中的表达水平和高表达组和低表达组的Kaplan-Meier生存曲线。值通过方差分析进行计算。样本按照的表达中值分为高表达和低表达组。组间差异采用秩和检验。B:在TCGA多种癌症的肿瘤及TCGA和GTEx非肿瘤组织中的表达水平。值通过方差分析进行计算。*:<0.01。C:TCGA泛癌队列中基于的表达水平的患者生存分析。HR和值由Cox比例风险回归分析确定。D:TCGA肝癌组织中基因拷贝数与其表达水平间的相关性。采用Spearman相关分析确定和值。E:TCGA肝癌组织中表达水平与缺氧评分间的相关性。采用Spearman相关分析确定和值。F:缺氧条件下和肿瘤缺氧相关基因表达水平显著增加。分别在常氧和氧气浓度2%条件下培养24 h。G:TCGA肝癌组织中表达水平与表达水平间的相关性。T,肿瘤样本;N,癌旁对照样本;ACC,肾上腺皮质癌;BLCA,膀胱尿路上皮癌;BRCA,乳腺浸润癌;CESC,宫颈鳞癌和腺癌;CHOL,胆管癌;COAD,结肠癌;DLBC,弥漫性大B细胞淋巴瘤;ESCA,食管癌;GBM,多形成性胶质细胞瘤;HNSC,头颈鳞状细胞癌;KICH,肾嫌色细胞癌;KIRC,肾透明细胞癌;KIRP,肾乳头状细胞癌;LAML,急性髓细胞样白血病;LGG,脑低级别胶质瘤;LIHC,肝细胞肝癌;LUAD,肺腺癌;LUSC,肺鳞癌;MESO,间皮瘤;OV,卵巢浆液性囊腺癌;PAAD,胰腺癌;PCPG,嗜铬细胞瘤和副神经节瘤;PRAD,前列腺癌;READ,直肠腺癌;SARC,肉瘤;SKCM,皮肤黑色素瘤;STAD,胃癌;TGCT,睾丸癌;THCA,甲状腺癌;THYM,胸腺癌;UCEC,子宫内膜癌;UCS,子宫肉瘤;UVM,葡萄膜黑色素瘤。
本研究也存在一定的局限性。首先,本研究的预后模型是基于公共数据库的回顾性数据集所构建,因此,还需要更多的前瞻性的数据来验证其潜在的临床价值。其次,仅仅考虑单一特征(比如缺氧)来构建预后模型的内在弱点是不可避免的,因为肝癌中的许多突出的预后基因可能已被排除在外。再次,本研究主要基于TCGA的数据集,其中大多数患者是白人或亚洲人,将本研究的发现拓展到其他种族的患者需要非常谨慎。最后,本研究的所有分析都是描述性的,需要进一步的功能实验来阐明这12个与肝癌缺氧相关的lncRNA的潜在机制。
总之,本研究构建了一个基于缺氧相关lncRNA的肝癌患者的预后预测模型,该模型初步显示了较好的临床应用潜力。为了进一步证实其预后预测效果,该模型未来还需要在更大的队列中进行验证。
附加材料见文章电子版www.chinagene.cn。
[1] Torre LA, Bray F, Siegel RL, Ferlay J, Lortet-Tieulent J, Jemal A. Global cancer statistics, 2012., 2015, 65(2): 87–108.S
[2] Yuen VW, Wong CC. Hypoxia-inducible factors and innate immunity in liver cancer., 2020, 130(10): 5052–5062.
[3] Shimamoto K, Tanimoto K, Fukazawa T, Nakamura H, Kanai A, Bono H, Ono H, Eguchi H, Hirohashi N. GLIS1, a novel hypoxia-inducible transcription factor, promotes breast cancer cell motility via activation of WNT5A., 2020, 41(9): 1184–1194.
[4] Huang JL, Zheng L, Hu YW, Wang Q. Characteristics of long non-coding RNA and its relation to hepatocellular carcinoma., 2014, 35(3): 507–514.
[5] Standaert L, Adriaens C, Radaelli E, Van Keymeulen A, Blanpain C, Hirose T, Nakagawa S, Marine JC. The long noncoding RNA Neat1 is required for mammary gland development and lactation., 2014, 20(12): 1844–1849.
[6] Wang Y, Chen WY, Lian JY, Zhang HB, Yu B, Zhang MJ, Wei FQ, Wu JH, Jiang JX, Jia YS, Mo F, Zhang SR, Liang XD, Mou XZ, Tang JM. The lncRNA PVT1 regulates nasopharyngeal carcinoma cell proliferation via activating the KAT2A acetyltransferase and stabilizing HIF-1α., 2020, 27(2): 695–710.
[7] Wang XW, Li L, Zhao KM, Lin QY, Li HY, Xue XT, Ge WJ, He HJ, Liu D, Xie H, Wu Q, Hu Y. A novel LncRNA HITT forms a regulatory loop with HIF-1α to modulate angiogenesis and tumor growth., 2020, 27(4): 1431–1446.
[8] Zeng Z, Xu FY, Zheng H, Cheng P, Chen QY, Ye Z, Zhong JX, Deng SJ, Liu ML, Huang K, Li Q, Li W, Hu YH, Wang F, Wang CY, Zhao G. LncRNA-MTA2TR functions as a promoter in pancreatic cancer via driving deacetylation-dependent accumulation of HIF-1α., 2019, 9(18): 5298–5314.
[9] Deng SJ, Chen HY, Ye Z, Deng SC, Zhu S, Zeng Z, He C, Liu ML, Huang K, Zhong JX, Xu FY, Li Q, Liu Y, Wang CY, Zhao G. Hypoxia-induced LncRNA-BX111 promotes metastasis and progression of pancreatic cancer through regulating ZEB1 transcription., 2018, 37(44): 5811–5828.
[10] Liang YR, Song XJ, Li YM, Chen B, Zhao WJ, Wang LJ, Zhang HW, Liu Y, Han DW, Zhang N, Ma TT, Wang YJ, Ye FZ, Luo D, Li XY, Yang QF. LncRNA BCRT1 promotes breast cancer progression by targeting miR-1303/PTBP3 axis., 2020, 19(1): 85.
[11] Dong L, Cao X, Luo Y, Zhang GQ, Zhang DD. A positive feedback loop of lncRNA DSCR8/miR-98-5p/STAT3/HIF- 1α plays a role in the progression of ovarian cancer., 2020, 10: 1713.
[12] Hua Q, Mi BM, Xu F, Wen J, Zhao L, Liu JJ, Huang G. Hypoxia-induced lncRNA-AC020978 promotes proliferation and glycolytic metabolism of non-small cell lung cancer by regulating PKM2/HIF-1α axis., 2020, 10(11): 4762–4778.
[13] Winter SC, Buffa FM, Silva P, Miller C, Valentine HR, Turley H, Shah KA, Cox GJ, Corbridge RJ, Homer JJ, Musgrove B, Slevin N, Sloan P, Price P, West CM, Harris AL. Relation of a hypoxia metagene derived from head and neck cancer to prognosis of multiple cancers., 2007, 67(7): 3441–3449.
[14] Eustace A, Mani N, Span PN, Irlam JJ, Taylor J, Betts GNJ, Denley H, Miller CJ, Homer JJ, Rojas AM, Hoskin PJ, Buffa FM, Harris AL, Kaanders JHAM, West CML. A 26-gene hypoxia signature predicts benefit from hypoxia- modifying therapy in laryngeal cancer but not bladder cancer., 2013, 19(17): 4879–4888.
[15] Hänzelmann S, Castelo R, Guinney J. GSVA: gene set variation analysis for microarray and RNA-seq data., 2013, 14: 7.
[16] Sun ZL, Jing CY, Xiao CT, Li TC. An autophagy-related long non-coding RNA prognostic signature accurately predicts survival outcomes in bladder urothelial carcinoma patients., 2020, 12(15): 15624–15637.
[17] Steyerberg EW, Vergouwe Y. Towards better clinical prediction models: seven steps for development and an ABCD for validation., 2014, 35(29): 1925–1931.
[18] Blanche P, Dartigues JF, Jacqmin-Gadda H. Estimating and comparing time-dependent areas under receiver operating characteristic curves for censored event times with competing risks., 2013, 32(30): 5381–5397.
[19] Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES, Mesirov JP. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles., 2005, 102(43): 15545–15550.
[20] Zhang CJ, Zheng YX, Li X, Hu X, Qi F, Luo J. Genome-wide mutation profiling and related risk signature for prognosis of papillary renal cell carcinoma., 2019, 7(18): 427.
[21] Tang FZ, Li CW, Kang BX, Gao G, Li C, Zhang ZM. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses., 2017, 45(W1): W98–W102.
[22] Kim S, Kang D, Huo ZG, Park Y, Tseng GC. Meta- analytic principal component analysis in integrative omics application., 2018, 34(8): 1321–1328.
[23] Tang R, Wu JC, Zheng LM, Li ZR, Zhou KL, Zhang ZS, Xu DF, Chen C. Long noncoding RNA RUSC1-AS-N indicates poor prognosis and increases cell viability in hepatocellular carcinoma., 2018, 22(2): 388–396.
[24] Torgovnick A, Schumacher B. DNA repair mechanisms in cancer development and therapy., 2015, 6: 157.
[25] Fasanaro P, Greco S, Ivan M, Capogrossi MC, Martelli F. microRNA: emerging therapeutic targets in acute ischemic diseases., 2010, 125(1): 92–104.
[26] Feng S, He AB, Wang DL, Kang B. Diagnostic significance of miR-210 as a potential tumor biomarker of human cancer detection: an updated pooled analysis of 30 articles., 2019, 12: 479–493.
[27] Kim JH, Park SG, Song SY, Kim JK, Sung JH. Reactive oxygen species-responsive miR-210 regulates proliferation and migration of adipose-derived stem cells via PTPN2., 2013, 4(4): e588.
[28] Yang W, Sun T, Cao JP, Liu FJ, Tian Y, Zhu W. Downregulation of miR-210 expression inhibits proliferation, induces apoptosis and enhances radiosensitivity in hypoxic human hepatoma cells in vitro., 2012, 318(8): 944–954.
[29] Ying Q, Liang LH, Guo WJ, Zha RP, Tian Q, Huang SL, Yao J, Ding J, Bao MY, Ge C, Yao M, Li JJ, He XH. Hypoxia-inducible microRNA-210 augments the metastatic potential of tumor cells by targeting vacuole membrane protein 1 in hepatocellular carcinoma., 2011, 54(6): 2064–2075.
[30] Wu H, Wang T, Liu YQ, Li X, Xu SL, Wu CT, Zou HB, Cao MF, Jin GX, Lang JY, Wang B, Liu BH, Luo XL, Xu C. Mitophagy promotes sorafenib resistance through hypoxia-inducible ATAD3A dependent axis., 2020, 39(1): 274.
[31] Ebright RY, Zachariah MA, Micalizzi DS, Wittner BS, Niederhoffer KL, Nieman LT, Chirn B, Wiley DF, Wesley B, Shaw B, Nieblas-Bedolla E, Atlas L, Szabolcs A, Iafrate AJ, Toner M, Ting DT, Brastianos PK, Haber DA, Maheswaran S. HIF1A signaling selectively supports proliferation of breast cancer in the brain., 2020, 11(1): 6311.
[32] Lin YT, Wu KJ. Epigenetic regulation of epithelial- mesenchymal transition: focusing on hypoxia and TGF-β signaling., 2020, 27(1): 39.
[33] Wang XX, Che XF, Yu Y, Cheng Y, Bai M, Yang ZC, Guo QQ, Xie XC, Li DN, Guo M, Hou KZ, Guo WD, Qu XJ, Cao L. Hypoxia-autophagy axis induces VEGFA by peritoneal mesothelial cells to promote gastric cancer peritoneal metastasis through an integrin α5-fibronectin pathway., 2020, 39(1): 221.
[34] Ramapriyan R, Caetano MS, Barsoumian HB, Mafra ACP, Zambalde EP, Menon H, Tsouko E, Welsh JW, Cortez MA. Altered cancer metabolism in mechanisms of immunotherapy resistance., 2019, 195: 162–171.
[35] Chen BW, Zhou Y, Wei T, Wen L, Zhang YB, Shen SC, Zhang J, Ma T, Chen W, Ni L, Wang Y, Bai XL, Liang TB. lncRNA-POIR promotes epithelial-mesenchymal transition and suppresses sorafenib sensitivity simultaneously in hepatocellular carcinoma by sponging miR-182-5p., 2021, 122(1): 130–142.
[36] Sun Z, Xue SL, Zhang MY, Xu H, Hu XM, Chen SH, Liu YY, Guo MZ, Cui HM. Aberrant NSUN2-mediated m(5)C modification of H19 lncRNA is associated with poor differentiation of hepatocellular carcinoma., 2020, 39(45): 6906–6919.
[37] Cai HY, Zhang Y, Zhang HY, Cui C, Li CH, Lu SC. Prognostic role of tumor mutation burden in hepatocellular carcinoma after radical hepatectomy., 2020, 121(6): 1007–1014.
[38] Maleki Vareki S. High and low mutational burden tumors versus immunologically hot and cold tumors and response to immune checkpoint inhibitors., 2018, 6(1): 157.
[39] Liu GM, Zeng HD, Zhang CY, Xu JW. Identification of a six-gene signature predicting overall survival for hepatocellular carcinoma., 2019, 19: 138.
[40] Li XY, Jin F, Li Y. A novel autophagy-related lncRNA prognostic risk model for breast cancer., 2021, 25(1): 4–14.
[41] Li XY, Li Y, Yu XM, Jin F. Identification and validation of stemness-related lncRNA prognostic signature for breast cancer., 2020, 18(1): 331.
[42] Jin LP, Li CY, Liu T, Wang L. A potential prognostic prediction model of colon adenocarcinoma with recurrence based on prognostic lncRNA signatures., 2020, 14(1): 24.
[43] Niklasson CU, Fredlund E, Monni E, Lindvall JM, Kokaia Z, Hammarlund EU, Bronner ME, Mohlin S. Hypoxia inducible factor-2α importance for migration, proliferation, and self-renewal of trunk neural crest cells., 2021, 250(2): 191–236.
[44] Liu OH, Kiema M, Beter M, Ylä-Herttuala S, Laakkonen JP, Kaikkonen MU. Hypoxia-mediated regulation of histone demethylases affects angiogenesis-associated functions in endothelial cells., 2020, 40(11): 2665–2677.
[45] Renfrow JJ, Soike MH, West JL, Ramkissoon SH, Metheny-Barlow L, Mott RT, Kittel CA, D'agostino Jr RB, Tatter SB, Laxton AW, Frenkel MB, Hawkins GA, Herpai D, Sanders S, Sarkaria JN, Lesser GJ, Debinski W, Strowd RE. Attenuating hypoxia driven malignant behavior in glioblastoma with a novel hypoxia-inducible factor 2 alpha inhibitor., 2020, 10(1): 15195.
[46] Yu AJ, Zhao LW, Kang QM, Li J, Chen K, Fu H. Transcription factor HIF1α promotes proliferation, migration, and invasion of cholangiocarcinoma via long noncoding RNA H19/microRNA-612/Bcl-2 axis., 2020, 224: 26–39.
[47] Wang L, Sun LK, Liu RY, Mo HY, Niu YS, Chen TX, Wang YF, Han SS, Tu KS, Liu QG. Long non-coding RNA MAPKAPK5-AS1/PLAGL2/HIF-1α signaling loop promotes hepatocellular carcinoma progression., 2021, 40(1): 72.
[48] Hu B, Yang XB, Yang X, Sang XT. LncRNA CYTOR affects the proliferation, cell cycle and apoptosis of hepatocellular carcinoma cells by regulating the miR-125b-5p/KIAA1522 axis., 2020, 13(2): 2626–2639.
[49] Wei F, Wang Y, Zhou Y, Li Y. Long noncoding RNA CYTOR triggers gastric cancer progression by targeting miR-103/RAB10., 2021, 53(8): 1044–1054.
[50] Yang JL, Ma Q, Zhang MM, Zhang WF. LncRNA CYTOR drives L-OHP resistance and facilitates the epithelial- mesenchymal transition of colon carcinoma cells via modulating miR-378a-5p/SERPINE1., 2021, 20(14): 1415–1430.
[51] Wang Y, Li WJ, Chen XY, Li Y, Wen PH, Xu F. MIR210HG predicts poor prognosis and functions as an oncogenic lncRNA in hepatocellular carcinoma., 2019, 111: 1297–1301.
[52] He ZY, Dang J, Song AL, Cui X, Ma ZJ, Zhang ZT. Identification of LINC01234 and MIR210HG as novel prognostic signature for colorectal adenocarcinoma., 2019, 234(5): 6769–6777.
[53] Ma J, Kong FF, Yang D, Yang H, Wang CC, Cong R, Ma XX. LncRNA MIR210HG promotes the progression of endometrial cancer by sponging miR-337-3p/137 via the HMGA2-TGF-β/Wnt pathway., 2021, 24: 905–922.
[54] Bu L, Zhang LB, Tian M, Zheng ZB, Tang HJ, Yang QJ. LncRNA MIR210HG facilitates non-small cell lung cancer progression through directly regulation of miR-874/STAT3 axis., 2020, 18(3): 1559325820918052.
[55] Wang AH, Jin CH, Cui GY, Li HY, Wang Y, Yu JJ, Wang RF, Tian XY. MIR210HG promotes cell proliferation and invasion by regulating miR-503-5p/TRAF4 axis in cervical cancer., 2020, 12(4): 3205– 3217.
[56] Yu TZ, Li GP, Wang CG, Gong GQ, Wang LW, Li CY, Chen Y, Wang XL. MIR210HG regulates glycolysis, cell proliferation, and metastasis of pancreatic cancer cells through miR-125b-5p/HK2/PKM2 axis., 2021, 18(12): 2513–2530.
[57] Du Y, Wei N, Ma RL, Jiang SH, Song D. Long noncoding RNA MIR210HG promotes the warburg effect and tumor growth by enhancing HIF-1α translation in triple-negative breast cancer., 2020, 10: 580176.
附表1 TCGA中肝癌患者的临床信息
Supplementary Table 1 Summary of clinical information of HCC patients in TCGA
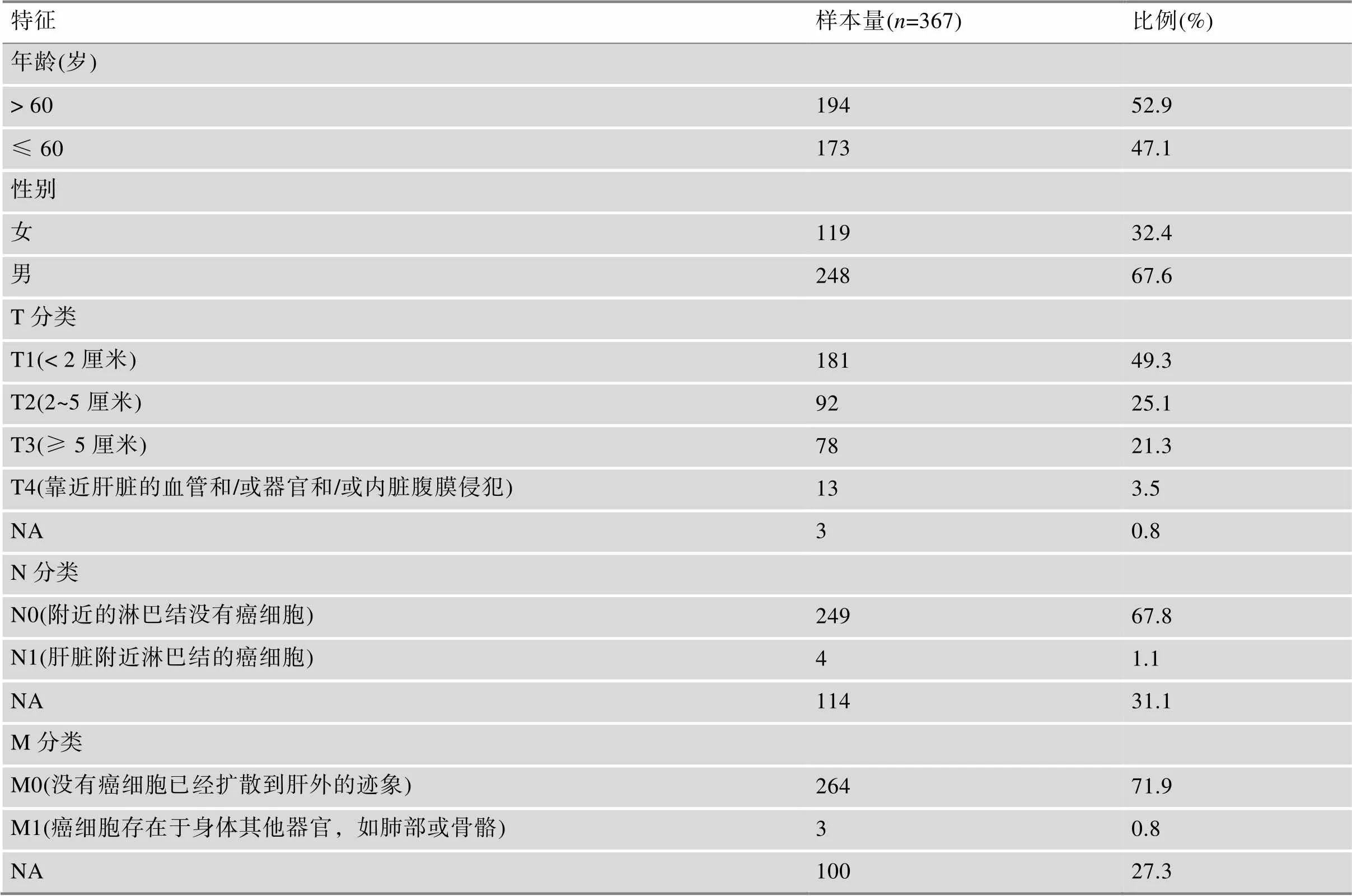
特征样本量(n=367)比例(%) 年龄(岁) > 6019452.9 ≤ 6017347.1 性别 女11932.4 男24867.6 T分类 T1(< 2厘米)18149.3 T2(2~5厘米)9225.1 T3(≥ 5厘米)7821.3 T4(靠近肝脏的血管和/或器官和/或内脏腹膜侵犯)133.5 NA30.8 N分类 N0(附近的淋巴结没有癌细胞)24967.8 N1(肝脏附近淋巴结的癌细胞)41.1 NA11431.1 M分类 M0(没有癌细胞已经扩散到肝外的迹象)26471.9 M1(癌细胞存在于身体其他器官,如肺部或骨骼)30.8 NA10027.3
T,肿瘤大小;N,淋巴结;M,远端转移;NA,缺乏数据(not available)。
附表2 肿瘤缺氧相关编码基因集
Supplementary Table 2 List of the hypoxia-related coding genes in tumors
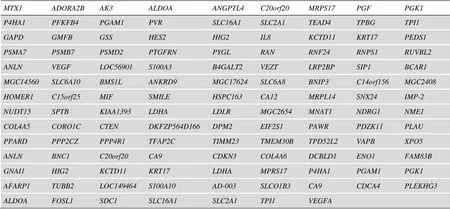
MTX1ADORA2BAK3ALDOAANGPTL4C20orf20MRPS17PGFPGK1 P4HA1PFKFB4PGAM1PVRSLC16A1SLC2A1TEAD4TPBGTPI1 GAPDGMFBGSSHES2HIG2 IL8KCTD11KRT17PEDS1 PSMA7PSMB7PSMD2PTGFRNPYGLRANRNF24RNPS1RUVBL2 ANLNVEGFLOC56901S100A3B4GALT2VEZTLRP2BPSIP1BCAR1 MGC14560SLC6A10BMS1LANKRD9MGC17624SLC6A8BNIP3C14orf156MGC2408 HOMER1C15orf25MIFSMILEHSPC163CA12MRPL14SNX24IMP-2 NUDT15SPTBKIAA1393LDHALDLRMGC2654MNAT1NDRG1NME1 COL4A5CORO1CCTENDKFZP564D166DPM2EIF2S1PAWRPDZK11PLAU PPARDPPP2CZPPP4R1TFAP2CTIMM23TMEM30BTPD52L2VAPBXPO5 ANLNBNC1C20orf20CA9CDKN3COL4A6DCBLD1ENO1FAM83B GNAI1HIG2KCTD11KRT17LDHAMPRS17P4HA1PGAM1PGK1 AFARP1TUBB2LOC149464S100A10AD-003SLCO1B3CA9CDCA4PLEKHG3 ALDOAFOSL1SDC1SLC16A1SLC2A1TPI1VEGFA
附表3 20个在肝癌中具有潜在预后价值的缺氧相关的lncRNAs
Supplementary Table 3 20 hypoxia-associated lncRNAs with prognostic value in HCCs
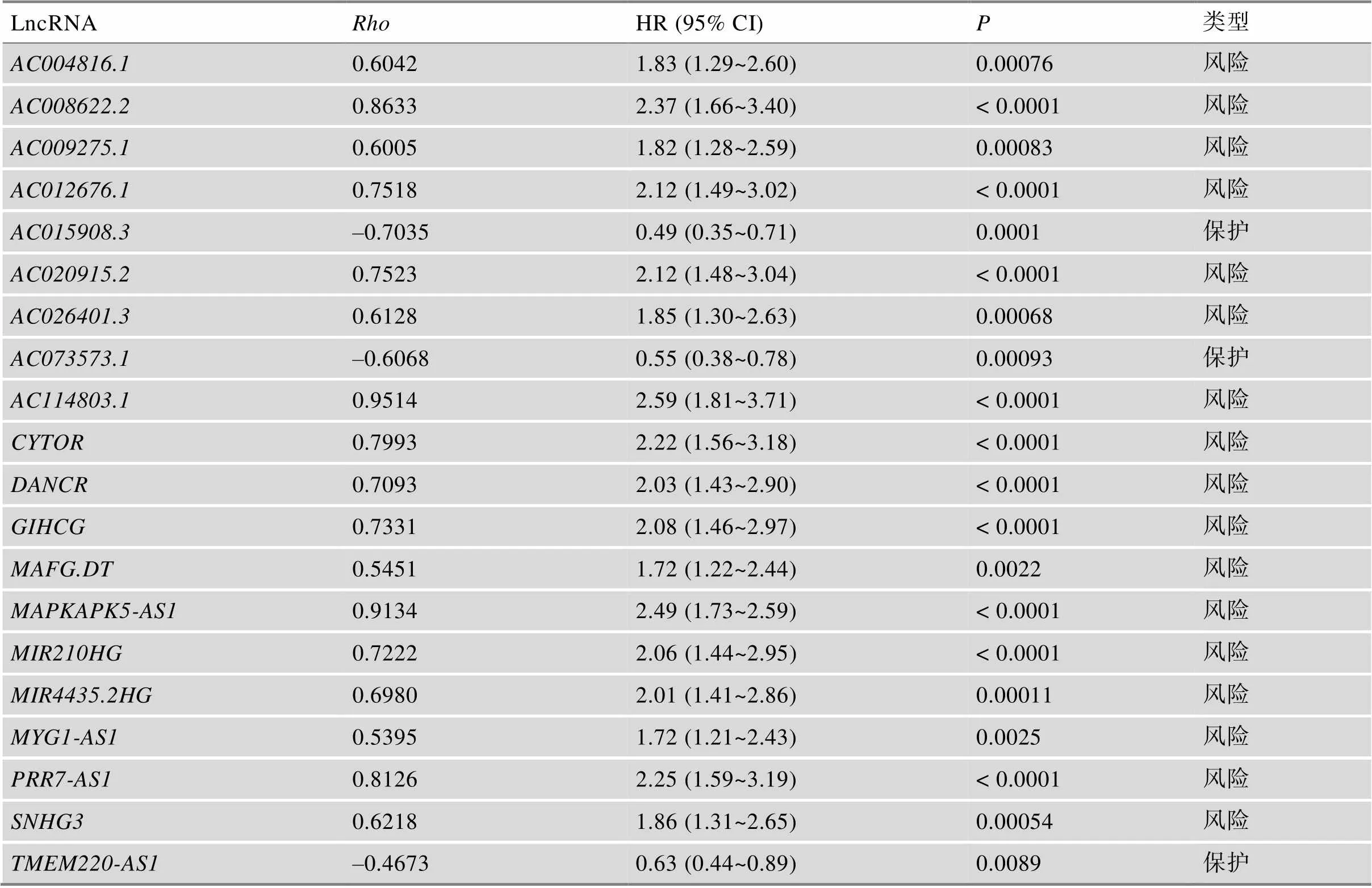
LncRNARhoHR (95% CI)P类型 AC004816.10.60421.83 (1.29~2.60)0.00076风险 AC008622.20.86332.37 (1.66~3.40)< 0.0001风险 AC009275.10.60051.82 (1.28~2.59)0.00083风险 AC012676.10.75182.12 (1.49~3.02)< 0.0001风险 AC015908.3–0.70350.49 (0.35~0.71)0.0001保护 AC020915.20.75232.12 (1.48~3.04)< 0.0001风险 AC026401.30.61281.85 (1.30~2.63)0.00068风险 AC073573.1–0.60680.55 (0.38~0.78)0.00093保护 AC114803.10.95142.59 (1.81~3.71)< 0.0001风险 CYTOR0.79932.22 (1.56~3.18)< 0.0001风险 DANCR0.70932.03 (1.43~2.90)< 0.0001风险 GIHCG0.73312.08 (1.46~2.97)< 0.0001风险 MAFG.DT0.54511.72 (1.22~2.44)0.0022风险 MAPKAPK5-AS10.91342.49 (1.73~2.59)< 0.0001风险 MIR210HG0.72222.06 (1.44~2.95)< 0.0001风险 MIR4435.2HG0.69802.01 (1.41~2.86)0.00011风险 MYG1-AS10.53951.72 (1.21~2.43)0.0025风险 PRR7-AS10.81262.25 (1.59~3.19)< 0.0001风险 SNHG30.62181.86 (1.31~2.65)0.00054风险 TMEM220-AS1–0.46730.63 (0.44~0.89)0.0089保护
LncRNA,长链非编码RNA(long non-coding RNA);,spearman相关性系数;HR,风险比(hazard ratio);CI,可信区间(confidence interval)。
附表4 基于TCGA中肝癌样本风险评分的GSEA结果
Supplementary Table 4 GSEA results based on the risk score in HCCs from TCGA
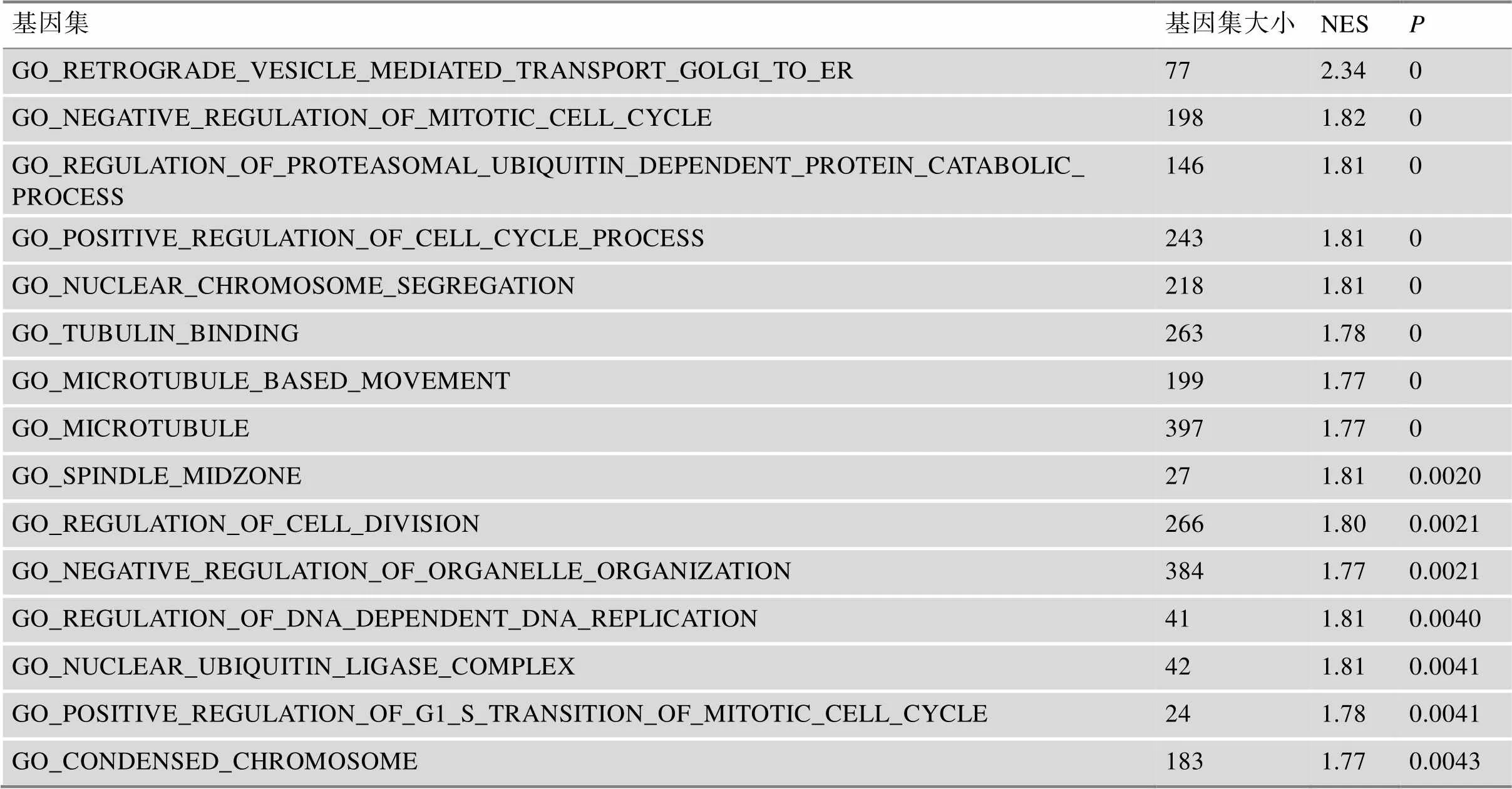
基因集基因集大小NESP GO_RETROGRADE_VESICLE_MEDIATED_TRANSPORT_GOLGI_TO_ER772.340 GO_NEGATIVE_REGULATION_OF_MITOTIC_CELL_CYCLE1981.820 GO_REGULATION_OF_PROTEASOMAL_UBIQUITIN_DEPENDENT_PROTEIN_CATABOLIC_ PROCESS1461.810 GO_POSITIVE_REGULATION_OF_CELL_CYCLE_PROCESS2431.810 GO_NUCLEAR_CHROMOSOME_SEGREGATION2181.810 GO_TUBULIN_BINDING2631.780 GO_MICROTUBULE_BASED_MOVEMENT1991.770 GO_MICROTUBULE3971.770 GO_SPINDLE_MIDZONE271.810.0020 GO_REGULATION_OF_CELL_DIVISION2661.800.0021 GO_NEGATIVE_REGULATION_OF_ORGANELLE_ORGANIZATION3841.770.0021 GO_REGULATION_OF_DNA_DEPENDENT_DNA_REPLICATION411.810.0040 GO_NUCLEAR_UBIQUITIN_LIGASE_COMPLEX421.810.0041 GO_POSITIVE_REGULATION_OF_G1_S_TRANSITION_OF_MITOTIC_CELL_CYCLE241.780.0041 GO_CONDENSED_CHROMOSOME1831.770.0043
续附表4

基因集基因集大小NESP GO_RECOMBINATIONAL_REPAIR701.780.0063 GO_CONDENSED_CHROMOSOME_CENTROMERIC_REGION941.780.0064 GO_POSITIVE_REGULATION_OF_MITOTIC_NUCLEAR_DIVISION511.770.0064 GO_SPINDLE_ASSEMBLY681.810.0081 GO_HETEROCHROMATIN661.770.0083 GO_CELL_CYCLE_G2_M_PHASE_TRANSITION1321.810.0084 GO_MITOTIC_CELL_CYCLE_CHECKPOINT1381.800.013 GO_SPINDLE_LOCALIZATION381.780.014 GO_POSITIVE_REGULATION_OF_CHROMOSOME_SEGREGATION251.770.014 GO_MEMBRANE_DISASSEMBLY461.790.021 GO_REGULATION_OF_TELOMERE_MAINTENANCE621.810.023 GO_CHROMOSOMAL_REGION3101.790.023 GO_SPLICEOSOMAL_COMPLEX1631.780.033 GO_RNA_SPLICING3351.790.038 GO_LIPID_OXIDATION68–2.150 GO_MICROBODY132–2.130 GO_FATTY_ACID_CATABOLIC_PROCESS71–2.130 GO_FATTY_ACID_BETA_OXIDATION49–2.100 GO_MICROBODY_PART92–2.080 GO_COENZYME_BINDING175–2.070 GO_ORGANIC_ACID_CATABOLIC_PROCESS202–2.030 GO_FLAVIN_ADENINE_DINUCLEOTIDE_BINDING73–2.020 GO_MICROBODY_MEMBRANE58–2.000 GO_METHIONINE_METABOLIC_PROCESS18–1.990 GO_COFACTOR_BINDING258–1.970 GO_MICROBODY_LUMEN44–1.970 GO_AMINO_ACID_BETAINE_METABOLIC_PROCESS18–1.940 GO_LIPID_HOMEOSTASIS107–1.890 GO_BILE_ACID_METABOLIC_PROCESS35–1.890 GO_REGULATION_OF_FATTY_ACID_OXIDATION27–1.840 GO_BILE_ACID_BIOSYNTHETIC_PROCESS20–1.820 GO_CELLULAR_ALDEHYDE_METABOLIC_PROCESS83–1.820 GO_ACYLGLYCEROL_HOMEOSTASIS29–1.820 GO_MONOOXYGENASE_ACTIVITY91–1.800 GO_DRUG_METABOLIC_PROCESS39–1.780 GO_POSITIVE_REGULATION_OF_FATTY_ACID_METABOLIC_PROCESS33–1.780 GO_NITROGEN_CYCLE_METABOLIC_PROCESS15–1.750 GO_STEROID_HYDROXYLASE_ACTIVITY31–1.700 GO_IRON_ION_BINDING158–1.700 GO_EPOXYGENASE_P450_PATHWAY18–1.690
续附表4
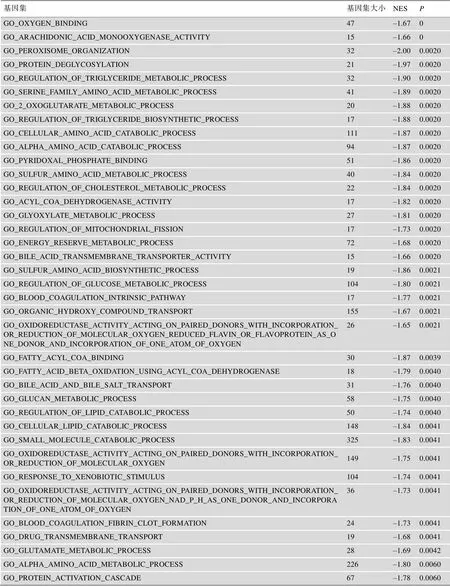
基因集基因集大小NESP GO_OXYGEN_BINDING47–1.670 GO_ARACHIDONIC_ACID_MONOOXYGENASE_ACTIVITY15–1.660 GO_PEROXISOME_ORGANIZATION32–2.000.0020 GO_PROTEIN_DEGLYCOSYLATION21–1.970.0020 GO_REGULATION_OF_TRIGLYCERIDE_METABOLIC_PROCESS32–1.900.0020 GO_SERINE_FAMILY_AMINO_ACID_METABOLIC_PROCESS41–1.890.0020 GO_2_OXOGLUTARATE_METABOLIC_PROCESS20–1.880.0020 GO_REGULATION_OF_TRIGLYCERIDE_BIOSYNTHETIC_PROCESS17–1.880.0020 GO_CELLULAR_AMINO_ACID_CATABOLIC_PROCESS111–1.870.0020 GO_ALPHA_AMINO_ACID_CATABOLIC_PROCESS94–1.870.0020 GO_PYRIDOXAL_PHOSPHATE_BINDING51–1.860.0020 GO_SULFUR_AMINO_ACID_METABOLIC_PROCESS40–1.840.0020 GO_REGULATION_OF_CHOLESTEROL_METABOLIC_PROCESS22–1.840.0020 GO_ACYL_COA_DEHYDROGENASE_ACTIVITY17–1.820.0020 GO_GLYOXYLATE_METABOLIC_PROCESS27–1.810.0020 GO_REGULATION_OF_MITOCHONDRIAL_FISSION17–1.730.0020 GO_ENERGY_RESERVE_METABOLIC_PROCESS72–1.680.0020 GO_BILE_ACID_TRANSMEMBRANE_TRANSPORTER_ACTIVITY15–1.660.0020 GO_SULFUR_AMINO_ACID_BIOSYNTHETIC_PROCESS19–1.860.0021 GO_REGULATION_OF_GLUCOSE_METABOLIC_PROCESS104–1.800.0021 GO_BLOOD_COAGULATION_INTRINSIC_PATHWAY17–1.770.0021 GO_ORGANIC_HYDROXY_COMPOUND_TRANSPORT155–1.670.0021 GO_OXIDOREDUCTASE_ACTIVITY_ACTING_ON_PAIRED_DONORS_WITH_INCORPORATION_OR_REDUCTION_OF_MOLECULAR_OXYGEN_REDUCED_FLAVIN_OR_FLAVOPROTEIN_AS_ONE_DONOR_AND_INCORPORATION_OF_ONE_ATOM_OF_OXYGEN26–1.650.0021 GO_FATTY_ACYL_COA_BINDING30–1.870.0039 GO_FATTY_ACID_BETA_OXIDATION_USING_ACYL_COA_DEHYDROGENASE18–1.790.0040 GO_BILE_ACID_AND_BILE_SALT_TRANSPORT31–1.760.0040 GO_GLUCAN_METABOLIC_PROCESS58–1.750.0040 GO_REGULATION_OF_LIPID_CATABOLIC_PROCESS50–1.740.0040 GO_CELLULAR_LIPID_CATABOLIC_PROCESS148–1.840.0041 GO_SMALL_MOLECULE_CATABOLIC_PROCESS325–1.830.0041 GO_OXIDOREDUCTASE_ACTIVITY_ACTING_ON_PAIRED_DONORS_WITH_INCORPORATION_OR_REDUCTION_OF_MOLECULAR_OXYGEN149–1.750.0041 GO_RESPONSE_TO_XENOBIOTIC_STIMULUS104–1.740.0041 GO_OXIDOREDUCTASE_ACTIVITY_ACTING_ON_PAIRED_DONORS_WITH_INCORPORATION_OR_REDUCTION_OF_MOLECULAR_OXYGEN_NAD_P_H_AS_ONE_DONOR_AND_INCORPORATION_OF_ONE_ATOM_OF_OXYGEN36–1.730.0041 GO_BLOOD_COAGULATION_FIBRIN_CLOT_FORMATION24–1.730.0041 GO_DRUG_TRANSMEMBRANE_TRANSPORT19–1.680.0041 GO_GLUTAMATE_METABOLIC_PROCESS28–1.690.0042 GO_ALPHA_AMINO_ACID_METABOLIC_PROCESS226–1.800.0060 GO_PROTEIN_ACTIVATION_CASCADE67–1.780.0060
续附表4
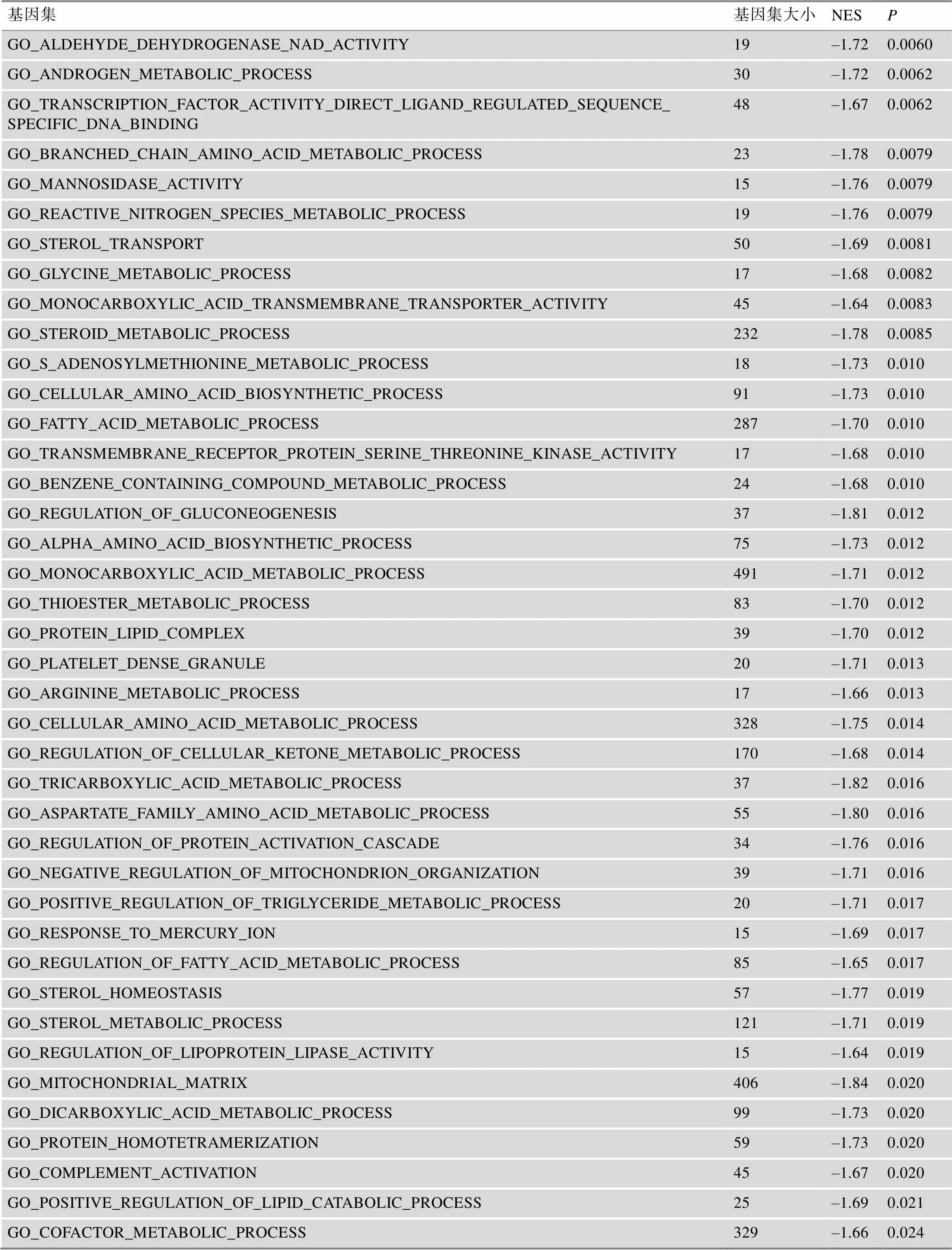
基因集基因集大小NESP GO_ALDEHYDE_DEHYDROGENASE_NAD_ACTIVITY19–1.720.0060 GO_ANDROGEN_METABOLIC_PROCESS30–1.720.0062 GO_TRANSCRIPTION_FACTOR_ACTIVITY_DIRECT_LIGAND_REGULATED_SEQUENCE_ SPECIFIC_DNA_BINDING48–1.670.0062 GO_BRANCHED_CHAIN_AMINO_ACID_METABOLIC_PROCESS23–1.780.0079 GO_MANNOSIDASE_ACTIVITY15–1.760.0079 GO_REACTIVE_NITROGEN_SPECIES_METABOLIC_PROCESS19–1.760.0079 GO_STEROL_TRANSPORT50–1.690.0081 GO_GLYCINE_METABOLIC_PROCESS17–1.680.0082 GO_MONOCARBOXYLIC_ACID_TRANSMEMBRANE_TRANSPORTER_ACTIVITY45–1.640.0083 GO_STEROID_METABOLIC_PROCESS232–1.780.0085 GO_S_ADENOSYLMETHIONINE_METABOLIC_PROCESS18–1.730.010 GO_CELLULAR_AMINO_ACID_BIOSYNTHETIC_PROCESS91–1.730.010 GO_FATTY_ACID_METABOLIC_PROCESS287–1.700.010 GO_TRANSMEMBRANE_RECEPTOR_PROTEIN_SERINE_THREONINE_KINASE_ACTIVITY17–1.680.010 GO_BENZENE_CONTAINING_COMPOUND_METABOLIC_PROCESS24–1.680.010 GO_REGULATION_OF_GLUCONEOGENESIS37–1.810.012 GO_ALPHA_AMINO_ACID_BIOSYNTHETIC_PROCESS75–1.730.012 GO_MONOCARBOXYLIC_ACID_METABOLIC_PROCESS491–1.710.012 GO_THIOESTER_METABOLIC_PROCESS83–1.700.012 GO_PROTEIN_LIPID_COMPLEX39–1.700.012 GO_PLATELET_DENSE_GRANULE20–1.710.013 GO_ARGININE_METABOLIC_PROCESS17–1.660.013 GO_CELLULAR_AMINO_ACID_METABOLIC_PROCESS328–1.750.014 GO_REGULATION_OF_CELLULAR_KETONE_METABOLIC_PROCESS170–1.680.014 GO_TRICARBOXYLIC_ACID_METABOLIC_PROCESS37–1.820.016 GO_ASPARTATE_FAMILY_AMINO_ACID_METABOLIC_PROCESS55–1.800.016 GO_REGULATION_OF_PROTEIN_ACTIVATION_CASCADE34–1.760.016 GO_NEGATIVE_REGULATION_OF_MITOCHONDRION_ORGANIZATION39–1.710.016 GO_POSITIVE_REGULATION_OF_TRIGLYCERIDE_METABOLIC_PROCESS20–1.710.017 GO_RESPONSE_TO_MERCURY_ION15–1.690.017 GO_REGULATION_OF_FATTY_ACID_METABOLIC_PROCESS85–1.650.017 GO_STEROL_HOMEOSTASIS57–1.770.019 GO_STEROL_METABOLIC_PROCESS121–1.710.019 GO_REGULATION_OF_LIPOPROTEIN_LIPASE_ACTIVITY15–1.640.019 GO_MITOCHONDRIAL_MATRIX406–1.840.020 GO_DICARBOXYLIC_ACID_METABOLIC_PROCESS99–1.730.020 GO_PROTEIN_HOMOTETRAMERIZATION59–1.730.020 GO_COMPLEMENT_ACTIVATION45–1.670.020 GO_POSITIVE_REGULATION_OF_LIPID_CATABOLIC_PROCESS25–1.690.021 GO_COFACTOR_METABOLIC_PROCESS329–1.660.024
续附表4

基因集基因集大小NESP GO_OXIDOREDUCTASE_ACTIVITY_ACTING_ON_THE_CH_CH_GROUP_OF_DONORS57–1.770.025 GO_QUATERNARY_AMMONIUM_GROUP_TRANSPORT18–1.740.025 GO_SIGNAL_PEPTIDE_PROCESSING24–1.710.027 GO_GLUCAN_BIOSYNTHETIC_PROCESS25–1.680.027 GO_RETROGRADE_TRANSPORT_VESICLE_RECYCLING_WITHIN_GOLGI23–1.720.028 GO_NAD_BINDING53–1.690.029 GO_NUCLEOSIDE_BISPHOSPHATE_METABOLIC_PROCESS37–1.700.030 GO_ENDOCYTIC_VESICLE_LUMEN17–1.640.030 GO_LIGASE_ACTIVITY_FORMING_CARBON_SULFUR_BONDS40–1.650.035 GO_TRIGLYCERIDE_RICH_LIPOPROTEIN_PARTICLE19–1.650.035 GO_ASPARTATE_FAMILY_AMINO_ACID_BIOSYNTHETIC_PROCESS23–1.660.036 GO_COENZYME_A_METABOLIC_PROCESS17–1.650.041 GO_2_IRON_2_SULFUR_CLUSTER_BINDING21–1.650.044
NES,校正后的富集分数(normalized enrichment score)。
附表5 PCR引物的序列信息
Supplementary Table 5 Primers for RT-qPCR assays
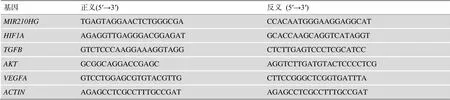
基因正义(5′→3′)反义 (5′→3′) MIR210HGTGAGTAGGAACTCTGGGCGACCACAATGGGAAGGAGGCAT HIF1AAGAGGTTGAGGGACGGAGATGCACCAAGCAGGTCATAGGT TGFBGTCTCCCAAGGAAAGGTAGGCTCTTGAGTCCCTCGCATCC AKTGCGGCAGGACCGAGCAGGTCTTGATGTACTCCCCTCG VEGFAGTCCTGGAGCGTGTACGTTGCTTCCGGGCTCGGTGATTTA ACTINAGAGCCTCGCCTTTGCCGATAGAGCCTCGCCTTTGCCGAT
引物根据人类参考基因组(基于hg19)设计。
Prognostic and predictive value of the hypoxia-associated long non-coding RNA signature in hepatocellular carcinoma
Min Cheng1,3, Jing Zhang4, Pengbo Cao2, Gangqiao Zhou1,2,3
Hepatocellular carcinoma (HCC) is a common cancer worldwide. Hypoxia is an important feature of solid tumors, including HCC, and is also an important factor involved in malignancy progression. However, the identification of hypoxia-related long non-coding RNA (lncRNAs) and their prognostic value in HCC have not been systematically investigated. The aim of this study is to identify the features based on the hypoxia-related lncRNAs and evaluate their predictive value for HCC prognosis. Based on the integrated analysis of HCC transcriptome data from The Cancer Genome Atlas (TCGA), we had identified 233 potential hypoxia-related lncRNAs. We further evaluated the prognostic value of these lncRNAs and optimally established a 12-lncRNA (,,,,,,,,,,and) prognostic risk model. The Cox proportional hazards regression analysis revealed that the hypoxia risk score is a novel independent prognostic predictor for HCC patients, which outperforms the traditional clinical pathological factors. Gene set enrichment analysis (GSEA) showed that the hypoxia risk score reflects the activation of biological features related to cell proliferation and the inactivation of lipid metabolism processes. In summary, we had constructed a risk score model based on 12 hypoxia-related lncRNAs, which might be a promising prognostic predictor for HCC patients and highlight their potential roles in the prevention and treatment of this malignancy.
hepatocellular carcinoma; hypoxia; long non-coding RNA; prognostic model
2021-12-02;
2022-01-11;
2022-01-19
国家重点研发计划(编号:2017YFA0504301)和国家自然科学基金重大研究计划重点项目(编号:91440206)资助[Supported by the National Key Research and Development Program of China (No. 2017YFA0504301) and the Major Research Plan of the National Natural Science Foundation of China (No. 91440206)]
程敏,硕士研究生,专业方向:流行病与卫生统计学。E-mail: 18351990262@139.com
曹鹏博,博士,副研究员,研究方向: 医学遗传与基因组学。E-mail: birchcpb@163.com
周钢桥,博士,研究员,研究方向:医学遗传与基因组学。E-mail: zhougq114@126.com
10.16288/j.yczz.21-416
(责任编委:宋旭)
