LincRNA AC027700.1在小鼠蜕膜化中的表达研究
2022-03-08谭丽萍高茹菲尹鑫陈雪梅李方方袁柳何俊琳
谭丽萍,高茹菲,尹鑫,陈雪梅,李方方,袁柳,何俊琳
研究报告
LincRNA AC027700.1在小鼠蜕膜化中的表达研究
谭丽萍,高茹菲,尹鑫,陈雪梅,李方方,袁柳,何俊琳
重庆医科大学,生殖与发育国际合作联合实验室, 重庆 400016
长链非编码RNA(long non-coding RNA, lncRNA)是一类长度大于200 nt、不具有蛋白编码潜能的RNA分子。在细胞生长发育、物质代谢以及疾病等的发生发展过程中起关键调控作用,但在蜕膜化相关领域研究报道较少。为了探究lincRNA AC027700.1在早孕小鼠子宫内膜中的表达规律,初步探讨AC027700.1在小鼠蜕膜化中的作用,本研究利用qRT-PCR检测AC027700.1在小鼠孕第6 d胚胎着床点及着床旁子宫组织中的表达;构建了假孕小鼠体内人工诱导蜕膜化模型和原代小鼠子宫内膜基质细胞体外诱导蜕膜化模型,qRT-PCR检测AC027700.1在人工诱导蜕膜化的组织和细胞中的表达;通过分离细胞核、细胞质RNA,qRT-PCR检测AC027700.1在胞核和胞质中的相对表达水平;利用GOseq和KOBAS软件对AC027700.1下游靶基因进行GO和KEGG分析。结果表明,AC027700.1在小鼠孕第6 d着床点子宫组织中的表达显著高于着床旁;AC027700.1在成功诱导蜕膜化的子宫内膜组织及细胞中的表达显著高于未诱导的组织及细胞;AC027700.1主要定位于细胞核内;AC027700.1下游靶基因主要富集于自噬通路、细胞周期以及RNA转运等通路。本研究初步揭示了lincRNA AC027700.1可能与早孕子宫内膜蜕膜化有关,但具体作用及调节机制还有待进一步研究。
lincRNA;AC027700.1;蜕膜化;子宫内膜;着床
尽管辅助生殖技术在不断进步并已成为目前治疗不孕症的最重要手段,但临床妊娠率和胚胎着床率仍然保持相对较低的水平[1]。根据世界卫生组织计算,大约50%的不孕症是由女性生殖缺陷引起的[2],而母体子宫内膜蜕膜化缺陷是导致不孕的重要原因之一[3,4]。正常生理状况下,子宫内膜基质细胞受到蜕膜化诱导因子的刺激,广泛增殖分化而形成一种特殊的蜕膜组织。蜕膜组织能为胚胎的发育提供营养物质,为胎盘滋养细胞对母体子宫的适度侵袭提供保障以及参与协调母胎免疫应答等,对于人类及所有侵入性着床动物(灵长类动物及啮齿类动物)的妊娠建立和维持都是不可或缺的[5,6]。
长链非编码RNA(long non-coding RNA, lncRNA)是指长度超过200 nt、不具有蛋白编码潜力的转录本[7]。根据lncRNA相对于蛋白编码基因的位置,可将其分为不与蛋白编码基因序列有重叠的长链基因间非编码RNA(long intergenic non-coding RNA, lincRNA),其余以某种方式与蛋白编码基因重叠的lncRNA被进一步分类为外显子lncRNA、内含子lncRNA和重叠子lncRNA[8]。越来越多的研究证据表明,lncRNA具有基因转录、RNA剪接、mRNA翻译以及表观遗传调节等功能[9~11],在细胞生长发育、物质代谢等过程中起关键调控作用[12,13]。除此之外,lncRNA还可以作为分子支架调节核酸-核酸、核酸-蛋白或蛋白-蛋白之间的相互作用[13,14]。LncRNA的异常表达与许多疾病的发病机制有关,如肿瘤[15]、神经系统疾病[16]、自身免疫性疾病[17]和心血管系统疾病[18]等。但迄今为止,lncRNA在蜕膜化相关领域的研究报道较少。本课题组前期通过RNA-seq测序技术对小鼠孕第6 d着床点及着床旁子宫组织中的RNA进行测序,首次发现lincRNA AC027700.1在着床点和着床旁子宫组织中的表达存在显著差异,提示AC027700.1可能参与小鼠子宫内膜蜕膜化过程。因此,本研究进一步探究了AC027700.1在小鼠子宫内膜蜕膜化过程中的表达规律,以期为后续深入探讨lincRNA在小鼠子宫内膜蜕膜化中的作用提供线索。
1 材料与方法
1.1 动物样本
本研究所使用的6~8周龄雌雄昆明小鼠购买并代养于重庆医科大学动物实验中心(实验动物生产许可号:SCXK(渝) 2018-0003;实验动物使用许可号:SYXK(渝) 2018-0003)。小鼠于温度(22±2)℃、12 h光照/12 h黑暗、自由摄水和饮食条件下饲养,所有动物实验均通过重庆医科大学伦理委员会审查批准。
1.2 正常孕鼠子宫内膜组织收取及转录组RNA测序
构建正常妊娠孕鼠模型,将雌鼠与雄鼠按3:1比例合笼,次日清晨有阴道栓的雌鼠视为妊娠第1 d (D1)。以脱颈法处死孕第6 d小鼠,收集胚胎着床点(implantation sites, IS)和着床旁(inter implantation sites, IIS)子宫组织用于后续实验,着床点和着床旁的区分见图1A。共收取4份小鼠着床点和着床旁子宫组织,建库测序及数据分析均由北京诺禾致源科技股份有限公司完成。
1.3 假孕小鼠体内人工诱导蜕膜化
参考文献[19]报道的方法构建假孕小鼠体内人工诱导蜕膜化模型:将雌鼠与双侧输精管结扎的雄鼠按3:1进行合笼,次日清晨有阴道栓的雌鼠记为假孕第1 d(PD1)。假孕第4 d(PD4)早上9:00对雌鼠一侧子宫给予子宫角注射玉米油(美国Sigma公司)处理(即诱导侧),另一侧不给予任何处理(即对照侧)。假孕第8 d (PD8)处死小鼠,分别收集小鼠双侧子宫内膜组织。
1.4 原代小鼠子宫内膜基质细胞分离及培养
按照文献[20]报道的方法分离原代小鼠子宫内膜基质细胞,具体步骤如下:(1)以脱颈法处死8周龄左右正常昆明雌鼠,在无菌环境下,取出小鼠双侧子宫,HBSS(上海博士德公司)清洗后剪碎;(2)加入适量胰蛋白酶,4℃中消化2 h,之后转移至37℃中继续消化30 min;(3)用枪头反复吹打组织10 min后,加入适量胶原酶(美国Sigma公司),于37℃中消化30 min;(4)将消化完毕的组织经70 μm滤器过滤,对下层细胞悬液进行离心并清洗。接种适量的细胞悬液于含有20%血清(以色列Biological Industries公司)、青链霉素及两性霉素(上海碧云天公司)DF12培养基(美国Sigma公司)培养;(5)大约1 h后更换培养基,置于5%CO2、37℃的孵箱中培养过夜。
次日更换培养基,同时依据文献[19]报道的方法,加入10 nmol/L 雌二醇和1 µmol/L 孕酮(美国Sigma公司)对细胞进行诱导蜕膜化处理,连续诱导72 h后,收集细胞用于后续实验。
1.5 鬼笔环肽-FITC染色
将分离得到的小鼠子宫内膜基质细胞接种到无菌细胞爬片上,按上述方法诱导其蜕膜化。72 h后,使用无菌PBS清洗爬片3次,每次5 min。使用异硫氰酸荧光素标记的鬼笔环肽(鬼笔环肽-FITC,美国Sigma公司)避光染色40~60 min。之后DAPI避光核染10 min,PBS清洗后,取出爬片,滴加抗荧光淬灭剂,封片后于荧光显微镜下观察拍照。
1.6 实时荧光定量PCR(qRT-PCR)
使用Trizol (美国Sigma公司)提取子宫内膜组织及细胞总RNA,并检测其浓度和纯度。使用PrimerScript RTTMMaster Mix (Perfect Real Time)逆转录试剂盒(日本TaKaRa公司)将RNA逆转录为cDNA。使用TB Green Premix Ex TaqTMⅡ(Tli RNaseH Plus)试剂盒(日本TaKaRa公司)在Real-time PCR仪(美国BioRad公司)上进行qRT-PCR反应,各基因引物序列见表1。以β-actin作为总RNA内参,采用2–ΔΔCT法计算各基因的相对表达量。
1.7 细胞核、质RNA分离
根据文献[13]的方法,使用RNA Subcellular Isolation Kit试剂盒(美国Active motif公司)提取细胞核RNA和细胞质RNA。其工作原理是利用完全裂解液裂解已诱导蜕膜化的基质细胞,然后高速离心分离裂解液,上清液即包含细胞质RNA,沉淀含细胞核RNA。之后利用纯化柱对RNA进行清洗纯化,纯化后的RNA用于qRT-PCR。以核小RNA U3及U4作为细胞核内参,β-actin作为细胞质内参,采用已报道的2−dCt[2−(NUC Ct−CYT Ct)]法[21]计算AC027700.1在细胞质和细胞核中的相对表达量,引物序列见表1。
1.8 生物信息学分析
利用iLoc-LncRNA(iLoc-LncRNA (lin-group.cn))对AC027700.1亚细胞定位进行预测,利用GOseq软件和KOBAS软件分别对AC027700.1下游靶基因进行基因本体富集(Gene Ontology, GO)分析和通路(Kyoto Encyclopedia of Genes and Genomes, KEGG)分析。
1.9 统计分析
采用SPSS 26.0软件进行统计分析,两组之间数据比较均采用配对检验,数据结果以均数±标准差形式表示,以*< 0.05表示差异具有统计学意义。

表1 qRT-PCR引物序列
2 结果与分析
2.1 LincRNA AC027700.1在早孕小鼠子宫组织中的表达
根据转录组RNA测序结果,本研究发现lincRNA AC027700.1在小鼠孕第6 d着床点和着床旁子宫组织中的表达存在显著差异(图1B),提示AC027700.1可能在早孕小鼠子宫内膜蜕膜化中发挥调控作用。LincRNA AC207700.1基因位于小鼠第10号染色体上,转录本全长2587 bp,包含3个外显子(图1C)。为了进一步验证AC207700.1在早孕小鼠子宫组织中的表达,本研究进一步分离了小鼠孕第6 d着床点和着床旁子宫组织(图1A),并通过qRT-PCR检测了AC027700.1在着床点和着床旁子宫组织中的表达。结果显示,AC027700.1在着床点的表达显著高于着床旁(图1D)。
2.2 假孕小鼠体内人工诱导蜕膜化后AC027700.1表达上调
为排除胚胎对AC027700.1表达的影响以及进一步确定AC027700.1在小鼠子宫内膜蜕膜化中的作用,本研究构建了假孕小鼠体内人工诱导蜕膜化模型。结果显示,和对照侧相比,假孕小鼠诱导侧子宫明显肿大、重量显著增加、蜕膜标志物蜕膜/滋养层催乳素相关蛋白(decidual/trophoblastic prolactin related proteins, Dtprp) mRNA水平显著增高(图2:A~C),以上结果提示假孕小鼠体内人工诱导蜕膜化模型构建成功。qRT-PCR结果显示,AC027700.1在诱导侧子宫内膜组织中的表达显著高于对照侧(图2D)。
2.3 体外诱导原代小鼠子宫内膜基质细胞蜕膜化后AC027700.1表达上调
子宫内膜基质细胞向具有分泌功能的上皮样蜕膜细胞的转变是蜕膜化最显著的特征,也是目前研究蜕膜化最主要的对象[22]。为进一步探索AC027700.1在基质细胞蜕膜化中的作用,本研究分离和培养了原代小鼠子宫内膜基质细胞,联合应用雌二醇和孕酮对细胞进行诱导蜕膜化。利用异硫氰酸荧光素标记的鬼笔环肽对细胞的F-肌动蛋白(F-actin)进行荧光染色后发现,与对照组细胞相比,诱导组细胞胞体变大变圆、双核细胞数量增加(图3A)。同时qRT-PCR结果显示,蜕膜化标志物Dtprp在诱导组细胞中的表达显著高于对照组(图3B),提示原代小鼠子宫内膜基质细胞体外诱导蜕膜化模型成功构建。qRT-PCR检测AC027700.1在子宫内膜基质细胞体外蜕膜化过程中的表达发现,AC027700.1在诱导组细胞中的表达显著高于对照组(图3C),提示AC027700.1可能在基质细胞蜕膜化中发挥重要调控作用。

图1 AC027700.1在小鼠孕第6 d子宫中的表达
A:小鼠孕第6 d子宫;B:AC027700.1在小鼠孕第6 d的IIS和IS子宫组织中的FPKM值;C:AC027700.1基因组位点;D:qRT-PCR检测AC027700.1在小鼠孕第6 d的IIS和IS子宫组织中的表达。IIS:着床旁;IS:着床点。*< 0.05,***< 0.001。
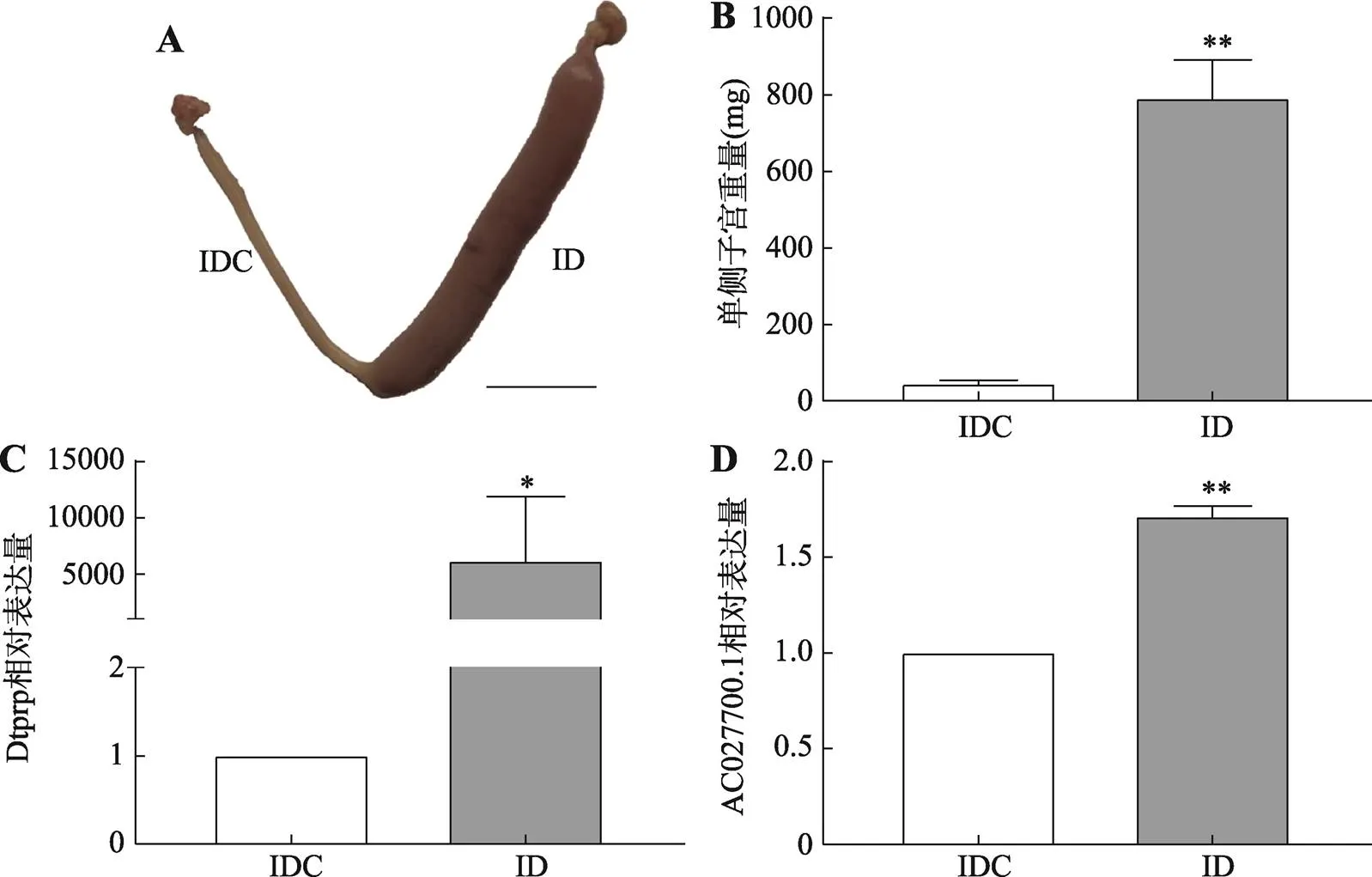
图2 AC027700.1在假孕小鼠人工体内诱导蜕膜化模型中的表达
A:假孕小鼠人工体内诱导蜕膜化模型;B:双侧子宫重量比较;C:qRT-PCR检测Dtprp在IDC及ID子宫内膜组织中的表达;D:qRT-PCR检测AC027700.1在IDC及ID子宫内膜组织中的表达。IDC:对照侧;ID:诱导侧。*< 0.05,**< 0.01。
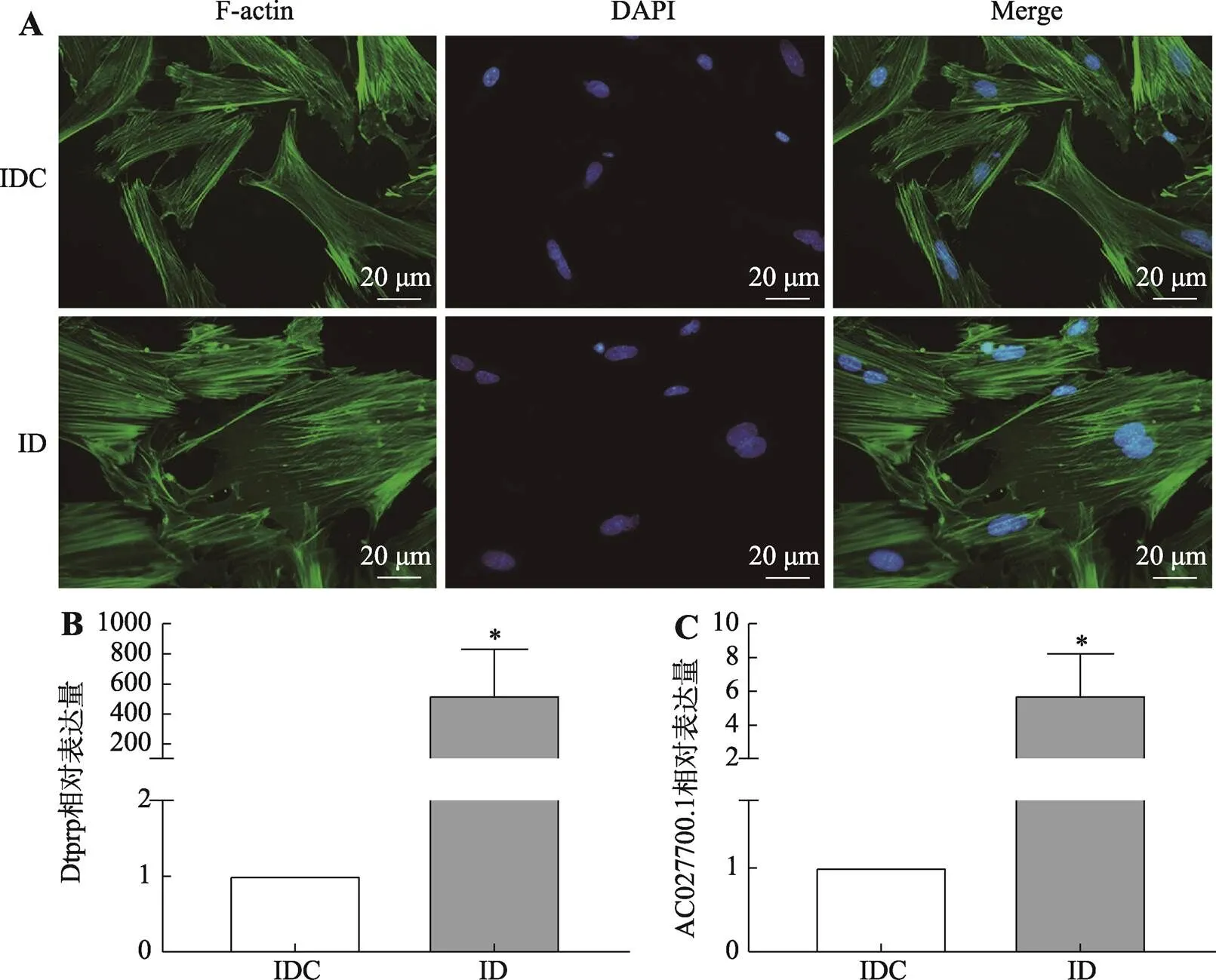
图3 AC027700.1在原代小鼠子宫内膜基质细胞体外人工诱导蜕膜化模型中的表达
A:异硫氰酸荧光素标记的鬼笔环肽对IDC及ID细胞的肌动蛋白染色;B:qRT-PCR检测Dtprp在IDC及ID细胞中的表达;C:qRT-PCR检测AC027700.1在IDC及ID细胞中的表达。IDC:对照组;ID:诱导组。*< 0.05。
2.4 AC027700.1主要定位在基质细胞的细胞核
LncRNA的功能取决于其亚细胞结构定位:核富集lncRNA的功能包括染色质相互作用、转录调控等;而富集于细胞质的lncRNA主要参与调节mRNA的稳定性或翻译等[15]。因此,为探究AC027700.1可能发挥的功能,本研究首先利用在线预测工具iLoc-LncRNA对AC027700.1细胞定位进行了预测。预测结果显示AC027700.1主要定位于细胞核,可能性约为0.65 (图4A)。为验证AC027700.1亚细胞定位,本研究对对照组及诱导组细胞进行细胞核、细胞质RNA分离,以只存在于细胞核的核小RNA U3、U4作为胞核内参,以主要存在于胞质的β-actin作为胞质内参。利用qRT-PCR分别检测了U3、U4、β-actin及AC027700.1在细胞核和细胞质中的相对表达水平,结果显示,核小RNA U3、U4在胞核中的表达显著高于胞质,β-actin在胞质中的表达显著高于胞核(图4B),证明细胞核、细胞质RNA分离成功。AC027700.1主要富集在细胞核中,与预测结果一致(图4C)。
2.5 AC027700.1下游靶基因预测
根据lncRNA与下游靶基因位置关系可以将lncRNA功能大致分为影响附近基因表达或染色质状态的顺式调节和在整个细胞中执行功能的反式调节[23]。基于全转录组RNA测序数据,本研究根据AC027700.1与基因的位置关系(上下游100 kb以内)进行顺式共定位靶基因预测,根据与基因的表达相关性(相关系数大于0.95)进行反式共表达靶基因预测。最终预测得到:AC027700.1下游共定位靶基因2个,共表达靶基因598个,共计600个(图5A)。分别利用GOseq和KOBAS软件对AC027700.1靶基因进行GO分析和KEGG分析。结果显示,AC027700.1共定位靶基因主要富集于T细胞激活、T细胞聚集、淋巴细胞聚集等生物学过程,主要参与调控自噬通路(图5:B,C);AC027700.1共表达靶基因主要富集于基础代谢、大分子代谢等细胞生物学过程,主要参与调控自噬、细胞周期和RNA转运等通路(图5:D,E)。以上结果提示,主要定位于细胞核的AC027700.1可能通过顺式作用或反式作用调节下游靶基因,从而通过调控自噬、细胞周期以及RNA转运等通路在蜕膜化中发挥用。
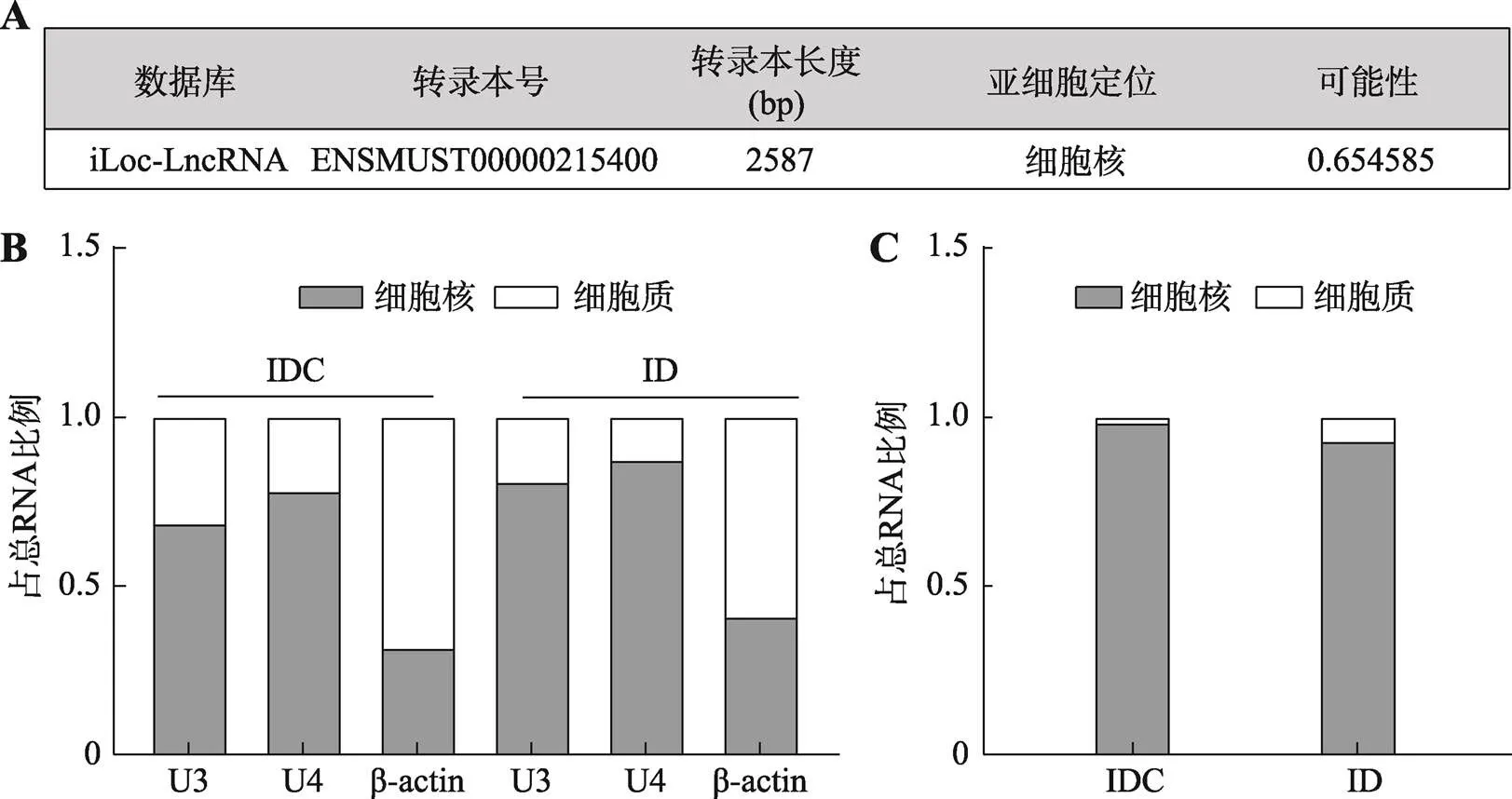
图4 AC027700.1细胞定位
A:iLoc-LncRNA预测AC027700.1在细胞中的定位;B:qRT-PCR检测U3、U4和β-actin在细胞核和细胞质RNA中的相对表达;C:qRT-PCR检测AC027700.1在细胞核和细胞质RNA中的相对表达。IDC:对照组;ID:诱导组。
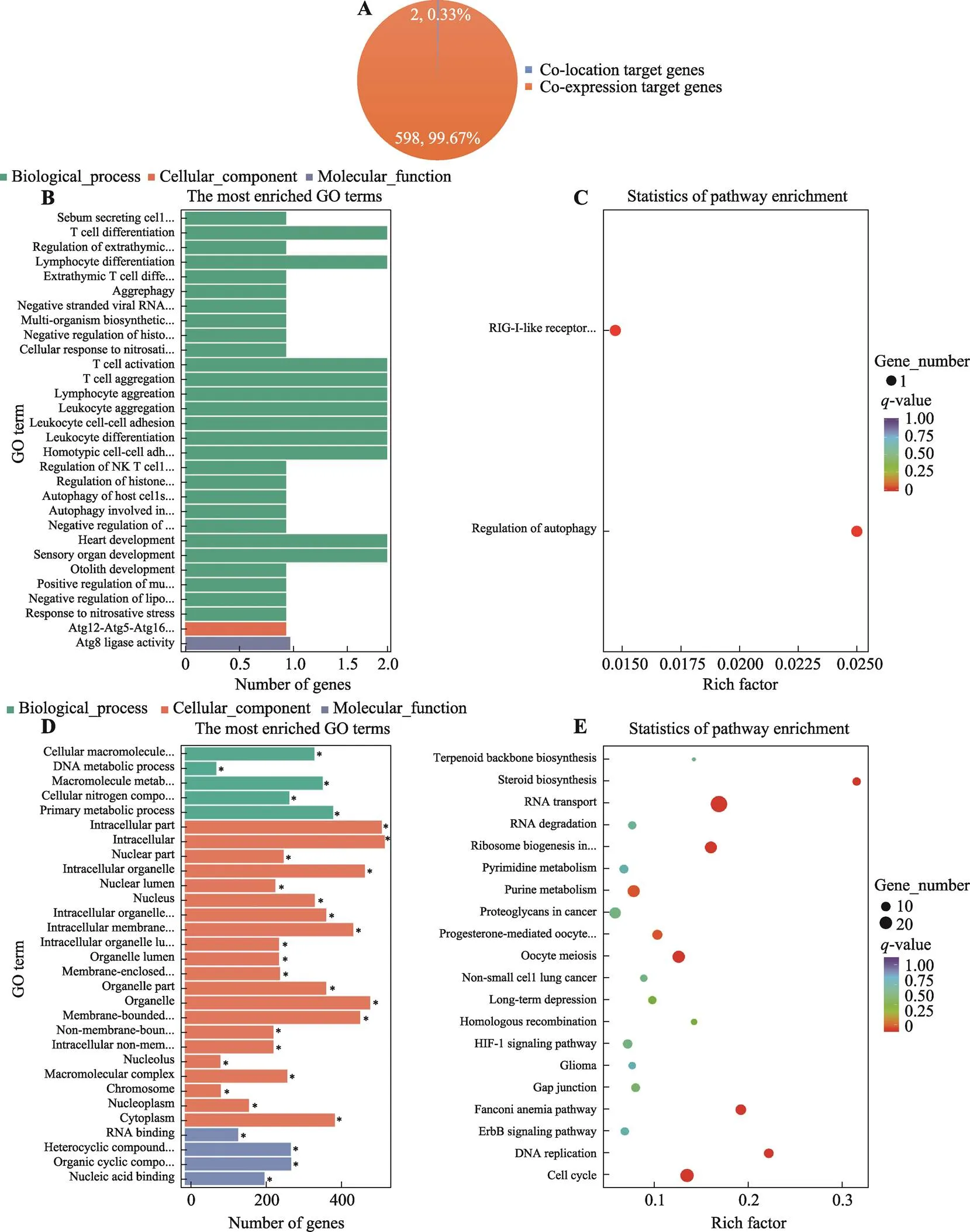
图5 AC027700.1靶基因GO和KEGG分析
A:AC027700.1下游靶基因分类;B:AC027700.1共定位靶基因GO分析;C:AC027700.1共定位靶基因KEGG分析;D:AC027700.1共表达靶基因GO分析;E:AC027700.1共表达靶基因KEGG分析。*< 0.05。
3 讨论
人类和哺乳动物的妊娠是一个复杂的、受多种因素精细调控的过程,包括受精、着床、子宫内膜蜕膜化、胎盘形成、分娩等事件,任何一个阶段异常或失败都会影响妊娠结局[3,24]。母体子宫内膜处于容受状态、胚胎与母体子宫内膜同步对话是成熟胚胎成功着床的决定因素[25]。在母体一侧,由于早期妊娠中蜕膜化异常导致的蜕膜缺陷可促使不孕、反复植入失败、复发性流产、子痫前期、早产等妊娠相关疾病的发生[3,4]。例如,Aghajanova等[26]认为子宫内膜蜕膜化受损是子宫内膜异位症患者不孕的病理基础。研究报道,蜕膜缺陷是复发性流产的重要发生机制[27]。Hirota等[28]研究发现子宫转化相关蛋白P53(Trp53)缺失的雌鼠在排卵、受精及胚胎着床阶段均正常。然而,胚胎植入后子宫蜕膜细胞表现出终末分化和生长受限,诱发了胎儿早产、难产和死亡。因此,蜕膜化的正常进行是成功妊娠的关键。深刻理解蜕膜化发展过程及其调节机制,对于预防、治疗妊娠相关疾病具有重要的意义。
子宫内膜蜕膜化同样受众多因素的调控,包括甾体激素、细胞因子、生长因子和转录调节因子等[29]。以往研究发现,lncRNA可能在胚胎着床和子宫内膜基质细胞的增殖分化中起着重要的调控作用[30,31]。例如,Liu等[32]对人子宫内膜基质细胞(human endometrial stromal cells, HESCs)进行cAMP处理后,lncRNA LINC00473表达显著增加。在HESCs的体外诱导蜕膜化实验中发现,LINC00473表达下调导致人蜕膜PRL和IGFBP1的mRNA及蛋白水平降低,提示蜕膜化进程受到抑制。Zhang等[33]研究发现lncRNA CD36-005在PCOS大鼠卵巢及子宫内膜组织中表达显著上调,lncRNA CD36-005可改变大鼠子宫内膜基质细胞中的mRNA分子表达谱,降低大鼠子宫内膜基质细胞的增殖活性。
本研究经过RNA测序,发现lincRNA AC027700.1在小鼠孕第6 d着床点子宫组织中的表达显著高于着床旁。在后续实验中,本研究成功构建了假孕小鼠体内人工诱导蜕膜化模型和原代小鼠子宫内膜基质细胞体外诱导蜕膜化模型。研究结果显示,AC027700.1在成功诱导蜕膜化的小鼠子宫内膜组织及细胞中的表达显著增高。同时,为了探索AC027700.1的功能,本研究首先对AC027700.1的亚细胞定位进行了探索。iLoc-LncRNA预测结果和qRT-PCR检测细胞核、质RNA中AC027700.1的相对表达水平,发现AC027700.1主要定位于细胞核内。AC027700.1下游靶基因GO和KEGG分析结果提示,AC027700.1下游靶基因可能通过调控自噬、细胞周期以及RNA转运等通路在蜕膜化中发挥调控作用,但仍需通过实验进一步确定。
综上所述,本研究首次发现lincRNA AC027700.1在小鼠孕第6 d着床点子宫组织中高表达,并探讨了AC027700.1在小鼠蜕膜化过程中的表达规律及其亚细胞定位,初步预测和筛选了可能参与调控蜕膜化的下游通路。总之,lincRNA AC027700.1的表达谱鉴定和初步的功能分析强调了lincRNA在早孕子宫内膜蜕膜化过程中的重要性,为胚胎着床的研究提供了新的思路和理论依据,亦为女性妊娠及妊娠相关疾病提供新的标志物及潜在治疗靶点。
[1] Wang Y, Hu SG, Yao GX, Zhu QL, He YQ, Lu Y, Qi J, Xu R, Ding Y, Li JX, Li XY, Sun Y. A novel molecule in human cyclic endometrium: lncRNA TUNAR is involved in embryo implantation., 2020, 11: 587448.
[2] Guerri G, Maniscalchi T, Barati S, Gerli S, Di Renzo GC, Della Morte C, Marceddu G, Casadei A, Laganà AS, Sturla D, Ghezzi F, Garzon S, Unfer V, Bertelli M. Non-syndromic monogenic female infertility., 2019, 90(10-s): 68–74.
[3] Cha J, Sun XF, Dey SK. Mechanisms of implantation: strategies for successful pregnancy., 2012, 18(12): 1754–1767.
[4] Wang HB, Dey SK. Roadmap to embryo implantation: clues from mouse models.,2006, 7(3): 185–199.
[5] Cross JC. The genetics of pre-eclampsia: a feto-placental or maternal problem?, 2003, 64(2): 96–103.
[6] Dey SK, Lim H, Das SK, Reese J, Paria BC, Daikoku T, Wang HB. Molecular cues to implantation., 2004, 25(3): 341–373.
[7] Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs., 2009, 136(4): 629–641.
[8] Derrien T, Johnson R, Bussotti G, Tanzer A, Djebali S, Tilgner H, Guernec G, Martin D, Merkel A, Knowles DG, Lagarde J, Veeravalli L, Ruan XA, Ruan YJ, Lassmann T, Carninci P, Brown JB, Lipovich L, Gonzalez JM, Thomas M, Davis CA, Shiekhattar R, Gingeras TR, Hubbard TJ, Notredame C, Harrow J, Guigó R. The gencode v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression., 2012, 22(9): 1775–1789.
[9] Halley P, Kadakkuzha BM, Faghihi MA, Magistri M, Zeier Z, Khorkova O, Coito C, Hsiao J, Lawrence M, Wahlestedt C. Regulation of the apolipoprotein gene cluster by a long noncoding RNA., 2014, 6(1): 222–230.
[10] Hua Q, Jin MM, Mi BM, Xu F, Li T, Zhao L, Liu JJ, Huang G. Linc01123, a c-Myc-activated long non-coding RNA, promotes proliferation and aerobic glycolysis of non-small cell lung cancer through miR-199a-5p/c-Myc axis., 2019, 12(1): 91
[11] Ji Q, Zhang L, Liu X, Zhou L, Wang W, Han Z, Sui H, Tang Y, Wang Y, Liu N, Ren J, Hou F, Li Q. Long non-coding RNA MALAT1 promotes tumour growth and metastasis in colorectal cancer through binding to SFPQ and releasing oncogene PTBP2 from SFPQ/PTBP2 complex., 2014, 111(4): 736–748.
[12] ENCODE Project Consortium, Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, Margulies EH, Weng ZP, Snyder M, Dermitzakis ET, Thurman RE, Kuehn MS, Taylor CM, Neph S, Koch CM, Asthana S, Malhotra A, Adzhubei I, Greenbaum JA, Andrews RM, Flicek P, Boyle PJ, Cao H, Carter NP, Clelland GK, Davis S, Day N, Dhami P, Dillon SC, Dorschner MO, Fiegler H, Giresi PG, Goldy J, Hawrylycz M, Haydock A, Humbert R, James KD, Johnson BE, Johnson EM, Frum TT, Rosenzweig ER, Karnani N, Lee K, Lefebvre GC, Navas PA, Neri F, Parker SC, Sabo PJ, Sandstrom R, Shafer A, Vetrie D, Weaver M, Wilcox S, Yu M, Collins FS, Dekker J, Lieb JD, Tullius TD, Crawford GE, Sunyaev S, Noble WS, Dunham I, Denoeud F, Reymond A, Kapranov P, Rozowsky J, Zheng DY, Castelo R, Frankish A, Harrow J, Ghosh S, Sandelin A, Hofacker IL, Baertsch R, Keefe D, Dike S, Cheng J, Hirsch HA, Sekinger EA, Lagarde J, Abril JF, Shahab A, Flamm C, Fried C, Hackermüller J, Hertel J, Lindemeyer M, Missal K, Tanzer A, Washietl S, Korbel J, Emanuelsson O, Pedersen JS, Holroyd N, Taylor R, Swarbreck D, Matthews N, Dickson MC, Thomas DJ, Weirauch MT, Gilbert J, Drenkow J, Bell I, Zhao XD, Srinivasan KG, Sung WK, Ooi HS, Chiu KP, Foissac S, Alioto T, Brent M, Pachter L, Tress ML, Valencia A, Choo SW, Choo CY, Ucla C, Manzano C, Wyss C, Cheung E, Clark TG, Brown JB, Ganesh M, Patel S, Tammana H, Chrast J, Henrichsen CN, Kai C, Kawai J, Nagalakshmi U, Wu JQ, Lian Z, Lian J, Newburger P, Zhang XQ, Bickel P, Mattick JS, Carninci P, Hayashizaki Y, Weissman S, Hubbard T, Myers RM, Rogers J, Stadler PF, Lowe TM, Wei CL, Ruan YJ, Struhl K, Gerstein M, Antonarakis SE, Fu YT, Green ED, Karaöz U, Siepel A, Taylor J, Liefer LA, Wetterstrand KA, Good PJ, Feingold EA, Guyer MS, Cooper GM, Asimenos G, Dewey CN, Hou MM, Nikolaev S, Montoya-Burgos JI, Löytynoja A, Whelan S, Pardi F, Massingham T, Huang HY, Zhang NR, Holmes I, Mullikin JC, Ureta-Vidal A, Paten B, Seringhaus M, Church D, Rosenbloom K, Kent WJ, Stone EA, NISC Comparative Sequencing Program, Baylor College of Medicine Human Genome Sequencing Center, Washington University Genome Sequencing Center, Broad Institute, Children's Hospital Oakland Research Institute, Batzoglou S, Goldman N, Hardison RC, Haussler D, Miller W, Sidow A, Trinklein ND, Zhang ZD, Barrera L, Stuart R, King DC, Ameur A, Enroth S, Bieda MC, Kim J, Bhinge AA, Jiang N, Liu J, Yao F, Vega VB, Lee CW, Ng P, Shahab A, Yang AN, Moqtaderi Z, Zhu Z, Xu XQ, Squazzo S, Oberley MJ, Inman D, Singer MA, Richmond TA, Munn KJ, Rada-Iglesias A, Wallerman O, Komorowski J, Fowler JC, Couttet P, Bruce AW, Dovey OM, Ellis PD, Langford CF, Nix DA, Euskirchen G, Hartman S, Urban AE, Kraus P, Van Calcar S, Heintzman N, Kim TH, Wang K, Qu CX, Hon G, Luna R, Glass CK, Rosenfeld MG, Aldred SF, Cooper SJ, Halees A, Lin JM, Shulha HP, Zhang XL, Xu MS, Haidar JN, Yu Y, Ruan YJ, Iyer VR, Green RD, Wadelius C, Farnham PJ, Ren B, Harte RA, Hinrichs AS, Trumbower H, Clawson H, Hillman-Jackson J, Zweig AS, Smith K, Thakkapallayil A, Barber G, Kuhn RM, Karolchik D, Armengol L, Bird CP, de Bakker PIW, Kern AD, Lopez-Bigas N, Martin JD, Stranger BE, Woodroffe A, Davydov E, Dimas A, Eyras E, Hallgrímsdóttir IB, Huppert J, Zody MC, Abecasis GR, Estivill X, Bouffard GG, Guan XB, Hansen NF, Idol JR, Maduro VVB, Maskeri B, McDowell JC, Park M, Thomas PJ, Young AC, Blakesley RW, Muzny DM, Sodergren E, Wheeler DA, Worley KC, Jiang HY, Weinstock GM, Gibbs RA, Graves T, Fulton R, Mardis ER, Wilson RK, Clamp M, Cuff J, Gnerre S, Jaffe DB, Chang JL, Lindblad-Toh K, Lander ES, Koriabine M, Nefedov M, Osoegawa K, Yoshinaga Y, Zhu BL, de Jong PJ. Identification and analysis of functional elements in 1% of the human genome by the encode pilot project., 2007, 447(7146): 799–816.
[13] Ding XY, Jia XM, Wang C, Xu JY, Gao SJ, Lu C. A DHX9-lncRNA-MDM2 interaction regulates cell invasion and angiogenesis of cervical cancer., 2019, 26(9):1750–1765.
[14] Yoon JH, Abdelmohsen K, Kim J, Yang XL, Martindale JL, Tominaga-Yamanaka K, White EJ, Orjalo AV, Rinn JL, Kreft SG, Wilson GM, Gorospe M. Scaffold function of long non-coding RNA HOTAIR in protein ubiquitination., 2013, 4: 2939.
[15] Schmitt AM, Chang HY. Long noncoding RNAs in cancer pathways., 2016, 29(4): 452–463.
[16] Pastori C, Wahlestedt C. Involvement of long noncoding RNAs in diseases affecting the central nervous system., 2012, 9(6): 860–870.
[17] Tang YJ, Zhou T, Yu X, Xue ZX, Shen N. The role of long non-coding RNAs in rheumatic diseases., 2017, 13(11): 657–669.
[18] Li D, Wang DX, Liang JY. The role and mechanism of lncRNA in cardiovascular diseases., 2020, 49(6): 777–783.
李鼎, 王大新, 梁景岩. 长链非编码RNA在心血管疾病中的作用及机制. 华中科技大学学报(医学版), 2020, 49(6): 777–783.
[19] Long J, Yang CS, He JL, Liu XQ, Ding YB, Chen XM, Tong C, Peng C, Wang YX, Gao RF. Foxo3a is essential for murine endometrial decidualization through cell apoptosis during early pregnancy., 2019, 234(4): 4154–4166.
[20] Li R, He JL, Chen XM, Ding YB, Wang YX, Long CL, Shen LJ, Liu XQ. Mmu-miR-193 is involved in embryo implantation in mouse uterus by regulating GRB7 gene expression., 2014, 21(6): 733–742.
[21] Khudayberdiev SA, Zampa F, Rajman M, Schratt G. A comprehensive characterization of the nuclear microRNA repertoire of post-mitotic neurons., 2013, 6: 43.
[22] Zhang XH, Liang X, Liang XH, Wang TS, Qi QR, Deng WB, Sha AG, Yang ZM. The mesenchymal-epithelial transition duringdecidualization., 2013, 20(4): 354–360.
[23] Kopp F, Mendell JT. Functional classification and experimental dissection of long noncoding RNAs., 2018, 172(3): 393–407.
[24] Carson DD, Bagchi I, Dey SK, Enders AC, Fazleabas AT, Lessey BA, Yoshinaga K. Embryo implantation., 2000, 223(2): 217–237
[25] Feng C, Shen JM, Lv PP, Jin M, Wang LQ, Rao JP, Feng L. Construction of implantation failure related lncRNA- mRNA network and identification of lncRNA biomarkers for predicting endometrial receptivity., 2018, 14(10): 1361–1377.
[26] Aghajanova L, Velarde MC, Giudice LC. Altered gene expression profiling in endometrium: evidence for progesterone resistance., 2010, 28(1): 51–58.
[27] Salker M, Teklenburg G, Molokhia M, Lavery S, Trew G, Aojanepong T, Mardon HJ, Lokugamage AU, Rai R, Landles C, Roelen BAJ, Quenby S, Kuijk EW, Kavelaars A, Heijnen CJ, Regan L, Macklon NS, Brosens JJ. Natural selection of human embryos: impaired decidualization of endometrium disables embryo-maternal interactions and causes recurrent pregnancy loss., 2010, 5(4): e10287.
[28] Hirota Y, Daikoku T, Tranguch S, Xie HR, Bradshaw HB, Dey SK. Uterine-specific p53 deficiency confers premature uterine senescence and promotes preterm birth in mice., 2010, 120(3): 803–815.
[29] Han BC, Yang ZM. The characteristic and regulation of endometrial decidualization., 2007, 29(5): 666–670.
韩丙辰, 杨增明. 子宫内膜蜕膜化的特征及其调节因素. 细胞生物学杂志, 2007, 29(5): 666–670.
[30] Khorkova O, Hsiao J, Wahlestedt C. Basic biology and therapeutic implications of lncRNA., 2015, 87: 15–24.
[31] Taylor DH, Chu ETJ, Spektor R, Soloway PD. Long non-coding RNA regulation of reproduction and development., 2015, 82(12): 932–956.
[32] Liu JL, Yang ZM. Non-coding RNAs and embryo implantation., 2011, 3: 1092–1099.
[33] Zhang XY, Xu Y, Fu LL, Li DD, Dai XW, Liu LL, Zhang JS, Zheng LW, Cui MH. Identification of mRNAs related to endometrium function regulated by lncRNA CD36-005 in rat endometrial stromal cells., 2018, 16(1): 96.
The expression of lincRNA AC027700.1 in mouse decidualization
Liping Tan, Rufei Gao, Xin Yin, Xuemei Chen, Fangfang Li, Liu Yuan, Junlin He
Long non-coding RNAs (lncRNAs), which belong to the non-protein-coding RNAs, are greater than 200 nt in length. Although they have been found to play crucial roles in the regulation of cell growth and development, cell metabolism and the development of diseases, they are rarely reported in decidualization. The objective of our study is to explore the expression of lincRNA AC027700.1 in the endometrium of early pregnant mice and its role in decidualization. The expression of AC027700.1 in uterine tissues at implantation sites and inter implantation sites on the 6th day of pregnancy were detected by qRT-PCR. The relative expression of AC027700.1 in anmodel of induced decidualization in pseudopregnant mice and inmodel of induced decidualization in primary stromal cells and nucleus/cytoplasmic fractions were detected by qRT-PCR. GO and KEGG analysis of downstream target genes were performed by GOseq and KOBAS, respectively. The results show that AC027700.1 expression is significantly increased in tissues at implantation sites on the 6th day of pregnancy and in decidualized endometrial tissues and stromal cells. Furthermore, AC027700.1 localizes in the nuclear fraction and the downstream targeted genes are mainly involved in autophagy, cell cycle and RNA transport pathways. This study revealed that lincRNA AC027700.1 may be involved in decidualization of endometrium in early pregnancy, but the specific role and regulatory mechanism remain to be further studied.
lincRNA; AC027700.1; decidualization; endometrium; implantation
2021-10-15;
2022-01-17;
2022-01-24
国家自然科学基金项目(编号:31571554),重庆市自然科学基金面上项目(编号:cstc2020jcy j-msxmX0041)和重庆市研究生导师团队建设项目(编号:dstd201809)资助[Supported by the National Natural Science Foundation of China (No.31571554), General Project of Chongqing Natural Science Foundation (No.cstc2020jcy j-msxmX0041) and Postgraduate Tutor Team Construction Project of Chongqing (No.dstd201809)]
谭丽萍,在读硕士研究生,专业方向:生殖遗传学。E-mail: 2957066450@qq.com
何俊琳,博士生导师,研究方向:胚胎着床分子机理,雌性生殖损伤机制。E-mail: hejunlin@cqmu.edu.cn
10.16288/j.yczz.21-356
(责任编委: 单革)
