天人菊matK基因克隆及生物信息学分析
2022-02-12徐丽娟
徐丽娟,熊 勇
(云南民族大学 民族医药学院,云南 昆明 650500)
天人菊(GaillardiapulchellaFoug.)属于菊科(Asteraceae)天人菊属(Gaillardia)植物,别名虎皮菊、老虎皮菊,原产北美,中国广泛栽培并归化[1].天人菊作为推广的绿化景观植物,抗旱能力强,是良好的防风固沙植物.研究和培育耐旱、耐贫瘠的天人菊,不仅可以节约资源成本,扩大城市绿化面积, 还能提高生态效益和社会效益[2-3].
叶绿体中的matK(maturase kinase)基因进化速度快、具有高度多态性,是植物物种进化和鉴定过程中的一种重要的分子标记[4].研究表明,matK基因快速进化过程中,会存在碱基替换、碱基缺失和插入突变,从而导致遗传变异,这也是物种进化的重要动力[5-7].基因组测序数据从核苷酸水平上提供了变异和突变的详细信息,通过同源性比较,可进行物种鉴定和判断基因组中是否存在特殊突变从而影响其表型改变[8].
植物的系统进化分析和亲缘关系鉴定一直是植物学的研究热点.菊科植物进化过程中存在杂交和分化等特点,使得其科内种类繁多,其系统发育关系的建立需要大量的遗传信息数据支持[9].其中,天人菊的具体基因信息研究较少,有待进一步的完善.本研究通基因克隆技术,获取完整的天人菊基因matK信息,为后续天人菊的分子生物学研究提供数据.再通过一系列的生物信息学方法将天人菊的氨基酸序列与同源性较高的12条氨基酸序列进行比较,从遗传进化角度探讨天人菊matK基因序列变异对其蛋白分子功能和生物学过程的影响,进一步探索matK基因的特征与进化规律,评估叶绿体基因组对于植物系统进化研究的贡献.
1 材料与方法
1.1 实验材料
8月份获取天人菊种子,采摘地点为云南省呈贡区,于次年4月初在实验室进行天人菊种子萌发培养,20~24 ℃ 条件下10~15 d 出苗[10],待发芽后将幼苗移栽到盆中,长出嫩叶时,方可采摘天人菊嫩叶部分进行DNA提取.
根据相关文献[11],设计3对引物用于扩增天人菊matK基因的完整基因,具体的引物信息如表1所示.

表1 引物信息
1.2 试验方法
1.2.1 天人菊DNA提取及检测
将天人菊嫩叶清洗干净,加入液氮快速研磨使细胞破碎,用改良SDS法提取DNA,然后用1.5%琼脂糖凝胶电泳检测DNA质量及纯度[12].
1.2.2 天人菊matK基因PCR扩增
反应体系及反应条件如下表2所示,待反应结束后,各取产物 8 μL 于1.5%胶进行琼脂糖凝胶电泳检测[13].

表2 PCR反应体系及反应条件的设定
1.2.3 PCR产物回收、克隆及测序
用多功能DNA纯化回收试剂盒对PCR扩增产物进行胶回收纯化,用Pbm16A pBM16A-ToPo Cloning Smart Kit试剂盒进行克隆,将回收的DNA片段连接到pBM16A-ToPo载体上,室温下(20~30 ℃)反应5~30 min,取连接产物转化到融化的DH5α感受态细胞中,加入无菌的不含抗生素的LB于 37 ℃ 下培养,再将菌液涂布在含有IPTG,X-gal的LB氨苄平板上,挑选克隆子,菌落PCR方法鉴定阳性克隆,用M13通用引物对阳性质粒进行测序分析[14].
1.3 数据分析
1.3.1 测序结果处理
采用MEGA 6.0将天人菊matK基因碱基序列翻译成蛋白质氨基酸序列,在NCBI中进行blast比较,获得到了12条同源性较高的氨基酸序列.
1.3.2 生物信息学分析
利用下表3中的一系列生物信息学软件,对实验中所有的基因序列进行分析.
表3 生物信息学分析软件
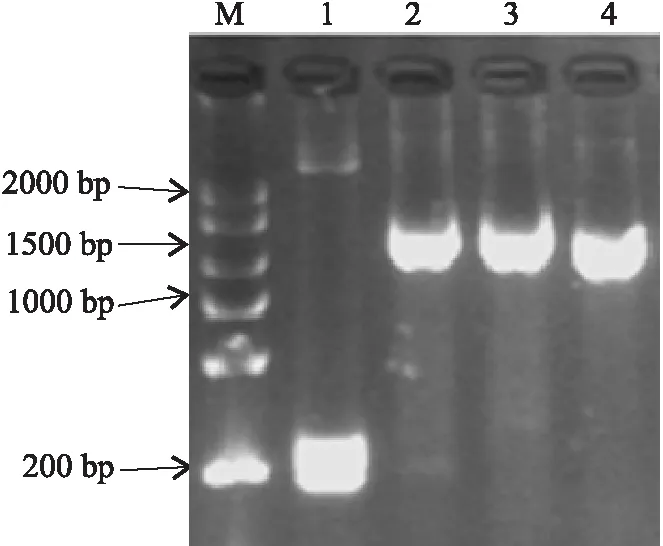
图1 重组质粒M13PCR检测电泳结果
2 结果与分析
2.1 天人菊matK基因的基因克隆
重组质粒M13PCR检测电泳结果如图1所示(M为DNA标准物DL2000+1500;1为空载质粒;2~4为引物1、2、3重组质粒M13 PCR检测结果),空载质粒PCR条带大小约为 200 bp,天人菊重组质粒PCR产物条带大小约为 1 000~1 500 bp[5],说明转化成功,2、3、4处的条带为目标基因,对重组子PCR产物进行测序,获取重组子的基因序列.
2.2 测序结果分析
经测序获取天人菊碱基序列 2 631 bp(见图2),利用在线工具ORFfinder-NCBI分析,结果显示matK基因 801 bp 的ATG为起始密码子,2 096 bp 的TGA为终止密码子,总长度为 1 296 bp,获得432个氨基酸序列(包含一个终止子),如下图3所示.

图2 天人菊matK基因碱基序列

图3 天人菊matK基因氨基酸序列
2.3 生物信息学分析
2.3.1 天人菊matK基因氨基酸序列二级结构
应用软件包DNAStarLasergene 7.1中protean分析天人菊matK蛋白二级结构,结果如图4所示,采用Chou-Fasman预测方法得到该氨基酸序列约有12个α-螺旋、20个β-折叠和31个无规则卷曲组成.
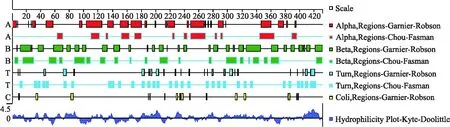
图4 matK蛋白二级结构组成
使用SOPMA软件对13条matK蛋白进行分析(图5),结果显示构成α-螺旋的氨基酸数量最多,占总氨基酸数量的42.89%~50.59%;无规则卷曲数量次之,占26.74%~32.02%;延伸链占比18.18%~22.40%;最少的是β-转角,仅有3.36%~5.59%.
2.3.2 天人菊matK基因氨基酸序列三级结构
应用Swiss-Model对天人菊matK蛋白构建三级结构[15-16],其序列一致性为17.77%,下载PDB格式,并在PyMOL中打开,三级结构模型见图6.matK蛋白三级模型的拉氏图(见图7).图中A,B,L区显示有273个氨基酸,占总氨基酸数的70.4%; a,b,l,p区显示有98个氨基酸,占25.3%;允许区为8个,非允许区为9个;末端残基(不包括Gly和Pro)为2个;甘氨酸残基16个;脯氨酸残基12个.
2.3.3 天人菊matK氨基酸序列的理化特征分析
使用在线软件PredictProtein分析天人菊matK基因的亚细胞定位(见图8),图中绿色部分为叶绿体亚细胞定位,发现matK基因只存在于真核生物的叶绿体中,符合目前matK基因的相关研究结果[17].
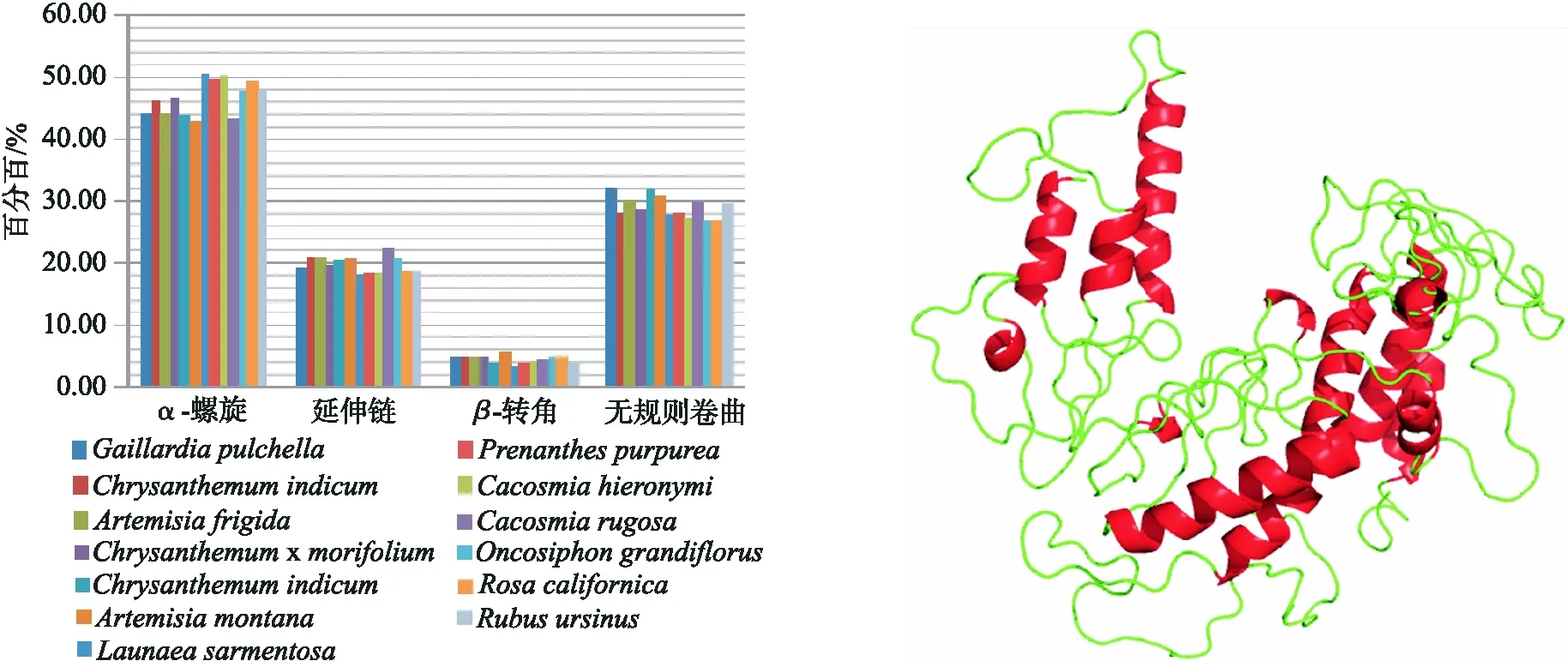
图5 天人菊matK蛋白与其他12种蛋白的二级结构 图6 matK蛋白三级结构
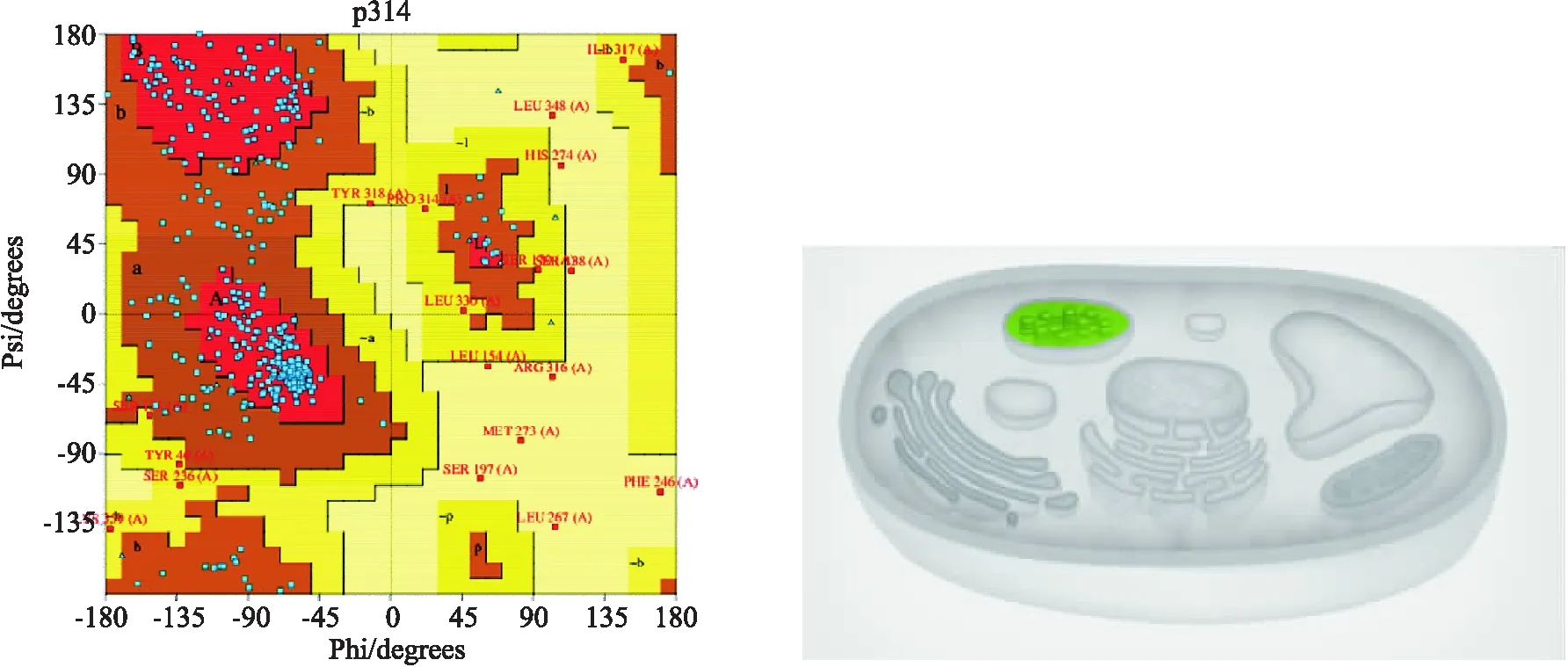
图7 matK蛋白三级模型的拉氏图 图8 matK蛋白进行亚细胞定位结果
靶位点分析表明有7个蛋白结合区,分别位于83、114、182、202、203、325、406、426位氨基酸,无多核苷酸结合区(图9).
利用软件Protparam和TMHMM2.0[18-19],对天人菊与其他12种不同蛋白的理化性质进行比较,表4显示,matK基因全长为 1 290 ~1 500 bp,编码430~500个氨基酸,理论等电点为大于9.6,不稳定指数大于40,平均疏水性为负值,matK蛋白是碱性不稳定亲水性蛋白,无跨膜结构,无信号肽,亚细胞定位于真核生物的叶绿体中.
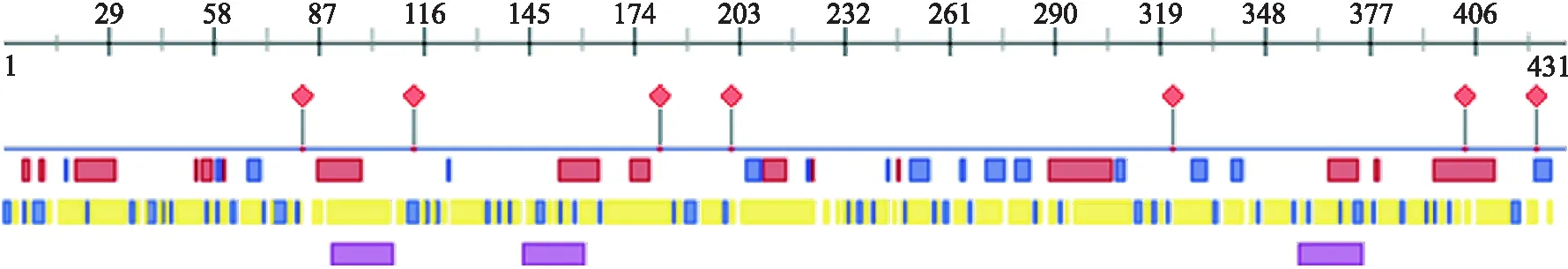
图9 matK蛋白氨基酸序列结合区结果
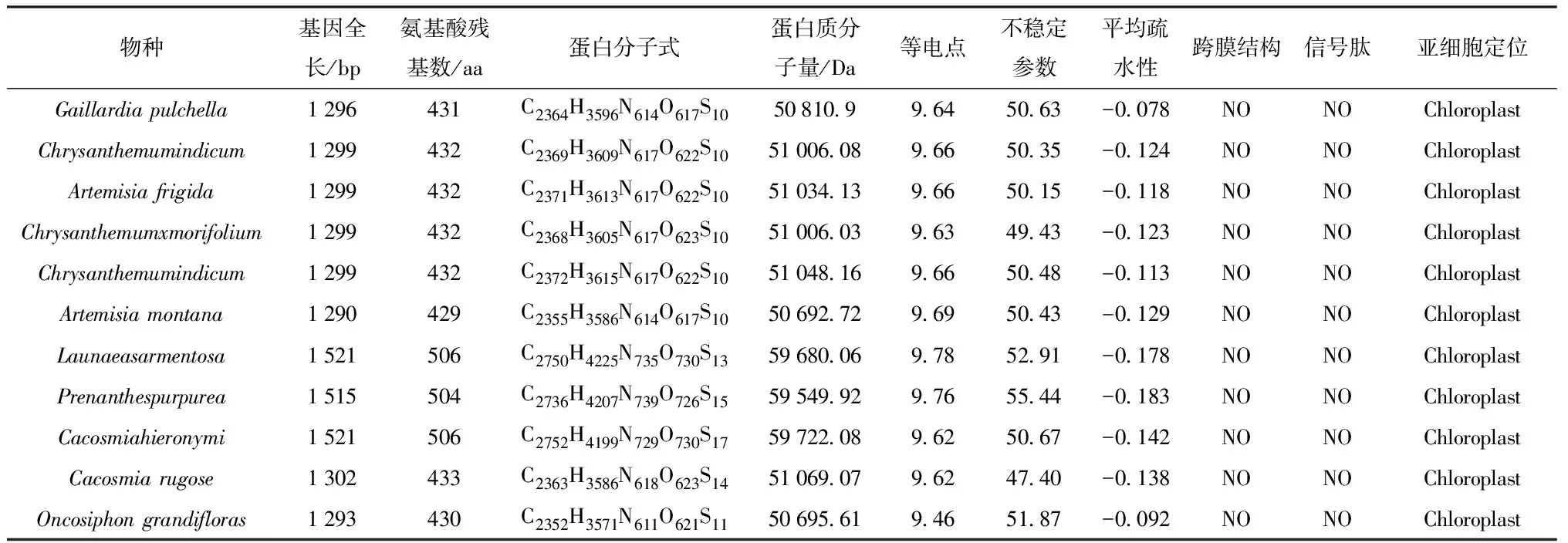
表4 天人菊matK蛋白与其他12种蛋白的理化特征
2.3.4 同源性分析
天人菊matK蛋白保守结构域预测分析见图10.图10表明,本实验所克隆的天人菊matK蛋白属于matK家族.将天人菊matK氨基酸序列输入NCBI进行blast 比对,与之相似性在96%以上的基因皆来自于菊科植物,从遗传角度说明matK基因在菊科植物中具有一定的保守性,得到该蛋白的同源性(见表5).
通过BLAST获取氨基酸序列,选择同源性排序较高的菊科物种8个(Chrysanthemumindicum,Chrysanthemumxmorifolium,Chrysanthemumindicum,Artemisiafrigida,Artemisiamontana,Launaeasarmentosa,Prenanthespurpurea,Oncosiphongrandifloras),和同源性排序较高的非菊科物种4个(Cacosmiahieronymi,Cacosmiarugose,Rosacalifornica,Rubusursinus),用于鉴定天人菊的亲缘关系.

图10 天人菊matK蛋白保守结构域预测分析

表5 天人菊matK基因氨基酸同源性比较
利用MEGA 6.0软件将天人菊matK氨基酸序列与12种氨基酸序列一起构建N~J系统进化树(图11).并在表6中展示了它们之间的遗传距离,其中,天人菊与野菊花的遗传距离最近为0.006,与加州玫瑰的遗传距离最远为0.032.图11分类显示,第1大类是天人菊matK氨基酸序列与3个菊属、2个艾属及1个大翅蓟属的matK氨基酸序列很好地聚成了一类,这6个菊科物种聚为一类符合形态学的分类研究结果[9],其中,大翅蓟属下的Oncosiphon grandifloras(遗传距离为0.009)在第1大类下单独成为一个分支.第2大类由遗传距离同为0.013的蔓茎栓果菊和盘果菊组成一支与另一支遗传距离同为0.014的Cacosmiahieronymi和Cacosmia rugose 2个物种一起构成姐妹类群关系,表明同一科内物种的属间聚类并不一致,因此在构建系统进化树时不能只选取单个基因序列或2个基因序列,而应选择多个属间、属内物种共同构建系统进化树,才能使鉴定结果更准确.第3大类为遗传距离较远的加州玫瑰和北美黑莓聚为一类.分类结果证实了实验获得的天人菊matK基因归属于菊科植物,还表明了matK氨基酸序列在不同的生物种类中具有高度的保守性[5],因此才能在叶绿体中能表达出相似的生物学功能.

图11 天人菊与12个物种matK氨基酸序列的系统进化树
2.3.5 天人菊matK基因氨基酸变异位点分析
使用Clustal X 1.83软件和MEGA 6.0软件对天人菊matK基因氨基酸序列和其他12条氨基酸序列进行比对,图12结果显示,总共有163处差异氨基酸.其中,天人菊与遗传距离较近的5条菊科植物氨基酸序列(Chrysanthemumindicum、Artemisiafrigida、Chrysanthemumxmorifolium、Chrysanthemumindicum、Artemisiamontana)比对,发现天人菊在第14和15位缺失2个谷氨酰胺(Q),而在第80、81和82位多出了苏氨酸(T)、脯氨酸(P)、苯丙氨酸(F)这3个氨基酸,对比发现,有且仅有15处差异氨基酸,且基因的5′端氨基酸变异频率低于3′端,说明matK基因在属间和属内差异性不大.而在相隔较远的加州玫瑰(Rosacalifornica)和北美黑莓(Rubusursinus)中,存在138处差异氨基酸,说明matK基因在不同科的生物中存在一定程度的分化.
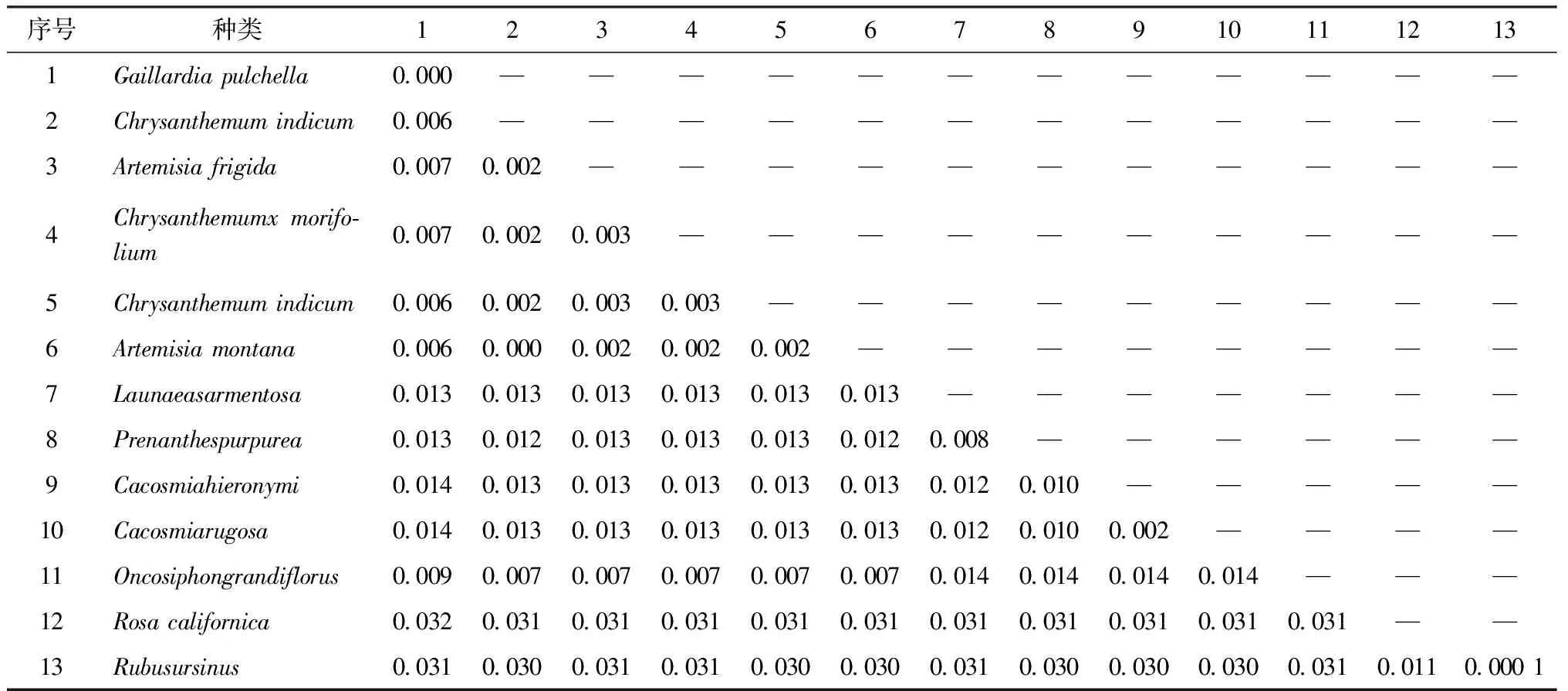
表6 天人菊与其他12种不同基因之间的遗传距离
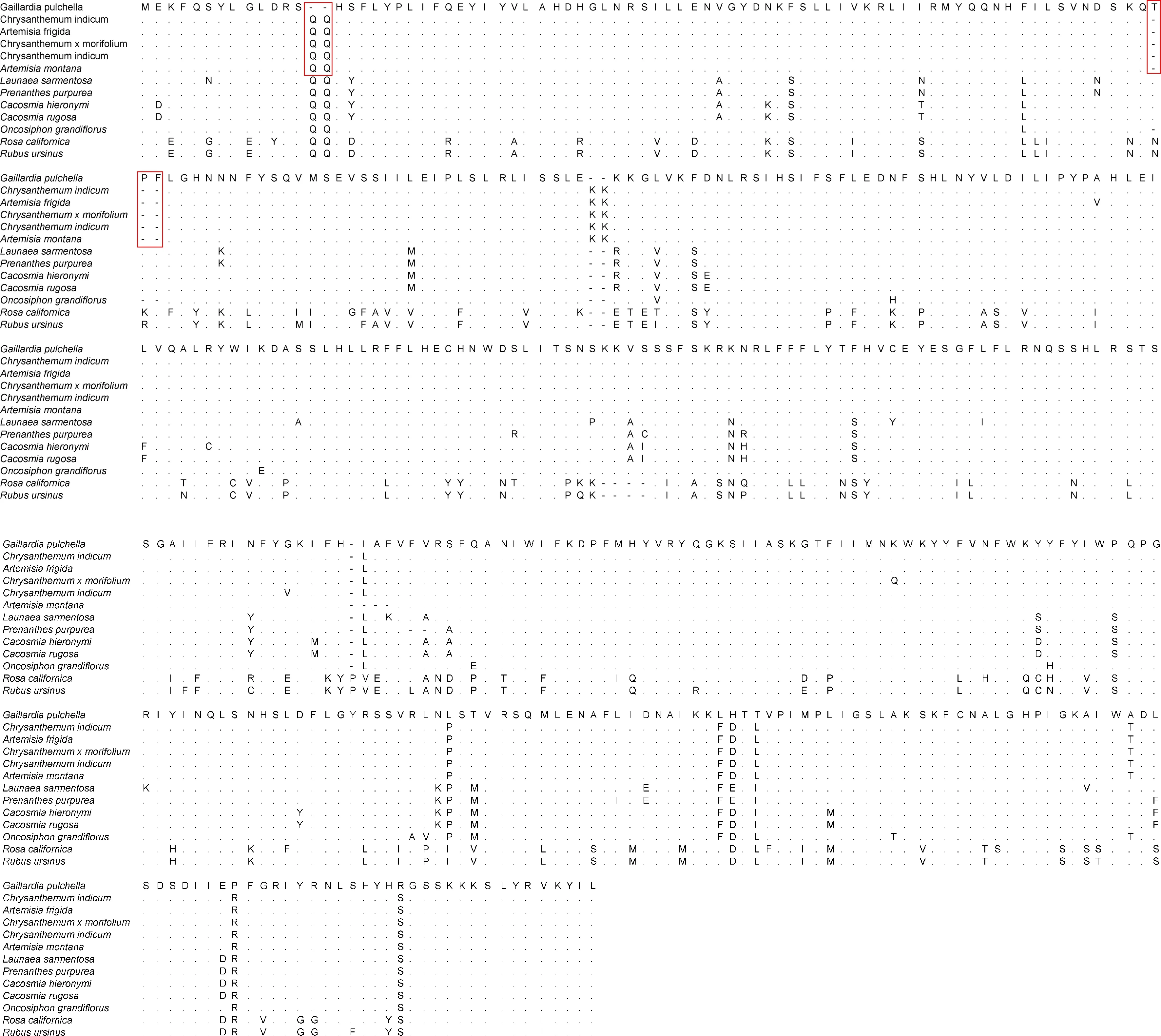
图12 天人菊matK基因氨基酸变异位点分析
2.3.6matK蛋白基因本体论分析
通过PredictProtein软件分别对天人菊matK蛋白以及其他12种来自不同物种的matK蛋白进行分析[20],其分子功能(表7)和生物学过程(表8)的可靠度区间为60%~70%,说明matK蛋白功能在不同生物中具有较高的一致性.分子功能表现为结合活性,包括:杂环化合物结合、有机环化合物结合、核酸结合、RNA结合.生物学过程包括:细胞过程、细胞代谢过程、细胞氮化合物代谢过程、细胞内芳香族化合物代谢过程、代谢过程、主要代谢过程、杂环代谢过程、含核酸的化合物代谢过程、氮化合物代谢过程、有机物代谢过程、有机环化合物代谢过程、大分子代谢过程、核酸代谢过程、基因表达、RNA剪接、RNA过程、RNA代谢过程、ncRNA过程、ncRNA代谢过程、mRNA过程、mRNA代谢过程、tRNA过程、tRNA代谢过程.matK蛋白参与了众多生物学过程,说明它在叶绿体执行功能的过程中扮演着极其重要的角色[21].
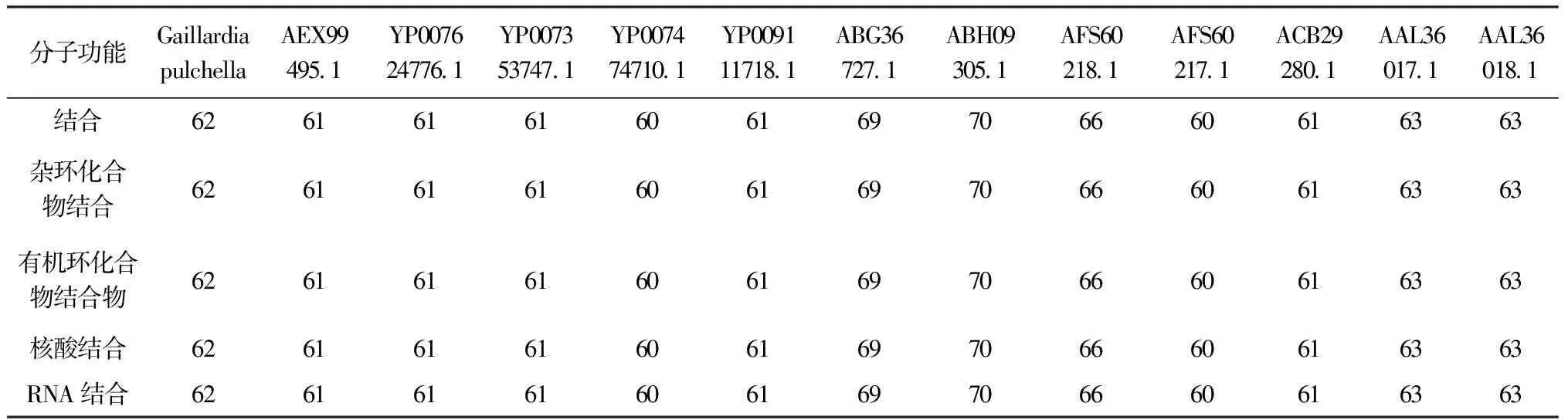
表7 天人菊matK蛋白分子功能本体分析及可靠度 %
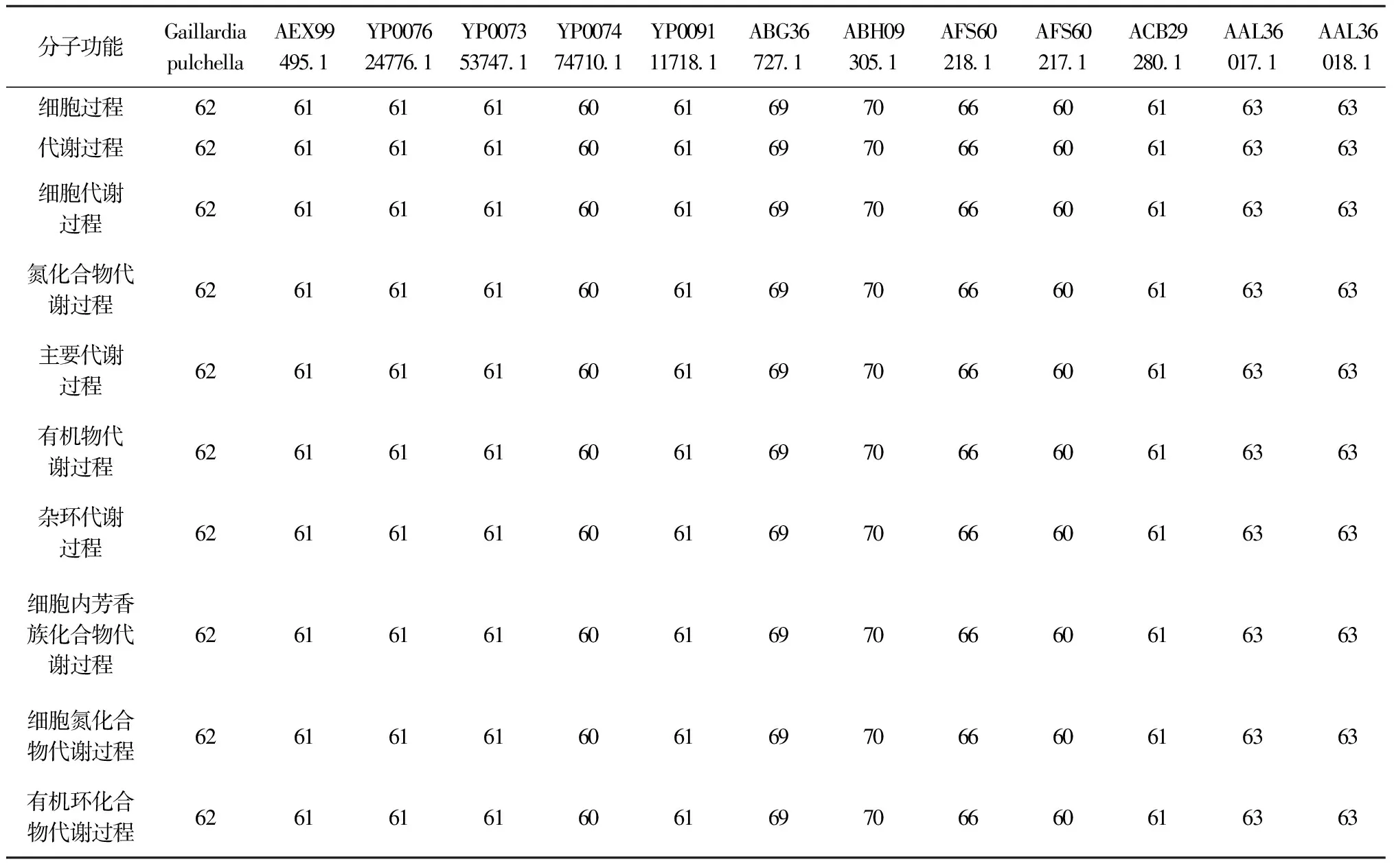
表8 天人菊matK蛋白生物学过程本体分析及可靠度 %
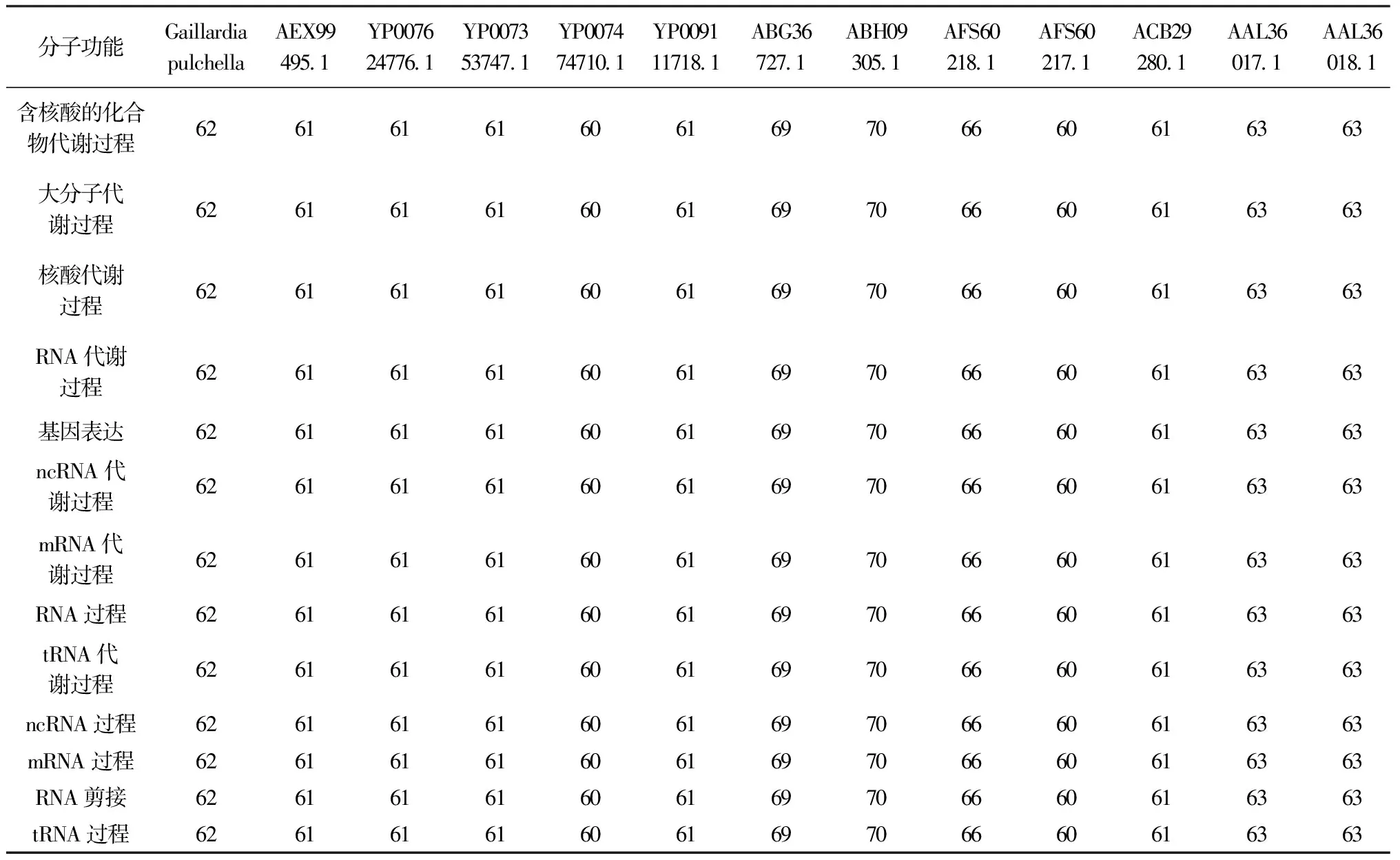
续表8
3 讨论
天人菊具有较强的抗旱性,能够适应山地及荒野等贫瘠土地.目前,对天人菊的研究主要集中于种子萌发、抗旱性评价、栽培管理等方面.潘立雪等[22]对天人菊种子盐碱胁迫的耐受性研究,以及抗寒性天人菊品种的育种研究,都是从宏观进化的层面来选育天人菊,若从微观进化的层面着手,可探索不同地区、不同抗寒抗旱能力的天人菊基因变化规律,追踪其分子功能和生物学过程,为优化育种和种植资源保护提供新思路.本研究通过对天人菊matK基因克隆并测序,获得了一条总长度为 1 296 bp的核苷酸序列,编码432个氨基酸,并建立其三级结构模型,为后续研究其他品种天人菊基因并进行遗传变异的规律性探究提供了数据支撑.将天人菊与其他12种不同matK蛋白进行比较,得知天人菊matK氨基酸序列与遗传距离较近的菊科植物相比,存在15处氨基酸变异位点,科内氨基酸变异位点较少.唐萍等[6]研究通过indels和核苷酸替代方式可实现基因突变,假设优化天人菊育种时,寻找优良品种的变异位点,人为定向干预天人菊基因突变,使天人菊性状随之发生变化,最终可达到筛选优良品种的目的.而天人菊与遗传距离较远的玫瑰等其他的生物相比,氨基酸变异位点大于30%,与以往研究结果一致,表明matK氨基酸序列在不同的生物中存在一定程度的分化[21].
植物的matK基因作为叶绿体基因组中的重要基因,是植物DNA条形码中的核心序列,广泛的用于植物的物种亲缘关系研究和物种的鉴定,matK基因结构相对保守,在同属类植物中同源性较高而进化速率较慢,可应用于下一级种的分类与鉴定[23].朱斌等[9]用CDS、rbcL基因序列、rbcL+matK基因序列对20个属50种菊科植物亲缘关系进行了分析.黄琼林等[24]将matK基因用于有效地鉴别高良姜及其同属混伪品.戚华沙等[25]利用trnH-psbA序列和matK序列结合对普通油茶和越南油茶与其他供试油茶物种进行了有效的鉴别.菊科是双子叶植物纲下面的第一大科,其科内等级划分和系统学研究存在巨大挑战[9],陈芙蓉等[26]将psbA-trnH+matK+trnL组合序列作为DNA条形码,建立了一种快速鉴定野菊和药用菊的方法.本研究在天人菊中克隆得到的matK基因,可有效应用于天人菊属的下一级种的分类研究,解决天人菊种植资源鉴定问题,为天人菊的分子鉴定提供了数据,为天人菊育种提供了新思路,也为菊科植物分子生物学研究提供了相关的理论依据.
