环境DNA方法对进口亲虾携带水中宿主物种的鉴定
2022-02-08谢艳辉郑舒尹李家侨
张 娜 , 刘 骁 , 郑 枢 , 谢艳辉 , 刘 荭 , 郑舒尹 , 李家侨
(1.湛江海关技术中心 , 广东 湛江 524000 ; 2.湛江海关 , 广东 湛江 524000;3.深圳海关动植物检验检疫技术中心 , 广东 深圳 518045)
环境DNA(Environmental DNA,eDNA)是环境样本中发现的,许多不同生物体基因组DNA的复杂混合物[1]。环境样本包括土壤、沉淀物、水和粪便等,也包括过滤空气或水、过滤沉积物或大块状样品所产生的物质。eDNA的研究对象是环境样本中提取的DNA,基于环境样本DNA的收集为所研究的体系获得尽可能全面的分类或功能信息[2]。传统的生物多样性评估方法是利用捕获或记录的个体来确定生物的存在或丰度,与直接取样的传统生物多样性评估方法不同,eDNA检测方法具有非侵入性和敏感性。一些研究证实,eDNA已经成功应用于单一物种生物入侵、难以捕获或威胁性较大的动物物种监测中[3-4]。eDNA检测方法在检测濒危、低密度入侵的生物监测时比传统的生物监测方法更准确、更灵敏[5-7]。Biggs等采用eDNA方法对英国濒危大冠蝾螈的监控效率比传统方法高出许多,并且不会产生假阳性[8]。eDNA研究具有易野外取样和能探测隐秘物种等优点,对于广泛直接取样的生态系统监测来说是一种有吸引力的替代方法[9-10]。
种虾入境不仅具有外来物种入侵风险,而且存在传播疫病风险,容易给养殖户及企业造成重大经济损失。养殖水作为水生动物生存的重要环境因素,也可成为一种外来物种入侵和疫病生物风险因素之一。本试验以进口亲虾携带水作为研究对象,运用eDNA检测方法进行凡纳滨对虾物种检测分析,并对携带水进行宏基因组测序,分析进口亲虾携带水中凡纳滨对虾物种含量,为水生动物外来物种入侵技术的探讨提供基础数据,为eDNA技术在检测外来物种入侵中的应用提供一定的科学依据。
1 材料与方法
1.1 试验材料 进境亲虾及水样品,由本实验室保存;DNeasy Blood and Tissue Kit,购自德国QIAGEN有限公司;ExTaq酶,购自宝生物工程(大连)有限公司;硝酸纤维素膜、玻璃纤维膜和聚碳酸酯膜,均购自北京优尼康生物科技有限公司;PCR管,购自爱思进生物技术(杭州)有限公司;UltraTMDNA Library Prep Kit,购自美国NEB生物公司;GenNextTMNGS Library Quantification Kit,购自日本东洋纺生物科技有限公司。
1.2 主要仪器设备 PCR仪(ABI ProFlex,美国ABI),高速冷冻离心机(Eppendorf 5417R,德国艾本德),真空泵(GM-0-33A,天津津腾实验设备有限公司),Qubit荧光仪(Qubit 4,美国Invitrogen),超声波基因组剪切仪 (M220,美国Covaris)。
1.3 试验方法
1.3.1 水样品eDNA收集和DNA提取 以进口亲虾携带水样为研究对象,每个水样取1~2 L,使用孔径为0.45 μm的过滤膜,采用无菌真空抽滤装置进行过滤。每次过滤之前用无菌水、0.5 mol/L氢氧化钠、3%次氯酸钠冲洗过滤漏斗,防止交叉污染。过滤完成后,将每张过滤膜单独放入1.5 mL离心管中,-20 ℃保存备用。
按照DNeasy Blood and Tissue Kit试剂盒说明书提取 DNA,为了利于过滤膜中的DNA释放,对样品处理稍加改进。将过滤膜取出解冻后,用剪刀将过滤膜剪成细条状,放入 2 mL 的离心管中,加入试剂盒中裂解液 ATL 550 μL和蛋白酶 K 80 μL,涡旋混匀,恒温水浴 3 h (期间每隔 15 min 轻轻颠倒混匀);加入试剂盒中Buffer AL 600 μL,涡旋振荡混匀。之后的操作步骤按照试剂盒说明书进行。提取的核酸置于-20 ℃备用。
1.3.2 不同过滤膜、不同样本体积对于eDNA获取的影响 以20个进口亲虾携带水样为研究对象,分别取硝酸纤维素膜、玻璃纤维膜和聚碳酸酯膜3种过滤膜进行过滤,每种过滤膜的孔径均为0.45 μm。每个样本、每个过滤膜分别过滤水样1 L、2 L,对应每个过滤膜分别做2个重复。过滤后的过滤膜按照1.3.1方法进行DNA提取,通过Quibt荧光计测定DNA浓度。
1.3.3 凡纳滨对虾宿主物种鉴定 在 GenBank 数据库中检索凡纳滨对虾 EF584003.1线粒体保守基因序列,使用 Primer Premier 6软件设计引物。引物由宝生物工程(大连)有限公司合成,预计扩增片段为347 bp。引物序列为:F2:5′-AACAGCCCTCACTA-TTTCAAG-3′;R2:5′-ATCGTTGCGTTGGATAAAATG-3′。
提取凡纳滨对虾组织 DNA进行PCR扩增。扩增体系:10×PCR buffer(Mg2+Plus) 2.5 μL,上、下游引物(10 μmol/L)各1 μL,dNTP(10 mmol/L)2 μL,TaqDNA聚合酶0.5 μL,DNA 5 μL,加水至25 μL。扩增程序:94 ℃ 3 min;94 ℃ 1 min,58 ℃ 1 min,72 ℃ 1 min,35个循环;72 ℃ 10 min。将10 μL PCR扩增产物经1%的琼脂糖凝胶电泳,切下目的条带送生工生物工程(上海)股份有限公司进行目的条带测序和质粒构建。目的条带序列在NCBI网站进行比对分析,确定为凡纳滨对虾 EF584003.1线粒体基因序列,质粒放置于-20 ℃保存备用。
1.3.4 不同来源水样中的宿主物种鉴定比对 设立3个不同来源的水样进行比对试验,分别为:成虾养殖池塘水样(10份样品)、进口亲虾携带水水样(10份样品)、亲虾养殖池塘水样(10份样品)。每份水样分别取1.5 L并用硝酸纤维膜过滤,提取过滤膜DNA,按照1.3.3方法进行宿主物种鉴定。
1.3.5 不同过滤膜中的宿主物种鉴定比对 根据1.3.2结果分析,聚碳酸酯膜过滤易发生堵塞,过滤水样体积达不到1.5 L,所以本阶段过滤分别使用硝酸纤维素膜和玻璃纤维膜对20份进口亲虾携带水样进行过滤,每个样品过滤1.5 L,按照1.3.3方法进行宿主物种鉴定。
1.3.6 不同取样时间的宿主物种鉴定比对 取4份进口亲虾水样(设为水样1、水样2、水样3、水样4),每份水样设置2个重复,每份水样分别是放置0 h、0.5 d、1 d、2 d、3 d、4 d、5 d和6 d后过滤。按1.3.1方法提取过滤膜DNA,通过1%琼脂糖凝胶方法观察DNA降解情况。按1.3.3方法进行宿主物种鉴定。
1.3.7 水过滤膜eDNA的宏基因组测序分析 对进口亲虾携带水过滤膜提取的DNA使用Quibt荧光计进行浓度测定,送往广州美格生物科技有限公司进行宏基因组测序。取质量合格的样品构建基因文库。利用超声波基因组剪切仪将各基因组 DNA 样本随机打断成300 bp左右的片段,随后进行文库构建。进行数据分析,首先对原始序列进行去接头、质量剪切以及去除污染等优化处理,然后使用优质序列进行拼接组装和基因预测,对得到的基因进行物种和功能上的注释以及分类。
2 结果
2.1 不同过滤膜、不同样本体积对于eDNA获取的影响 分别用硝酸纤维素膜、玻璃纤维膜、聚碳酸酯膜3种过滤膜过滤水样1 L、2 L。结果如图1和图2所示,使用硝酸纤维素膜过滤水样获得的eDNA的浓度大于相同体积的玻璃纤维膜、聚碳酸酯膜。在过滤过程中玻璃纤维膜过滤速度最快,而聚碳酸酯膜过滤最慢,并且容易发生堵塞。在过滤2 L水样时,聚碳酸酯膜发生堵塞的情况最多,影响后续试验结果。从结果还可以看出,过滤2 L水样比过滤1 L水样时获得的eDNA的浓度高,但是低于1 L水样得到的eDNA浓度的2倍,说明水样中eDNA的获取达不到与体积成正比关系,会受到过滤膜中DNA提取、水样过滤时损失等因素的影响。
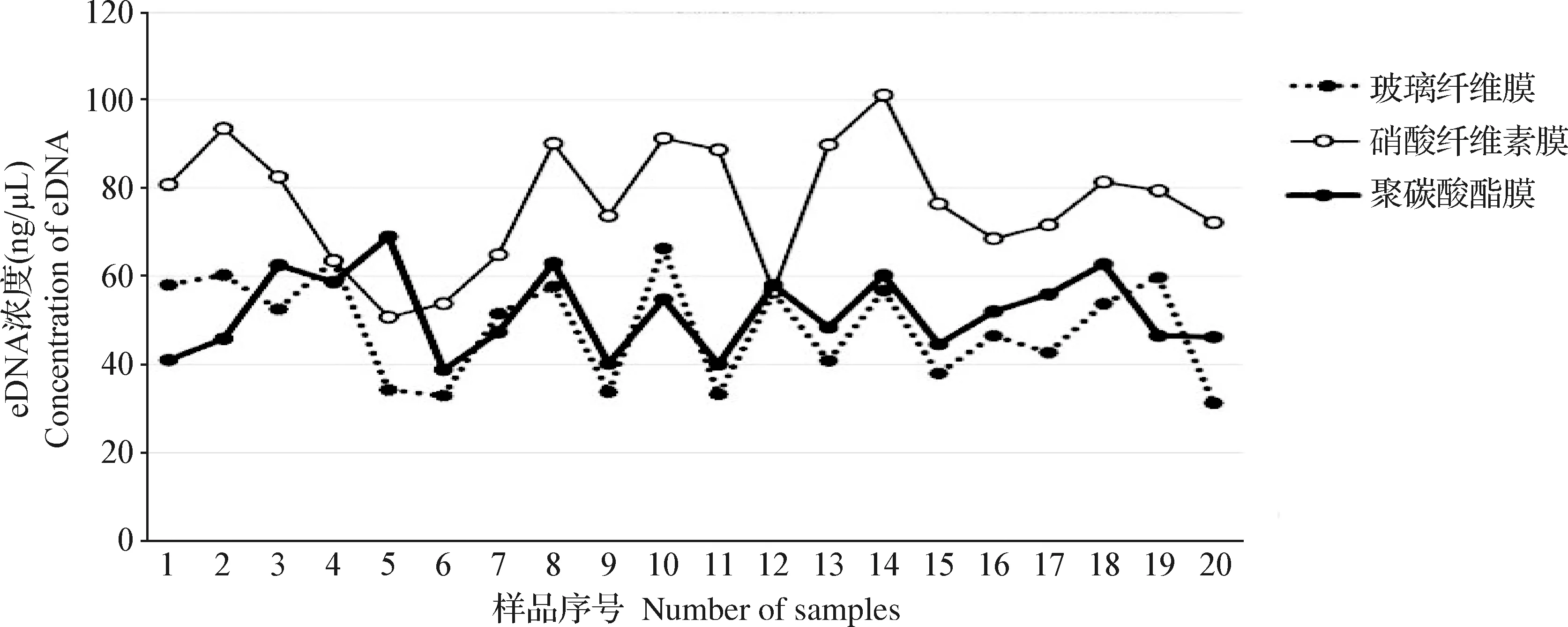
图1 3种过滤膜分别过滤1 L亲虾携带水的eDNA浓度对比 Fig.1 Comparison of eDNA concentration in 1 L parent shrimp carrying water filtered by three kinds of filtration membranes
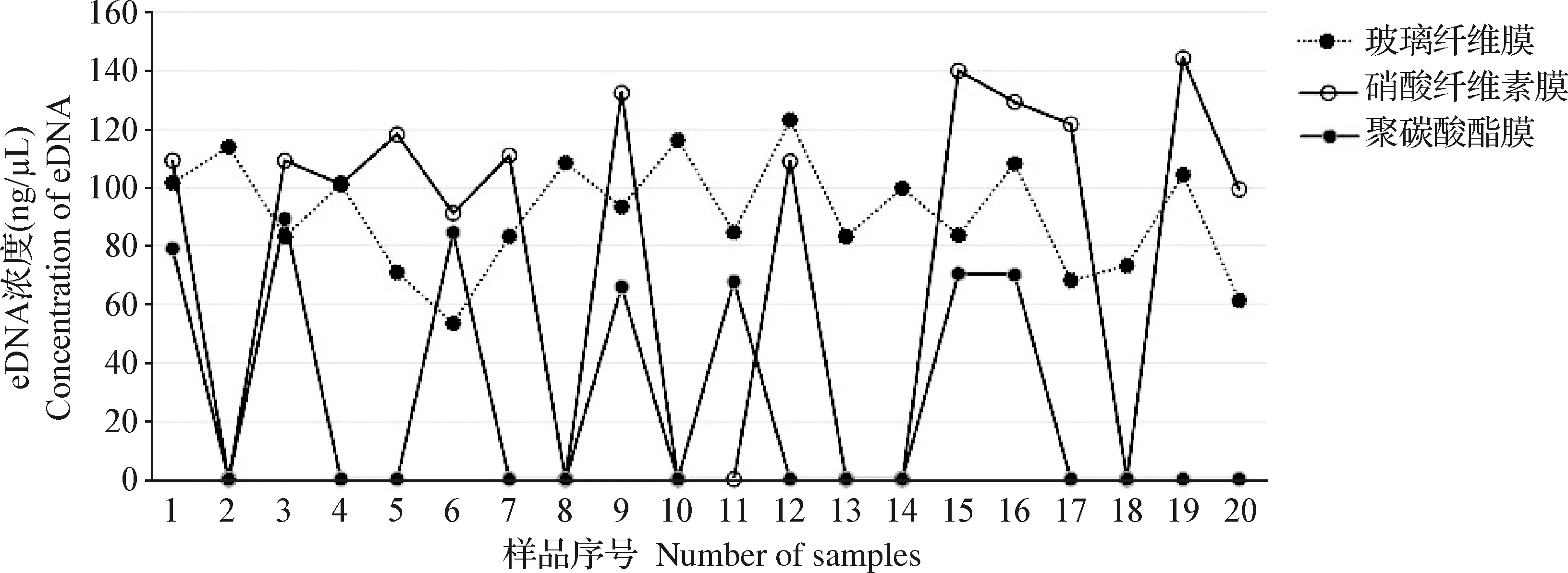
图2 用3种过滤膜分别过滤2 L亲虾携带水的eDNA浓度对比 Fig.2 Comparison of eDNA concentration in 2 L parent shrimp carrying water filtered by three kinds of filtration membranes
2.2 不同来源水样中的宿主物种鉴定比对 如图3所示,本试验成功地从10份成虾养殖池塘水样中扩增出347 bp的特异性目的片段,目的条带单一且明亮,与预期大小一致;如图4所示,10份亲虾养殖池塘水样(饲养超过15 d)中有8份水样成功扩增出特异性目的片段;如图5所示,10份进口亲虾携带水水样中有4份水样成功扩增出特异性目的片段。测序结果在 NCBI 网站上进行比对分析,结果显示,扩增条带的序列与EF584003.1线粒体基因片段同源性达100%。
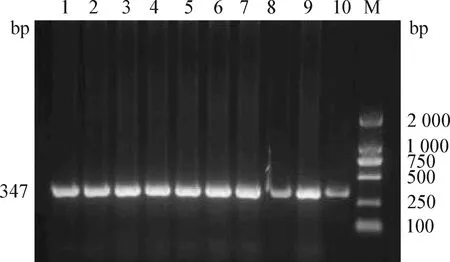
图3 成虾养殖池塘水样凡纳滨对虾物种鉴定Fig.3 Species identification of Penaeus vannamei in water samples from adult shrimp culture ponds1~10:10个成虾养殖池塘水样鉴定的PCR扩增产物; M:DL-2 000 DNA Marker1-10:PCR amplification products of 10 water samples from adult shrimp culture ponds; M:DL-2 000 DNA Marker
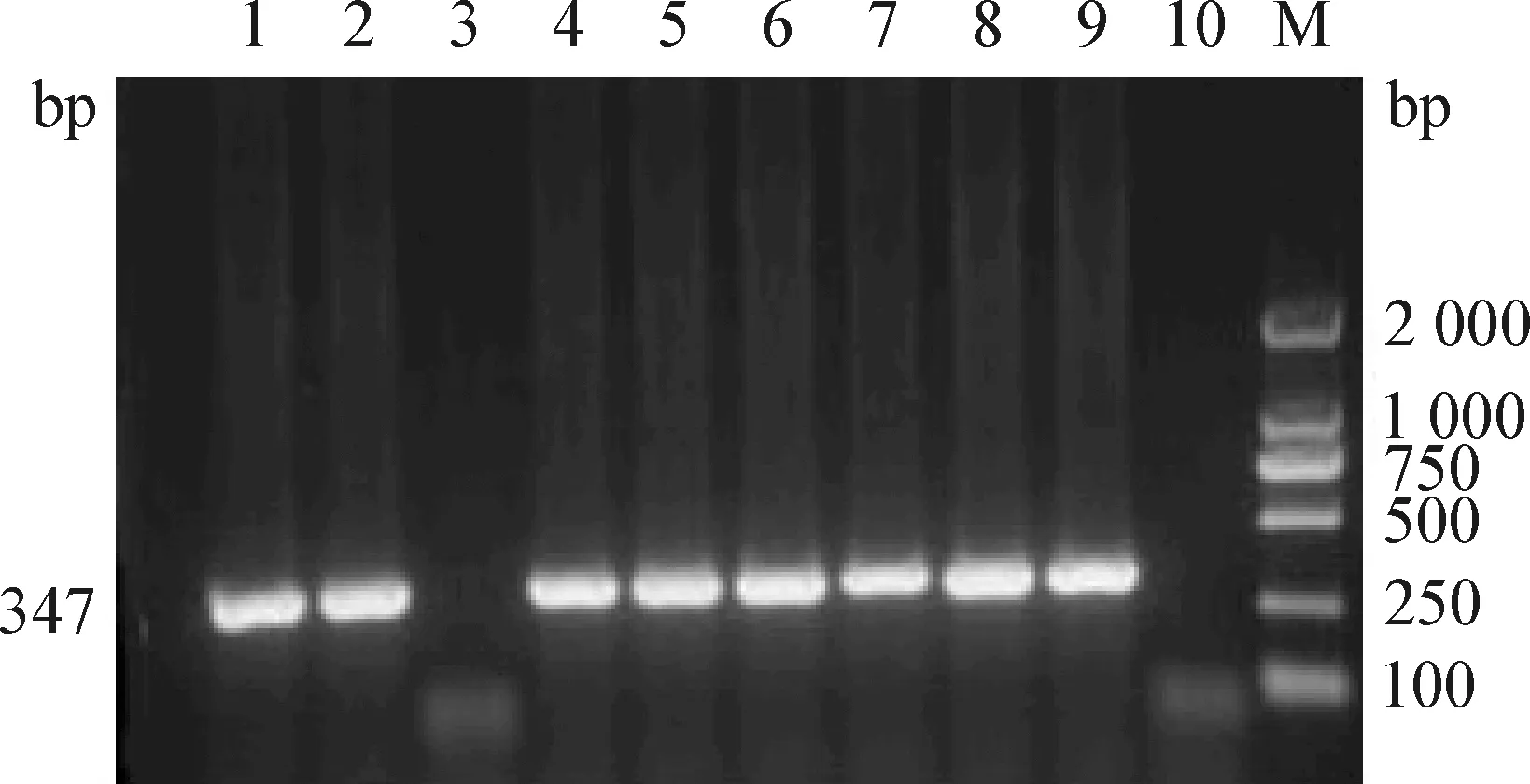
图4 亲虾养殖池塘水样凡纳滨对虾物种鉴定Fig.4 Species identification of Penaeus vannamei in water samples from broodstock culture ponds1~10:10个亲虾养殖池塘水样鉴定的PCR扩增产物; M:DL-2 000 DNA Marker 1-10:PCR amplification products of 10 water samples of broodstock culture ponds; M:DL-2 000 DNA Marker
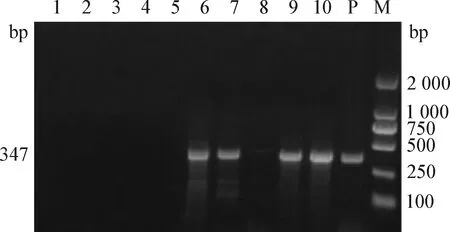
图5 进口亲虾水样凡纳滨对虾物种鉴定 Fig.5 Species identification of Penaeus vannamei in water samples from imported parent shrimps1~10:10个进口亲虾水样鉴定的PCR扩增产物; P:阳性对照; M:DL-2 000 DNA Marker1-10:PCR amplification products of 10 water samples from imported parent shrimps; P: Positive control; M:DL-2 000 DNA Marker
2.3 不同过滤膜中的宿主物种鉴定比对 如图6所示,硝酸纤维素膜过滤后,20份水样中有12份水样成功扩增出特异性目的片段;如图7所示,玻璃纤维膜过滤后,20份水样中有9份样品成功扩增出特异性目的片段。此结果与2.1结果一致,同样体积的水样使用硝酸纤维素膜过滤后获得的eDNA浓度较高,扩增效果好。
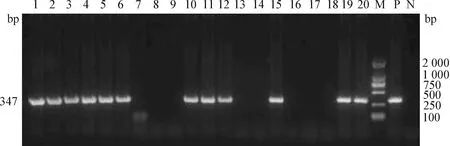
图6 硝酸纤维素膜过滤进口亲虾水样的凡纳滨对虾物种鉴定Fig.6 Species identification of Penaeus vannamei in water samples from imported parent shrimps filtered by nitrocellulose membrane1~20:20个经硝酸纤维素膜过滤的进口亲虾水样的PCR扩增产物; P:阳性对照;N:阴性对照; M:DL-2 000 DNA Marker1-20:PCR amplification products of 20 water samples from imported parent shrimps filtered by nitrocellulose membrane; P:Positive control; N:Negative control; M:DL-2 000 DNA Marker
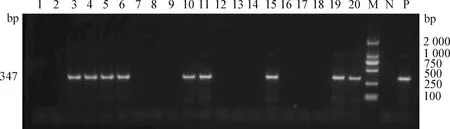
图7 玻璃纤维膜过滤进口亲虾水样的凡纳滨对虾物种鉴定Fig.7 Species identification of Penaeus vannamei in water samples from imported parent shrimps filtered by glass fiber membrane1~20:20个经玻璃纤维膜过滤的进口亲虾水样的PCR扩增产物; N:阴性对照; P:阳性对照; M:DL-2 000 DNA Marker1-20:PCR amplification products of 20 water samples from imported parent shrimps filtered by glass fiber membrane; N:Negative control; P:Positive control; M:DL-2 000 DNA Marker
2.4 不同取样时间的宿主物种鉴定比对 如图8所示,样品放置时间越长,DNA降解越多,在放置0.5 d后过滤已经有DNA降解,在放置3 d过滤获得的DNA降解非常多,在放置6 d时DNA已经全部降解。
凡纳滨对虾特异性鉴定结果如图9~图11所示,样品不放置(0 h)时,所有样品PCR扩增条带均非常亮;在水样放置2 d后,PCR扩增条带已非常弱。但由图8结果对比可知,放置2 d后的样品过滤得到的DNA降解不严重。过滤后可以鉴定出宿主的水样最长放置时间分别为:水样1为3 d,水样2为4 d,水样3为4 d,水样4为3 d。
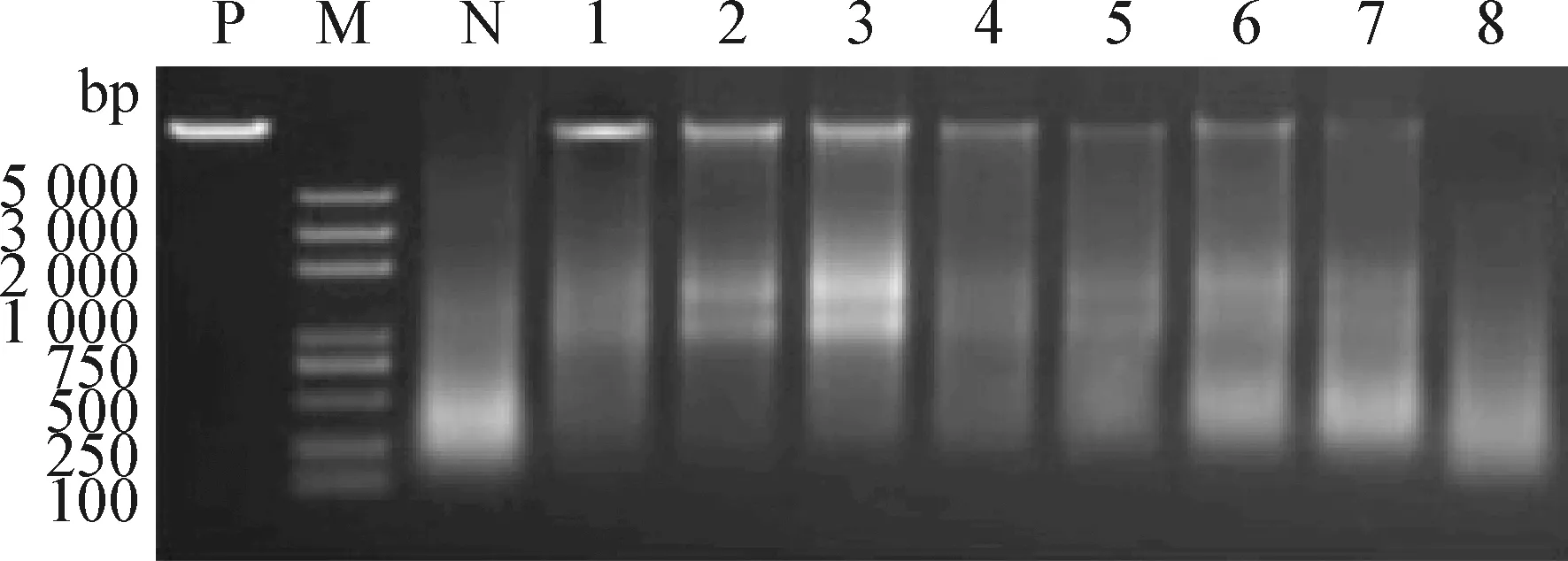
图8 进口亲虾水样放置不同时间进行过滤后DNA电泳降解图 Fig.8 DNA electrophoretic degradation of water samples from imported parent shrimps after filtration at different time1~8:样品放置0 h、0.5 d、1 d、2 d、3 d、4 d、5 d、6 d; P:阳性对照; N:阴性对照; M:DL-5 000 DNA Marker 1-8:Samples were placed for 0 h,0.5 d,1 d,2 d,3 d,4 d,5 d,6 d; P:Positive control; N:Negative control; M:DL-5 000 DNA Marker
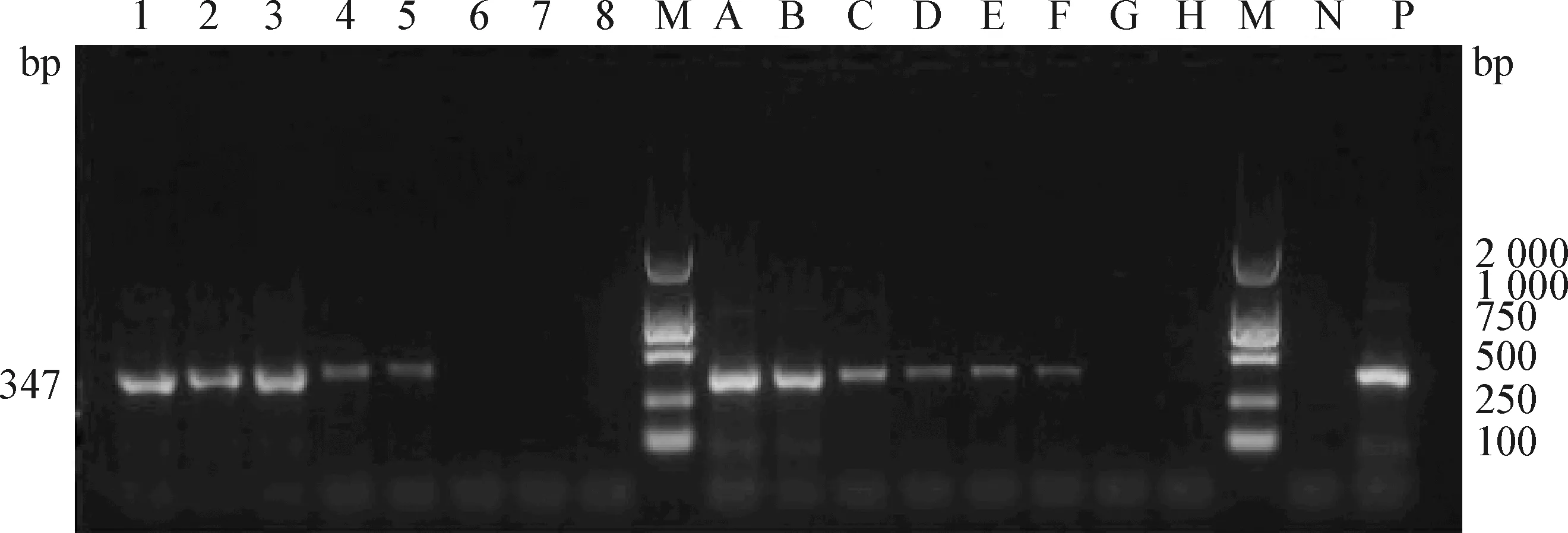
图9 进口亲虾水样1和水样2分别在放置不同时间过滤后凡纳滨对虾物种鉴定 Fig.9 Species identification of Penaeus vannamei in water sample 1 and sample 2 from imported parent shrimps after filtration at different time1~8和A~H:样品1和样品2分别放置0 h、0.5 d、1 d、2 d、3 d、4 d、5 d、6 d;N:阴性对照; P:阳性对照; M: DL-2 000 DNA Marker1-8 and A-H:sample 1 and sample 2 were placed for 0 h,0.5 d,1 d,2 d,3 d,4 d,5 d,6 d; N:Negative control; P:Positive control; M:DL-2 000 DNA Marker
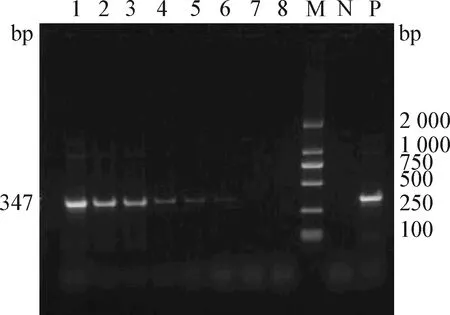
图10 进口亲虾水样3在放置不同时间过滤后凡纳滨对虾物种鉴定Fig.10 Species identification of Penaeus vannamei in water sample 3 from imported parent shrimps after filtration at different time1~8:样品3放置0 h、0.5 d、1 d、2 d、3 d、4 d、5 d、6 d; N:阴性对照; P:阳性对照; M:DL-2 000 DNA Marker1-8:Sample 3 was placed for 0 h,0.5 d,1 d,2 d,3 d,4 d,5 d,6 d; N:Negative control; P:Positive control; M:DL-2 000 DNA Marker
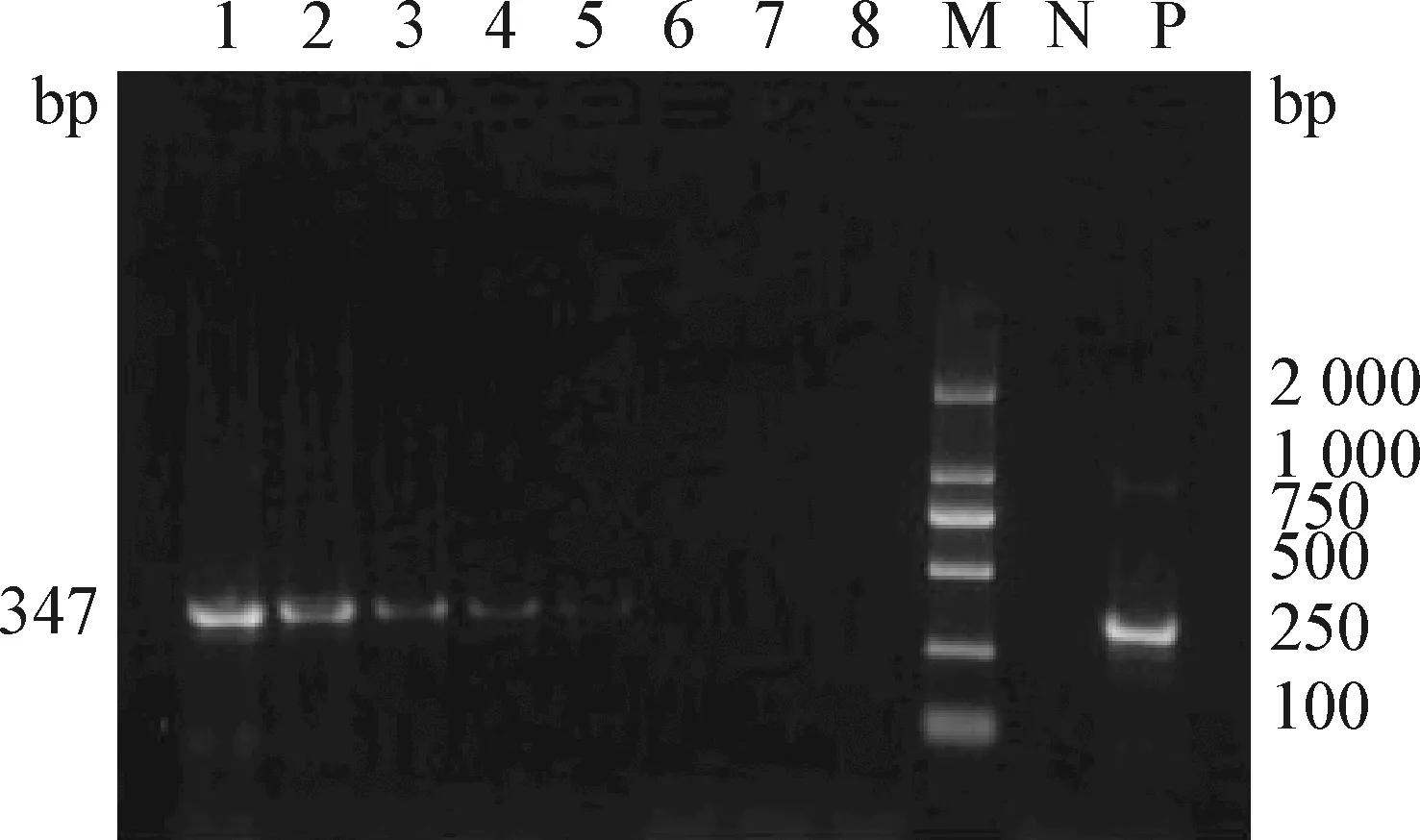
图11 进口亲虾水样4在放置不同时间过滤后凡纳滨对虾物种鉴定Fig.11 Species identification of Penaeus vannamei in water sample 4 from imported parent shrimps after filtration at different time1~8:样品4放置0 h、0.5 d、1 d、2 d、3 d、4 d、5 d、6 d; N:阴性对照; P:阳性对照; M:DL-2 000 DNA Marker1-8:Sample 4 was placed for 0 h,0.5 d,1 d,2 d,3 d,4 d,5 d,6 d; N:Negative control; P:Positive control; M:DL-2 000 DNA Marker
2.5 水过滤膜eDNA的宏基因组测序分析 对进口亲虾的环境水过滤膜进行宏基因组测序,使用 DIAMOND 软件将 UniqGeneSet 与 NCBI 的NR 序列进行比对(Blastp 比对参数设置期望值为1e-5);取相似度最高的比对序列作为该序列的物种注释信息,基因丰度总和计算该物种的丰度,并在各个分类学水平上统计物种在各个样品中的丰度,从而构建相应分类学水平上的丰度谱(Abundance profile)。为了综合而直观的展示各样品中不同分类层级的物种相对丰度,采用 Krona 对物种注释结果进行可视化展示,十足目(Decapoda)对虾科(Penaeidae)对虾属(Penaeus)的总序列丰度占比如图12所示,凡纳滨对虾(Penaeusvannamei)物种序列占对虾属的40%,日本对虾(Penaeusjaponicus)占38%,斑节对虾(Penaeusmonodon)占10%,加州对虾(Penaeuscaliforniensis)占3%,印度对虾(Penaeusindicus)占3%,中国对虾(Penaeuschinensis)占2%,其他对虾属(Penaeussp.)序列占3%。在宏基因组测序得到的所有物种序列中,宿主凡纳滨对虾测序序列物种丰度占比最高。
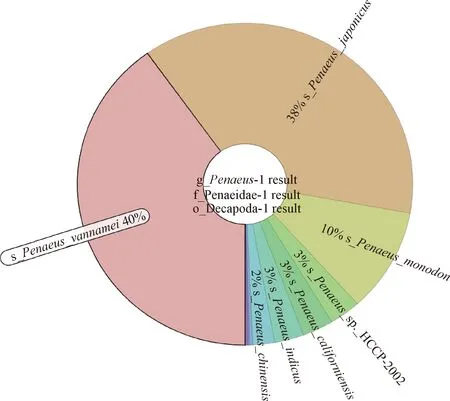
图12 水滤膜eDNA的宏基因组测序 Krona 示例图Fig.12 Krona map after metagenome sequencing of water filtration membrane eDNA
3 讨论
3.1 不同过滤膜、不同样品体积对于eDNA获取的影响 有文献报道已经说明,eDNA浓度和过滤样本体积呈正增长关系[11],但是,样本量过大时容易发生过滤膜堵塞情况。本试验中,对20份样本分别用硝酸纤维素膜、玻璃纤维膜、聚碳酸酯膜进行过滤,结果显示,玻璃纤维膜不容易堵,流速也快,过滤所用时间最短,过滤的样品量也最多,最多的时候可以达到4 L;聚碳酸酯膜过滤效果不好,且容易发生堵塞。所以水样浑浊或有大量浮游植物时,先用大孔径(如100目)尼龙膜预过滤后再用0.45 μm孔径过滤膜过滤效果更好。
3.2 凡纳滨对虾的特异性引物设计 大多数eDNA可能来源于排放的尿液和粪便物质[12-13]、从外部黏液层脱落的上皮细胞[14]和从分解有机体中脱落的组织[15]。研究显示,鱼类、两栖动物和软体动物向环境中释放大量的eDNA[16]。Tréguier等[17]在低丰度条件下检测小龙虾eDNA时遇到了困难,节肢动物的eDNA似乎很难检测,可能是因为它们的外骨骼减少了表皮细胞的脱落。本试验设计验证了多个凡纳滨对虾物种鉴定引物,在验证不同引物时,有的引物的PCR扩增产物在测序时会得到很多非特异性结果,分析原因应是eDNA 的成分较为复杂,含有许多其他物种和细菌、病毒的DNA,所以对引物的特异性要求更高。本试验设计并验证后选用的凡纳滨对虾物种鉴定引物的特异性较强,没有扩增出其他物种序列,且该引物灵敏度较好。
3.3 不同条件对凡纳滨对虾物种鉴定的影响 虾体养殖时间较长的虾塘水样,更容易进行宿主物种鉴定。已有多个研究证实,eDNA的含量与对虾的生存时间和排泄能力有关[18-21]。本试验中部分进口亲虾携带水水样没有扩增出凡纳滨对虾的特异性条带,分析原因是携带水中虾的数量较少或虾在携带水中的生存时间较短,所释放的eDNA含量太低。这个情况也是eDNA方法用于鉴定水中物种的缺点之一,需进一步加强eDNA浓缩和富集方法的研究。
eDNA衰变的主要途径是组织和颗粒的物理降解。脱落的生物组织快速从多细胞组织碎片降解为单个细胞、分离的细胞器(如线粒体),并最终释放DNA,然后通过外源酶或自发的化学反应进一步降解DNA,降解的程度影响eDNA是否可以被捕获[22-24]。本试验在对比放置时间对于物种鉴定的影响时,结果显示,样本放置时间越长,eDNA的降解越多,物种鉴定越困难,故建议采集样本后马上进行过滤和收集处理。本试验在进行过滤时发现,如果样本马上过滤则不易出现过滤膜堵塞情况,如果样品放置12 h后再进行过滤,则过滤膜很容易堵塞,水样中会出现一些黏液状物质,分析应该是细胞破裂后的黏液。同时,本试验还发现,样品马上过滤时鉴定引物扩增得到的PCR条带最亮,而在水样放置2 d后过滤,鉴定扩增条带非常弱,但是样品马上过滤和放置2 d后过滤所得到的核酸浓度相差不大,所以不能只以核酸浓度来判断样本所获得的eDNA是否有效。
3.4 宏基因组测序在eDNA方法中的应用 宏基因组学是以生态环境中全部DNA作为研究对象,通过克隆、异源表达来筛选有用基因及其产物,研究其功能和彼此之间的关系和相互作用,并揭示其规律的一门科学,它决定了生物群体的生命现象[5,25]。目前,宏基因组测序是eDNA研究的一个重要方法[26]。本试验对亲虾携带水进行宏基因组测序并进行数据分析,采用Virus Refseq、Virus NT和Acalme 三个数据库进行比对,最终的注释结果是选取的最优注释。本试验在进行NCBI网站比对时,测序序列是与NCBI中全库序列进行比对,由于测序片段较短,序列比对很可能比对到其他物种上,且在线比对的参数与流程中的参数不一致,NCBI上的数据库实时更新,本试验数据无法实时更新,数据库的差异也会导致比对结果的差异。DNA宏基因组编码还面临着几个重要的技术难题,主要涉及引物偏差和PCR错误,更普遍的是需要在所有水平上进行试验标准化,以便进行交叉试验比较。因此,eDNA研究不仅在试验方法上,而且在记录相关单元分析数据方面都非常需要标准化[27]。
3.5 eDNA检测方法的应用与发展 eDNA检测方法的高灵敏性和可远程检测是其具有吸引力的主要原因,国外使用该方法在生物入侵方面的研究已经很多。该方法非常适合于检测濒危、低密度入侵、瞬时和隐秘物种,特别是当低密度物种的采样难以控制时,eDNA检测方法的敏感性、简单性和减少破坏的优势愈加显现出来。eDNA检测方法仍存在许多不足,比如:如何抵抗水的流动性对于eDNA收集的影响;eDNA收集的标准化和一致性;宏基因组测序的有效性等等,这些方面仍需要更深入地研究。
