基于计算的蛋白质复合物预测方法综述
2022-01-18潘玉亮关佶红石运佳周水庚
潘玉亮,关佶红+,姚 恒,石运佳,周水庚
1.同济大学 电子与信息工程学院,上海201804
2.同济大学 嵌入式系统与服务计算教育部重点实验室,上海201804
3.复旦大学 计算机科学技术学院,上海200433
4.复旦大学 上海市智能信息处理重点实验室,上海200433
作为生命活动的物质基础,蛋白质(proteins)存在于所有的生物细胞中,参与了几乎所有的生命活动过程。大多数蛋白质不是单独地行使生物学功能,而是通过与其他蛋白质相互作用形成蛋白质复合物(protein complexes)来完成。因此,蛋白质复合物预测有助于更加深入地理解细胞的组成及其生命过程。
尽管一些生物实验技术,例如串联亲和纯化与质谱(TAP-MS)和酵母菌双杂交技术(Y2H)可以直接探测蛋白质复合物,但实验结果不仅存在较高的假阳性和假阴性,而且还存在诸多弊端,譬如在串联亲和纯化与质谱实验中,吸附性较低的蛋白质在多次冲洗后很难再次被检测到;由于非稳态蛋白质复合物中蛋白质之间相互作用动态可变,实验方法也很难检测到;有些复合物的合成需要某种特定的生物环境,如果生物实验方法无法模拟相应的环境,则相应复合物将不能被检测到;由于蛋白质相互作用发生的时间、空间等信息难以捕获,这也将影响复合物检测的准确性;生物实验方法存在耗时长、成本高等问题,不能满足后基因组时代相关研究的实际需要。
随着高通量实验技术的发展,蛋白质相互作用(protein-protein interaction,PPI)数据日益增多,这使得通过计算方法预测蛋白质复合物成为了可能。针对生物实验技术中存在的诸多问题,计算方法逐渐被应用到该领域。计算方法具有速度快、成本低等优势,可以在短时间内预测一些高置信度的蛋白质复合物,有效地弥补了生物实验的不足。利用大量的蛋白质相互作用数据,可以构建蛋白质相互作用网络(PPI network,PIN)。其中,网络中的节点表示蛋白质,节点之间的连接表示对应蛋白质之间的相互作用,进而可以通过复杂网络理论和机器学习方法在PIN 上预测蛋白质复合物。
目前,针对基于计算的蛋白质复合物预测问题,国内外已有大量相关研究。最初,人们将图聚类算法应用到PPI 网络中,通过挖掘局部密集子图来预测蛋白质复合物。这种方法不仅简单直观,而且取得了较好的效果,但预测准确率有待提高。鉴于PPI网络存在较高的假阴性、假阳性问题,不少研究开始使用网络拓扑结构特征、蛋白质功能、基因表达等信息为PPI 网络进行加权以提升网络的可靠性,进而提高蛋白质复合物预测的准确性。另外,通过对已有蛋白质复合物结构的研究,人们发现复合物具有核心-附属结构,因而提出了基于核心-附属结构的预测方法。部分研究者针对蛋白质间互作的动态性,提出了基于动态网络的预测方法。有些研究团队利用已知的蛋白质复合物作为先验知识,提出基于监督学习的算法。还有的研究者提出从功能到互作的研究思路,从新的角度预测蛋白质复合物。
针对蛋白质复合物预测问题,这些年来国内外已有少量相关评述和比较研究,但这些工作未能系统地将该领域的方法进行有效归类和指出该领域有待解决的问题。其中,Chen 等人的综述对静态蛋白质相互作用网络到动态蛋白质相互作用网络的复合物预测算法进行了总结,但由于其发表时间较早,缺乏近些年的预测方法;Wu 等人的综述虽然涉及比较多的复合物预测方法,但是对于这些方法的分类界限较为模糊,不能有效区分和总结所述算法;于杨的综述只囊括了基于静态网络的复合物预测方法,没有涵盖基于动态网络的方法;代启国和郭茂祖的综述涉及方法较少,且只关注于方法本身,未对性能评估指标和测试数据集进行介绍,也没有进行主要方法的性能比较。总的来说,目前的综述工作存在如下三方面的不足:第一,涵盖方法较少,未能对该领域进行全面描述。第二,对已有方法的分类标准模糊,不能有效将各个方法按类别分开,无法为研究者提供清晰的领域视野。第三,由于每年都会有不少基于计算的蛋白质复合物预测新算法发表,鉴于之前综述涵盖的方法已经比较陈旧,因此有必要对该领域的方法进行全新的梳理和评述以及性能比较。
本文旨在对现有蛋白质复合物预测方法进行综述,除介绍各种方法的技术特点外,还对比、分析了各类方法的优、缺点,并指出蛋白质复合物预测中的一些挑战和开放性问题。此外,利用酵母菌PPI 数据,对一些代表性方法的性能进行了测试和比较分析。本文希望通过对现有方法的全面、深入的阐述与分析,为该领域的研究者和用户在使用这些方法和开发新方法方面提供一些有价值的参考意见和方向指导。
1 蛋白质复合物及其预测
蛋白质复合物是一组在特定的时间和空间上通过彼此相互作用聚集在一起完成特定生物功能或生物过程的蛋白质集合。因此,蛋白质复合物对于生物体正常生命运转有着至关重要的作用。常见的蛋白质复合物有在转录过程中用于合成RNA 的RNA聚合酶和用于分子降解的蛋白酶体等。图1 所示为新型冠状病毒RNA 依赖的RNA 聚合酶(PDB ID:6M71),其作为新冠病毒转录复制的核心部件,被认为是重要的抗病毒药物靶标,目前紧急获批的瑞德西韦便是基于此靶标的抗病毒药物。图中的不同颜色代表不同的肽链,绿色、橘色、紫色和蓝色分别代表复合物中的A 链、B 链、C 链和D 链,不同链之间通过相互作用构成了蛋白质复合物。

图1 RNA 聚合酶复合物(PDB ID:6M71)Fig.1 RNA polymerase complex(PDB ID:6M71)
目前,检测蛋白质复合物的生物学方法主要包括串联亲和纯化与质谱技术和酵母菌双杂交技术。下面,对这两种生物方法作简要介绍。
串联亲和纯化与质谱技术是当前蛋白质组学研究中的重要工具。其主要步骤是嵌入一段蛋白质标记并导入目标蛋白质,在生理条件下与目标蛋白质发生相互作用的蛋白质就可以一起被洗脱下来,然后通过质谱技术进行鉴定,如此便可以快速地得到生理条件下真实的蛋白质复合物。
酵母菌双杂交技术检测蛋白质复合物,首先是把已知编码的蛋白质DNA 序列连接到带有转录调控因子DNA 的表达载体上;然后将导入的酵母菌细胞与报告基因上游的启动调控区相结合作为“诱饵”蛋白质;接下来将已知编码转录激活结构域的DNA 与待筛选cDNA 文库中的不同片段连接获得“猎物”载体;最后激活报告基因表达并获得蛋白质复合物。
虽然可以在生物实验中使用串联亲和纯化与质谱和酵母菌双杂交等技术直接探测蛋白质复合物,但实验结果存在较严重的假阳性和假阴性。另外,由于实验技术限制,部分蛋白质复合物很难被检测到,而且存在实验耗时长、成本高等问题,无法满足后基因组时代相关研究的实际需求。随着高通量实验方法的发展,全基因组蛋白质相互作用数据日益增多,为通过计算方法来预测蛋白质复合物创造了条件。
基于计算的方法预测蛋白质复合物可以有效弥补生物实验的不足,短时间内可在大型生物网络上预测出许多高置信度的蛋白质复合物。在现有的计算方法中,通常用无向网络来表示蛋白质之间相互作用关系,记为=(,)。其中,表示蛋白质相互作用网络,代表蛋白质集合,代表蛋白质之间相互作用集合。图2 所示为酵母菌蛋白质相互作用网络。基于计算的方法预测蛋白质复合物,主要是利用网络所包含的拓扑结构和节点所包含的生物属性为特征,采用聚类方法在PPI 网络上挖掘密集子图,将得到的密集子图作为最终的蛋白质复合物。结果表明,基于计算的方法对于分析PPI 网络、预测蛋白质复合物等效果显著。

图2 蛋白质相互作用网络Fig.2 Protein-protein interaction network
2 基于计算的蛋白质复合物预测方法
目前,国内外研究学者已经提出多种基于计算的蛋白质复合物预测算法,本文将这些方法分为如下七类:基于局部密集子图的预测算法、基于核心-附属结构的预测算法、基于动态网络的预测算法、基于监督学习的预测算法、从功能到互作的预测算法、基于多源数据的预测算法以及其他方法。下面对以上七类方法分别加以阐述。
2.1 基于局部密集子图的预测算法
在现有七类预测算法中,基于局部密集子图的预测算法诞生最早且数量最多。由于大部分蛋白质需要与其他蛋白质通过相互作用形成复合物才能完成相应的生物功能,复合物中的蛋白质组对应于相互作用网络中联系紧密的若干个节点,即局部密集子图。同时,大量针对蛋白质相互作用网络的研究表明,蛋白质相互作用网络具有模块性。从网络拓扑结构的角度来看,PPI 网络中的模块是由联系紧密的蛋白质构成。从生物学的角度看,PPI 网络中的模块代表了共同执行某项生物功能的蛋白质集合。由此可以通过挖掘PPI 网络中的模块结构(即密集子图或子网络)来预测蛋白质复合物。根据PPI 网络中的边是否加权,可将基于局部密集子图的方法大致分为两类:基于非加权网络的预测算法和基于加权网络的预测算法。
2003 年Bader 和Hogue 提出的MCODE方法,作为早期预测蛋白质复合物的计算方法之一,分三个步骤完成对蛋白质复合物的预测。首先,通过计算节点的-core 值和局部子图密度的乘积得到节点的局部邻居密度。然后将密度值较大的节点选为种子节点,并从种子节点开始遍历其邻居节点并进行扩展,将满足相应阈值的节点依次加入当前子图中,直到子图不再扩展即得到初期的蛋白质复合物。当复合物预测完成后,若网络中还有未被处理的节点,则作为新的种子节点,重复上述过程。最后,MCODE为提升预测结果的准确性,对上述预测出的初期复合物进行相应后处理操作。第一,将节点数少于2 的复合物直接移除。第二,对于候选复合物中每个节点,若其直接邻居(包括节点)所构成子图的密度高于给定参数,则的所有邻居节点依次加入到当前复合物中并生成最终的蛋白质复合物。
基于网络的聚类算法被应用到蛋白质相互作用网络中以挖掘密集子图作为蛋白质复合物,例如,MCL算法通过在蛋白质相互作用网络上模拟随机游走,进而提取密集子图来预测蛋白质复合物。随机游走在PPI 网络构建的邻接矩阵上迭代执行“扩展”和“膨胀”两个操作,使得PPI 网络中原本密集的区域更加密集,原本稀疏的区域更加稀疏,从而将连接紧密的节点组作为复合物输出。由于该算法直接进行矩阵运算,是一个快速且可扩展的聚类算法。
根据蛋白质相互作用网络的特点,基于代价函数来识别复合物的算法被提出。其中,Nepusz 等人提出的ClusterONE 算法,是近几年提出的经典算法之一。作者为评估当前子图构成蛋白质复合物的概率,定义一个有效的“紧密度”函数。此函数描述了蛋白质复合物应该满足的两个基本结构特征:复合物内部的节点之间应该连接紧密;不同复合物之间的节点应该连接稀疏。“紧密度”函数定义如下:

其中,()代表子图内所有边上权重之和,()表示当前子图与子图外节点之间连边的权重之和,对于非加权网络,即为边数之和,最后()作为惩罚因子,用来模拟蛋白质互作网络中存在但未被发现的互作连边的不确定性。ClusterONE 选取度数最高的节点作为种子节点向外扩展,但异于其他只增加节点的扩展方法,它会同时进行添加和删除节点两项操作,直到没有节点再加入或离开当前子图为止,以保证当前子图“紧密度”函数值最优。然后将未处理的节点中以度数最高的节点选作种子节点,重复以上步骤,直到所有节点被处理完。该算法不但可预测出具有重叠性质的蛋白质复合物,而且具有很高的预测准确性。同样GraphEntropy也是采用代函数,利用种子点增长策略找到图熵值最小的子图作为复合物输出。
基于局部密集子图以及复合物内部节点间应紧密连接的思想,“完全图”的概念被应用到蛋白质复合物预测中。Clique算法分别通过穷举法、超顺磁性聚类和蒙特卡洛模拟三种方法从PPI 网络中提取完全图,按照一定规则对完全图进行进一步的后处理,包括舍弃、合并和选择等操作。Li 等人提出的LCMA 算法首先为每个蛋白质节点找到局部完全图,然后将其中重叠率高的子图合并生成极大密集区域,从而得到蛋白质复合物。CFinder算法首先从PPI 网络中找到k-完全图,然后通过合并所有相邻的k-完全图生成更大的子图,以此预测蛋白质复合物。
除以上算法外,针对局部密集子图的算法层出不穷。其中包括大量从种子节点出发,按照一定规则向外扩展的算法。如Ucar 等人提出的Hub Duplication 方法选取度数大于25 的蛋白质节点作为Hub 蛋白质,并通过加入其邻居节点建立密集子图以生成满足要求的蛋白质复合物。SCAN算法将公共邻居数大于给定阈值的两个蛋白质认为是结构可达的,以结构可达节点最多的节点作为种子节点向外扩展,逐步将结构可达的邻居节点纳入聚簇。Zhang 等人提出从计算子图中三节点连通图个数的角度来评价局部子图的联通紧密性,借鉴ClusterONE 方法的思想,从度数最大的节点开始扩展,通过加入新节点、删除内部节点两个方向的操作,使子图紧密度最大化。Ren 等人考虑蛋白质复合物可能存在低密度高模块化或高密度低模块化的情况,对子图定义了一个新的适应度函数,同时提出了LF_PIN 算法,通过局部最大适应度值来扩展种子边,从而预测蛋白质复合物。
此外,Navlakha 等人将原PPI 网络压缩成概要图,并在其上进行蛋白质复合物预测。Geva和Sharan提出的CODEC 方法使用质谱实验获取的数据建立二分图,节点集合分别为诱饵、靶标蛋白质。CODEC算法先从靶标及其邻居中寻找潜在的复合物组成,再通过增减节点最大化子图得分获得最终的预测结果。此外,Jia 等人提出基于Co-Graph 社区概念的复合物预测算法。Hu 等人针对具有重叠性质的蛋白质复合物,采用模糊聚类的算法进行预测。Rahman 等人定义了点到点的聚类值概念,公式如下:

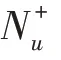
非加权蛋白质相互作用网络仅考虑蛋白质间是否存在相互作用。但由于目前的生物实验尚不完备,蛋白质相互作用数据中其实存在着一定的假阳性和假阴性,以此构建的非加权网络置信度低。因此,部分研究通过将PPI 网络的拓扑结构、基因表达数据、蛋白质功能等信息对边进行加权,提高PPI 网络的可靠性,并在此基础上提出相应的基于加权网络的复合物预测算法。
早期加权网络的构建是通过计算存在相互作用的一对蛋白质之间的公共邻居数为其连边加权。Altaf-Ul-Amin 等人提出的DPClus 算法根据节点对的公共邻居数给节点对之间的边赋值,节点的权重为邻接边的权重之和。DPClus 将权重最大的节点作为种子,使用聚簇属性将邻居节点中与之连接较为紧密的节点加入聚簇。另外,IPCA在DPClus 的基础上做出改进,提出了一种新的拓扑结构用于预测蛋白质复合物。Liu 等人提出基于最大完全图的聚类方法CMC,首先根据节点的公共邻居数来衡量相应边的权重,并迭代修改此值,然后找到PPI 网络中的完全图并对边权打分,最后根据分数对其合并生成复合物。MKE算法根据每对蛋白质的公共邻居数生成有向加权图,首先选取度数较高的蛋白质作为第一层核心,然后将权值均高于给定阈值的邻居节点与第一层核心相连,生成第二层核心,最后通过继续扩展,生成最终的蛋白质复合物。Ni等人也利用蛋白质对的公共邻居数为边加权,提出了WN-PC方法来预测蛋白质复合物。ProRank+应用轮辐模型根据节点类型和重要性值生成团簇,然后根据内聚性合并得到最终的蛋白质复合物。SPICi基于复合物的加权密度和团簇大小定义阈值,采取和DPClus 类似的策略生成复合物,可以在大型的生物网络上快速生成聚类结果。
近几年,涌现出大量利用拓扑结构特征构建目标函数,并利用遗传算法进行蛋白质复合物预测的算法。其中,Cao 等人提出的MOEPGA 算法,根据PPI 网络的多种网络拓扑特征构造目标函数,利用遗传算法的三个主要步骤,种群初始化、子图突变和子图选择,迭代计算,实现蛋白质复合物的识别。此外,Arnau 等人根据两个节点之间的最短路径长度为其边加权。Ma 等人重新定义高阶边聚类系数概念对网络进行加权,具体计算公式如下:
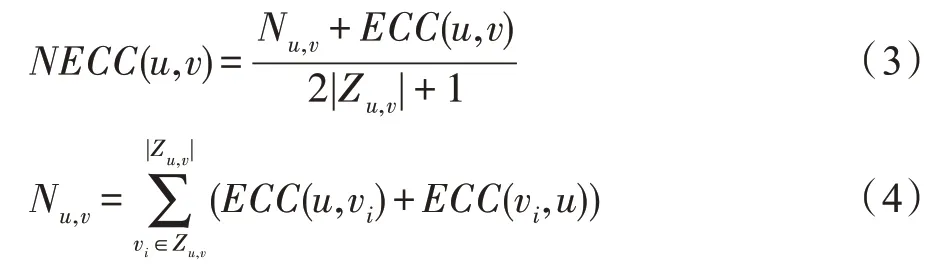
其中,(,)表示边聚集系数,Z表示节点、的共同邻居。Li 等人利用蛋白质间连接亲和度构建加权蛋白质相互作用网络,同时提出了CACE 算法进行蛋白质复合物预测。Chua 等人提出的PCP 方法使用拓扑结构来衡量PPI 网络中互作的可信度,将可靠度较低的边剔除并通过有效的完全图发现算法来预测复合物。Friedel 等人使用Bootstrap 采样法寻找复合物,首先从PPI 数据中做有放回的采样,通过计算蛋白质形成相互作用的倾向程度为边加权,并采用MCL 算法生成蛋白质聚簇,根据多次聚类的结果建立“Bootstrap 网络”,其中节点为蛋白质,各边的权重由相邻蛋白质在同一个初步聚类结果中出现的次数计算得到,最后在此网络中使用MCL 算法生成复合物。Wu 等人提出的idenPC-MIIP方法通过在有权网络上定义相互重要性邻居来改变原始网络权重,进而采用贪心算法识别蛋白质复合物。随着图神经网络的兴起,PPI 网络的拓扑结构信息可以通过图神经网络来获得更为高阶的信息。Yao 等人通过使用图自编码器重构PPI 网络,并使用一些经典的聚类算法在重构后的PPI 网络上识别蛋白质复合物。实验发现,经过图自编码器重构后的PPI 网络可以有效去除噪声数据,使预测结果更为准确。
一般来说,形成功能团的蛋白质具有相同的基因表达。Feng 等人提出的图分裂算法GFA 使用最密子图算法找到PPI 网络中密度最大的子图。其中,子图密度通过基因表达数据计算,同一子图内蛋白质表达量之和越高则子图密度越大。Maraziotis 等人提出的DMSP 算法首先对基因表达数据进行模糊聚类,两个蛋白质的相互作用可以通过它们到各自聚类中心的距离与两个聚类中心的距离之和估计。DMSP 从种子节点开始聚类,通过加入它的直接、间接邻居使聚类密度大于一定阈值来预测功能团。
一些预测蛋白质复合物的方法通过借助基因表达数据来估计PPI网络中边的权重以提高准确率。Ulitsky 等人提出的MATISEE 算法使用基因表达数据的相关度来衡量一对蛋白质的互作强度,并以此得到蛋白质节点权重。聚类算法从种子节点开始,通过加入、删除其邻居节点找到子图,并根据一定条件合并子图得到最终的预测结果。Ou-Yang 等人提出的SGNMF方法则是利用基因表达数据构建带符号的PPI 网络,并在此基础上预测复合物。SEDMTG由Wang 等人提出,其在网络的权重上既考虑拓扑结构也加入了Go 基因信息,最后的聚类算法采用高内聚低耦合的思想设计代价函数来识别蛋白质复合物。
蛋白质功能也为蛋白质复合物预测提供了重要的信息。King 等人提出的RNSC 算法从 一个随机聚簇开始,启发式地改变其中的节点使该聚簇满足代价最小,再利用蛋白质功能注释数据评估聚簇是否为蛋白质复合物。Lubovac等人提出的SWEMODE算法,首先计算两个蛋白质功能的语义相似度,公式如下:

其中,p(t,t)是t,t最小子集的概率,然后构建加权网络,节点权重通过加权聚类系数以及最近邻居数给出,最后使用与MCODE类似的聚类算法从加权网络中识别蛋白质复合物。Cho 等人首先根据功能相似度给边加权,节点的权重为其连边权重之和。随后采用流模拟算法,从信息量(即节点权重)较大的节点开始,向邻居节点发散出模拟的流,流的影响会根据先后经过节点的相似度逐步衰减,直到小于某一阈值时停止,由此将PPI 网络分割成多个子图。
一个蛋白质互作界面上的重合区域可能会阻止多对蛋白质同时作用,结合蛋白质结构域互作信息可以考察多对蛋白质组合是兼容还是互斥。Jung等人首先使用了MCODE和LCMA两种方法生成聚簇。从两个结果集中排除可能有冲突的互作,得到并发蛋白质互作集合。Will和Helms提出的基于结构域的紧密优化算法DACO 将蛋白质相互作用与结构域相互作用结合,在PPI 网络中使用图聚类算法预测蛋白质复合物时,要求预测到的蛋白质集合内的蛋白质间存在结构域互作。Maruyama 等人提出 的PPSampler2-PIME 算 法,其 在PPSampler2 算 法基础之上,加入相互排斥边对的考虑。根据作者的假设:一个蛋白质复合物中的边与边之间不能存在相互排斥现象,并据此设计目标函数,以此达到最优目标。实验结果表明,通过引入相互排斥边对的思想能够有效提升蛋白质复合物预测准确度。
基于局部密集子图的蛋白质复合物预测方法简单直观,但是受网络本身的噪声影响较大,而且无法有效预测小复合物(蛋白质数量<3)及内部连接稀疏的复合物。
2.2 基于核心-附属结构的预测算法
除了基于局部密集子图的预测算法,研究学者通过研究已知复合物的内部结构特征,提出了基于核心-附属结构的预测算法。Gavin 等人通过研究酵母菌蛋白质复合物的结构,发现每个复合物由两部分组成:存在大量互作的蛋白质集合构成复合物的核心结构,与核心结构相连接且相对稀疏的蛋白质构成附属结构。
Leung 等人提出的CORE 算法通过两个蛋白之间以及它们与公共邻居的互作情况计算出它们为同一复合物的核心蛋白质的概率,具体计算公式如下:
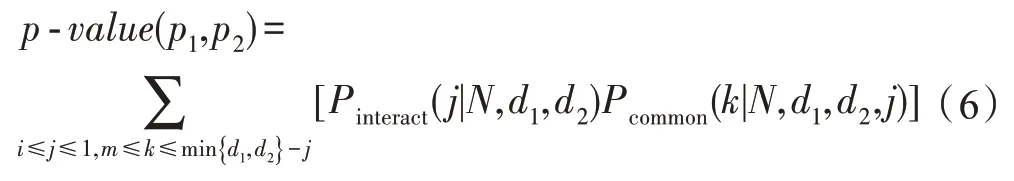
其中,、是节点、的度,(|,,)是节点、具有相互作用的概率,(|,,,)是节点、具有相互作用并且共同邻居数为的概率。之后,通过合并大小为2、3 的核心集合以获得更大的核心集合,直到整合条件不再满足为止,以此构成最终的复合物核心结构。之后根据其他节点与核心蛋白质的连接强度添加附属蛋白质,形成最终的蛋白质复合物。
Wu 等人提出的COACH 算法则根据蛋白质及其邻居节点在网络中的重要性(权重大小)寻找核心蛋白质,再添加其连接的附属蛋白质。WCOACH算法在COACH 算法的基础之上,通过GO 语义相似性对边进行加权,将其改进为可在加权网络上进行复合物预测的新算法,预测结果显示加权网络的预测效果要好于非加权网络。Peng 等人提出的WPNCA 算法,首先在加权PPI网络中利用PageRank-Nibble 算法,将PPI 网络划分为多个连接紧密的子图,然后在每个子图中,通过基于核心-附属结构的思想来预测蛋白质复合物分子。Luo 等人提出的动态核心-附属结构(DCA)算法,从动态PPI 网络(DPN)中挖掘连接紧密且在多个动态子网络中共同活跃的蛋白质集合作为核心结构,进而通过向核心添加紧密连接的直接邻居节点以构成最终的复合物集合。Mehranfar等人提出采用区间二型模糊表决模型融合GO 注释、基因表达等生物数据,建立可靠性比较高的加权蛋白质相互作用网络,并基于核心-附属结构预测蛋白质复合物。EWCA算法首先为蛋白质网络重新设置边权,然后基于结构相似性识别复合物的核,随后以核为基础辨别附属蛋白和外围蛋白,最后将核心蛋白和附属蛋白结合形成复合物。
基于核心-附属结构的预测方法从生物角度出发,考虑了复合物的结构特征,在一定程度上提升了蛋白质复合物预测的准确性,但泛化能力差,因为不是所有复合物均满足核心-附属结构,例如,小复合物、稀疏复合物等依然无法被有效预测。
2.3 基于动态网络的预测算法
早期的预测算法多是从静态PPI 网络中预测蛋白质复合物,但蛋白质之间的相互作用是动态的且随时间改变,因此在静态PPI 网络上预测将会限制预测的准确性。近几年,基于动态网络的预测算法越来越多,研究者将基因表达等时序信息加入到蛋白质复合物预测工作中,利用时序等信息帮助识别PPI 网络中的静态互作和动态互作,通过建立静态网络、动态网络等工作有效提升了复合物预测的准确性。由此可见,基于动态网络的预测算法大有可为。
Tang 等人利用时序表达数据建立多个动态的蛋白质互作网络,其中时序表达数据包含了每种基因产物在36 个测试时间段的表达量,以此表示各基因、蛋白质在各时间段的活跃程度。首先,为获得各时间段内活跃的蛋白质,作者通过设定全局阈值来过滤表达量低的蛋白质,即在某时间段内表达量高于该阈值的蛋白质属于活跃蛋白质,反之亦然。该步骤得到36 个蛋白质集合,每个集合对应该时间段内所有活跃的蛋白质。然后,若一对在静态PPI 网络中有连边的蛋白质在某一时间段内共同活跃,那么它们将构成相应时序动态网络的一条边,以此类推可以构建得到36 个时序网络。最后,作者将多种经典算法,如MCL、RNSC、MCODE 等分别在静态网络和时序动态网络上做实验,通过显著性分析发现,其建立的动态网络比静态网络、随机网络更具有生物意义。
鉴于不同蛋白质表达水平的差异性,用全局阈值来筛选活跃蛋白质显然不太合理。基于此,Wang等人通过自定的three-sigma 模型,基于每种蛋白质的表达曲线,为其计算属于自己的活跃阈值。实验结果表明,针对蛋白质特异性构造出的动态网络较全局阈值构造出的网络能更好地反映蛋白质互作网络的生物意义,在复合物预测上也取得更好的预测效果。
Ou-Yang 等人使用基因表达数据识别瞬态、稳态蛋白质互作。使用不同时间的表达数据建立动态互作网络,运用概率模型从各动态网络中预测动态蛋白质复合物以及在各时刻都存在的稳定蛋白质复合物。Mucha等人通过计算、比较蛋白质组合在静态网络内、临近动态网络间、全部动态网络间的连边数量来预测蛋白质复合物。Jin 等人在预测蛋白质复合物时要求其在静态图中联通,且组成同一复合物的蛋白质在不同时刻的表达水平具有相关性。Shen 等人基于核心-附属结构在动态PPI 网络上预测复合物。Zhang 等人使用基因表达数据、通过改良后的three-sigma 方法识别瞬态、稳定的蛋白质互作,计算蛋白质活跃概率,并利用核心-附属结构预测复合物,计算公式如下:
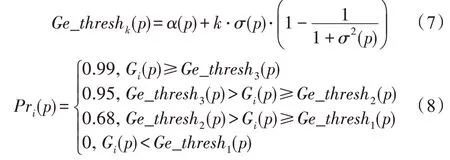
其中,取值为1、2、3,()与()分别是基因的算法平均值和标准差,G()表示在时刻基因的表达值,Pr()表示在时刻基因的活跃概率。如果在时刻基因的表达值大于等于(),那么时刻基因的活跃概率就是0.99。Lei 等人也是使用three-sigma 方法识别瞬态、稳定蛋白质互作,建立动态互作网络,然后采用萤火虫算法对马尔科夫聚类相关参数进行优化。另外,Lei 等人提出的TP-WDPIN 算法也是先利用three-sigma 模型构建动态PPI 网络,然后在每个子网络上基于拓扑势等信息为蛋白质节点加权,通过选取权值较高的部分节点作为种子节点进行扩展来预测蛋白质复合物。CPredictor4.0基于动态加权网络预测蛋白质复合物,其在动态PPI 网络上采用核心-附属结构的方式来寻找蛋白质复合物,通过合并重合率较高的复合物得到最终的预测结果。另外,李敏和Chen 等人的蛋白质复合物预测综述也对动态PPI 网络上的复合物预测做了比较全面的评述,可见基于动态网络的蛋白质互作网络是个值得深入研究的方向。
动态PPI网络相较于静态PPI网络能够更加准确反映蛋白质相互作用的动态特性,但是目前构建动态PPI网络的方法较少,仍有待进一步研究。
2.4 基于监督学习的预测算法
除了无监督方法外,有些人提出基于监督学习的复合物预测算法,其本质是借助标准蛋白质复合物集合中的信息来预测潜在的蛋白质复合物。Qi等人将真实的蛋白质复合物作为训练集,选取了生物及拓扑结构作为特征,使用概率贝叶斯模型对从PPI 网络中随机生成的子图进行分类,判断其是否为复合物,再使用模拟退火算法对候选子图进行修改以预测复合物。Shi 等人使用神经网络,采用与Qi等人类似的策略进行复合物预测。为了能够预测仅由2、3 个蛋白质组成的小复合物,Yong 等人以真实复合物作为训练数据,融合了互作数据、功能、文本信息以及拓扑结构来计算特征,使用贝叶斯模型估计所有蛋白质互作对组成的小复合物、大复合物以及非复合物成员的概率,再从中提取大小为2、3 的小复合物。Yu 等人构建正、负、中三种样本作为训练集,且分别为非加权、加权网络选取多种特征,利用以上训练集与特征集训练回归模型来判断当前子图为蛋白质复合物的概率,并通过Clique 算法初始化复合物集合,以回归模型为基础,选择性向当前子图添加邻居节点以达到回归模型值最优。SLPC首先将生物文献中提取的PPI数据与现有PPI数据集进行整合,然后剔除置信度较低的相互作用实现网络重构,最后通过基于监督学习的算法完成重构PPI 网络上的蛋白质复合物预测。SIKARNDAR 等人提出的IoMT方法认为蛋白质的氨基酸序列决定蛋白质的形成且对预测蛋白质间的相互作用性质具有重要意义。因此,在预测蛋白质复合物时不仅使用了拓扑特征还计算了氨基酸序列的生物学特征,即离散小波系数、长度和熵值。最后基于部分树和非嵌套广义样本等关联规则,训练监督学习方法来识别蛋白质复合物。
基于监督学习的预测方法有别于非监督学习算法,其充分利用已知复合物信息作为先验知识,能有效预测蛋白质复合物,尤其在小复合物、稀疏复合物的预测上贡献很大。但该类方法受特征选取影响较大,目前选取可以充分描述蛋白质复合物的生物特征还有待进一步研究。
2.5 从功能到互作的预测算法
与上述研究思路不同,Xu 等人提出的CPredictor系列算法,提出从功能到互作的研究新思路。CPredictor首先根据基因本体中的生物过程项计算蛋白质之间的功能相似度,然后使用谱聚类算法从中获取功能相似的蛋白质聚簇,最后利用蛋白质相互作用数据从中依次提取联通子图,并扩展、合并子图得到蛋白质复合物。其中,子图扩展时,对每个提取的联通子图,统计其在原蛋白质相互作用网络中的邻居与子图中节点的连接数量,并通过设定参数控制子图扩展。
针对基于密集子图的预测算法难以预测小复合物,而少数预测小复合物的方法很难用于大复合物预测的问题,Xu 等人在CPredictor算法基础上提出兼顾大小复合物的CPredictor 2.0 算法。该算法采用更直观的FunCat(function catalogue)功能目录对蛋白质进行功能分组,然后根据蛋白质相互作用信息在每一个分组内建立相互作用子网络,最后使用马尔科夫聚类算法从中获取蛋白质复合物。实验表明,从功能到互作的预测算法在预测兼顾大、小复合物上取得了良好的效果。随着图嵌入的兴起,Yao 等人利用图嵌入方法加权蛋白质互作网络,并融合蛋白质的功能信息来预测复合物。
2.6 基于多源数据的预测算法
鉴于生物实验自身的局限性,蛋白质相互作用数据存在假阳性和假阴性的问题。为解决这一问题,同时提高蛋白质相互作用网络的置信度,研究者们开始通过融合PPI 数据和其他多种相关数据资源来弥补蛋白质互作实验中缺失的信息,例如基因表达数据、蛋白质功能注释、蛋白质域及蛋白质序列等。目前,大多数方法通过融合多源数据构建加权网络,在加权网络上进行蛋白质复合物预测。
基因表达数据代表了不同时刻编码蛋白质的相关基因表达水平。文献[79-82]等方法,通过融合PPI数据和基因表达数据构建加权蛋白质相互作用网络,以基因表达数据的相关度衡量蛋白质相互作用强度。文献[22-23,105]等方法,利用基因表达数据构建动态蛋白质相互作用网络,更加准确描述了蛋白质的动态特性。蛋白质复合物通常由功能相似的一组蛋白质构成,通过蛋白质功能注释数据计算蛋白质功能相似性,以此作为网络权值,在一定程度上提升了网络置信度。Xu 等人利用功能注释数据对蛋白质进行聚类或分组,然后再预测蛋白质复合物。Wu 等人提出的idenPC-CAP方法在RNA-蛋白质相互作用的异质网络上进行蛋白质复合物的识别,这样可以有效消除蛋白质互作网络的假阳性,提高预测的准确率。融合多源数据提升了蛋白质相互作用网络置信度,也在一定程度上提高了蛋白质复合物预测的准确率。
2.7 其他预测算法
除了上述几类预测算法外,还有一些其他的预测算法。例如,Sharan 等人基于不同物种中复合物的组成具有保守性这一发现,通过建立酿酒酵母菌和幽门螺杆菌的同源图来寻找共有的保守蛋白质复合物。Wu 等人对传统的聚类集成进行了扩展,提出了EnsemHC 框架进行蛋白质复合物预测。首先,将不同聚类结果结合构建共簇矩阵来描述每对蛋白质是否属于同一复合物;然后,利用迭代加权方法对共簇矩阵进行处理后得到一个集成矩阵;最后,对集成矩阵层次聚类得到蛋白质复合物。
为了便于方法的复现及扩展,根据不同的类别将可获得代码链接的方法展示在表1 中。可以看出,基于局部密集子图的方法拥有较多公开代码,可为读者提供思路和实践上的参考。由于归类于“其他预测算法”的工作较少且没有可获得的方法链接,并未在表中列出具体方法链接。

表1 蛋白质复合物预测主要方法及其代码链接汇总Table 1 Summary of main methods and source code for protein complex prediction
尽管基于计算的蛋白质复合物预测已经取得了显著的进展,且各种预测方法层出不穷,但是仍然存在一些不足。基于局部密集子图的预测算法简单直观,但是对于小复合物(蛋白质数量不超过3)的预测结果有待提高,而且这类方法非常依赖蛋白质相互作用数据的准确度,而现有数据中过高的假阴性、假阳性使得网络置信度低,导致预测结果准确率不足。基于核心-附属结构的预测算法从蛋白质复合物自身的生物结构特性出发,在一定程度上提高了实验的准确率,然而不是所有的复合物都具有核心-附属结构,这类算法无法预测具有其他结构的蛋白质复合物,因此泛化能力较差。基于动态网络的预测算法是近年来一个大有可为的研究方向,但是目前工作比较少,现有的构建动态蛋白质相互作用网络的方法也比较少,无法准确描述蛋白质的动态特性。基于监督学习的预测算法主要利用已知的蛋白质复合物及其相关的结构特征来训练分类模型,对提取的蛋白质相互作用网络中的子图候选项进行预测,因此这类方法受复合物的特征表示影响较大,寻找和筛选真正符合要求的特征也太过耗时费力。
3 性能评估方法与测试数据集
本章将对蛋白质复合物预测问题中所用到的评估准则、数据集进行介绍,并在此基础上比较不同的算法在同一数据集下的预测结果,并对其结果进行分析。
3.1 评估准则
在介绍评估准则之前,首先要得到预测的复合物与标准蛋白质复合物的匹配程度。假设预测复合物标记为=(V,E),标准复合物记为=(V,E),那么两个蛋白质复合物的匹配度(overlapping score,OS)可以用下面的公式表示,其中代表复合物中的蛋白质,表示蛋白质间的相互作用。公式中的分子代表两个复合物中公共蛋白质节点个数的平方,分母为两个复合物中蛋白质个数的乘积。

当两个蛋白质复合物的匹配度(,)≥(阈值),就可认为二者是匹配的,反之亦然。常见的匹配度阈值选择包括0.20 和0.25 两种。
目前,评估蛋白质复合物识别算法性能的指标有很多种,下面对其中一些常见指标进行详细介绍。假设算法的预测结果集为={,,…,p},复合物的标准验证集表示为={,,…,r}。召回率()反映了算法对真实蛋白质复合物的预测覆盖率,值越大,说明算法能够预测出更多真实的蛋白质复合物。

其中,N为与至少一个预测复合物匹配的真实复合物的个数。精准度()则反映了一个预测算法的预测结果的精度,值越高,预测结果的精度越高。

其中,N为与至少一个真实复合物匹配的预测复合物的个数。1 值(1-measure)是与的调和平均值,用来评估一个蛋白质复合物预测算法的整体性能。
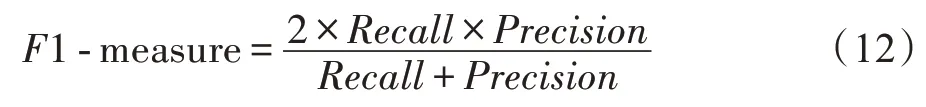
敏感度()用来评估实验结果中包含的蛋白质占标准复合物中所包含蛋白质的比例。
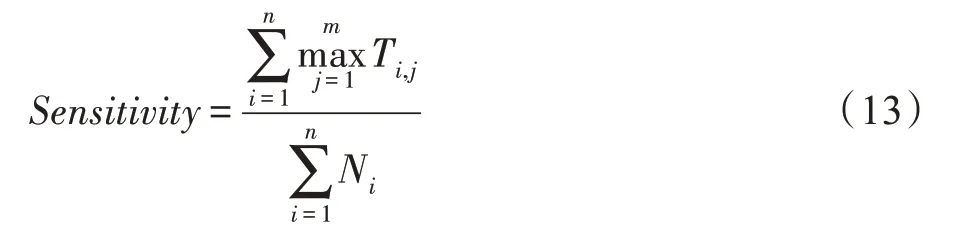
其中,T表示第个标准复合物与第个预测复合物的公共蛋白质数量,N表示第个标准复合物中蛋白质的数量。
表示实验结果中被正确预测的蛋白质复合物所占的比例。

准确度()是和的几何平均值,当或者某一值低时,那么值也低。因此,值高时表示和值均比较高。
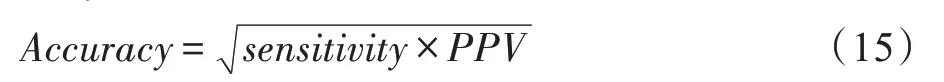
3.2 数据集
随着实验技术的发展和数据的积累,蛋白质相互作用数据不断增加。这里主要介绍蛋白质复合物预测实验中常用的数据库及相关数据集。
STRING 数据库是一个包含大量蛋白质相互作用的数据库,覆盖了2 000 多个物种,不仅整合了已被实验验证的蛋白质相互作用数据,还包括了通过生物信息学方法预测得到的蛋白质相互作用数据。
DIP 数据库主要存储经过实验验证的蛋白质相互作用数据,包括蛋白质信息、蛋白质相互作用信息和描述预测蛋白质相互作用的实验技术细节三部分内容。DIP 数据库的主要数据来源是各种科学杂志和其他蛋白质数据库,是研究蛋白质相互作用的主要数据库之一。
BioGRID 数据库收集的蛋白质和遗传相互作用数据主要来自酵母菌、果蝇和人等。BioGRID 数据库的主要数据来源是文献研究和其他高通量相互作用数据库,其中每一条相互作用数据都包括实验验证编码,并且可以链接到支持的出版期刊。目前,最新的版本4.2.192 中包含了1 997 840 个蛋白质和基因相互作用。
IntAct 数据库是一个开源的分子相互作用数据库,由高质量文献或用户直接提交的蛋白质相互作用数据组成。到目前为止,IntAct 数据库包含了来自22 037篇出版物的1 130 596个精选的相互作用。上述4 个数据库的链接地址如表2 所示。

表2 蛋白质相互作用数据库Table 2 Protein-protein interaction databases
上述4 个数据库中均涉及多个物种的蛋白质相互作用数据,但酵母菌中的蛋白质复合物被研究得更为广泛,下面对常见的酵母菌蛋白质相互作用数据集进行详细介绍。
在酵母菌蛋白质复合物预测实验中,数据集被分为蛋白质互作数据集和标准复合物数据集。Gavin数据集、Krogan数据集和Collins数据集是常用的蛋白质相互作用数据集。其中,Gavin 数据集可从BioGRID 数据库下载,包含1 855 个蛋白质和7 669个相互作用。该数据集是Gavin 等人通过亲和纯化与质谱技术检测蛋白质相互作用得到的,过程中采用socio-affinity 指数计算两个蛋白质之间相互作用被检测到的概率。Krogan 数据集可由BioGRID 数据库进行下载,含2 674 个蛋白质和7 075 个相互作用。该数据集是Krogan 等人使用LC-MS/MS 技术检测蛋白质相互作用,并借助机器学习的方法评估蛋白质相互作用的可信度。Collins 数据集也可由BioGRID数据库进行下载,其中包含1 622 个蛋白质,9 074 个相互作用。上述3 个酵母菌数据集的具体信息如表3所示。
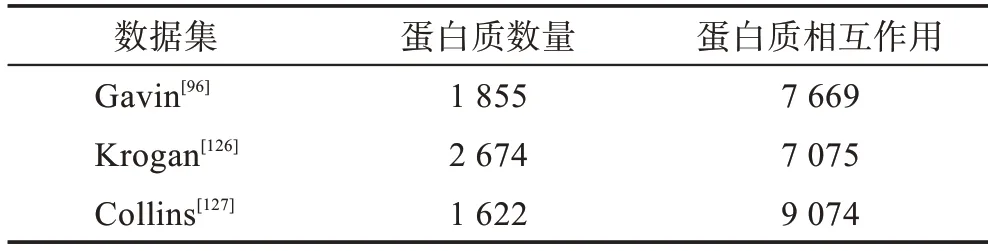
表3 蛋白质相互作用数据集Table 3 Protein-protein interaction data sets
除了之前介绍的蛋白质相互作用数据集,蛋白质的标准复合物数据集通常采用MIPS和CYC2008数据集。其中,MIPS 数据集是常用的蛋白质功能注释数据集,包含了313 个蛋白质复合物。CYC2008 数据集包含349 个通过生物方法检测得到的蛋白质复合物,每个复合物包含2 个或2 个以上的蛋白质,具体信息如表4 所示。
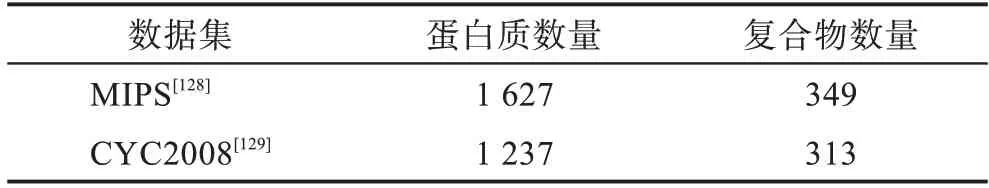
表4 蛋白质复合物数据集Table 4 Protein complex data sets
4 性能比较与分析
为了比较各类算法在不同数据集下的预测效果,本章选取了21 种经典且常用的蛋白质复合物识别算法,如MCODE、ClusterOne 和CPredictor 算法等,并对其结果进行了比较分析。
实验中的蛋白质相互作用数据采用Collins 和Gavin 数据集,将CYC2008 数据集作为标准复合物数据集。表5 和表6 显示了不同算法在Collins 与Gavin相互作用数据集上预测蛋白质复合物的基本情况,其中小复合物表示构成复合物的蛋白质数量不大于3,而包含3 个以上蛋白质的复合物称为大复合物。复合物平均大小是指预测复合物中所含蛋白质数量的平均值。从表5 中可以明显看出,CPredictor2.0 算法预测到的蛋白质复合物数量最多(764 个),并且小复合物数量在所列方法中数量最多。主要是因为CPredictor2.0 通过对蛋白质进行分组后再采用马尔科夫聚类算法将每个蛋白质聚类到复合物中,更有利于产生小规模团簇。另外,EWCA 算法得到大复合物数量最多(588 个),且复合物平均尺寸最大,达到了21.6。表6 显示的是Gavin 数据集上各种方法预测复合物属性的结果比较。Clique方法在Gavin 数据集上预测的复合物数量最多(1 148 个),并且大复合物数量也是所列方法中最多的,这主要是由于Clique算法是基于局部密集子图识别蛋白质复合物。该类算法倾向于将图中的密集团簇预测为复合物,因此倾向于输出较大体积的团簇。
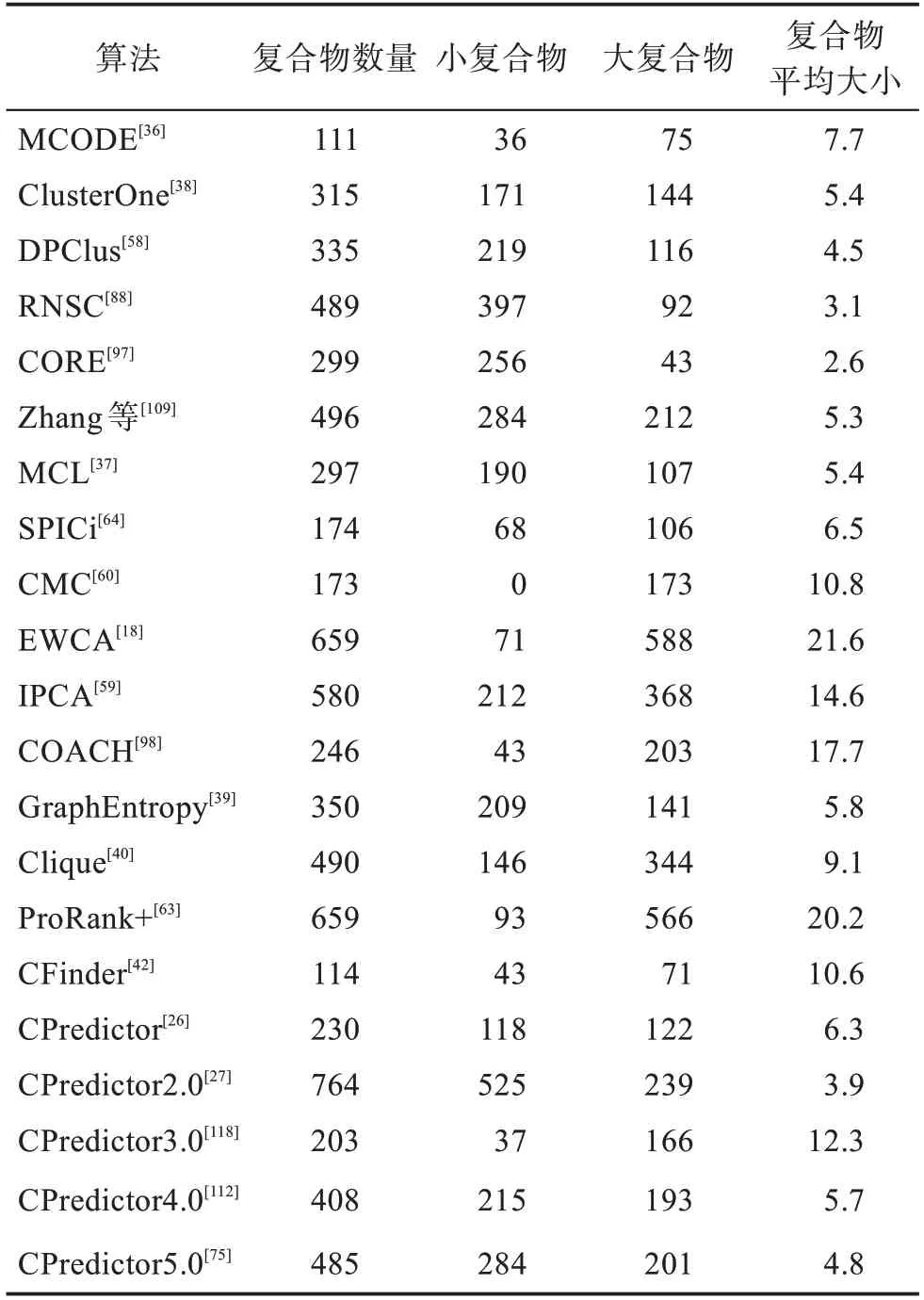
表5 Collins数据集上各种方法预测结果中蛋白质复合物的属性比较Table 5 Attribute comparison of protein complexes for different computational methods on Collins data set

表6 Gavin 数据集上各种方法预测结果中蛋白质复合物的属性比较Table 6 Attribute comparison of protein complexes for different computational methods on Gavin data set
表7 是CYC2008 作为标准复合物数据集时,不同算法在Collins 和Gavin 数据集上性能比较结果,这里使用了召回率()、精确率()和1 值来评估预测结果。在Collins 数据集中,召回率最高的方法是CPredictor5.0,达到了0.60,说明CYC2008中许多蛋白质复合物大部分都可以匹配到预测的复合物,因而在真实蛋白质复合物集合中被正确预测出的复合物比例较高。CPredictor4.0 算法通过采用动态网络预测蛋白质复合物,并在这些方法中精确率达到最高的0.73,并且1 值也最高为0.63。1 值主要得益于较高的召回率和精确率,说明其预测结果质量很高,预测得到的复合物基本和标准集中的复合物相匹配。在Gavin 数据集中,IPCA 算法以0.54的召回率高居第一,同时,发现CPredictor3.0 与CPredictor5.0 算法的召回率同样具有竞争力,而且CPredictor 算法预测的复合物数量远小于IPCA 算法。在精确率与1 值上发现CPredictor4.0 取得了最优结果,这与在Collins 数据集上的发现相一致,说明基于动态网络和从功能到互作的预测算法相比于其他方法更为准确。
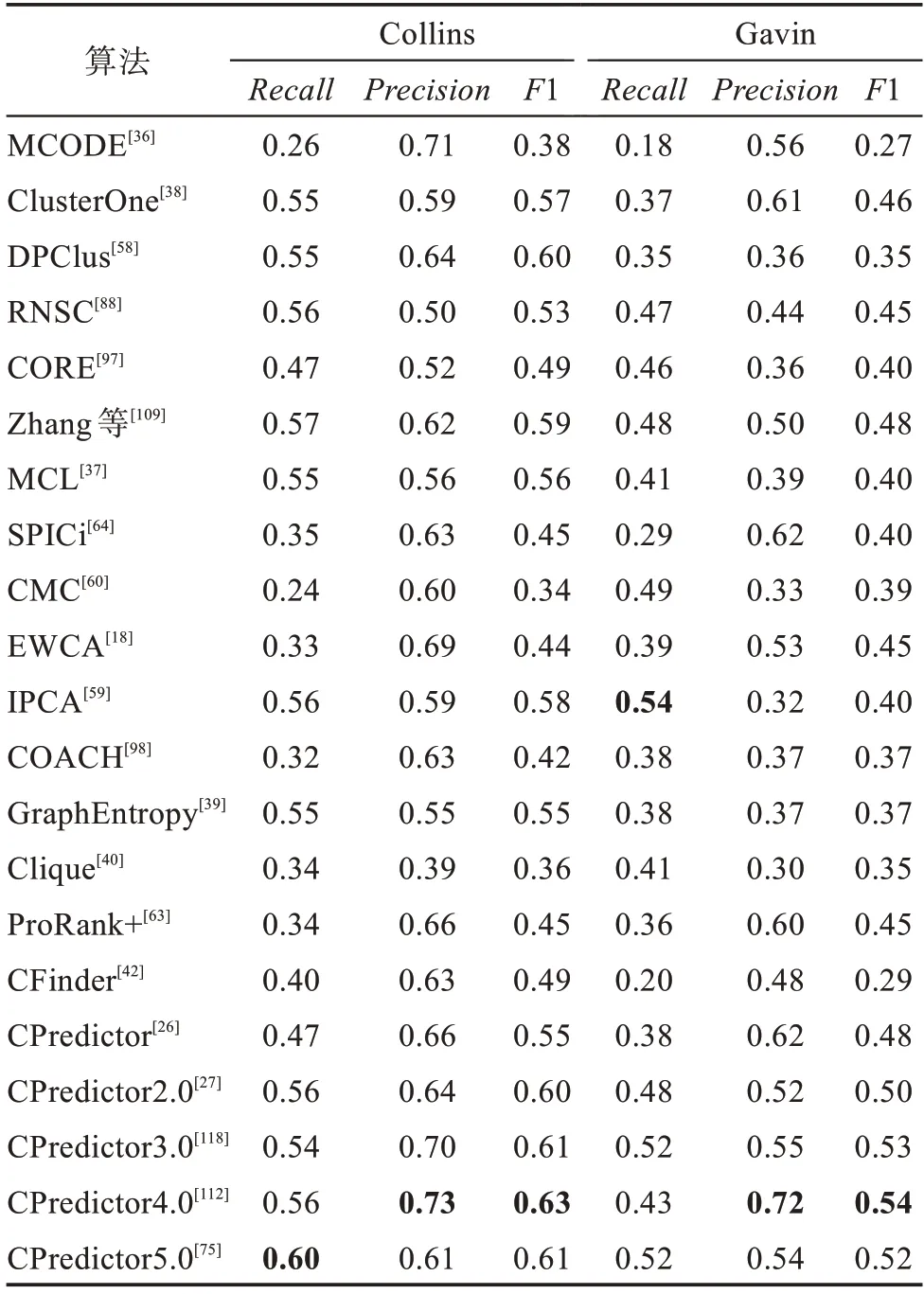
表7 CYC2008 标准库上各种方法的蛋白质复合物预测结果比较Table 7 Comparison of protein complex prediction results for various methods on CYC2008 standard set
综合上述比较,可以发现各种算法都存在各自的优势及不足。基于局部密集子图的算法更加倾向于输出大复合物,但对于小复合物的预测不尽如人意;基于核心-附属结构的算法虽然解决了重叠蛋白质的预测问题,但如何定义核心蛋白质和附属蛋白质仍需要进一步研究;基于动态网络的预测方法取得了较好的预测结果,主要是由于其在多个静态网络上进行复合物的预测,使得其结果更加鲁棒;从结构到互作的预测算法预测的复合物大、小比例更加接近,相比其他算法得到的复合物数量较少,而且预测结果较好。目前,各类方法既有自己的优势也存在自己的劣势,因此基于计算的蛋白质复合物预测算法仍存在很大的发展空间。
5 总结与展望
蛋白质复合物预测作为计算生物学和生物信息学领域的重要课题已被广泛关注。该问题的研究有助于了解生命科学中细胞的功能是如何实现的,从而帮助医学人员了解复杂疾病的各个阶段并最终找到治疗方案。随着高通量技术的发展,蛋白质相互作用数据日益增多,使基于计算的蛋白质复合物预测方法成为可能。本文对基于计算的蛋白质复合物预测算法进行了分类评述与性能比较,将已有的基于计算的蛋白质复合物预测方法大致分为如下七个类别:基于局部密集子图的预测算法、基于核心-附属结构的预测算法、基于动态网络的预测算法、基于监督学习的预测算法、从功能到互作的预测算法、基于多源数据的预测算法和其他方法。需要指出的是,在已有文献中,对于现有预测方法可能会有不同的分类体系,这里不作深究。另外,由于文献众多,力求对主要的工作进行评述,但难免挂一漏万。
尽管基于计算的蛋白质复合物预测已经经历了十多年的发展,取得显著的进展,但仍然存在诸多不足。基于密集子图挖掘的预测方法主要对较大(包含的蛋白质个数>3)的复合物效果较好;基于核心-附属结构的方法只能发现一部分复合物,泛化能力较差;基于动态PPI 网络的预测方法是一个大有可为的研究方向,但目前的工作还很少,有待深入研究;基于监督学习的方法受复合物表达属性影响很大,而且很难寻找到真正符合蛋白质复合物生物性质的属性,预测效果不尽如意。从高通量实验获得的蛋白质互作本身可靠度较低,有大量的假阳性、假阴性存在,在这样的数据上进行检测,即便是结合了像互作网络拓扑结构特征、基因表达数据、蛋白质功能和蛋白质域等某一类信息,也并不能取得令人满意的效果,这方面也需要进一步的探索。未来的研究可以从以下几个方面展开:
(1)具有重叠性质的蛋白质复合物预测
通常,一个蛋白质具有多种功能,即参与到不同的蛋白质复合物中。在PPI 网络中,这些存在相同蛋白质的复合物表现为具有重叠性质的复合物。这些复合物可能形成于不同的细胞周期,出现在不同的位置,即使同时出现,它们仍属于单独的个体,即重叠复合物中的共有蛋白质与不同复合物中的连边不可同时出现。但是在静态PPI 网络中,这些信息并未被反映,因此如何有效预测出这些具有重叠性质的复合物,有待进一步研究。
针对以上问题,研究者们利用结构域(DDI)信息为互作信息构建兼容、互斥数据,并假设一个蛋白质复合物中的边与边之间不能存在相互排斥现象。Ozawa 等人在原有经典算法MCL 和MCODE 的实验结果上,利用以上假设对实验结果进行优化,剔除复合物中具有互斥关系的边。另外,Jung 等人利用结构域信息将PPI 网络划分为多个子网络,其中每个子网络中只包含没有互斥关系的互作。由于复合物的形成要求其中的蛋白质同时同地活跃,Liu 等人利用细胞定位基因本体术语对蛋白质互作网络进行划分,其中每个子网络中的蛋白质出现在同一细胞空间。实验结果表明,这些方法相较于其他方法提升了预测准确率,但是仍需要进一步的研究。
(2)稀疏性复合物的预测
目前,大部分预测算法以预测PPI 网络中的密集子图为出发点,进行蛋白质复合物的预测。但是很多内部连接稀疏的复合物不能被有效预测,加之PPI网络的高假阴性,导致网络中重要互作信息的缺失,这也为预测稀疏复合物增加了难度。PPI 网络中互作信息的缺失源于多个方面。首先,某些在特定生物环境中才出现的互作信息,在生物实验条件有限的情况下很容易被漏检。其次,譬如在串联亲和纯化与质谱实验中,吸附性较低的蛋白质在多次冲洗后很难再被预测到。因此,基于不完整的互作网络预测稀疏性复合物仍然是个挑战。
鉴于以上问题,Srihari 等人在现有预测复合物方法的基础上,结合蛋白质功能等信息向初始复合物中添加具有强功能相似性的蛋白质互作以帮助识别稀疏复合物,虽然在一定程度上提升了稀疏性复合物预测的准确性,但是并未取得令人满意的效果,因此这方面还需进一步的探索。
(3)兼顾大、小复合物的预测
由2、3 个蛋白质组成的复合物被称为小复合物,研究表明,蛋白质复合物大小的分布遵循幂律分布,即小复合物占据很大一部分比例。以目前较成熟的复合物参照集CYC2008 和MIPS 为例,小复合物的数量分别占总数的37.7%与63.6%。由此可见,小复合物的预测对于提升复合物预测的准确性至关重要。
目前,基于局部密集子图的预测算法一般通过预测完全联通子图的方法预测小复合物,但是预测结果中假阳性很高,因此很难准确预测小复合物。此外,PPI 网络的高假阴性导致互作信息的缺失,仅拥有较少连边的小复合物预测依然极具挑战。
针对这个问题,Yong 等人提出基于监督学习的SSS 算法。通过选取互作数据、功能、文本信息以及拓扑结构等多种数据设计特征向量,利用现有复合物参照集构建分类模型来预测任意两个蛋白质属于同一小复合物概率。Xu 等人提出的CPredictor2.0算法为小复合物的预测提供了新的思路。该方法认为复合物是完成某一生物功能的蛋白质集合,因此依据功能信息将蛋白质进行分组,将相同功能的蛋白质分为一组,而没有相同功能的蛋白质被分离。通过使用功能对蛋白质进行分组不仅更加直观、快速、准确,而且得到的复合物也更具有生物意义。上述两个方法在预测小复合物方面略优于现存的其他方法,但是针对小复合物预测的工作还比较少,未来复合物预测方法可以从兼顾不同大小的蛋白质复合物方向进行改进。
(4)动态蛋白质相互作用网络构建
细胞作为生物体的基本结构和功能单位,具有高度的动态性和对环境刺激的响应。同时,蛋白质也随着细胞周期的变化而不断变化。相较于静态蛋白质相互作用网络,动态蛋白质相互作用网络能够更加准确地描述蛋白质相互作用的动态特性。然而目前构建蛋白质相互作用网络的主要方法就是利用基因表达数据,基于three-sigma 准则,根据蛋白质在不同时刻的活跃状态将静态相互作用网络划分为动态的蛋白质相互作用网络。但是这种方法未充分考虑到某些处于活跃状态的蛋白质其基因表达值低的情况,因此如何有效构建动态蛋白质相互作用网络仍是一个研究热点。
(5)基于深度学习算法的蛋白质复合物预测
随着深度学习在图像领域的成功应用,深度学习算法已被运用到了各行各业。近期,谷歌旗下DeepMind 团队推出的Alphafold2,运用深度学习算法在蛋白质结构预测上达到了可与生物实验相匹敌的准确度,改变了人们对计算生物学的认知。而在蛋白质复合物预测领域,基于深度学习算法预测蛋白质复合物的方法还很少。目前主要是运用图嵌入方法,如node2vec、图自编码器等算法,根据PPI网络拓扑结构为网络中的每个节点计算特征,然后对PPI 网络进行降噪处理,以此提高网络质量,进而为后期的相关聚类算法服务。这些方法都是将深度学习算法作为复合物预测的其中一步,并未完全基于深度学习算法端到端地输出蛋白质复合物。因此,将来可望针对蛋白质复合物预测开发一套深度学习模型,用户只需要输入PPI 网络即可得到较好的预测结果。
(6)蛋白质复合物在线预测平台
在线预测平台的开发是生物信息学的重要研究内容,目前蛋白质复合物预测算法越来越多,但是缺少一个高效且直观的在线预测平台。未来可以融合多种公开数据资源,部署高性能计算模型,构建蛋白质复合物在线预测的平台。
