AtNUDX6/7基因缺失型拟南芥基因芯片表达谱的生物信息学分析
2021-12-30尹虹
尹虹
(南方医科大学基础医学院,广东 广州 510515)
Nudix水解酶(nucleoside diphosphates linked to some moiety X,Nudix)广泛存在于真核生物、古细菌、细菌和病毒中,是一类需要Mg2+的酶,Nudix水解酶拥有一个保守的Nudix基序-GX5EX7REVXEEXGU,其中U通常是Ile,Leu或Val[1-3]。Nudix水解酶根据其水解底物的不同进行分类,其水解底物包含有核苷酸糖,二核苷酸聚磷酸, NADH,ADP-核酸,脱氧核糖核苷酸三磷酸,核糖核苷酸三磷酸等[3]。所以,Nudix水解酶不仅能调节各生化途径的中间代谢产物的积累,还可以清除细胞内产生的有毒代谢产物[4-5]。目前,已发现28个拟南芥Nudix水解酶基因,分别分布于细胞质(AtNUDT1-11、AtNUDT25、AtDCP2)、叶绿体(AtNUDT12-18)和线粒体(AtNUDT19-24、AtNUDT26-27)中[2,6]164-168。Ishikawa等[7]发现拟南芥AtNUDT6基因能通过调节NADH正向调控NPR1依赖的水杨酸信号通路。AtNUDT7基因不仅是EDS1信号通路的负调控因子,同时也是2个不同防御途径的负调节因子,既参与植物细胞程序性死亡,也与保持内稳态的平衡相关[8-10]。以往的研究集中在单个拟南芥Nudix水解酶基因缺失或过表达对植株造成的影响,很少有研究探讨2个或多个以上拟南芥Nudix水解酶基因缺失时的分子机制,因此本研究利用BRB-Array Tools基因芯片数据分析软件对来源于公共基因芯片数据库(gene expression omnibus,GEO)的AtNUDX6和AtNUDX7基因共缺失型植株的基因表达谱芯片数据进行差异表达基因筛选,通过生物信息学分析,从整体上了解其信号通路和基因组的变化,进而了解其分子机制[11-13]。
1 材料和方法
1.1 研究材料
本文所用的数据集来自NCBI里的GEO数据库,这个数据集的序列号为GSE74346。所用芯片都是Affymetrix第1代拟南芥基因组芯片,其中包含22 746个拟南芥基因,实验中,总RNA均来自植株的子叶。GSE74346数据集总共含有8张拟南芥芯片样本,分别为两张哥伦比亚生态型拟南芥植株(野生型,GSM1917702和GSM1917703),两张AtNUDX6基因缺失型植株(KO-nudx6,GSM1917704和GSM1917705),两张AtNUDX7基因缺失型植株(KO-nudx7,GSM1917706和 GSM1917707),以及两张AtNUDX6和AtNUDX7基因共缺失型植株(KO-nudx6/7,GSM1917708和GSM1917709)。
1.2 研究方法
1.2.1 数据过滤和标准化以及差异基因的筛选
从NCBI中的GEO数据库中下载以上 8 个GSM, 解压后放入同一文件夹中。利用BRB-Array Tools Version 3.8.0(http:/ /linus.nei.nih.gov/BRB-ArrayTools.html)软件对芯片数据进行统计学分析,为获得表达信号强且质量高的基因,我们采用中位值的方法对数据进行标准化[11-12]852-855,242-249。对基因过滤时选取基因的标准包括:(1)基因表达数据的缺失数≤50%;(2)基因中位数值至少发生1.5倍的改变,且这种改变≥20%的样本数。对通过过滤标准的基因进行t检验,通过Class comparison对两组样本进行分类比较,找到不同基因缺失型拟南芥植株的差异表达基因(P<0.01)[11]852-855。
1.2.2 不同基因缺失型植株的差异基因分析
利用Venny 2.0在线软件(http://bioinfogp.cnb.csic.es/tools/venny/index.html)对拟南芥不同基因缺失型植株的差异表达基因做交集分析。
1.2.3 AtNUDX6和AtNUDX7基因共缺失型植株特有差异基因的生物信息学分析
利用Metascape在线工具[9]204-215对AtNUDX6和AtNUDX7基因共缺失型植株特有差异表达基因进行 GO 本体分析与 KEGG 通路分析,其中P为阈值。GO本体 Cluster 分为分子功能(molecular function,MF)、细胞组分(cellular component,CC)、生物学过程(biological process,BP)[12]242-249。
1.2.4 AtNUDX6和AtNUDX7基因共缺失型植株特有差异基因的蛋白相互作用网络分析
利用Cytoscape 3.2.1(http://cytoscape.org)中的GENEMANIA软件包[10]1523对AtNUDX6和AtNUDX7基因共缺失型植株特有差异基因进行蛋白相互作用网络的构建,并利用Cytoscape中的Network Analysis插件对蛋白相互作用网络进行分析,得到网络的相关拓扑系数,如各节点的度(degree),挑选出在网络中节点(蛋白)度≥10的中心节点,蛋白质相互作用网络的中心节点即是该网络的核心蛋白[14]。本研究中,我们将d≥10的节点定为核心蛋白。
2 结 果
2.1 数据过滤和标准化以及差异表达基因的筛选
通过设定的数据过滤标准及中位值标准化处理后,22 746个探针中有2247个通过了过滤标准,可进行后续的生物信息学分析。应用 BRB-Array Tools 软件在上千种表达差异显著的基因中对两组样本进行了分类比较(P<0.01),其中,AtNUDX6基因缺失型植株相对于野生型植株共有3个上调基因和3个下调基因。AtNUDX7基因缺失型植株相对于野生型植株共有8个上调基因和13个下调基因。AtNUDX6和AtNUDX7基因共缺失型植株相对于野生型植株共有161个上调基因和152个下调基因,见图1。

A:AtNUDX6基因缺失型;B:AtNUDX7基因缺失型;C:AtNUDX6和AtNUDX7基因共缺失型
2.2 不同基因缺失型植株的差异基因分析
应用Venny 2.0在线软件对AtNUDX6基因缺失型植株,AtNUDX7基因缺失型植株以及AtNUDX6和AtNUDX7基因共缺失型植株的差异基因(相对于野生型植株)的韦恩图进行分析,我们发现,基因AT1G26920在3种缺失型的植株中均呈现出高表达,且均在10倍以上。另外,基因AT2G40260和ATNUDT6基因在AtNUDX6基因缺失型植株和AtNUDX6和AtNUDX7基因共缺失型植株中均下调表达;基因AT4G17470、NATA1、ARP9、COR78和AtNUDT7(探针264450_s_at未找到注释基因)在AtNUDX7基因缺失型植株和AtNUDX6和AtNUDX7基因共缺失型植株中均下调表达。AtNUDX6和AtNUDX7基因共缺失型植株特有的差异基因有304个,其中上调基因160个,下调基因144个,见图2。

图2 3种不同缺失植株的差异表达基因韦恩图
2.3 AtNUDX6和AtNUDX7基因共缺失型植株特有差异基因的生物信息学分析
Metascape在线分析结果显示AtNUDX6和AtNUDX7基因共缺失型植株差异基因显著参与免疫系统过程、真菌应对反应、缺水应对反应、次级代谢过程、茉莉酸刺激的反应、药物反应、防御反应的调控、活性氧反应、次生代谢物生物合成过程、对光强度的响应等多个生物过程(P<0.01),见图3。KEGG通路分析发现(P<0.05),差异基因主要参与植物-病原菌相互作用、α-亚麻酸代谢、苯丙烷类化合物的生物合成以及植物激素信号转导,见图4。Metascape的MCODE插件共寻找到5个密集连接的蛋白质子网络集群motif,见图5。

前20个,P<0.01

图4 AtNUDX6和AtNUDX7基因共缺失型植株差异表达基因生物学过程的 KEGG通路分析
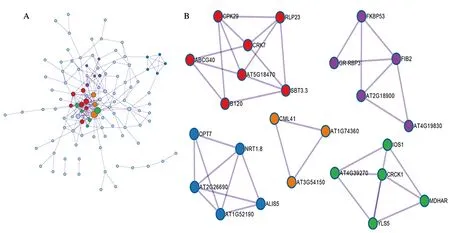
A:PPI网络;B:5种重要motif
2.4 AtNUDX6和AtNUDX7基因共缺失型植株特有差异基因的蛋白相互作用网络分析
AtNUDX6和AtNUDX7基因共缺失型植株特有差异基因的蛋白相互作用网络图,见图6,304个差异探针中只有282个差异基因找到,GENEMANIA数据库中找到517对蛋白相互作用对。AHB1、F13K9.15、NF-YC12以及AKT1是节点度10以上的蛋白,在构建的网络中处于中心位置,属于核心蛋白,其中AHB1、F13K9.15和AKT1为3个上调核心蛋白,NF-YC12为下调核心蛋白。图7可以看出AHB1在AtNUDX6和AtNUDX7基因共缺失型植株中表达量最高。
3 讨 论
本研究从GEO数据库中下载了 8个源自拟南芥植株样本的表达谱数据,进行差异表达分析后显示,ATNUDT6基因在AtNUDX6基因缺失型植株和AtNUDX6和AtNUDX7基因共缺失型植株中均下调表达,AtNUDT7在AtNUDX7基因缺失型植株和AtNUDX6和AtNUDX7基因共缺失型植株中均下调表达,说明构建的模型可信度高,可以反应AtNUDX6和AtNUDX7基因缺失后拟南芥植株的基因表达情况。
AtNUDX6和AtNUDX7基因共缺失型植株特有的差异基因参与植株体内植物防卫反应、次级代谢过程、茉莉酸刺激的反应、损伤反应、真菌应对反应、氧化应激反应、芳香族化合物生物合成过程、活性氧反应等多个生物过程,表明AtNUDX6和AtNUDX7基因能够共同协作参与拟南芥的多种防御反应,并能参与降解细胞的中间代谢物,这与之前的体外研究结果一致[15-16]。
在发现的4个核心蛋白中,AHB1是非共生血红蛋白1,可以被低氧胁迫、蔗糖诱导表达,与氧有很高的亲和性[17-18]。Nudix水解酶可能通过抑制AHB1基因的表达发挥作用。F13K9.15是B-box型锌指蛋白13,目前,对拟南芥B-box型锌指蛋白的研究集中在开花调控、花发育、光形态发生以及光周期调控等方面[19-21],而在植株防御反应的研究报道较少。AKT1是一个钾离子通道蛋白,对钾的吸收积累有重要作用[22-23]。高表达的AKT1能增加拟南芥的逆境胁迫能力[24],可能是AtNUDX6和AtNUDX7基因的下游基因。NF-YC12是 NF-Y 转录因子复合物的一个重要组成成分[25],其详细功能尚未见报道。因此,以上4个核心蛋白在AtNUDX6和AtNUDX7基因共缺失型拟南芥中的具体分子作用机制还需进一步验证。

方格代表AtNUDX6和AtNUDX7基因共缺失型植株特有差异蛋白;黄色方格代表核心蛋白;圆圈代表数据库中与特有差异蛋白相互作用的蛋白

颜色越红,表达越高;颜色越蓝,表达越低
