新疆哈萨克族传统风干肉中细菌多样性分析及安全评价
2021-12-17王俊钢李宇辉刘成江蒲顺昌
王俊钢,李宇辉,刘成江,王 刚,蒲顺昌*
(1 亳州学院生物与食品工程系 安徽亳州236800 2 新疆农垦科学院农产品加工研究所 新疆石河子832000 3 新疆农垦科学院 农产品加工重点实验室 新疆石河子832000)
风干肉是新疆伊犁州哈萨克游牧民族的传统食品,主要是山区牧民为了过冬而制作的生肉制品[1],在气温较低的秋冬季牧民转场时进行制作。将屠宰后的牛、马、羊肉切成条状,用盐涂抹均匀后自然风干而成,其风味品质形成过程与传统发酵肉制品火腿[2]、pastirma[3]、bresaola[4]类似。课题组前期研究发现,新疆传统哈萨克风干肉中的发酵微生物主要包括乳酸菌、微球菌和霉菌[5]。Nam 等[6-8]对韩国当地的传统发酵食品“kochujang”、“cheonggukjang”、“doenjang” 进行了系统的研究,发现传统发酵食品中微生物菌群结构比工业化生产的产品要复杂的多。为深入研究传统发酵食品中微生态体系,可借鉴微生物生态学中研究细菌多样性的技术和方法[9]。
食品微生态学研究方法主要包括传统培养法、基于代谢特征差异的非培养生理生化法、基于DNA 或者RNA 等遗传信息的分子生物学技术和以微生物代谢产物为信息的现代分析技术[10]。其中,高通量测序技术更加适用于自然发酵食品中微生物群落结构的分析,主要优点是能够同时分析多个样品,目前在食品发酵过程的微生物多样性分析和菌群结构分析的研究中应用较为广泛[11]。Greppi 等[12]利用RT-PCR-DGGE 技术能够清晰地分析出意大利salami 香肠在不同发酵阶段细菌的菌群结构变化。Wang 等[13]利用宏基因组学技术,研究中国南北方泡菜中微生物菌群结构,发现南北方泡菜中微生物菌群结构明显不同。Quijada 等[14]利用高通量测序技术对“Chorizo de León”在自然发酵过程中的微生物进行分析,结果表明,不同地区手工制作的传统肉制品在细菌多样性方面存在显著差异。Rebecchi 等[15]采用Illumina 高通量测序技术对不同原料肉制成的腊肠中微生物多样性进行研究,发现不同原料肉制成的腊肠细菌多样性存在明显差异。
新疆传统风干肉是一类自然发酵肉制品,其微生物菌群结构复杂,目前的研究主要为通过传统培养法研究分离自传统风干肉的优势菌的发酵特性,对新疆传统风干肉的细菌多样性研究不够全面。另外,地理环境和生活习惯对不同地区的相同产品中微生物菌群结构影响也较大[16],因此有必要对新疆传统风干肉中的细菌多样性进行研究,明确细菌菌群结构,进一步探究菌群构成与风味品质的形成关系,为传统手工制作风干肉的工业化提供基础数据。
1 材料与方法
1.1 样品
风干肉样品全部来自于新疆哈萨克牧民聚居区,均是牧民手工制作,制作时间为当年11月份,经自然风干40 d 后,用无菌袋逐一进行收集并标记采样时间地点,0~4 ℃冷藏不超过24 h,运回后置于-76~-80 ℃的实验室冰箱中备用,具体如表1所示。

表1 样品来源及分组Table 1 Sample source and grouping
1.2 试剂
细菌DNA 提取试剂盒,美国Biomiga 公司;2×Taq PCR 预混液,南京诺唯赞生物科技有限公司;磁珠法土壤和粪便基因组DNA 提取试剂盒,天根生化科技(北京)有限公司;琼脂糖,范德北京生物科技有限责任公司。
1.3 仪器与设备
TGL-20M 台式高速冷冻离心机,上海卢湘仪离心机仪器有限公司;FA-1004 电子分析天平,天津天有利科技有限公司;Bio-Rad 电泳仪、T100 型PCR 仪、HP1020 凝胶成像系统,美国Bio-Rad 伯乐公司;Axygen 凝胶回收试剂盒,美国Axygen 公司;YX600W 卧式高温灭菌器,上海三申医疗器械有限公司。
1.4 方法
1.4.1 细菌DNA 提取与检测 使用细菌DNA 试剂盒提取样品总DNA。参考田建军等[16]的方法,将180~220 mg 样品于2 mL 置于离心管中,加入1.4 mL 缓冲液涡旋1 min,直到样品充分混匀,按照试剂盒说明书进行细菌DNA 提取,每个样品提取3次,并用乙醇沉淀DNA 进行提纯。
1.4.2 细菌多样性分析 高通量测序分析选取细菌的16S rRNA V4-V5 区序列进行分析[16]。PCR扩增使用T100 型PCR 仪,引物选择16S rRNA V4-V5 区通用引物,515F(5’-GTGCCAGCMGCCGCGG-3’)和926R(5’-CCGTCAATTCMTTTGAG TTT-3’)。采用1.2%琼脂糖电泳仪检测PCR 扩增产物,凝胶成像仪观测后对符合条件的条带进行切胶,用胶回收试剂盒回收后,进行二次PCR 扩增。将接头、测序引物、标签序列添加到目的片段两端,便于Illumina 平台测序。将PCR 产物通过使用专用试剂盒回收后,用RT-PCR 仪对产物进行荧光定量,在IlluminaMiSeq 2×300 bp 平台测序。
PCR 反应体系如下所示[17]:4 μL 5×FastPfu 缓冲液,2 μL 2.5 mmol/L 脱氧核糖核苷三磷酸(Deoxy-ribonucleoside triphosphate,dNTPs),0.8 μL正向引物(5 μmol/L),0.8 μL 反向引物(5 μmol/L),0.4 μL FastPfu 聚合酶,0.2 μL 牛血清蛋白(Bovine serum albumin,BSA),10 ng 模板 DNA补ddH2O 至20 μL。程序如下所示[17]:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,循环数为27;72 ℃最终延伸10 min。PCR 试验结束后,在质量分数为2%琼脂糖凝胶上电泳检测其扩增效果。PCR 产物使用胶回收试剂盒回收,构建文库,之后在IlluminaMiSeqPE 300 bp 平台测序。
1.5 数据处理及分析
使用QIIME 中的PyNAST[18]对拼接得到的原始基因序列条带进行过滤,得到高质量的基因序列条带数据,用FLASH[19]鉴定并去除嵌合体序列[20-21],得到最终的有效数据。用UPARSE[22]软件对基因序列条带在97%的相似度水平下进行聚类分析,并获得OTU[23],并基于细菌分类学数据库对所得到的的信息进行分类学注释。采用R 语言和Python(https://www.python.org/)进行数据分析,通过利用热图或聚类分析对样品中物种丰度与环境因子的相关性进行计算[24]。
2 结果与分析
2.1 样品16S rRNA 基因序列质量评估及Alpha 多样性分析
高通量测序结果显示,26 个样品测序共获得1 788 749 对有效的基因序列条带,平均每个样品产生68 798 条。根据97%相似性水平下的OTU信息,采用Alpha 多样性指标的Chao1、Ace、Shannon、Simpson 指数以及群落覆盖度指数等对得到的有效优化序列进行分析[25],同时评估样品的物种丰度和多样性(表2)。从表2 中可以看出,全部样品的OTU 覆盖率为99.89%~99.99%,说明测序结果基本涵盖了样品中细菌种类,能够准确反应全部样品中的细菌组成。OTU 数量与样品中的细菌丰度呈正相关[26],样品H1、H2、I1 和I2 的OTU数量明显偏少,只有50 左右,其它样品中的OTU数量基本均在100 以上,说明这4 个样品中细菌种类较少,而这4 种样品均源自于富蕴县牧区的风干牛肉样品,可能是由于富蕴地处高纬度高寒地带,环境相对恶劣,因此细菌结构相对简单。图2 表示细菌群落OTU 数目的维恩图,3 组中有237个相同的OTU 数目,全部样品中仅有13 个相同的OTU 数目(图1b),3 组样品中OTU 数目组间差异不显著(P>0.05)。

图1 样品中细菌群落OTU 的维恩图Fig.1 Venn diagram of the bacterial community OTU in the samples

表2 各样品Alpha 多样性指数统计Table 2 Alpha diversity index statistics for each sample
稀释曲线(Rarefaction curve)可以反映检测样品中新物种的出现速度,曲线上某一点的斜率越大,表示出现新物种被检测的速率越快,斜率越小则表示新物种出现的速率越慢。香农指数表示样品中OTU 种类的多少,指数越大样品中微生物种类越多,曲线平坦说明检测完成,OTU 种类不会增加。由图2 可知,对26 个不同地区新疆风干肉中细菌高通量测序分析时,样品测序量低于20 000,OTU 数量还有明显增加,说明此时的样品中还有较多的物种没有被检测。当测序量继续增加到40 000 条时,大部分样品的OTU 虽仍有增加,但是趋势平缓,说明细菌的多样性增加已经不明显,测序量已经可以反映样品中的细菌丰度信息。

图2 样品稀释性曲线(a)和香农指数(b)曲线Fig.2 Sample dilution curves(a)and Shannon index(b)curve
2.2 不同来源风干肉中细菌群落组成分析
不同样品中细菌群落构成可以从高通量测序的分类学分析结果得出,一种颜色代表一个物种。图3a 和4a 为样品中基于门分类水平下丰度在0.1%以上的细菌群落构成。其中变形菌门(Proteobacteria)和蓝细菌门(Cyanobacteria)是第1 组和第3 组的优势菌,而厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)则在第2 组中占比较多。虽然不同样品中菌群结构差异较大,但优势菌门都是变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和蓝细菌门(Cyanobacteria),平均丰度分别为43.24%,26.96%和25.38%,而G1、G2 和J2 3 个样品中的放线菌门(Actinobacteria)丰度比较高,分别达到了9.11%,10.5%和19.15%。这与田建军等[16]、宁亚丽等[27]的研究结果相似。尽管研究对象不同,却有相似的细菌群落结构,主要是自然发酵的食品其主要微生物来源为环境或土壤。经前人研究和本试验测序结果(图3a)可以得出,自然发酵食品的微生物基本上都源自于以上3 个门。从每组的菌群构成来看,变形菌门、厚壁菌门和蓝细菌门仍是新疆风干肉中最主要的细菌,几乎占了样品中细菌丰度的93%以上(图4a),在门分类水平条件下第1 组、第2 组和第3 组3 组样品中的菌群组成有差异,其中第1 组中蓝细菌门占比最大,为44.51%,而第2 组和第3 组中变形菌门占比最大,分别为58.08%和51.02%。变形杆菌门中包括大肠杆菌、沙门氏菌和假单胞菌等病原菌,因此可以判断新疆传统风干肉存在一定的食用安全隐患。厚壁杆菌门主要包括一些乳酸菌和葡萄球菌等全部的革兰氏阳性菌,其中有有益菌也有有害菌,因此必须进一步研究细菌特性才能确定其安全性。
在属水平下,26 个样品中共检测出171 个细菌物种,图3b 显示的是丰度排名在前30 的物种。从图3b 可以看出,样品中检测出的物种平均丰度大于1%的有14 个,分别是嗜冷杆菌(Psychrobacter,30.76%)、Allium_ampeloprasum_leek(24.64%)、环丝菌属(Brochothrix,7.82%)、弧菌属(Vibrio,2.60%)、肉杆菌属(Carnobacterium,2.59%)、芽孢杆菌(Bacillus,2.44% )、Orontium_aquaticum(2.43%)、明串珠菌属(Leuconostoc,2.20%)、假单胞菌属(Pseudomonas,2.04%)、微球菌(Macrococcus,1.69%)、葡萄球菌(Staphylococcus,1.60%)、假交替单胞菌属(Pseudoalteromonas,1.58%)、Clostridium_sensu_stricto_13(1.52%)、肠球菌属(Enterococcus,1.06%),田建军等[16]在研究风干肉中细菌多样性的过程中也发现了类似的结果,这可能是因为选取的研究对象都是传统自然风干肉,且内蒙和新疆生态环境比较类似所致。黄郑朝等[28]在对我国不同地区传统发酵香肠细菌多样性的研究中发现乳杆菌属(Lactobacillus)、葡萄球菌和乳球菌属(Lactococcus)是发酵香肠中的优势菌,其相对丰度都在10%以上,样品中乳杆菌最高丰度可以达到80%,而本试验中采集样品中乳杆菌最多的样品是D1,其相对丰度也只有9.22%,其它的样品中含量较少,所有样品中乳杆菌属的平均丰度只有0.92%;乳球菌属丰度最高的是L2,其相对丰度为4.54%,所有样品的平均丰度仅为0.42%,这可能由于新疆独特的地理环境造成的,另外还可能受样品成熟时间的影响,本试验中的样品风干时间只有40 d,而香肠的发酵时间一般都在90 d 以上。有研究表明,在自然发酵食品中的不同发酵阶段,其微生物结构不同[29-32]。由图4b可知,第1 组和第3 组中嗜冷杆菌和环丝菌属是优势菌群,嗜冷杆菌、环丝菌属和肉杆菌属是第2组的优势菌群,且3 组样品在属水平细菌菌群多样性差异显著(P<0.05)。乳酸菌和凝乳酶阴性葡萄球菌以及部分微球菌被公认为是发酵肉制品的主要微生物,对发酵肉制品品质形成至关重要[33-34],然而本试验中优势的细菌菌群大部分属于条件致病菌,严重影响新疆自然风干肉的安全性。

图3 各样品细菌群落在门(a)和属(b)水平下的结构分布Fig.3 Structure distributed of bacterial in each sample at the phylum(a)and genus(b)level

图4 3 组样品在门(a)和属(b)水平下的细菌群落结构Fig.4 Bacterial community structure of three samples at the phylum(a)and genus(b)levels
聚类热图(Heatmap)能够更加直观地看出不同来源样品之间细菌菌群结构的相似程度(图5)。3 组样品在细菌属水平上的聚类分析相互交叉且有差异,其中只有J2、L1 和L2 3 个样品与其它样品相比群落组成差异较大,这可能是由于样品所处纬度和生态环境相似,采样地点均属于准格尔盆地。试验样品均为哈萨克族传统自然发酵风干肉,其中有4 个风干马肉,22 个风干牛肉,表明使用不同原料制作风干肉,其菌群多样性有不同但差异不大,推测制作地区风干期间的气候因素以及手工操作者的不同是导致样品间细菌群落差异的主要原因。
PCoA 分析结果(图6)可知,26 个样品的细菌群落组成主要在3 个区域,第1 组主要分布区域为一、二区域,第2 组样品主要分布在二、三区域,2 组样品分布较为集中,这2 组间的细菌群落结构相似,第3 组在一、二、三区域均有分布,且两两一组,样品分布位置相对距离远,其组内的细菌群落结构差异大,这可能是因为3 组样品采自3个不同牧民驻地的6 个家庭,这也说明手工操作者的不同是导致样品间细菌群落差异的主要原因之一。
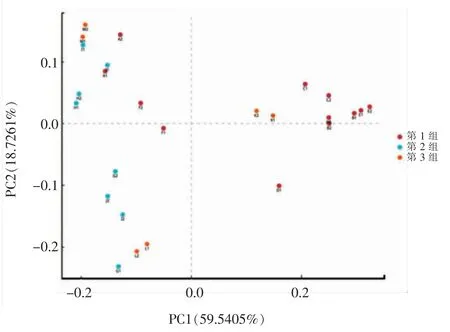
图6 不同样品PCoA 分析图Fig.6 PCoA analysis plots for different samples
3 讨论
传统风干肉由于采用自然发酵方式,整个成熟过程没有添加任何发酵剂,微生物全部来自于原料、环境以及操作者,因此微生物结构复杂,而且存在一定的安全隐患。本试验采用高通量测序技术对3 个地区(第1 组、第2 组和第3 组)26 个自然风干肉样品的细菌群落结构进行分析,就细菌群落的OTU 数目来看,第1 组、第2 组和第3组的组间差异不大。OTU 数目是衡量样品中细菌丰度的指标,这3 组样品均来自于新疆,自然环境类似,3 组样品的OTU 数目差别不大。然而,26 个样品之间的OTU 数目差异较大,这是由于新疆传统风干肉全部为手工制作,操作者的操作方式对样品中的菌群的影响很大,王春艳等[35]在研究新疆传统手工奶酪菌群变化过程中也有类似的发现,因此可以推断即使是在同一地区制作的风干肉,其细菌丰度也会有差别。另外,本试验中选取的26 个样品中有4 个是风干马肉,细菌多样性分析结果表明,这4 个样品中只有J2 和其它样品的细菌菌群多样性差异较大(P<0.05)(图5 和6),J1、M1 和M2 3 个样品与其它样品中的菌群多样性差异不大,说明在本地区影响风干肉中细菌多样性的主要因素应该是环境和风干肉的制作方式。
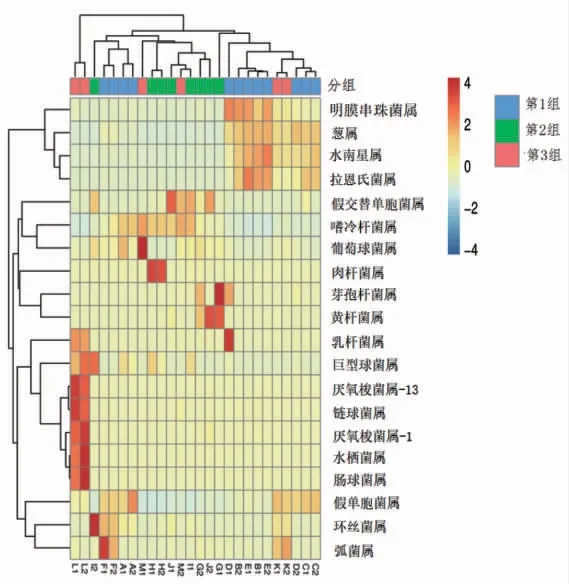
图5 样品在属水平物种丰度聚类热图Fig.5 Heatmap of species abundance at genus level
菌群多样性方面,变形菌门、厚壁菌门、蓝细菌门、放线菌门和拟杆菌门为样品中的主要菌门。变形菌门主要包括一些条件致病菌,影响着食品的安全性。蓝细菌门为样品中的主要菌门之一,说明当地环境比较湿润;放线菌门主要分布在土壤中;拟杆菌门主要分布在动物的肠道中,说明风干肉中这些微生物来源与环境密切有关。厚壁菌门在本试验中检测相对较低,其中的乳酸菌和葡萄球等微生物在发酵过程中可以使肉制品中产生特殊的风味,提升产品品质[36]。田建军等[16]对内蒙、西藏等地风干肉中菌群多样性的研究表明,厚壁菌门约占整个细菌的92%,而本试验中厚壁菌门只占到26.96%。属水平上,26 个样品中检出171 个细菌属,其中嗜冷杆菌是绝对优势菌群,占到整个细菌菌群的30%以上。前人的研究表明,发酵肉制品中的乳酸菌应该是主要的优势菌群[37-39],然而本试验样品中的乳酸菌平均丰度检出量超过1%的只有3 种,分别是肉杆菌属(2.59%)、肠球菌属(1.06%)和明串珠菌属(2.20%)。乳酸菌在肉制品发酵过程中至关重要,不仅可以提供酸性环境,而且还可以产生一些脂肪酶和蛋白酶,对肉制品的风味品质具有一定的提升作用[39]。另外,凝固酶阴性的葡萄球菌也有较强的蛋白酶和脂肪酶活性,可以分解脂肪和蛋白质,产生小分子的游离脂肪酸和多肽,对肉的滋味影响较大。本试验所有样品中葡萄球菌的平均丰度为1.06%,相对较少,然而也不排除这部分葡萄球菌中含有致病性的菌株。为保障食品安全,还需要将高通量测序的结果结合其它技术对菌群进行更加深入的分析和研究。
哈萨克族传统风干肉制作条件相对粗放,原料肉腌制以及风干过程都是在室外进行,卫生质量很难保证,产品容易滋生其它腐败菌。传统风干肉是哈萨克族牧民在冬、春两季的主要食物,其食用的安全性应引起重视。
4 结论
26 个样品高通量测序后共获得1 788 749对有效的基因序列条带,平均每个样品产生68 798 条有效序列,所有样品共检测出的OTU 数目为308 个,单个样品中OTU 数目最多的是J1(237 个);最少的是H1(34 个)。样品中细菌多样性分析表明,变形菌门(43.24%)、厚壁菌门(26.96%)、蓝细菌门(25.38%)丰度最高。细菌属水平结果表明,嗜冷杆菌属(30.76%)、环丝菌属(7.82%)为优势菌群,样品中肉杆菌属(2.59%)、明串珠菌属(2.20%)、微球菌(1.69%)、葡萄球菌(1.60%)、肠球菌属(1.06%)也有检出。丰度聚类热图和Alpha 多样性分析表明,J2、L1 和L2 3 个样品和其它样品的群落组成差异较大,其它样品间群落结构差异不大。菌群多样性分析表明,新疆哈萨克族传统风干肉中含有大量的条件致病菌,具有一定的安全隐患。
