基于脂代谢相关基因的肝癌预后预测模型的构建与评价
2021-10-29曾裕文吴帆谭国钳张芳雍
曾裕文,吴帆,,谭国钳,张芳雍
1 暨南大学附属广州红十字会医院肝胆外科,广州510220;2 广州市红十字会医院肝胆外科
肝癌是我国高发的、危害极大的恶性肿瘤,其近年来的发病率持续上升[1]。肝癌的高异质性和放化疗耐药,以及早期诊断困难、疾病进展迅速和缺乏高效的靶向药物,导致其整体治疗效果并不理想。因此,开发肝癌早期诊断、治疗和预后的评估方法对提高肝癌患者的生存率具有重要意义。研究表明,脂质代谢失调是癌症中最显著的代谢改变之一,不仅影响原发性肿瘤的生长,而且介导肿瘤的进展和转移[2]。JIANG 等[3]通过组织微阵列芯片分析显示,胆固醇酯在254 例肝癌组织中的表达量明显高于癌旁组织,提示脂质代谢可能参与了肝癌的发生发展过程。但迄今为止,脂质代谢在肝癌中的作用尚不明确。2021年1 月—5 月,本研究通过生物信息学分析,筛选在肝癌中可能发挥重要作用的脂代谢基因,构建肝癌患者预后预测模型,并通过该模型分析脂代谢相关基因的表达与肝癌预后的相关性,以期探索脂代谢基因在肝癌诊治中的潜在价值。
1 资料与方法
1.1 mRNA 表达数据和患者临床资料收集 从癌症基因组图谱数据库(TCGA,https://portal.gdc.cancer.gov)和国际癌症基因组联合体数据库(IC⁃GC,https://daco.icgc.org/)下载截至2020年11 月收录的肝癌患者肿瘤组织和正常肝组织的mRNA转录组数据和临床资料(TCGA 库数据以欧美人种为主、ICGC数据仅为亚洲人种)。TCGA数据库共收集到374 个肝癌组织、50 个正常肝组织的mRNA 表达数据,370 例肝癌患者的临床资料,内容包括性别、年龄、生存时间、生存状态、组织学分级和TNM 分期。ICGC 数据库共收集到240 个肝癌组织、202 个正常肝组织的mRNA 表达数据,231 例肝癌患者的临床资料,内容包括患者的生存时间和生存状态。
1.2 脂质代谢基因获取 从基因集合富集分析网站(GSEA,http://www.gsea- msigdb.org/gsea/msig⁃db/index.jsp)下载与脂质代谢相关的基因集,包括脂蛋白代谢(GO_LIPOPROTEIN_METABOL⁃IC_PROCESS)、脂蛋白生物合成(GO_LIPOPRO⁃TEIN_BIOSYNTHETIC_PROCESS) 、膜脂代谢(GO_MEMBRANE_LIPID_METABOLIC_PROESS)、磷脂生物合成(GO_PHOSPHOLIPID_BIOSYNTHET⁃IC_PROCESS)、磷脂代谢(GO_PHOSPHOLIP⁃ID_METABOLIC_PROCESS)、甘油脂质代谢(GOBP_GLYCEROLIPID_METABOLIC_PROCESS)6个脂代谢相关信号通路,以TCGA 数据库下载的374个肝癌组织、50 个正常肝组织的mRNA 表达数据为输入文件,通过GSEA 软件(4.0.1 版本)对所选6 个信号通路进行基因富集分析。
1.3 肝癌组织中差异表达脂质代谢基因筛选 采用perl 软件(5.26.3 版)提取6 个脂代谢相关信号通路中的基因,然后通过R 软件(4.0.2 版)对所提取的基因按照P<0.01,logFC>1.5 的标准进行过滤,筛选出在肝癌组织中差异表达的脂质代谢基因。
1.4 肝癌预后相关关键脂代谢基因筛选 通过R软件的limma 包将所有表达上调脂代谢基因的表达数据与生存数据合并,引用survival 包进行单因素Cox 分析,分别计算表达上调脂代谢基因的风险比(HR)和P,按照P<0.05的标准进行过滤。将筛选得到的基因纳入多因素Cox 回归分析,筛选能独立影响肝癌预后的关键脂代谢基因。
1.5 基于脂代谢相关基因的肝癌预后预测模型建立 基于关键脂代谢相关基因的表达构建肝癌患者预后预测模型,模型公式:风险评分=基因1的表达量×基因1的β1系数+基因2的表达量×基因2的β 2系数+……+基因n 的表达值×基因n 的βn 系数,其中β 系数由R 软件在多因素Cox 回归分析时直接得出。
1.6 基于脂代谢相关基因的肝癌预后预测模型评价
1.6.1 TCGA 数据库 按照公式计算TCGA 数据库中370 例肝癌患者的风险评分,取其中位数作为截断值,将肝癌患者分为高风险组和低风险组。先利用R 软件“survival”和“survminer”包对两组进行Ka⁃plan-Meier 生存分析并绘制生存曲线,利用R 软件“survivalROC”包绘制受试者工作特征(ROC)曲线,计算曲线下面积(AUC)。根据患者的年龄、性别、TNM分期和风险评分,利用R软件“rms”包构建诺莫列线图并计算C值。将患者的年龄、性别、组织学分级、TNM 分期和风险评分分别纳入单因素和多因素Cox回归分析并计算各因素的P和HR。
1.6.2 ICGC 数据库 使用ICGC 数据库中下载的肝癌转录组数据,通过R软件的“limma”包将基因的表达数据与生存数据进行合并,按照风险评分计算公式计算出每个样本的风险评分,取其中位数作为截断值,将肝癌患者分为高、低风险组。利用R软件“survival”和“survminer”包对两组患者进行Kaplan-Meier 生存分析并绘制生存曲线,利用R 软件“sur⁃vivalROC”包绘制ROC曲线,计算AUC。
2 结果
2.1 肝癌组织中差异表达脂质代谢基因筛选结果 6 个脂代谢信号通路基因集基因富集分析结果显示,6 个基因集在肝癌组织中表达均上调,P<0.01。见表1。因为6 个基因集均在肝癌组织中富集,故只选择表达上调的基因作为肝癌预后相关脂代谢基因。利用perl 软件提取6 个基因集中的不同基因共757 个,按照P<0.01、logFC>1.5标准过滤后,筛选出在肝癌组织表达上调的基因109 个。

表1 基因集在肝癌中的富集结果
2.2 肝癌预后相关关键脂代谢基因筛选结果 单因素Cox回归分析显示,共有21个P<0.05的肝癌预后相关脂代谢基因。将21个基因纳入多因素Cox回归分析,筛选出6 个能独立影响肝癌预后的关键脂代谢基因,分别为DAGLA、PCSK9、PIGU、FABP6、GLA、ESYT3。
2.3 基于脂代谢相关基因的肝癌预后预测模型构建结果 根据6 个关键脂代谢基因的mRNA 表达水平和β系数计算每个患者的风险评分。预后预测模型为:风险评分=DAGLA×0.161 878 597 + PCSK9×0.014 967 28 + PIGU×0.043 461 843 + FABP6×0.078 505 362 + GLA×0.019 799 065 + ESYT3×0.278 063 77。
2.4 基于脂代谢相关基因的肝癌预后预测模型评价结果
2.4.1 TCGA 数据库 根据预后预测模型计算风险评分,取风险评分中位数(0.85)为截断值,将TC⁃GA 数据库中的370 例肝癌患者分为低风险者(185例)、高风险者(185 例)。Kaplan-Meier 生存分析显示,高风险者的生存时间为(1.79 ± 1.95)年,低风险者的生存时间为(2.46 ± 1.95)年,高风险者的生存时间低于低风险者(P<0.01)。ROC 曲线分析显示,预测肝癌患者生存率的AUC 为0.732。利用R软件构建诺莫列线图,见图1。评价得到C 值为0.706,提示该模型对肝癌患者生存率的预测能力良好。单因素和多因素Cox 回归分析结果显示,风险评分及TNM 分期是影响肝癌患者预后的独立影响因子(P均<0.01),而年龄、性别和组织学分级对患者的预后无明显影响(P均>0.05)。
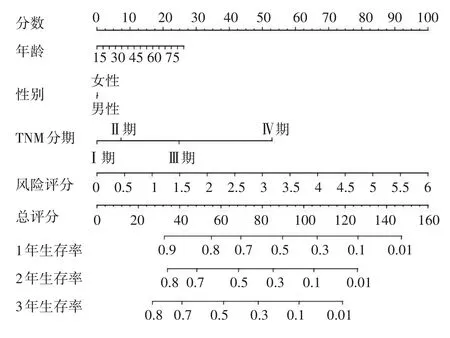
图1 基于脂代谢相关基因的肝癌预后预测模型诺莫列线图
2.4.2 基于ICGC 数据库的预后预测模型评价结果 取风险评分中位数(15.21)为截断值,将ICGC数据库中的231 例肝癌患者分为高风险者(115 例)和低风险者(116 例),Kaplan-Meier 生存分析显示,高风险者的生存时间为(2.05 ± 1.22)年,低风险者的生存时间为(2.39 ± 1.04)年,高风险者的生存时间低于低风险者(P<0.01)。ROC 曲线分析显示,预测肝癌患者生存率的AUC为0.693。
3 讨论
脂质代谢异常是近年来肿瘤研究领域的热点,研究发现脂类从头合成的增加是人类癌症的共同特征[4]。例如,脂肪酸合成酶及催化脂肪酸合成的关键酶在人类恶性肿瘤中存在过表达,水解单酰甘油释放游离脂肪酸的脂解酶在侵袭性肿瘤细胞中过表达,而通过抑制脂肪酸合成酶可抑制多种恶性肿瘤细胞系的增殖活性[5]。研究显示,与非恶性肿瘤性肝组织相比,参与脂肪酸生物合成的基因在大多数肝癌组织中普遍上调,但脂质代谢及其相关基因与肝癌预后的关系尚不清楚[6]。
为此,本研究通过GSEA 数据库下载与脂质代谢相关的信号通路,每个信号通路含有数十至数百个不等的基因,最后整理出所有信号通路中的不同基因共757个,以此作为脂质代谢相关基因。从TC⁃GA 数据库下载肝癌患者的mRNA 转录组数据和临床资料,利用生物信息学技术筛选出在肝癌组织中表达上调的脂代谢相关基因共109 个。为探究这些差异基因的表达水平与肝癌预后的关系,先利用R软件中的survival 包进行单因素Cox 分析,初步筛选出21 个与肝癌预后相关的脂代谢基因。考虑到单因素Cox 回归分析每一次只纳入一个变量,可能存在过度拟合的现象,因此对单因素Cox 回归筛选出的21 个基因进行多因素Cox 回归分析,最终筛选得到了6 个与肝癌预后密切相关的脂代谢基因,分别为DAGLA、PCSK9、PIGU、FABP6、GLA、ESYT3,本研究即以这6 个基因作为模型构建基因构建了肝癌预后预测模型。
为评估该模型的预测价值及可靠性,本研究通过肝癌预后预测模型公式计算出TCGA 数据库中370 例肝癌患者的风险评分,并根据风险评分的中位数将肝癌患者分为高风险者和低风险者。生存分析结果显示,高风险组的预后劣于低风险组;ROC曲线显示,预测肝癌患者生存率的AUC 大于0.7,说明该模型对肝癌预后具有较好的预测能力[7]。本研究根据风险评分和肝癌患者临床信息构建的诺莫列线图可以判断该模型的取分能力,根据诺莫列线图得出的C 值越接近1,说明列线图的预测能力越准确。我们构建的预测模型C值为0.706,表示该模型具有较好的区分能力[8]。本研究还将风险评分和肝癌临床病理特征一起进行单因素和多因素Cox 回归分析,结果显示风险评分和TNM 分期同为影响肝癌患者预后的独立影响因子。上述结果提示,本研究模型的风险评分与肝癌预后密切相关且可较好的预测肝癌患者预后。考虑到TCGA 数据库中主要为欧美人种数据,为进一步验证该模型的预测价值,本研究通过ICGC 数据库下载了亚洲人种的肝癌转录组数据和临床数据,通过肝癌预后预测模型公式计算出ICGC 数据库中231 例肝癌患者的风险评分,发现高风险者的生存时间低于低风险者,预测肝癌患者生存率AUC 为0.662,该结果TCGA 数据库的结果基本一致。
在构成本模型的6 个脂代谢基因中,PCSK9 和PIGU 已被证明可促进肝癌的发展,PCSK9通过抑制肝癌细胞的凋亡来促进肿瘤生长,是肝癌患者预后不良的指标[9];PIGU 可通过激活NF-κB 途径和增加免疫逃逸来促进肝癌的进展[10]。DAGLA 和FABP6在其他肿瘤中有被报道,是致癌基因。DAGLA 可催化二酰甘油水解为2-花生四烯酰甘油和游离脂肪酸,通过促进细胞周期进程影响口腔癌的发生和发展[11]。OHMACHI等[12]通过对78例结直肠癌的肿瘤与癌旁组织进行免疫组化发现,FABP6 在肿瘤组织中的表达明显高于正常组织,且与肿瘤细胞的增值和侵袭密切相关。GLA 和ESYT3 在肿瘤中少有报道,但均有研究发现其与脂质代谢有关,比如ESYT3在脂质稳态和细胞内信号传导中起着重要作用,敲减ESYT3 可改善血脂代谢异常[13]。尽管其中一些基因在肝癌中的作用尚未有研究报道,但本研究显示它们均在肝癌中过表达且能影响肝癌患者预后,提示这些基因在肝癌发生发展过程中发挥了重要作用,有必要进一步研究其在肝癌中的具体功能及分子机制。肿瘤可通过多种机制逃避机体的免疫监视,其中一种机制即涉及肿瘤脂质库的改变以及脂质和脂肪酸的积累[14]。LUO 等[15]研究发现,荷瘤小鼠肿瘤相关巨噬细胞中的脂质含量明显高于无肿瘤小鼠的巨噬细胞,且较高的脂质含量降低了巨噬细胞的吞噬能力。本研究中的6个基因均在脂质代谢信号通路中高表达且与患者预后呈负相关,提示肝癌细胞可能通过上调这些基因的表达改变其自身的脂代谢过程,从而降低其周围环境中免疫细胞的抗肿瘤能力并得以逃避免疫监视。
综上所述,本研究通过生物信息学分析手段,筛选出6 个肝癌预后相关关键脂代谢基因,分别为DAGLA、PCSK9、PIGU、FABP6、GLA 及ESYT3。以这6个基因作为模型构建的肝癌预后预测模型对肝癌患者的预后有较好的预测价值及可靠性,提示脂代谢基因可能是潜在的肝癌标志物或治疗靶点。
