中华猕猴桃WOX转录因子的生物信息学分析
2021-10-16付春黄春莉江纳杨瑶君
付春 黄春莉 江纳 杨瑶君

摘 要:WOX基因家族是植物特有的一類转录因子,在植物发育关键时期,如胚的形成、维持干细胞稳定性和器官形成过程中发挥重要调控作用。为探究WOX转录因子调控中华猕猴桃的生长发育的分子理论基础,该文利用ProtParam、Cell-PLoc2.0、SignalP4.1 Server等在线软件对中华猕猴桃的16个WOX转录因子进行理化特性、亚细胞定位和信号肽等详细的生物信息学分析。结果表明:(1)中华猕猴WOX转录因子的所有蛋白成员的氨基酸数目在138~371之间,分子量为16.222~42.185 kD,均定位于细胞核的不稳定亲水蛋白。(2)中华猕猴桃的16个WOX蛋白成员均属于Homodomain超家族,仅Achn362451是分泌蛋白,且仅Achn362451和Achn141001具有跨膜区。(3)大部分WOX蛋白成员的潜在磷酸化位点都位于丝氨酸处。(4)其二级结构主要以无规则卷曲为主,其次为α-螺旋。(5)保守基序分析结果表明中华猕猴桃的16个WOX蛋白成员均含有motif1。(6)系统进化分析结果表明中华猕猴桃与菠萝的亲缘关系最近,与番茄的亲缘关系最远。(7)在中华猕猴桃果实成熟过程中不同时期的转录组分析结果表明其WOX基因家族中4个成员Achn131681、Achn145561、Achn336591、Achn362451基因在授粉后20、120、127 d表达含量都较高,其他12个WOX基因成员表达含量低甚至不表达。该研究为进一步研究中华猕猴桃WOX转录因子在其生长发育过程中的分子机理提供了一定的参考依据。
关键词:中华猕猴桃,WOX,转录因子,基因家族,生物信息学分析
中图分类号:Q943.2
文献标识码:A
文章编号:1000-3142(2021)09-1561-15
Abstract:WOX gene family is a kind of transcription factor peculiar to plants,and plays an important regulatory role in the critical stages of plant development,such as embryo formation,stem cell stability and organ formation. In order to explore the molecular basis of WOX transcription factors regulating the growth and development of Actinidia chinensis,the physical and chemical characteristics,subcellular localization and signal peptide of 16 WOX transcription factors in A. chinensis were analyzed by ProtParam,Cell-PLoc2.0,SignalP4.1 Server online software in detail in this study. The results were as follows:(1)The amino acid number of all protein members of WOX transcription factors in A. chinensis ranged from 138 to 371,with molecular weight ranging from 16.222 to 42.185 kD,all of which were unstable hydrophilic proteins located in nucleus. (2)The 16 WOX protein members of A. chinensis belonged to Homodomain superfamily,only Achn362451 was secreted protein,and only Achn362451 and Achn141001 had transmembrane regions. (3)The potential phosphorylation sites of most WOX members were located in serine site. (4)The secondary structure of WOX proteins was mainly random coil,followed by α-helix. (5)Conservative motifs analysis results showed that all 16 WOX protein members of A. chinensis contained motif1. (6)Phylogenetic analysis results showed that the genetic relationship between A. chinensis and Ananas comosus was the closest,and that between Actinidia chinensis and Solanum lycopersicum was the farthest. (7)The results of transcriptome analysis in different stages of fruit ripening process of Actinidia chinensis showed that four members of the WOX gene family,Achn131681,Achn145561,Achn336591,Achn362451,were highly expressed on 20,120,127 d after pollination,while those of the other 12 WOX gene members were low or even not expressed. This study provides a reference basis for further study on the molecular mechanism of WOX transcription factors in the growth and development of A. chinensis.
Key words:Actinidia chinensis,WOX,transcription factor,gene family,bioinformatics analysis
中华猕猴桃(Actinidia chinensis),多年生藤本植物,是杜鹃花目(Ericales)猕猴桃科(Actinidiaceae)的一种经济树木。中华猕猴桃原产自我国,果实富含维生素C,且维生素C在人体中的利用率高达94%(宋圃菊等,1984),一个成年人每天只摄食一个鲜猕猴桃就可满足人体对维生素C的需求,因而被誉为“维C之王”。果实酸甜可口,除了鲜食,还可加工成各种食品和饮料。临床试验发现,猕猴桃汁能有效抑制麻风杆菌,从而明显改善麻风病的临床症状。除此之外,猕猴桃汁所含的一种抗突变成分谷胱甘肽,可抑制诱发致癌基因的突变,并有效阻断致癌物质亚硝酸基吗啉的合成,从而防止癌症的发生(《湖北林业科技》编辑部,2007)。
WUSCHEL(WUS)相关同源框(WOX)蛋白家族成员特异存在于植物中,在茎顶端分生组织、根顶端分生组织和形成层中干细胞的维持和增殖等生长发育中发挥重要作用。根据系统进化分析,模式植物拟南芥(Arabidopsis thaliana)中15个WOX转录因子被分为以ATWOX10,13,14在内的遠古进化支,以ATWOX8,9,11,12在内的中间进化支,和以WUS,ATWOX1-7在内的现代进化支/WUS进化支(高丽等,2015)。其中,远古进化支起源于藻类植物,中间进化支起源于维管植物,现代进化支起源于种子植物(Lian et al.,2014)。随着对WOX转录因子的深入研究,WOX蛋白家族在各个植物生长发育中的功能及其作用机理逐渐明确,特别是在模式植物拟南芥中,大多数WOX蛋白成员的功能及作用机理都已明确。ATWOX1的过度表达导致拟南芥分生组织发育和多胺稳态的缺陷(Zhang et al.,2011)。ATWOX2和ATWOX8在卵细胞和合子中共表达,但由于其不对称分裂分别仅局限于合子的顶端和基部子细胞(Lie et al.,2012)。ATWOX3调控旁侧器官(叶和花)的发育,其突变体会出现花瓣缺失或变窄(Shimizu et al.,2008)。ATWOX4涉及形成层中维管束的干细胞的维持。ATWOX5在根尖静止中心特异性表达,主要维持根尖分生组织(RAM)中干细胞的活性(Haecker,2004)。ATWOX6主要在胚珠中表达,在珠被和卵细胞的形成过程中可防止细胞提前分化,进而调控胚珠的生长发育(Park SO,2005);Zhu et al.(2004)指出ATWOX6可影响冷胁迫的应答。ATWOX9可识别发育信号并调控细胞周期(Wu et al.,2005)。ATWOX10的具体作用虽未明确,但已有研究发现ATWOX10可能受到光信号、生长素、赤霉素和脱落酸等植物激素以及胁迫等多种信号途径的协同作用(王占军等,2017)。ATWOX11和ATWOX12共同影响外植体根的发育(Liu et al.,2014)。ATWOX13和ATWOX14在根部及花药中表达,主要防止其提前分化(Deveaux et al.,2008)。
关于植物基因组中的WOX基因家族鉴定和生物信息学分析的报道较多。利用隐马尔科夫模型对番茄(Solanum lycopersicum)基因组进行WOX基因家族鉴定并发现有10个WOX基因成员,同时分析其WOX蛋白结构域和基因结构及系统进化关系(李晓旭等,2016)。蝴蝶兰(Phalaenopsis aphrodite)和石斛兰(Dendrobium catenatum)基因组中分别含有14个和10个WOX基因成员,其中,蝴蝶兰有3个WOX基因成员出现基因重复,而在石斛兰基因组中没有出现WOX基因重复(Ramkumar et al.,2018)。茶树(Camellia sinensis)基因组中有18个WOX基因成员,且其系统进化树分为3组,分别是古老分支、中间分支和现代分支三类(Wang et al.,2019)。在甘蓝型油菜、白菜和甘蓝基因组序列中分别鉴定出52、25、29个WOX基因成员(Li et al.,2019)。大豆(Glycine max)基因组中含有33个WOX基因成员,并定位在19条染色体上,而16号染色体没有WOX基因(Hao et al.,2019)。对毛白杨(Populus tomentosa)的PtoWUSa、PtoWOX4a和PtoWOX5a三个基因编码蛋白质的结构域和在不同组织中的表达进行了详细分析(李建波,2017;王爽等,2019)。在玉米(Zea mays)基因组中鉴定出31个WOX基因成员,且分析了其编码蛋白的理化特性和系统进化关系(郑玲等,2019)。毛竹(Phyllostachys heterocycla)基因组中有27个WOX基因成员(李晨曦等,2016),而在其他竹类植物基因组中,俄莉竹(Olyra latifolia)有12个WOX基因成员,Raddia guianensis含有9个WOX基因成员,瓜多竹(Guadua angustifolia)含有18个WOX基因成员以及芸香竹(Bonia amplexicaulis)含有26个WOX基因成员(Li et al.,2020)。在葡萄(Vitis vinifera)基因组中鉴定出10个WOX基因成员,且其与拟南芥的进化树分为祖先分支、中间分支和现代分支三类(王鹏飞等,2018)。小麦(Triticum aestivum)基因组中含有80个WOX基因成员,其中定位在A、B、D基因组上的分别有23、26、29个,另有2个WOX基因成员定位不明确(李仲卿,2018)。苹果(Malus pumila)基因组中含有12个WOX基因成员,且在果实中的表达含量较高(王楚堃等,2020)。小桐子(Jatropha curcas)基因组中有12个WOX基因成员,且在不同组织中的表达呈显著性差异(唐跃辉等,2019)。棉花(Gossypium hirsutum)基因组中有37个WOX基因成员,其中,26个WOX基因在棉花根、茎、叶、花等器官中都有表达(吕有军等,2017)。簸箕柳(Salix suchowensis)基因组中被鉴定出15个WOX基因成员,且定位在20条染色体上(Wang et al.,2019)。在人参(Panax ginseng)基因组中鉴定出10个WOX基因成员(Liu et al.,2019)。虽然关于植物基因组中WOX基因家族的鉴定、功能分析的研究较多,但对于中华猕猴桃基因组中WOX基因家族的鉴定及其分子功能研究的文献报道较少。
关于中华猕猴桃基因组中转录因子的研究报道有R1R2R3-MYB转录因子增强其干旱胁迫反应(Ya et al.,2019),EIN3/EIL转录因子的鉴定,系统进化及其基因表达分析(陈强等,2018),MADS-box转录因子与中华猕猴桃乙烯促成熟相关(Mcatee et al.,2015),还有bZIP家族的相关报道(于丰源等,2020),但未见中华猕猴桃基因组中WOX转录因子鉴定和生物信息学研究的报道。为研究中华猕猴桃WOX基因家族编码蛋白质的分子功能及其在生长发育过程中的作用如何和非生物胁迫条件下的应激反应如何。本研究拟对中华猕猴桃16个WOX基因成员进行理化特性、亚细胞定位、蛋白高级结构及系统进化等详细的生物信息学分析,为后续进一步研究中华猕猴桃的生长发育和分子功能等提供一定的参考依据和理论基础。
1 材料与方法
1.1 材料
中华猕猴桃的16个WOX转录因子的蛋白序列来自于PlantTFDB(http://planttfdb.cbi.pku.edu.cn/)数据库,并通过该数据库下载与中华猕猴桃WOX转录因子成员Achn172381同源的番茄、矮牵牛花(Petunia inflata)、辣椒(Capsicum annuum)、拟南芥、油菜(Brassica napus)、葡萄、菠菜(Spinacia oleracea)、大豆、菠萝(Ananas comosus)、小麦、毛竹、玉米、水稻(Oryza rufipogon)、花生(Arachis hypogaea)、馬铃薯(Solanum tuberosum)的WOX转录因子成员的蛋白序列用于不同物种间WOX转录因子的系统进化关系分析研究。
1.2 方法
1.2.1 理化性质分析 利用在线程序ProtParam (https://web.expasy.org/protparam/)对中华猕猴桃的WOX基因进行理化性质分析,包括分子量、氨基酸数、等电点、带负电荷残基数、带正电荷残基数、不稳定指数、脂肪指数,以及亲疏水性的预测分析。并利用数据库PlantTFDB(http://planttfdb.cbi.pku.edu.cn/blast.php)进行Blastp,分别找出中华猕猴桃16个WOX转录因子在模式植物拟南芥对应的基因。通过Cell-PLoc2.0 (http://www.csbio.sjtu.edu.cn/bioinf/ Cell-PLoc-2/)对该家族成员进行亚细胞定位分析。
1.2.2 保守结构域、信号肽的分析及跨膜域的预测 利用NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测WOX蛋白的保守结构域;利用SignalP4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)分析WOX蛋白的信号肽;在TMHMM Severv.2.0 (http://www.cbs.dtu.dk/services/TMHMM/)在线程序输入中华猕猴桃16个WOX转录因子蛋白序列进行跨膜域预测分析。
1.2.3 磷酸化位点的预测分析 通过在线工具NetPhos 3.1 Server (http://www.cbs.dtu.dk/services/NetPhos/)对中华猕猴桃的WOX转录因子潜在磷酸化位点进行预测分析。
1.2.4 二级结构、三级结构的分析预测 使用SOPMA程序(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对中华猕猴桃WOX转录因子的蛋白质二级结构进行预测分析;利用在线工具SWISS-MODEL(https://swissmodel.expasy.org/)对其蛋白质的三级结构进行预测分析。
1.2.5 保守基序分析 利用在线工具MEME(http://meme-suite.org/tools/meme)对中华猕猴桃的WOX蛋白家族进行保守基序分析,参数设置:motifs的数值设为10,其余参数默认。
1.2.6 序列的多重比对及进化树的构建 利用中华猕猴桃的Achn172381蛋白序列在PlantTFDB(http://planttfdb.cbi.pku.edu.cn/blast.php)数据库进行Blastp,分别找到番茄、辣椒、油菜、葡萄、玉米、拟南芥等其他15物种中的同源蛋白序列,并分别下载下来。通过软件MEGA-X(Sudhir et al.,2018)中的ClutalW比对功能对中华猕猴桃的Achn172381蛋白序列与拟南芥等其他15种植物的同源蛋白序列进行多重比对,参数默认;并用邻接法(nerghbor-join,NJ)构建进化树,Bootstrap replications值设定为1 000。
1.2.7 中华猕猴桃在不同生长发育时期的WOX基因表达分析 中华猕猴桃授粉后20、120、127 d的转录组原始数据下载于NCBI(https://www.ncbi.nlm.nih.gov/)数据库(SRA No.SRA065642)(Huang et al.,2013),利用Tang et al.(2016)对该转录组原始数据进行注释分析其授粉20、120、127 d后的表达谱结果,通过筛选中华猕猴桃WOX基因家族的16个基因成员表达水平FPKM值分析其WOX基因表达水平情况。
2 结果与分析
2.1 中华猕猴桃WOX蛋白的理化性质分析
利用ProtParam预测分析中华猕猴桃16个WOX转录因子的理化性质,分析结果显示中华猕猴桃WOX转录因子的氨基酸范围为138(Achn107771)~371(Achn363401);分子量大小范围为16.222(Achn107771)~42.185 kD(Achn363401);等电点为5.14(Achn362341)~9.79(Achn141001),其中,Achn053291、Achn362341、Achn104531、Achn362451、Achn145561的等电点均小于7,它们可能编码弱酸性蛋白,在酸性亚细胞环境中发挥作用;不稳定指数为46.01~74.15,表明均为不稳定蛋白(不稳定指数>40为不稳定蛋白);脂肪指数为44.53(Achn053291)~79.49(Achn141001),说明蛋白成员间热稳定性差异较大。负电荷的氨基酸残基数(Asp + Glu)为13(Achn107771)~37(Achn362451),正电荷的氨基酸残基数(Arg + Lys)为14(Achn107771)~42(Achn363401),其中,除Achn053291、Achn362341、Achn104531、Achn362451、Achn145561这5个成员的负电荷的氨基酸残基总数大于正电荷的氨基酸残基总数外,其余13个WOX蛋白成员的正电荷的氨基酸残基总数大于负电荷的氨基酸残基总数。亚细胞定位分析表明,中华猕猴桃的16个WOX蛋白成员均位于细胞核(表1)。
根据各个WOX蛋白成员的氨基酸组成不同,主要分为a、b、c、d、e 五类:a类以丝氨酸为主,包括Achn172381、Achn220151、Achn285781、Achn207451、
Achn053291、Achn104531、Achn145561、Achn172571、Achn362341、Achn141001這10个成员,其中Achn053291丝氨酸的含量最高,为15.20%;b类中仅有Achn362451这1个成员,其氨基酸组成以丝氨酸和谷氨酰胺为主,丝氨酸和谷氨酰各占9.30%;c类以亮氨酸为主,包括Achn131681、Achn107771、Achn336591这3个成员,其中Achn107771亮氨酸最高,占10.90%;d类中以亮氨酸和苏氨酸为主,仅含Achn299661这1个成员,亮氨酸和苏氨酸各占10.40%;e类中以苏氨酸为主,只含Achn363401这1个成员,苏氨酸占为11.10%(图1)。
对中华猕猴桃WOX转录子进行氨基酸亲疏水性Protscole预测发现中华猕猴桃的16个WOX蛋白成员的GRAVY值均为负,即这16个成员均为亲水蛋白。其中总平均亲水指数最大的为Achn053291,其值为-0.954,该成员的第8、第9、第10、第11、第12、第13、第14位氨基酸表现为最大亲水性,其值为-3.5;第152位氨基酸表现为最大疏水性,其值为1.533。总平均亲水指数最小的是Achn104531,其值为-0.402,该成员的第84、第85、第86位氨基酸表现为最大亲水性,其值为-3.567;第250位氨基酸表现为最大疏水性,其值为2.5(表2,图2)。
2.2 保守结构域预测、信号肽的分析及跨膜域的预测分析
中华猕猴桃的16个WOX转录因子的保守结构域预测分析表明中华猕猴桃的16个WOX转录因子中,仅Achn145561有Homodomain superfamily和PIG-H superfamily两个保守结构域,其余15个WOX蛋白成员只含Homodomain superfamily这一个保守结构域。中华猕猴桃WOX蛋白成员的保守结构域长度为38~61,其中,Achn172381、Achn131681、Achn299661、Achn363401这4个成员的保守结构域等长,均为59,但位置各异;Achn207451与Achn053291的保守结构域等长,均为60,且位置吻合;Achn362341与Achn104531的保守结构域长度和位置均吻合;Achn362451与Achn145561中的Homodomain superfamily保守结构域等长,均为58,但位置不同(表3,图3)。
SignalP4.1 Server信号肽预测分析结果显示除Achn141001外,其余15个中华猕猴桃转录因子均无信号肽。因此,推测Achn141001为分泌蛋白,其余均为非分泌蛋白(图4)。TMHMM Severv.2.0跨膜域分析结果显示,除Achn362451、Achn141001有明显的跨膜结构域外,其余14个WOX转录因子均无明显的跨膜结构域。因此,推测Achn362451、Achn141001为跨膜蛋白(图5)。
2.3 磷酸化位点的预测分析
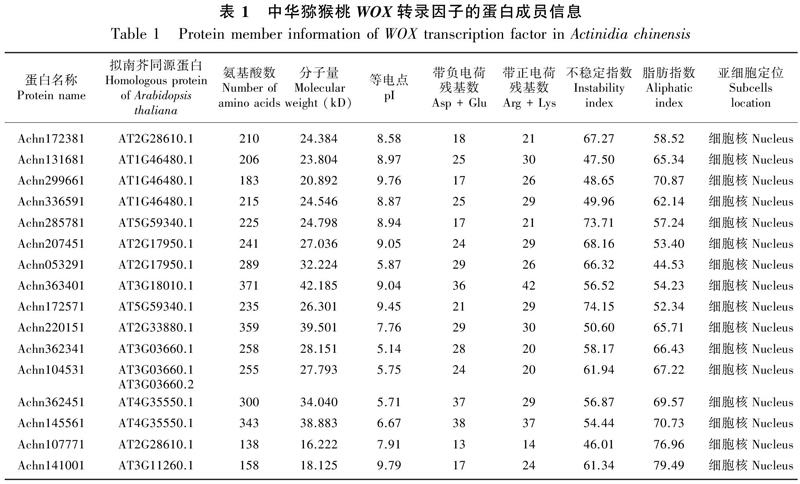
NetPhos 3.1磷酸化位点分析表明16个WOX蛋白成员中,Achn172381、Achn336591、Achn285781、Achn207451、Achn053291等13个WOX蛋白成员中磷酸化位点的总数:丝氨酸>苏氨酸>酪氨酸;Achn131681和Achn299661中磷酸化位点总数:苏氨酸>丝氨酸>酪氨酸;Achn363401中磷酸化位点总数:丝氨酸=苏氨酸>酪氨酸。就单个成员而言,除Achn299661的潜在磷酸化位点为苏氨酸外,其余15个WOX蛋白成员潜在磷酸化位点均为丝氨酸。在16个WOX蛋白成员中,极易在丝氨酸处磷酸化的成员为Achn207451、Achn053291、Achn172571(0.998),极易在苏氨酸处磷酸化的成员为Achn362341和Achn104531(0.995),极易在酪氨酸处磷酸化的成员为Achn362451(0.978)(表4,图6)。
2.4 二级结构和三级结构的预测分析
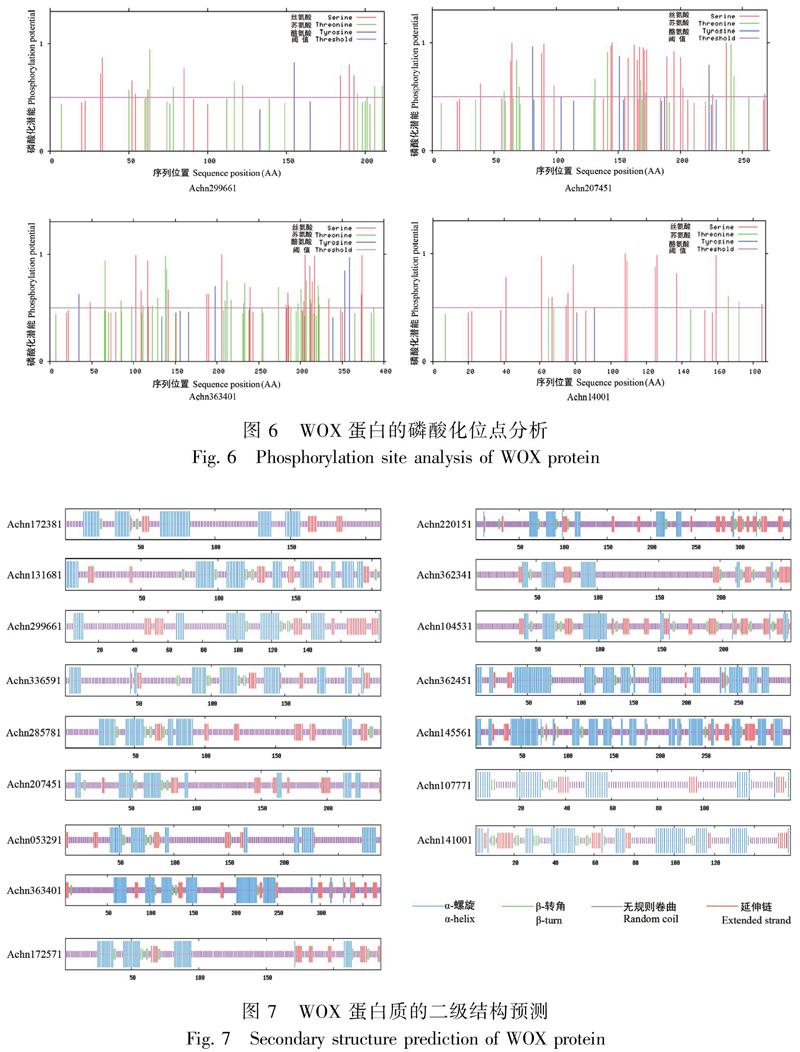
使用SOPMA程序对中华猕猴桃WOX转录因子的二级结构进行预测分析。结果显示,大部分WOX蛋白成员的二级结构都以无规则卷曲为主,其次为α-螺旋和延伸链,β-转角含量最少。其中,Achn172381、Achn131681、Achn299661、Achn336591、Achn285781等12个成员的二级结构含量为无规则卷曲>α-螺旋>延伸链>β-转角;Achn220151、Achn220151和Achn104531三个成员的二级结构含量为无规则卷曲>延伸链>α-螺旋>β-转角;Achn141001的二级结构含量为无规则卷曲=α-螺旋>延伸链>β-转角。α-螺旋在成员中占比为12.79%(Achn362341)~42.00%(Achn362451);延伸链在成员中占比为4.00%(Achn362451)~21.18%(Achn104531);β-转角在成员中占比为2.38%(Achn172381)~9.49%(Achn141001);无规则卷曲在成员中占比为69.38%(Achn362341)~37.34%(Achn141001)(表5,图7)。
通过在线工具SWISS-MODEL对中华猕猴桃WOX转录因子进行蛋白质的三级结构预测,结果显示,16个WOX蛋白成员的蛋白质三级结构可分为11类。Achn172381与Achn220151为一类,其代表为Achn220151;Achn131681与Achn336591为一类,其代表为Achn131681;Achn207451、Achn053291、Achn172571为一类,其代表为Achn053291;Achn362341与Achn104531为一类,其代表为Achn362341;Achn299661、Achn285781、Achn363401、Achn362451、Achn145561、Achn107771、Achn141001各代表一类(图8)。
2.5 保守基序分析
利用在线工具MEME对中华猕猴桃的WOX家族进行保守基序分析,结果表明:motif1出现在中华猕猴桃的所有的WOX蛋白成员中,motif2出现在除Achn107771和Achn141001外的其他WOX蛋白成员中。根据不同成员中所含motif不同,将中华猕猴桃的16个WOX蛋白成员分为A、B、C三个组。A组主要含motif1、motif2、motif3、motif5和motif10的蛋白成员有Achn104531与Achn362341,只含motif1、motif2、motif3和motif5的有Achn362451与Achn145561,只含motif1、motif2、motif3和motif10的有Achn220151;B组主要含motif1、motif2、motif4、motif7、motif8和motif9的有Achn131681与Achn336591,只含motif1、motif2、motif7和motif9的有Achn299661,只含motif1、motif2和motif4的有Achn172381与Achn363401;C组主要含motif1、motif2、motif4和motif6的有Achn285781与Achn172571,含有motif1、motif2、motif4和motif7的有Achn053291与Achn207451,只含motif1和motif4的有Achn141001,只含motif1的为Achn107771(图9)。
2.6 中华猕猴桃WOX基因家族在不同时期的表达水平分析
通过对中华猕猴桃授粉后20、120、127 d的转录组表达分析发现其WOX基因家族中Achn131681、Achn145561、Achn336591和Achn362451成员在中华猕猴桃授粉后20 d的基因表达水平显著提高,成员Achn362451在中华猕猴桃16个WOX基因成员中表达含量最高。在授粉后120 d,成员Achn131681、Achn145561和Achn362451基因表达水平显著提高,Achn362451表达含量最高。在授粉后127 d,成员Achn145561和Achn362451的表达含量较高,且Achn362451表达含量在所有WOX基因中表达含量中最高。从中华猕猴桃授粉后20、120、127 d的生长发育成熟过程来看,成员Achn131681基因表达水平是先上升后下降;成员Achn145561基因表达水平一直上升,在授粉后127 d达最高水平;成员Achn336591基因表达水平是先上升后下降,在授粉后127 d几乎不表达;成员Achn362451基因表达水平是先上升后下降,但在授粉127 d后表达含量也较高。而除以上4个基因成员外,其他12个WOX基因在其授粉后20、120、127 d表达含量很低甚至不表达(图10)。
2.7 系统发育树构建
中華猕猴桃与拟南芥WOX转录因子构建的系统进化树结果表明,其进化树分为三大分支,与拟南芥的三大进化支即远古进化支、中间进化支和现代进化支/WUS进化支相吻合。因此,推测Achn145561和Achn362451为古代进化支,Achn220151、Achn362341、Achn104531为中间进化支,Achn172381、Achn107771、Achn285761、Achn172571、Acn299661、Achn131681、Achn336591、Achn363401、Achn207451、Achn053291、Achn141001为现代进化支,即WUS进化支(图11)。
对中华猕猴桃、拟南芥、番茄等16种植物的WOX基因家族的蛋白序列进行多重比对并构建进化树。其进化树可分为GroupⅠ、GroupⅡ、Group Ⅲ三组,分别包括番茄、辣椒、矮牵牛花、拟南芥、油菜、葡萄、菠菜、大豆在内的Group I,包括中华猕猴桃、菠萝、玉米、水稻、小麦以及毛竹在内的Group Ⅱ,包括马铃薯及花生在内的Group Ⅲ。分支的远近及距离长短表明,中华猕猴与菠萝的亲缘关系最近,与番茄的亲缘关系最远(图12)。
3 讨论与结论
本研究对中华猕猴桃的16个WOX转录因子进行了蛋白质理化性质、保守结构域预测、信号肽、跨膜结构域预测、磷酸化位点、保守基序、二级结构分析、三级结构分析和进化树等详细生物信息学分析。王楚堃等(2019)对苹果WOX转录因子研究发现苹果中WOX蛋白成员的氨基酸数量为176~381,分子量为20.122~45.337 kD。李雪垠等(2019)对谷子和狗尾草WOX转录因子研究发现,谷子中WOX蛋白成员的氨基酸数量为212~533,分子量为20.011~54.998 kD;狗尾草中WOX蛋白成员的氨基酸数量为212~531,分子量为23.969~54.788 kD。郭亚静等(2020)对星油藤WOX转录因子的研究中发现星油藤WOX蛋白成员氨基酸数量为220~420,分子量为24.95~45.37 kD。同时,在对小桐子(唐跃辉等,2019)、番茄(李晓旭等,2016)、棉花(吕有军等,2017)、乌拉尔图小麦(单强强等,2019)、玉米(郑玲等,2019)、簸箕柳(Wang et al.,2019)、蒺藜苜蓿和鹰嘴豆(张洪涛,2018)中WOX蛋白成员的研究中发现,其WOX蛋白成员的氨基酸数量和分子量差异很大。其中,对棉花(吕有军等,2017)、苹果(王楚堃等,2019)和乌拉尔图小麦(单强强等,2019)WOX蛋白成员进行稳定性和亲疏水性预测分析发现其WOX蛋白成员均为不稳定的亲水蛋白,张洪涛等(2018)对蒺藜苜蓿和鹰嘴豆WOX蛋白成员的亲疏水性分析发现蒺藜苜蓿和鹰嘴豆中WOX蛋白成员均为亲水蛋白。本研究结果表明,中华猕猴桃的16个WOX蛋白成员氨基酸数量为138~371,分子量为16.222~42.185 kD的不稳定亲水蛋白。结合前人以及本研究中WOX蛋白质理化特性分析结果可以推测植物大部分WOX蛋白成员的氨基酸数量和分子量差异性很大且为不稳定的亲水蛋白。对乌拉尔图小麦(单强强等,2019)、玉米(郑玲等,2019)、鹰嘴豆(张洪涛,2018)、簸箕柳(Wang et al.,2019)、谷子和狗尾草(李雪垠等,2019)基因组中的WOX蛋白理化特性分析结果发现,其大部分WOX蛋白成员为定位在细胞核的碱性蛋白,而在蒺藜苜蓿(张洪涛,2018)、棉花(吕有军等,2017)、苹果(王楚堃等,2019)、星油藤(郭亚静等,2020)中WOX蛋白成员以酸性蛋白为主,在番茄的WOX蛋白成员中酸性蛋白和碱性蛋白各占一半。棉花WOX转录因子的亚细胞预测结果显示,棉花中的WOX蛋白成员大部分定位于细胞质和微体,少部分定位于细胞核。中华猕猴桃的WOX蛋白成员大多为定位于细胞核的碱性蛋白,极少数为定位于细胞核的酸性蛋白。此结果与乌拉尔图小麦、玉米鹰嘴豆、簸箕柳、谷子中WOX蛋白成员的分析结果有相似之处,但由于缺少具体实验数据的支撑,暂不能得出WOX蛋白成员中亚细胞定位与酸碱性的关系结论,但可以推测不同的物种中WOX蛋白成员由于发挥的功能不同造成其WOX蛋白成员的亚细胞定位和酸碱性也会有所差异。由氨基酸组成来看,中华猕猴桃中16个WOX蛋白成员主要以丝氨酸为主,其次为亮氨酸、苏氨酸和谷氨酰胺,而在其他物种中对WOX蛋白成员的氨基酸组成分析尚未见报道。
保守结构域分析表明,中华猕猴桃的16个WOX转录因子均属于Homodomain superfamily,此结果与玉米(郑玲等,2019)、番茄(李晓旭等,2016)、葡萄(王鹏飞等,2018)等研究结果一致,在玉米(郑玲等,2019)、番茄(李晓旭等,2016)、葡萄(王鹏飞等,2018)等研究中,所有的WOX蛋白成员都含有homeobox这一结构域。信号肽和跨膜结构域显示,在中华猕猴桃中,大多数成员无跨膜区和信号肽,仅Achn362451是分泌蛋白,且仅Achn362451和Achn141001具有跨膜区,为跨膜蛋白。而在陆地棉花(吕有军等,2017)中,所有的WOX都不含跨膜区和信号肽。故推测大多数WOX蛋白成员为非跨膜蛋白或非分泌蛋白。
中华猕猴桃大多数WOX蛋白二级结构主要以无规则卷曲为主,其次为α-螺旋和延伸链,β-转角含量最少。此结果与陆地棉中WOX转录因子的二级结构含量预测结果相似(吕有军等,2017;Hao et al.,2019)。保守基序分析结果显示,中华猕猴桃中所有的WOX蛋白成员均含有motif1。李晓旭等(2016)对番茄的WOX基因家族研究中发现所有成员都含有motif1,并指出motif1是由60个氨基酸残基组成的保守的同源异型结构域homeodomain。王鹏飞等(2018)在葡萄的WOX基因家族研究中发现所有WOX蛋白成员均含有motif1和motif2,并进一步分析指出这两个结构域序列是包含WOX蛋白的homeobox结构域的序列。王楚堃等(2019)在苹果WOX基因家族研究中发现,其WOX所有蛋白成员均含motif1,且motif1结构域包含WOX家族homeobox的结构域序列。李晨曦等(2016)在毛竹WOX基因家族研究中发现,所有WOX蛋白成员都含有motif1和motif3,且指出motif1和motif3共同组成了homeodamin。本研究的中华猕猴桃WOX蛋白基序分析结果与以上这些植物基因组中WOX蛋白的保守基序分析结果相似,故推测中华猕猴桃中motif1在所有基序中最为保守,可能包含有WOX蛋白的homeobox结构域序列。
系统进化研究揭示了中华猕猴桃的16个WOX蛋白成员被分为远古进化支、中间进化支和现代进化支。此结果与番茄、棉花、玉米、苹果、葡萄、乌拉尔图小麦、星油藤、欧洲油菜(Li et al.,2019)、人参(Liu et al.,2019)、玫瑰和野蔷薇、蒺藜苜蓿和鹰嘴豆中构建的WOX进化树的分支相同,但与毛竹构建的WOX进化树不同。李晨曦等(2016)在构建的毛竹WOX进化树中发现,其WOX蛋白成员分为现代/WUS进化分支和古老进化支,并指出由于缺少与10个拟南芥对应同源基因,且不含中间进化支,故与一般种子植物含有的3个分支不同。在中华猕猴桃与其他15个物种构建的进化树中,中华猕猴与菠萝的亲缘关系最近,与番茄的亲缘关系最远。中华猕猴桃授粉后20、120、127 d的转录组表达分析结果表明,其Achn131681、Achn145561、Achn336591和Achn362451基因表达水平显著提高,Achn131681、Achn336591和Achn362451基因表达水平呈先上升后下降的趋势,而Achn145561基因表达水平一直上升,除了这4个WOX基因外,中华猕猴桃其他12个WOX基因在其生长发育及其成熟过程中表达含量很低甚至不表达。本研究为进一步研究中华猕猴桃WOX转录因子的分子功能提供了一定的理论依据。
参考文献:
CHEN Q,LIU J,CHEN DY,et al.,2018. Genome-wide identification,phylogenetic evolution,and gene expression pattern of EIN3/EIL transcription factors in Actinidia chinensis [J]. Chin J Appl Environ Biol,24(2):315-321.[陈强,刘建,陈丹阳,等,2018. 中华猕猴桃EIN3/EIL转录因子家族的成员鉴定、系统进化和基因表达模式[J]. 应用与环境生物学报,24(2):315-321.]
DEVEAUX Y,TOFFANO-NIOCHE C,CLAISSE G,et al.,2008. Genes of the most conserved WOX clade in plants affect root and flower development in Arabidopsis[J]. BMC Evol Biol,8(1):291-0.
Editorial Department of Hubei Forestry Science and Technology. 2007. Nutritional value and health function of kiwifruit[J]. Hubei For Sci Technol,(2):9.[《湖北林業科技》编辑部,2007. 猕猴桃的营养价值与保健功能[J]. 湖北林业科技,(2):9.]
GAO L,SUN YM,SHAO TM,et al.,2015. Studies on WUSCHEL-related homebox(WOX)protein family[J]. Biotechnol Bull,31(5):7-12.[高丽,孙祎敏,邵铁梅,等,2015. 植物WUSCHEL-related homeobox (WOX)家族研究进展[J]. 生物技术通报,31(5):7-12.]
GUO YJ,FU QT,HU XD,et al.,2020. Identification and expression analysis of WOX family genes in sacha inchi (Plukenetia volubilis)[J]. Mol Plant Breed,18(12):3907-3915.[郭亚静,付乾堂,胡晓笛,等,2020. 星油藤WOX基因家族成员的鉴定及其表达谱分析[J]. 分子植物育种,18(12):3907-3915.]
HAECKER A,2004. Expression dynamics of WOX genes mark cell fate decisions during early embryonic patterning in Arabidopsis thaliana[J]. Development,131(3):657-668.
HAO QN,ZHANG L,YANG YY,et al.,2019. Genome-wide analysis of the WOX gene family and function exploration of GmWOX18 in soybean[J]. Plants (Basel,Switzerland),8(7).
HUANG SX,DING J,DENG DT,et al.,2013. Draft genome of the kiwifruit Actinidia chinensis[J]. Nat Comm,4(4): 2640.
LI CX,CHEN L,JIANG T,et al.,2016. Identification and expression analysis of WUSCHEL-related homeobox(WOX)gene family in moso bamboo[J]. Mol Plant Breed,14(8):2003-2011.[李晨曦,陈磊,江涛,等,2016. 毛竹WUSCHEL-related homeobox(WOX)基因家族鉴定及表达分析[J]. 分子植物育种,14(8):2003-2011. ]
LI JB,2017. Functional analysis of WOX and HSP17.8 in poplar development and stress resistance[D]. Beijing:Chinese Academy of Forestry.[李建波,2017. 楊树WOX和HSP17.8基因在植物生长发育和抗逆胁迫中的调控作用研究[D]. 北京:中国林业科学研究院.]
LI MD,WANG RH,LIU ZY,et al.,2019. Genome-wide identification and analysis of the WUSCHEL-related homeobox (WOX)gene family in allotetraploid Brassica napus reveals changes in WOX genes during polyploidization[J]. BMC Genomics,20(1).
LI XX,LIU C,LI W,et al.,2016. Genome-wide identification,phylogenetic analysis and expression profiling of the WOX family genes in Solanum lycopersicum[J]. Hereditas,38(5):444-460.[李晓旭,刘成,李伟,等,2016. 番茄WOX转录因子家族的鉴定及其进化、表达分析[J]. 遗传,38(5):444-460.
LI XY,LI J,CAI MM,et al.,2020. Identification and evolution of the WUSCHEL related homeobox protein family in Bambusoideae[J]. Biomolecules,10:739.
LI XY,WU YM,ZHANG FJ,2019. Genome-wide identification and analysis of WOX transcription factor genes in Setaria italic and Setaria viridis[J]. J Shanxi Agric Sci,47(6):957-962.[李雪垠,武懿茂,张凤洁,2019. 谷子和狗尾草WOX转录因子基因的全基因组鉴定与分析[J]. 山西农业科学,47(6):957-962. ]
LI ZQ,2018. Genome-wideanalysis of WOX gene family and functional research of TaWUS in wheat[D]. Shandong:Shandong Agricultural University.[李仲卿,2018. 小麦WOX基因家族的全基因组分析及TaWUS的功能研究[D]. 山东:山东农业大学.]
LIAN GB,DING ZW,WANG Q,et al.,2014. Origins and evolution of WUSCHEL-related homeobox protein family in plant Kingdom[J]. Sci World J,(1):1-12.
LIE C,KELSOM C,WU XL,2012. WOX2 and STIMPY-LIKE/WOX8 promote cotyledon boundary formation in Arabidopsis[J]. Plant J,72(4):674-682.
LIU J,JIANG C,CHEN T,et al.,2019. Identification and 3D gene expression patterns of WUSCEHEL-related homeobox (WOX)genes from Panax ginseng[J]. Plant Physiol Biochem,143(2019):257-264.
LIU JC,SHENG LH,XU YQ,et al.,2014. WOX11 and 12 are involved in the first-step cell fate transition during de novo root organogenesis in Arabidopsis[J]. Plant Cell,26(3):1081-1093.
L YJ,YANG WJ,ZHAO LJ,et al.,2017. Genome-wide identification and analysis of WOX family genes in Gossypium hirsutum L.[J]. Acta Agric Boreal-Sin,32(2):21-31.[吕有军,杨卫军,赵兰杰,等,2017. 棉花WOX转录因子家族基因的全基因組鉴定与分析[J]. 华北农学报,32(2): 21-31.]
MCATEE PA,RICHARDSON AC,NIEUWENHUIZEN NJ,et al.,2015. The hybrid non-ethylene and ethylene ripening response in kiwifruit (Actinidia chinensis)is associated with differential regulation of MADS-box transcription factors[J]. BMC Plant Biol,15(1):304.
PARK SO,2005. The PRETTY FEW SEEDS2 gene encodes an Arabidopsis homeodomain protein that regulates ovule development[J]. Development,132(4):841-849.
RAMKUMAR TR,KANCHAN M,UPADHYAY SK,et al.,2018. Identification and characterization
of WUSCHEL-related homeobox (WOX)gene family in economically important orchid species Phalaenopsis equestris and Dendrobium catenatum[J]. Plant Gene,14:37-45.
SHAN QQ,YANG WL,LI YJ,et al.,2019. Cloning and exoression analysis of WOX genes in Triticum urartu[J]. J Tritic Crops,39(8):903-911.[单强强,阳文龙,李英洁,等,2019. 乌拉尔图小麦WOX基因的克隆与表达分析[J]. 麦类作物学报,39(8):903-911.]
SHIMIZU R,JI J,KELSEY E,et al.,2008. Tissue specificity and evolution of meristematic WOX3 function[J]. Plant Physiol,149(2):841-850.
SONG PJ,TANG Y,YU XX,1984. Utilization of ascorbic acid in concentrated kiwifruit juice in human body[J]. Acta Nutr Sin,6(1):35-40.[宋圃菊,唐仪,俞锡璇,1984. 猕猴桃浓缩果汁中抗坏血酸在人体的利用[J]. 营养学报,6(1): 35-40.]
SUDHIR K,GLEN S,MICHAEL L,et al.,2018. MEGA X:Molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol,(6):6.
TANG W,ZHENG Y,DONG J,et al.,2016. Comprehensive transcriptome profiling reveals long noncoding RNA expression and alternative splicing regulation during fruit development and ripening in kiwifruit (Actinidia chinensis)[J]. Front Plant Sci,7:335.
TANG YH,BAO XX,WANG J,et al.,2019. Genome-wide identification and expression analysis of the WOX gene family in physicnut[J]. Mol Plant Breed,17(4):1154-1162.[唐跃辉,包欣欣,王健,等,2019. 小桐子WOX基因家族全基因组鉴定与表达分析[J]. 分子植物育种,17(4):1154-1162.]
WANG CK,HAN PM,WANG YM,et al.,2019. Genome-wide identification and analysis of apple WUSCHEL-related homeobox(WOX)family genes[J]. Acta Hortic Sin,46(6):1021-1032.[王楚堃,韩朋良,王咏梅,等,2019. 苹果WUSCHEL-related homeobox(WOX)家族基因的鉴定与分析[J]. 园艺学报,46(6):1021-1032.]
WANG P,GUO Y,CHEN X,et al.,2019. Genome-wide identification of WOX genes and their expression patterns under different hormone and abiotic stress treatments in tea plant (Camellia sinensis)[J]. Trees,4(33):1129-1142.
WANG PF,WU XY,YANG LY,et al.,2018. Identification and ananlysis of grape WUSCHEL-related homebox(WOX)family genes[J]. Sino-Overseas Grapevine Wine,(3):16-23.[王鹏飞,吴新颖,杨立英,等,2018. 葡萄WUSCHEL-related homeobox(WOX)家族基因鉴定与分析[J]. 中外葡萄与葡萄酒,(3):16-23.]
WANG S,ZHANG Y,REN MX,et al.,2019. Genome-wide analysis of the WOX family reveals their involvement in stem growth of Populus trichocarpa[J]. Bull Bot Res,(4):568-577.[王爽,张洋,任梦轩,等,2019. 全基因组分析杨树WOX基因家族在茎部发育中的作用[J]. 植物研究,(4):568-577.]
WANG XL,BI CW,WANG CY,et al.,2019. Genome-wide identification and characterization of WUSCHEL-related homeobox(WOX)genes in Salix suchowensis[J]. J For Res,30:1811-1822.
WANG ZJ,HUANG SP,OU ZL,et al.,2017. The cloning and sequence analysis of promoter of WOX10 gene family in Arabidopsis thaliana[J]. J Huangshan Univ,19(3):58-61.[王占军,黄士平,欧祖兰,等,2017. 拟南芥WOX10基因启动子的克隆与序列分析[J]. 黄山学院学报,19(3): 58-61.]
WU XL,DABI T,WEIGEL D,2005. Requirement of homeobox gene STIMPY/WOX9 for Arabidopsis meristem growth and maintenance[J]. Curr Biol,15(5):436-440.
YA BZ,TANG W,LI HW,et al.,2020. Kiwifruit(Actinidia chinensis)R1R2R3-MYB transcription factor AcMYB3R enhances drought and salinity tolerance in Arabidopsis thaliana[J]. J Integr Agric,(2):417-427.
YU FY,JIANG LL,JIA JQ,et al.,2020. Comparative genomics analyses of bZIP family members in Actinidia chinensis[J]. Chin J Appl Environ Biol,26(2):226-235.[于豐源,蒋礼玲,贾举庆,等,2020. 中华猕猴桃bZIP家族成员的比较基因组学分析[J]. 应用与环境生物学报,26(2):226-235.]
ZHANG HT,2018. Analysis of Medicago and Cicer arietinum WOX transcription factor gene family[M]. Harbin:Harbin Normal University.[张洪涛,2018. 蒺藜苜蓿和鹰嘴豆WOX转录因子基因家族分析[M]. 哈尔滨:哈尔滨师范大学.]
ZHANG YX,WU RH,QIN GT,et al.,2011. Over-expression of WOX1 leads to defects in meristem development and polyamine homeostasis in Arabidopsis[J]. J Integr Plant Biol,53(6):493-506.
ZHENG L,FENG MY,HAN JM,et al.,2019. Identification and analysis of WOX family genes in maize[J]. J Maize Sci,27(3):16-22.[郑玲,冯梦园,韩建明,2019. 玉米WOX家族基因的鉴定与分析[J]. 玉米科学,27(3):16-22.]
ZHU JH,SHI HZ,LEE BH,et al.,2004. An Arabidopsis homeodomain transcription factor gene,HOS9,mediates cold tolerance through a CBF-independent pathway[J]. Proc Natl Acad Sci USA,101(26):9873-9878.
(责任编辑 李 莉)
