融合肿瘤及腺体影像特征的乳腺癌分型预测
2021-09-29刘伟芬厉力华
刘伟芬,范 明,厉力华
(杭州电子科技大学自动化学院,浙江 杭州 310018)
0 引 言
世界卫生组织发表的2020年全球癌症报告显示,全球每年有六分之一的人死于癌症,其中乳腺癌是全球女性中最常被确诊的癌症类型,也是女性癌症死亡的主要原因[1]。乳腺癌作为一种高度异质性的疾病,不同患者个体间或者同一患者体内不同部位肿瘤细胞间从基因型到表型上都存在差异[2],这种复杂性与多样性给乳腺癌的诊疗带来巨大困难。针对乳腺癌异质性导致的治疗后复发、转移和耐药性,研究者运用基因表达分析方法对肿瘤进行分型。Perou等[3]首先依据基因谱表达方法对乳腺肿瘤进行不同亚型的分类,成为乳腺癌分子分型的基础。然而乳腺癌全基因组分析成本过高,随着分子生物医学研究的不断发展,临床上采取免疫组化方法来检测孕激素受体(Progesterone Receptor,PR)、雌激素受体(Estrogen Receptor,ER)、细胞增殖相关核抗原(Ki-67 抗原)、人类表皮生长因子受体2(Human Epidermal Growth factor 2,HER-2)、等生物分子标记信息,将乳腺癌分为4种分子分型,分别为管腔A型(Luminal A)、管腔B型(Luminal B)、HER-2过表达型(HER-2)、基底细胞样型(Basal-like)[4]。不同分子分型的乳腺癌在发病率、治疗反应、预后、复发转移风险等临床表现上存在很大差异,分子亚型是乳腺癌肿瘤异质性的体现[5-7]。
目前,临床检查主要通过活检穿刺获取患者的病理信息,但是活检对人体有侵入式伤害,易受其他因素干扰,无法对肿瘤内部的异质性进行描述,也无法反映肿瘤组织的整体情况[2]。随着医学影像学技术的不断发展,影像学检查作为非侵入式无创检测技术,能描述肿瘤组织的时间与空间性质,与病理学检查形成互补[8]。DCE-MRI是磁共振成像最主要的技术之一。DCE-MRI需要静脉注射造影剂钆喷酸葡胺(Gadolinium-diethylenetetramine Pentaacetic Acid,Gd-DTPA),对注射前中后不同时间点的同一位置进行多组扫描,全面描述造影剂流入和流出肿瘤的动力学过程,间接反映肿瘤内不同部位的血流特性[8-9]。研究表明影像学分析可用于乳腺癌分子亚型预测。Grimm等[10]用计算机视觉法从肿瘤DCE-MRI影像中提取了56个图像特征,揭示了特征与Luminal A型、Luminal B型之间的关联性。王世健等[11]使用计算机半自动化方法从肿瘤影像中提取了65维特征,表明DCE-MRI特征可作为潜在乳腺癌分子分型的影像学标记。Fan等[12]使用进化算法从肿瘤DCE-MRI中识别了24个重要影像特征,并结合2个临床病理学特征,对乳腺癌分子亚型进行预测,揭示了从肿瘤DCE-MRI获得的特征是预测乳腺癌分子亚型的潜在生物标志物。Mazurowski等[13]研究了肿瘤DCE-MRI影像及背景实质之间的增强率关系,指出Luminal B型与肿瘤强化动态及背景组织相关。乳腺影像特征与分子分型的关联性研究虽已取得一定进展,但相关研究多数只针对肿瘤影像进行分析,忽略了肿瘤周围腺体组织对肿瘤的新陈代谢及浸润生长的动态调控作用。例如,肿瘤间质干细胞能够分泌驱化因子用于应答癌细胞发出的信号,并反作用于癌细胞,激发侵袭行为[14];位于肿瘤周边的巨噬细胞能够通过间质降解酶促进癌细胞局部侵袭与生长[15]等。为充分利用肿瘤与腺体之间的相互关系,全面探索乳腺癌肿瘤及腺体影像特征对分子分型预测的重要作用,本文研究联合肿瘤及腺体影像特征,建立基于肿瘤和腺体融合的影像组学分子分型预测模型。
1 数据与方法
1.1 患者信息
本文实验所用的数据采自浙江省中医院,所有病例在DCE-MRI检查前均未进行任何乳腺手术或者化疗。对205例病例进行病理信息统计,患者均为女性,年龄分布在29~84岁,平均年龄52岁;绝经94例,未绝经111例;分子分型Luminal A型50例,Luminal B型75例,Basal-like型32例,HER-2型33例。以不同分子分型对经期状态进行卡方检验,对年龄进行方差分析,P值分别为0.096 7和0.154 5,表明经期状态及年龄在4种分子分型中没有统计差异。
1.2 影像采集及参数设置
DCE-MRI影像数据由德国西门子8通道双乳房线圈MAGNETOM Verio 3.0T超导型磁共振成像系统采集。患者俯卧于双侧乳腺线圈上,双乳对称自然垂悬于乳腺线圈中央。动态增强扫描参数设置如下:重复时间(TR)4.51 ms,回声时间(TE)1.61 ms,层厚(ST)1 mm,翻转角度(FA)10°,视野(FOV)340 mm×340 mm,采集矩阵(AM)448×448,层间距1 mm,像素分辨率0.759 mm×0.759 mm。使用0.2 mmol/kg的造影剂Gd-DTPA以4 ml/s的速度静脉注射,注射前首先扫描获取蒙片序列记作S0序列,注射后每隔60 s采集一个增强序列,记作S1,S2,S3,S4,S5序列。
1.3 影像感兴趣区域分割
由于造影剂的增强作用,在增强序列中,肿瘤区域信号明显高于背景组织,采用自适应的空间模糊C-均值聚类(Fuzzy C-Means,FCM)算法与马尔科夫随机场(Markov Random Field,MRF)相互结合的三维分割方法对乳腺肿瘤DCE-MRI影像进行分割[16]。首先,人工标注肿瘤区域起止切片、肿瘤最大截面切片的粗略轮廓及中心点;然后,根据DCE-MRI上下切片之间的强空间相似性,以肿瘤最大径所在图像切片两端为起始位置,以上一张切片的肿瘤分割结果作为下一张切片的分割起始模板,采用FCM算法对肿瘤进行级联分割粗分割;最后,运用MRF调整粗边缘实现精细分割。
乳腺腺体是指乳房组织内包括乳腺血管、输乳管、纤维结缔组织等的乳腺实质性部分。造影剂对乳腺血管有增强作用,但是对输乳管、纤维结缔组织等无增强作用,在增强序列中进行自适应聚类分割的方法不再适用;而蒙片序列中腺体组织信号平稳均匀,且高于脂肪信号,故在蒙片序列中进行腺体的分割。首先,综合灰度变换、均值滤波、边缘检测、连通区域分析、多角度差分、图像相似度比较等图像处理基本方法,确定乳房区域边缘;然后,采用FCM算法分离腺体与背景区域;最后,选取肿瘤外20 mm内的腺体区域作为感兴趣区域[17]。
1.4 影像组学特征提取
本文的研究针对DCE-MRI病灶影像选择1个蒙片序列(S0),2个减影序列,分别为第3增强序列减去蒙片序列(S3-S0)、第5增强序列减去蒙片序列(S5-S0),以及1个增强率序列,即第5增强序列减去第3增强序列,再除以第3增强序列减去蒙片序列(S5-S3)/(S3-S0),共4个序列进行分析,分别提取111维特征。具体包括:中位数、极差、标准差、信息熵等19维统计特征[18];体积、中心距、矩形度、粗糙度等17维形态特征[19];基于灰度共生矩阵(Gray-level Co-occurrence Matrix,GLCM)[20]的对比度、同质性、集群突出物、能量等24维纹理特征,基于灰度区域大小矩阵(Gray-Level Size Zone Matrix,GLSZM)[21]的小区域优势、大区域优势、区域大小非一致性、区域比等16维纹理特征,基于灰度游程矩阵(Gray-level Run-Length Matrix,GLRLM)[22]的短游程优势、长游程优势、游程分布、游程比等16维纹理特征,基于邻域灰度差矩阵(Neighboring Gray Tone Difference Matrix,NGTDM)[23]的粗糙度、复杂度等5维纹理特征,基于灰度相关矩阵(Gray Level Dependence Matrix,GLDM)[24]的小依赖因子、大依赖因子、依赖方差等14维纹理特征。
1.5 机器学习模型分析
为了探究DCE-MRI影像肿瘤和腺体的联合对乳腺癌分子分型预测性能的影响,将205例病例数据集按照2∶1的比例随机划分为训练集与测试集,即训练集137例,测试集68例,对肿瘤和腺体提取的特征分别建立单特征及多特征机器学习模型,最后使用不同的融合方法对肿瘤/腺体多特征模型进行融合,以预测乳腺癌分子分型。通过对已知数据的学习,实现未知数据的预测,预测未知数据时出现的误差被称为泛化误差。因此,需要制定合理的策略,本文选取十折交叉验证(10-fold cross-validation)进行模型性能验证,十折交叉验证结合网格搜索法(Grid Search Method,GSM)进行影像特征选择和模型参数选择。最后根据测试结果绘制接受者工作特征(Receiver Operating Characteristic,ROC)曲线评价模型的性能。为了对模型进行量化分析,计算ROC曲线下面积(Area Under the ROC,AUC),AUC的取值在0~1之间,AUC越大,说明模型预测性能越好。
1.5.1 基于肿瘤/腺体影像的单特征模型
对DCE-MRI影像肿瘤和腺体提取的特征进行单特征分析。分别对4种分子分型进行预测,如预测Luminal A型时,令Luminal A型为1,非Luminal A型为0。以训练集构建支持向量机(Support Vector Machine,SVM)模型进行训练和预测,计算AUC评价模型的性能,采用t检验比较单个特征在不同分子分型标签下的统计学差异,最后绘制4种分子分型的最优单特征盒形图。
1.5.2 基于肿瘤/腺体的多特征预测模型
由于肿瘤存在异质性,单个特征可能无法对其进行客观有效的描述,为分析多特征共同作用的效果,建立基于肿瘤/腺体的多特征预测模型。DCE-MRI影像经特征提取后,获得了相当数量的影像特征,但不是每个特征都对乳腺癌分子分型预测起重要作用,因此,使用无监督学习的特性选择法,即皮尔逊相关性分析对特征进行降维处理。通过计算特征之间的两两相关性,筛选出相关性大于0.9的特征对,比较该特征对中2个特征与其他特征相关性大于0.9的数量总和,剔除数量总和较大特征,完成去冗余过程。初步筛选后,在训练集中,采用有监督学习的SVM-RFE算法获得特征重要性排序,依据重要性排序,在训练集中逐个添加特征,依次建立SVM预测模型,运用留一法交叉验证计算AUC,最大AUC对应的特征子集即为最优特征子集。由于采用RBF核函数涉及到惩罚因子c和径向作用范围g参数的选取设定,故采用固定的参数组,针对每个特征子集,结合十折交叉验证法与网格搜索法,查找对应SVM模型最优参数。在训练集中,以最优特征子集及其对应的最优模型参数建立SVM多特征预测模型,使用测试集来评价模型的性能。
1.5.3 基于肿瘤和腺体的预测模型融合
基于肿瘤/腺体的多特征预测分析中,对训练集进行特征筛选,获得最优特征子集,建立SVM预测模型,用测试集AUC评价模型性能。由于基于肿瘤/腺体的多特征预测模型相互独立,可采用模型融合的方法结合2个预测模型以提升学习性能。本文研究采用概率平均法、概率加权平均法和Stacking学习法进行模型的融合。概率平均法将基于肿瘤/腺体影像特征的预测模型赋予相同权值,对肿瘤/腺体模型预测分析得到的概率预测值求平均值,作为融合模型的预测结果。概率加权平均法将肿瘤/腺体预测模型进行融合,对预测性能较好的模型设置更高的权值,相对表现较差的模型设置较低的权值,具体权值占比为(AUC肿瘤/腺体-0.5)/(AUC肿瘤-0.5+AUC腺体-0.5)。Stacking学习法通过留一法交叉验证来选择初始数据集,训练初级学习器,以初级学习器的预测结果作为新特征输入次级学习器,得到最终决策结果,学习器采用SVM模型。
2 实验结果与分析
2.1 肿瘤/腺体影像单特征模型预测结果
DCE-MRI单个影像特征对分子分型的预测结果如表1所示。从表1可以看出,对于肿瘤预测Luminal A型、Luminal B型、Basal-like型、HER-2型性能最优的单特征对应AUC分别为0.840 9,0.681 9,0.729 3,0.799 4。对于腺体预测Luminal A型、Luminal B型、Basal-like型、HER-2型性能最优的单特征对应AUC分别为0.704 8,0.653 2,0.743 6,0.737 4。Luminal A型、Luminal B型、HER-2型单序列最优单特征P值均小于0.05,说明分布差异具有统计学意义,Basal-like型单序列最优单特征P值大于0.05,分布差异没有统计学意义。比较肿瘤与腺体,肿瘤单特征模型总体效果更好。
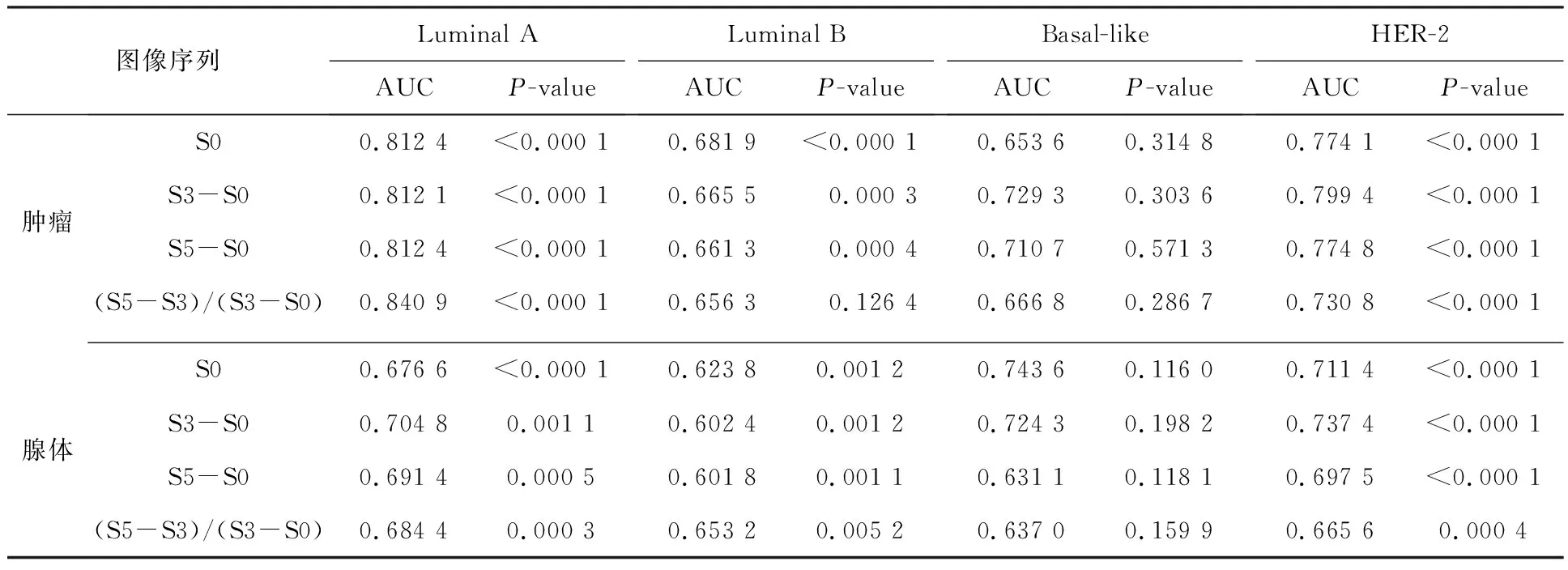
表1 最优单特征模型预测结果
乳腺癌分子分型预测任务的最优单特征盒形图如图1所示。图1(a)中,Luminal A型的肿瘤最优单特征为与区域具有相同标准二阶中心距的椭圆的短轴长度,腺体最优单特征为基于灰度区域大小矩阵的区域大小非一致性,图1(a)特征值大小表明,相对于其他分型,Luminal A型肿瘤体积小,腺体内部灰度分布均匀。图1(b)中,Luminal B型的肿瘤最优单特征为基于灰度共生矩阵的信息相关性度量,腺体最优单特征为基于灰度区域大小矩阵的区域比,图1(b)特征值大小表明,相对于其他分型,Luminal B型肿瘤内部相关性低,腺体内部差异性高。图1(c)中,Basal-like型的肿瘤最优单特征为基于灰度区域大小矩阵的灰度方差,腺体最优单特征为四分位距,图1(c)特征值大小表明,相对于其他分型,Basal-like型肿瘤灰度强度的变化小,腺体灰度级分布范围大。图1(d)中,HER-2型的肿瘤最优单特征为最大直径,腺体最优单特征为基于灰度游程矩阵的游程分布,图1(d)特征值大小表明,相对于其他分型,HER-2型肿瘤体积大,腺体灰度分布均匀。

注:“*”表示P<0.05,“**”表示P<0.01,“***”表示P<0.000 1。图1 乳腺癌分子分型预测任务的最优单特征盒形图
2.2 肿瘤/腺体多特征模型预测结果
对DEC-MRI各序列影像进行多特征预测分析,结果如表2所示。
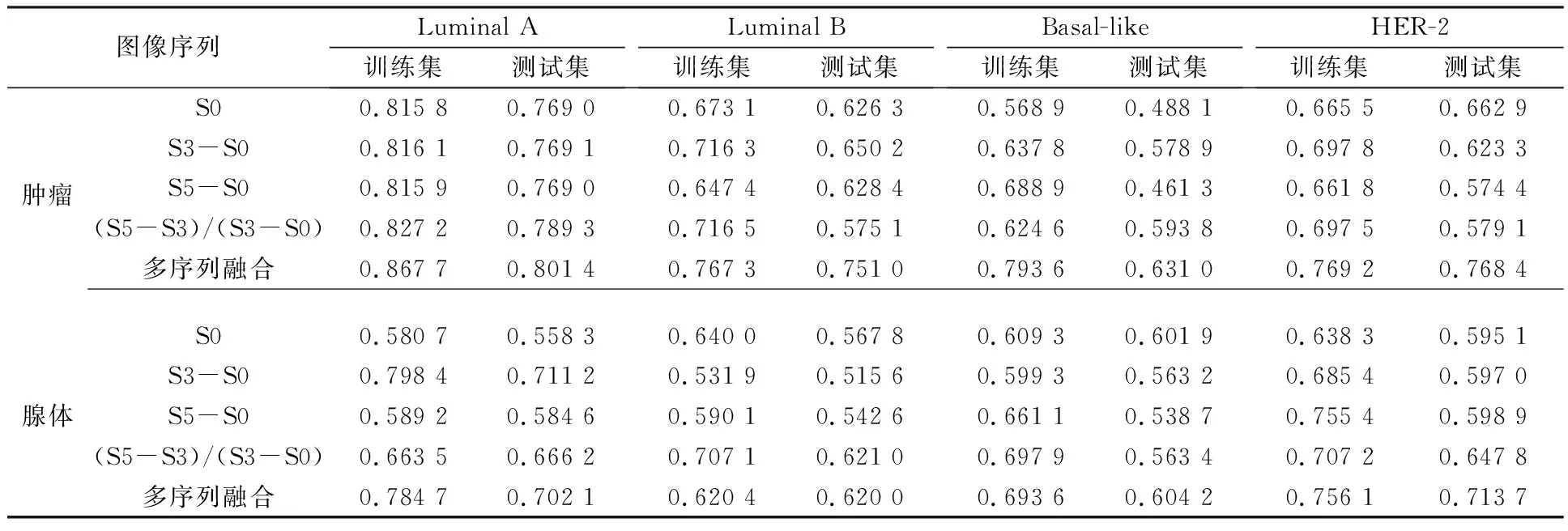
表2 肿瘤/腺体多特征模型AUC
从表2可以看出,(S5-S3)/(S3-S0)序列预测Luminal A型效果最佳,测试集AUC为0.801 4;S3-S0序列预测Luminal B型效果最佳,测试集AUC为0.650 2;(S5-S3)/(S3-S0)序列预测Basal-like型效果最佳,测试集AUC为0.593 8;S0序列预测HER-2型效果最佳,测试集AUC为0.662 9。对于腺体模型,S3-S0序列预测Luminal A型取得了最佳效果,测试集AUC为0.711 2;(S5-S3)/(S3-S0)序列预测Luminal B型取得了最佳效果,测试集AUC为0.621 0;S0序列预测Basal-like型取得了最佳效果,测试集AUC为0.601 9;(S5-S3)/(S3-S0)序列预测HER-2型取得了最佳效果,测试集AUC为0.647 8。
在肿瘤/腺体上分别将4个序列特征串联融合,模型预测效果得到明显提升。对于肿瘤模型,Luminal A型测试集AUC为0.801 4,优于所有单序列;Luminal B型测试集AUC为0.751 0,优于所有单序列;Basal-like型测试集AUC为0.631 0,优于所有单序列;HER-2型测试集AUC为0.768 4,优于所有单序列;对于腺体模型,Luminal A型测试集AUC为0.702 1,仅次于S3-S0序列;Luminal B型测试集AUC为0.620 0,仅次于(S5-S3)/(S3-S0)序列;Basal-like型测试集AUC为0.604 2,优于所有单序列;HER-2型测试集AUC为0.713 7,优于所有单序列。结果表明,相比单特征预测模型,多特征预测模型可以更大限度地利用从影像数据中提取的信息,提升了模型的泛化能力,提高了模型的预测性能。
2.3 基于肿瘤和腺体的预测模型融合结果
分别采用概率平均法、概率加权平均法和Stacking学习法这3种不同的影像组学特征联合方法,将基于肿瘤和腺体分别构建的预测模型进行融合,得到预测结果如表3所示。
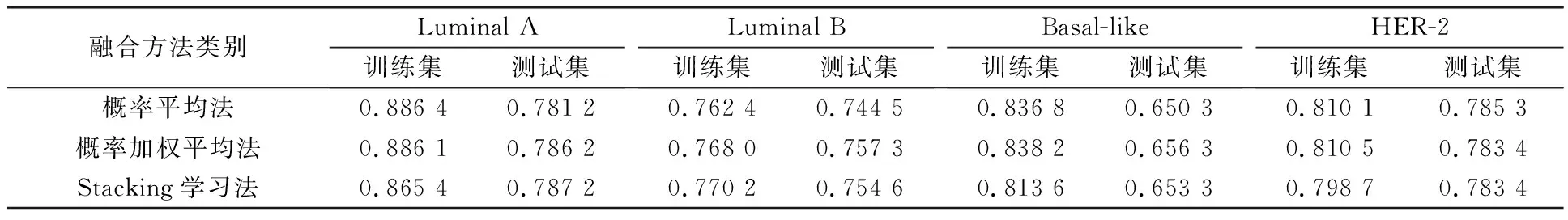
表3 肿瘤和腺体预测模型融合AUC
从表3可以看出,相对于肿瘤/腺体模型的多序列融合模型,肿瘤和腺体融合模型在Luminal B型、Basal-like型和HER-2型上的预测效果均得到提升。相比于肿瘤模型序列融合的Luminal B型最优AUC,概率加权平均法和Stacking学习法融合模型预测性能均有所提高,其中概率加权平均法预测Luminal B型的达到最优AUC,为0.757 3;相比于肿瘤模型序列融合的Basal-like型最优AUC,概率平均法、概率加权平均法和Stacking学习法融合模型预测性能均有所提高,其中概率加权平均法预测Basal-like型达到最优AUC,为0.656 3;相比于肿瘤模型序列融合的HER-2型最优AUC,概率平均法、概率加权平均法和Stacking学习法预融合模型预测性能也均有所提高,其中概率平均法预测HER-2型的达到最优AUC,为0.785 3。Luminal A型的预测在肿瘤多序列融合模型中达到最优AUC,为0.801 4。
综上可知,肿瘤和腺体融合模型对于Luminal A型、Luminal B型、和HER-2型均取得了良好的预测效果,Basal-like型稍差。Basal-like型是一种特殊的亚型,其肿瘤异质性程度高,肿瘤细胞转移存在异常机制,易于复发[25],预后差且无针对性的标准治疗方案[26],因此预测精度低于其他亚型[27]。
实验及分析表明,本文联合肿瘤和腺体影像进行乳腺癌分子分型预测基本达到预期效果。但是,也存在局限性,一是样本数据集规模较小,且分子分型样本分布不均衡,后续研究可以通过增加数据集来提高模型的鲁棒性;二是乳房腺体区域个体化差异大,分割精度相对粗糙,如何进行有效、精准、快速的腺体分割有待进一步研究。
3 结束语
针对现阶段肿瘤空间异质性的问题,本文提出一种融合肿瘤和周围腺体影像特征的乳腺癌分子分型预测研究方法。相对仅分析肿瘤影像特征的方法而言,有效提高了乳腺癌分子分型预测模型的预测性能,为乳腺癌的精准诊疗提供参考。后续将通过增加不同机型不同分辨率的数据进行验证,进一步提升模型的临床应用性。
