乳腺癌相关非编码RNA 的生物信息学分析
2021-09-02赵林林蒲婷钱婧雯沈红兰周学
赵林林,蒲婷,钱婧雯,沈红兰,周学
(南京师范大学泰州学院 化学与生物工程学院,江苏泰州 225300)
非编码RNA(ncRNA)是一类不编码蛋白质的RNA 统称,包括微小RNA(miRNA)和长链非编码RNA(LncRNA)。研究乳腺癌相关非编码RNA 的特征并预测其功能,对揭示ncRNA 在乳腺癌中的机制非常重要,也为临床诊疗提供新依据[1-5]。
miRNA 是一类长度约为22 nt 内源性非编码小分子RNA,miRNA 调控人类30%左右的基因。近年来,越来越多的研究显示miRNA 在包括乳腺癌在内的多种恶性肿瘤组织中均存在异常表达[2]。在某miRNA 发生突变后,可能会导致相应癌基因的表达,也可能会出现抑癌基因的缺失,从而使正常机体发生病变。已有研究结果表明,miRNA 可能不仅有助于乳腺癌的诊断、预后以及治疗效果的预测,也有望成为新的治疗靶标[6-10]。
LncRNA 是非编码RNA 中另外一个重要的成员,长度大于200 nt,缺乏开放的阅读框,没有编码蛋白质的功能,其分子内部具有特定而复杂的二级空间结构,它可以提供多个蛋白质结合位点,或通过碱基互补配对原理与DNA 和RNA 特异、动态地相互作用,形成由LncRNA 参与的复杂、精确、精细的基因表达调控网络[2]。LncRNA 具有5'端帽子结构和3'端聚腺苷酸尾巴,基因结构类似于mRNA,LncRNA 和miRNA 可相互作用,又可竞争结合mRNAs。在乳腺癌的发生发展过程中,LncRNA 起到了调节增殖、细胞表型等作用,一些特异性LncRNA 可作为乳腺癌发生发展的标志物[11-13]。
本文通过生物信息学方法研究乳腺癌相关ncRNA的特征并预测其功能,对揭示ncRNA 在乳腺癌中的机制非常重要,也为临床诊疗提供新依据[14-15]。
1 材料与方法
1.1 数据收集
通过文献调研以及数据库搜索,收集乳腺癌相关非编码RNA 的数据,并利用Perl 程序对下载的数据进行分析。miRNA 的数据从miRCancer(http://mircancer.ecu.edu/download.jsp)、HMDD(http://www.cuilab.cn/hmdd) 及miR2Disease(http://www.mir2disease.org/)获得;LncRNA 数据从Lnc2Cancer(http://www.bio-bigdata.net/lnc2cancer/) 和LncRNADisease(http://www.cuilab.cn/ lncrnadisease)数据库下载。
1.2 乳腺癌相关非编码RNA 保守性及SNP 位点分布分析
以miRNA 和LncRNA 各自基因组位置作为输入数据,在UCSC 数据库批量下载基于100 个脊椎动物比对的人类基因组中每个位点的保守值,并利用Perl 程序计算平均值。利用miRNA SNP 和LncRNA SNP2 对乳腺癌相关非编码RNA 上的SNP 位点进行分析。
1.3 乳腺癌相关非编码RNA 重复元件及转录调控分析
利用RepeatMasker 程序对乳腺癌相关非编码RNA 上分布的重复元件进行预测。从TransmiR(http://www.cuilab.cn/transmir)、miReg(http://www.diana.pcbi.upenn.edu/miRGen.html) 和AnnoLnc(http://annolnc.cbi.pku.edu.cn)数据库获取人miRNA和LncRNA 对应TF-miRNA、TF-LncRNA 转录调控数据。
1.4 乳腺癌相关非编码RNA 间相互作用及作用靶基因功能分析
利 用NPInter(http://bigdata.ibp.ac.cn/npinter4/)数据库对LncRNA 与miRNA、LncRNA 与蛋白质之间的相互作用进行分析。从miRTarBase 数据库批量下载乳腺癌相关miRNA 作用的靶基因,利用DAVID数据库分析miRNA 靶基因和LncRNA 作用蛋白质的功能。
2 结果与分析
2.1 乳腺癌相关非编码RNA 的鉴定
如图1 所示,通过搜索miRCancer 和HMDD 数据库,最终获得252 个乳腺癌相关的miRNA,包括hsalet-7a-1、hsa-mir-103a、hsa-mir-146a、hsa-mir-31、hsa-mir-99a 以 及hsa-mir-455 等。 从Lnc2Cancer 和LncRNADisease 数据库总计获得245 个乳腺癌相关的LncRNA,包括53BP1、7SL、ADARB2-AS1、BANCR、CASC2、DIRC3 以及XIST 等。
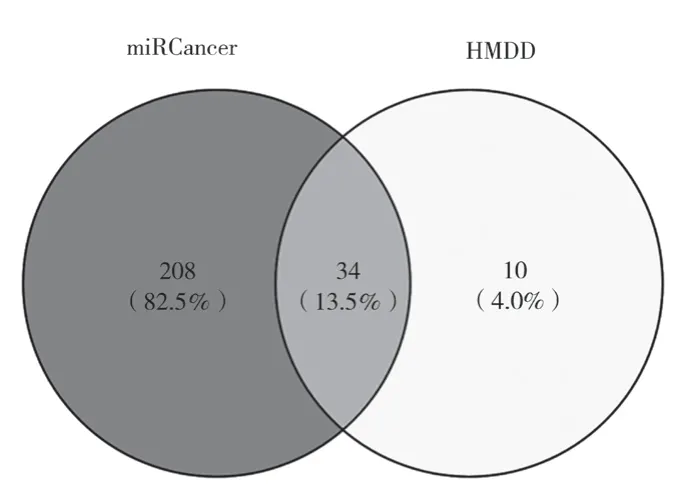
图1 miRCancer、HMDD 数据库中乳腺癌相关miRNA 分布图
2.2 乳腺癌相关非编码RNA 保守性及SNP 位点分布分析
利用UCSC 数据库提供的保守性数据,通过统计,最终获得215 个miRNA 的平均保守值为2.97,187 个LncRNA 平均保守值为0.24,发现乳腺癌相关miRNA 的保守性要显著高于LncRNA。
SNP 位点分布分析发现,在253 个miRNA 中,有115 个miRNA 分布了SNP 位点,总计分布的SNP 位点数目为215 个。每个miRNA 上分布的SNP 位点平均为1.87 个,数目为1 ~5 个,如hsa-mir-1228、hsa-mir-520g 和hsa-mir-630 这3 个miRNA 各 分布5 个SNP 位 点。245 个LncRNA 中,有115 个LncRNA分布SNP位点,总计分布21 359个SNP位点,平均每个LncRNA 分布的SNP 位点数目为185.73个,SNP 位点数目的分布范围在1 ~2 399,其中4个LncRNA(3.48%)上分布23 个SNP 位点,3 个LncRNA(2.60%)上分布66 个SNP 位点。
如图2 所示,对非编码RNA 分布的SNP 位点与保守值进行相关性分析,发现SNP 分布密度与miRNA 保守性成负相关,而SNP 分布密度与LncRNA保守性成正相关。

图2 miRNA 保守值与SNP 密度的相关性
2.3 乳腺癌相关非编码RNA 重复元件及转录调控分析
重复元件分析结果显示,在人15 个miRNA 前体中总计发现了1 个DNA/TcMar-Mariner 元件、2 个DNA/hAT-Charlie 元件、2 个简单重复元件和9 个LINE/L2 元件等。进一步分析发现,这些起源于重复元件的miRNA 在进化上不保守,具有物种特异性。在人117 个LncRNA 序列中总计发现了622 个重复元件,包括130个LINE/L1元件,59个LTR/ERVL元件,43 个LTR/ERVL-MaLR 元件,100 个简单重复元件,88 个SINE/MIR 元 件,72 个SINE/Alu 元 件 及36 个DNA/hAT-Charlie 元件等。每个LncRNA 涉及的重复元件数目为1 ~97 个,例如在KCNQ1OT1 上预测存在97 个重复序列,每个重复元件涉及LncRNA 数目为1 ~130 个,在130 个LncRNA 中均发现LINE/L1重复序列。
转录调控分析发现,总计334 个转录因子参与177 个miRNA 的表达,每个转录因子调控的miRNA的 数 目 为1 ~53,如MYC 参 与49 个miRNA 的 转录调控,包括hsa-let-7a-1、hsa-mir-100、hsa-mir-106a、hsa-mir-146a 以 及hsa-mir-148a 等;TP53参与53 个miRNA 的转录调控,它调控29.94%的miRNA。同时每个miRNA 涉及的转录因子数目为1 ~45 个,34 个miRNA(19.21%)只有1 个转录因子参与调控,剩余的80.79%miRNA 涉及2 种及2种以上转录因子的调控,例如hsa-mir-200c 有45 个转录因子参与调控,包括KLF5、LIN28A、MUC1、MYB 及NCOR1 等(见图3)。
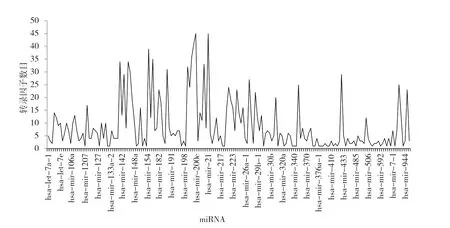
图3 每个miRNA 涉及调控的转录因子数目
对LncRNA 的转录因子进行分析,发现150 个LncRNA 涉及7 252 个转录因子的调控,每个LncRNA涉及的转录因子数目范围在1 ~139,MALAT1 在35 个细胞类型中受到139 个转录因子的调控,转录因 子 包 括AP-2alpha、AP-2gamma、ATF1、ATF2、ATF3、SP1、SP2 以 及p300 等,CCDC26 非 编 码RNA受113个转录因子参与调控,包括CEBPB、c-Myc、COREST、CTCF 和FOXA1 等;同时发现每个转录因子参与调控的LncRNA 的数目为1 ~119 个,Rad21参与119 个LncRNA 的调控,包括NNT-AS1、SRA1、DANCR、RP11-445H22.4、linc-TRIP11、lncFOXO1及CCAT1 等,Pol3 只 调 控1 个 非 编 码RNA——GHET1。
通过比较发现,有44 个转录因子为miRNA 和LncRNA 共 有 的,包 括 转 录 因 子ATF3、BRCA1、E2F6、FOS 和IRF3 等,例如BRCA1 参与调控hsamir-146a 及NKILA 等。
2.4 乳腺癌相关非编码RNA 间相互作用及作用靶基因功能分析
对LncRNA-miRNA 作用关系分析发现,151 个LncRNA 作用的miRNA 的数目范围在1 ~71,其中LncRNA XIST 与71 个miRNA 发生相互作用,作用方式包括调节作用、结合作用等;hsa-mir-214 与66个LncRNA 发生相互作用。进一步分析发现:83 个LncRNA 作用423 个蛋白质,每个LncRNA 作用的蛋白质数目为1 ~222 个,如MALAT1 与194 个蛋白质发生相互作用,包括AKAP8L、AQR、BCCIP、CASP3、DGCR8、EIF2C1 以及EIF3D 等蛋白。
靶基因分析结果显示:380 个miRNA 作用了13 983 个靶基因,miRNA 调控的靶基因数目为2 ~2 627 个,每个靶基因上作用的miRNA 数目为1 ~88 个,如 靶 基 因NUFIP2 上 作 用 了88 个miRNA,包 括hsa-miR-101-3p、hsa-miR-103a-3p、hsa-miR-106a-5p 及hsa-miR-106b-5p 等。靶 基 因功能分析结果显示,涉及的分子功能包括转录因子结合活性、钙通道活性等,参与的生物进程中包括细胞通讯调节、调节细胞凋亡等。
3 结论
本文利用文献调研及生物信息学的方法对乳腺癌相关非编码RNA 进行数据挖掘,并从保守性、SNP 位点分布、转录调控和作用靶基因等方面系统分析乳腺癌相关非编码RNA 的特征。
结果共获得乳腺癌相关的253 个miRNA 和245 个LncRNA。保守性分析结果显示乳腺癌相关miRNA 的保守性要显著高于LncRNA。转录调控分析共计发现334 个转录因子参与了177 个miRNA的表达,共计7 252 个转录因子涉及调控150 个LncRNA。靶基因预测结果显示,LncRNA 作用蛋白质同时为miRNA 作用的靶基因,靶基因的功能涉及金属离子结合活性等。乳腺癌相关的非编码RNA 的研究将为今后乳腺癌的治疗提供有效的治疗靶点。
