水稻苗期根部性状的遗传分析和最长根长QTL qLRL4的精细定位
2021-08-05丁仕林刘朝雷阮班普姜洪真董国军胡光莲郭龙彪高振宇
田 彪 丁仕林 刘朝雷 阮班普 姜洪真 郭 锐 董国军 胡光莲 郭龙彪 钱 前 高振宇
水稻苗期根部性状的遗传分析和最长根长QTL的精细定位
田 彪 丁仕林 刘朝雷 阮班普 姜洪真 郭 锐 董国军 胡光莲 郭龙彪 钱 前 高振宇*
中国水稻研究所, 浙江杭州 310006
为了解析水培苗期根系相关性状的遗传调控, 以籼稻9311和粳稻日本晴(Nipponbare, NPB)为亲本的148个株系构成的重组自交系群体为材料, 对水稻幼苗根系相关性状开展QTL分析。在2次重复中共检测到26个控制最长根长、总根系长、根表面积、根体积和根直径的QTL, 分布在水稻第1、2、4、7、9、10、11号共7条染色体上, 发现了水稻第2、4、7和10号染色体上的4个QTL簇, 包括第4号染色体上控制最长根长的QTL。为了精细定位该QTL, 我们构建了以9311为背景、插入缺失标记IND4-1和IND4-4间来自NPB的近等系NIL-。利用NIL-和9311构建的F2群体, 最终将精细定位在标记IND4-1和IND4-3之间约68.23 kb的区间内并预测了候选基因。此根长QTL的精细定位将有助于水稻根长遗传机理的研究, 为探究水稻根系形态建成的分子机制奠定了基础。
水稻; 根部性状; 最长根长;; 精细定位
水稻是人类最主要的粮食作物之一[1], 而根系作为水稻主要的器官, 具有固定水分、吸收养分、合成与分泌激素、酶和有机酸等功能[2]。虽然根系研究与其他性状相比相对薄弱[3], 但是植物学家对根系相关性状已开展了大量遗传研究。徐吉臣等[4]以窄叶青8号和京系17的F1代花培加倍的127个双单倍体(double haploid, DH)群体为材料, 测定了营养液培养条件下亲本及DH株系的最大根长、根干重和根茎干重比, 检测到4个最大根长QTL, 分别位于第2、4、9、10号染色体; 检测到水稻第2号染色体的1个根干重QTL; 根茎干重比共检测到3个QTL, 分别位于第3、5、6号染色体上。滕胜等[5]和胡兴明等[6]也用相同群体进行了根部性状的QTL分析。前者在抽穗期对根系活力进行QTL分析, 在第4号染色体的分子标记RG449和RG809间检测到1个QTL ()。后者对水稻苗期发根力进行了QTL和上位性分析, 在水稻第3号染色体的C63~CT125之间检测到1个发根力主效QTL; 同时检测到影响水稻发根力的5对上位性效应基因座, 分别位于第2、3、5、6、7、12号染色体上, 其中影响根长和根数的上位性效应各有2对QTL, 有一对QTL同时影响根长和根数。迄今, 国内外科学家已定位了多个水稻根长相关QTL。Mitsuhiro等[7]用粳稻品种Koshihikari和籼稻品种Kasalath杂交和回交得到的38个染色体片段替代系(CSSL)检测生长在5mmmol L–1或500mmmol L–1NH4+中的水稻幼苗根长QTL, 共检测到8个QTL, 分别位于第1、2、4、6、8、11和12号染色体, 并将6号染色体的根长主效QTL精细定位在长臂337 kb区间内。王汝慈等[8]利用协青早B与中恢9308杂交构建的RIL群体在水稻第4号染色体长臂的分子标记RM307与RM1205之间定位到一QTL簇, 控制不同时期的相对根长、相对根冠比、相对根干质量等性状, 其中包括控制水稻根长的主效QTL——。Obara等[9]以水稻WAB56-104和NERICA7品系的杂交F2群体为材料, 在高NH4+环境下于水稻第1号染色体检测到2个水稻根长相关QTL, 分别命名为和, 其中的定位区间进一步缩小至0.7 Mb。Kitomi等[10]为了鉴定与水稻最大根长相关的基因组区间, 用来自IR64和KinandangPatong间杂交回交得到的26个CSSL进行QTL分析, 得到2个最大根长QTL: 2号染色体的和6号染色体的。将精细定位在SSR标记RM5651和RM6107之间的1.7 Mb区间, 将定位在SSR标记RM20495和RM3430-1之间884 kb的区间。到目前为止, 科学家已成功克隆了一些控制水稻根系性状的基因[11-12], 如[13]和[14]基因调控水稻根长,[15]基因影响冠状根的发生和不定根的形成,[16]基因影响不定根的形成,[17]基因促进冠根的起始和伸长,[18]和[19]基因影响不定根的的数目和长度,[20]基因抑制初生根的发育,[21]基因控制侧根发生。为了深入了解水稻根系性状的遗传基础, 我们以9311和日本晴(Nipponbare, NPB)构建的重组自交系(recombinant inbred lines, RIL)群体为材料, 利用水培技术和数量遗传学手段对水稻根系性状相关QTL进行了分析和鉴定。
1 材料与方法
1.1 试验材料
研究采用籼稻品种9311和粳稻品种NPB杂交获得的148个株系构成的RIL群体。我们选择了区域具有NPB基因型的RIL与亲本9311回交, 并在插入缺失(InDel)标记IND4-1和IND4-4之间选择目标QTL区域携带NPB纯合等位基因的近等基因系(near isogenic lines, NIL) NIL-。
1.2 试验方法
在黑暗条件下, 水稻种子于37°C去离子水中浸泡2 d, 然后转移至漂浮在去离子水上的网格, 培养3 d。将幼苗培养在正常浓度的Kimura B营养液中(pH 5.4), 每3 d更换一次营养液。然后, 将幼苗转移至30°C温室中培养21 d, 用尺子测量5株幼苗的主根长, 然后用i800扫描仪扫描总根长, 用LA-S根分析系统(万深, 中国杭州)进行分析。用亚甲基蓝比色法(Leagene, 中国北京)测定根系活性(总吸收面积和比表面积), 每3株幼苗为1次重复, 3个重复, 总体进行2次重复实验, 记为重复I (Repeat I)和重复II (Repeat II)。
用均匀分布于水稻12条染色体的166个在9311和NPB之间存在多态的SSR和STS标记构建了RIL群体的遗传连锁图谱。使用MultiQTL软件(www. multiqtl.com)对RIL群体采用最大似然区间映射方法进行QTL分析, 根据每个数据集的排列测试(1000个排列,=0.05)获得LOD阈值。根据McCouch等[22]的方法命名QTL。利用Microsoft Excel软件分析根部形状的表型值与频数分布, 并绘制相关图表; 利用IBM SPSS Statistics 24软件进行显著性差异检验和相关性分析。
通过近等系NIL-与9311的杂交和自交, 构建了用于精细定位的830个植株构成的F2群体, 测量了单株根长表型, 在已定位的区间内开发了4个InDel标记(表1)检测单株基因型, 用于的精细定位。
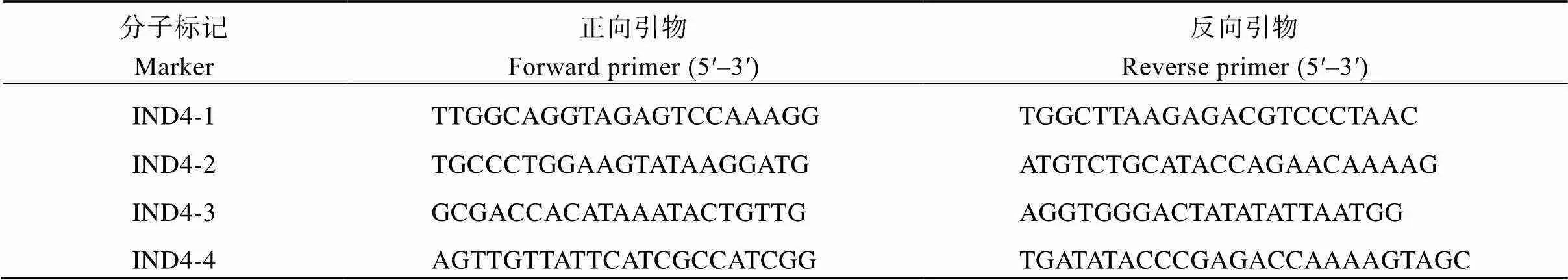
表1 qLRL4精细定位的InDel标记引物
采用Axygen公司的AxyPrep总RNA小量制备试剂盒(AP-MD-MS-RNA-50)提取9311、NPB和NIL-的根部RNA并使用TOYOBO公司的cDNA逆转录试剂盒(fsq-101)反转录得到cDNA后, 用候选基因引物和内参引物(表2)在ABI7900HT Real-time PCR仪上对样品的候选基因表达量进行相对定量检测。

表2 候选基因实时定量PCR引物
2 结果与分析
2.1 9311、NPB及其RIL群体的根系性状
亲本9311和NPB之间的根系形态存在显著差异(表3和图1)。9311的最长根长(the longest root length, LRL)、总根系长(total root length, TRL)、根表面积(root surface area, RSA)、根体积(root volume, RV)和根直径(root diameter, RD)均显著大于NPB。考察9311和NPB构建的RIL群体的最长根长、总根系长、根表面积、根体积和根直径这5个性状, 统计2次重复的平均值, 最长根长为14.8 cm和14.3 cm, 总根系长为296.35 cm和331.61 cm, 根表面积为25.26 cm2和25.62 cm2, 根体积为0.22 cm3和0.26 cm3, 根直径为0.25 mm和0.23 mm。RIL的5个性状与两亲本相比, 最长根长和平均直径均小于两亲本。RIL的平均总根系长、根表面积和根体积介于NPB和9311之间(表4)。在2次重复中, 5个根系性状在RIL群体中均呈连续正态分布(图2), 且大部分呈现出明显的超亲分离, 表明这些性状由多基因控制, 适合进行QTL分析。2次重复之间比较后发现, 重复I和重复II的最长根长、根表面积和根体积的分布范围基本一致。然而, 重复I的总根长和平均直径的变化范围大于重复II。
2.2 RIL群体各根系性状间的相关性分析
将RIL群体各根系性状进行相关性分析发现(表5), 在重复I中, 最长根长与总根系长、根表面积、根体积均呈显著正相关, 相关系数为0.359~0.399, 与根直径没有显著相关性。总根系长与根表面积和根体积的相关系数为0.946和0.859, 远大于与最长根长和根直径的0.359和0.408。同样, 根表面积与根体积间具有显著相关性。总的来说, 这5个性状, 最长根长与根直径没有显著的相关性, 其他性状之间都存在显著正相关。而在重复II中, 最长根长与其他4个性状间均无明显相关性, 其他性状相互之间都存在显著正相关。
NPB: 日本晴; 标尺为3 cm。NPB: Nipponbare; Bar: 3 cm.
2.3 RIL群体根系性状的QTL分析
利用一些SSR和STS标记引物进行QTL定位(表6), 然后对9311和NPB构建的RIL群体的最长根长、总根系长、根表面积、根体积和根直径这5个性状进行QTL分析(图3和表7)。重复I共检测到15个QTL, 分布在水稻第1、2、4、7、10和11染色体上。控制最长根长的QTL有3个, 分布在第1、4和10号染色体, 变异贡献率幅度为4.8%~10.3%。其中, 贡献率超过5%的位点有2个:和, 贡献率分别为10.3%和6.3%, 前者效应来自NPB, 后者效应来自9311。我们检测到控制总根系长的QTL 4个, 分布在水稻第1、2、7和11号染色体上, LOD值为2.92~8.47。这些QTL的表型变异贡献率均超过5%, 幅度为7.8%~42.5%。的效应来自NPB, 其余3个QTL的效应来自9311。控制根表面积的QTL有3个, 分布于水稻第2、7和11号染色体, 3个QTL的表型变异贡献率均超过5%。控制根体积的QTL有4个, 分布于水稻第2、7、10和11号染色体, 贡献率(除外)都超过5%。控制根直径的QTL有2个, 分布在水稻第1和4号染色体, 这2个QTL的贡献率均超过5%。的效应来自9311,的效应来自NPB。
NPB: 日本晴; 倒三角指示亲本的平均值; LRL: 最长根长; TRL: 总根系长; RSV: 根表面积; RV: 根体积; RD: 根直径。
NPB: Nipponbare; the inverted triangle indicates the average value of the parent. LRL: the longest root length; TRL: total root length; RSV: root surface area; RV: root volume; RD: root diameter.
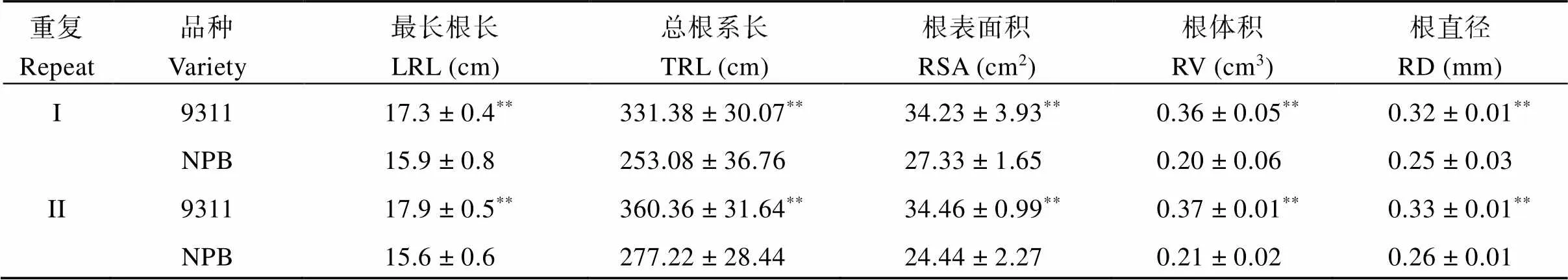
表3 9311和NPB的根系性状
NPB: 日本晴; 值表示均值± SD;**表示达0.01显著水平差异。LRL: 最长根长; TRL: 总根系长; RSV: 根表面积; RV: 根体积; RD: 根直径。
NPB: Nipponbare; values represent means ± SD;**indicates significant difference at the 0.01 probability level. LRL: the longest root length; TRL: total root length; RSV: root surface area; RV: root volume; RD: root diameter.

表4 RIL群体的根系性状
LRL: 最长根长; TRL: 总根系长; RSV: 根表面积; RV: 根体积; RD: 根直径。
LRL: the longest root length; TRL: total root length; RSV: root surface area; RV: root volume; RD: root diameter.
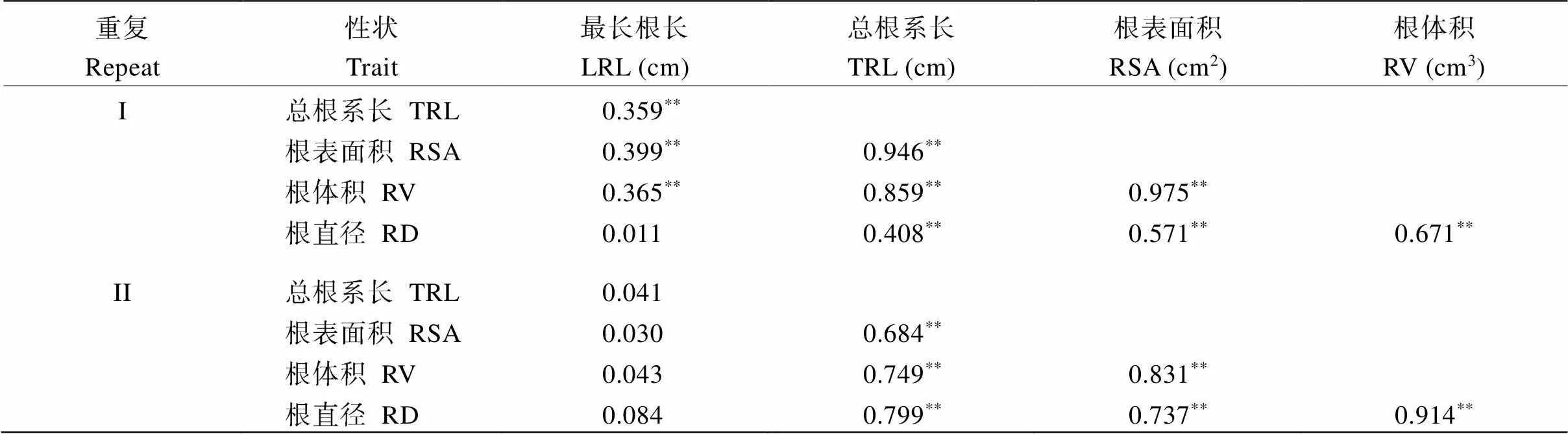
表5 RIL群体根系性状间的相关系数
**表示0.01显著水平相关。LRL: 最长根长; TRL: 总根系长; RSV: 根表面积; RV: 根体积; RD: 根直径。
**indicates significant difference at the 0.01 probability level. LRL: the longest root length; TRL: total root length; RSV: root surface area; RV: root volume; RD: root diameter.
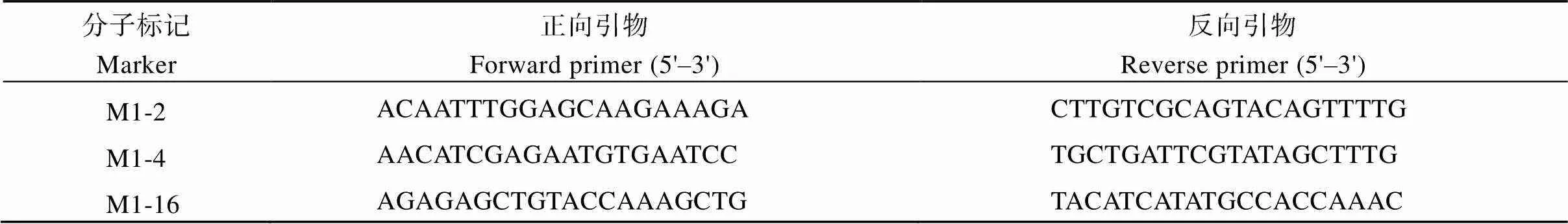
表6 QTL定位的SSR和STS标记引物
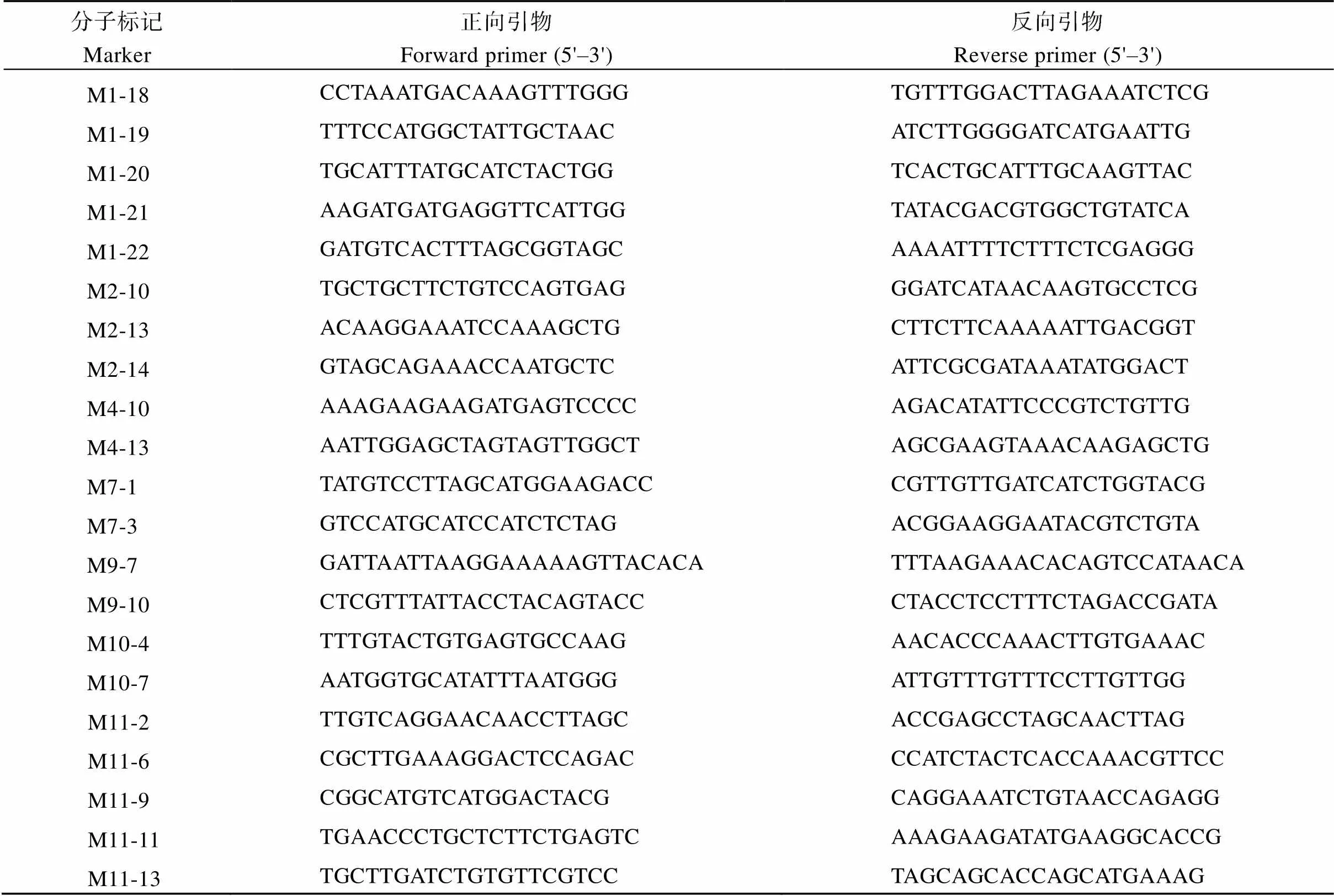
(续表6)

表7 RIL群体的根系性状QTL
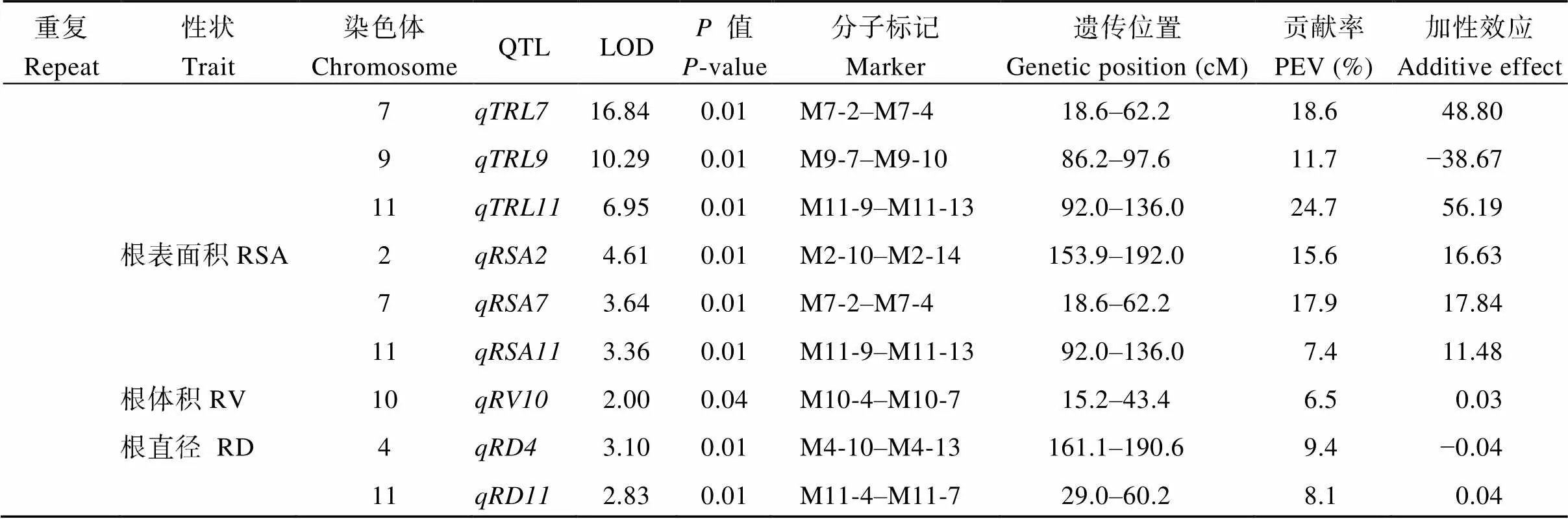
(续表7)
LRL: 最长根长; TRL: 总根系长; RSV: 根表面积; RV: 根体积; RD: 根直径。
LRL: the longest root length; TRL: total root length; RSV: root surface area; RV: root volume; RD: root diameter.
每条染色体的左边列出为分子标记。
Molecular markers for QTLs are listed on the left along each chromosome.
重复II共检测到11个QTL, 分布在第1、2、4、7、9、10和11号染色体上。我们只在4号染色体上发现了一个控制最长根长的QTL, 贡献率为8.3%, 加性效应来自于9311。控制总根长的QTL有4个, 分布在水稻第1、7、9和11号染色体上, LOD值为6.95~16.84。这些QTL的表型变异贡献率均超过5%, 幅度为11.7%~24.7%。和的效应来自NPB, 其余2个QTL的效应来自9311。控制根表面积的QTL有3个, 分布于水稻第2、7和11号染色体, 3个QTL的表型变异贡献率均超过5%。控制根体积的QTL有1个, 在10号染色体上。控制根直径的QTL有2个, 分布在水稻第4和11号染色体, 这2个QTL的贡献率均超过5%。其中,的效应来自NPB,的效应来自9311。
对2个重复进行分析发现, 有5个控制相同性状的QTL在同一位置被重复检测到。包括4号染色体上的和, 7号染色体上的, 10号染色体上的和11号染色体上的。、、和这4个QTL的效应在2个重复中均来自9311, 说明RIL的最长根长、根表面积和根体积的正效应来自9311等位型。的效应在2个重复中来自NPB, 说明RIL根直径受NPB等位型的正调控。综合2个重复鉴定的QTL来看, 在7条染色体上共分布有4个QTL簇, 分别位于水稻第2、4、7和10号染色体, 只有4号染色体上检测到的最长根长和根直径相关QTL, 在2次重复中定位的染色体相同位置。因此, 我们对QTL开展精细定位。
2.4 最长根长QTL qLRL4的精细定位和候选基因确定
位于水稻第4号染色体的QTL是控制最长根长的QTL。9311背景下InDel标记IND4-1和IND4-4间约158 kb来自NPB的近等系NIL与9311相比, 最长根长缩短, 根直径略微变大, 总吸收面积和活跃吸收面积减少, 其他性状基本无变化(图4和表8)。利用近等系NIL-和9311构建的大规模F2群体, 结合个体的InDel标记基因型和根长表型, 我们进一步精细定位这一最长根长QTL, 最终将其定位于标记IND4-1和IND4-3之间约68.23 kb的物理距离内(图5和表1)。依据水稻基因组注释数据库(http://rice.plantbiology.msu.edu/ cgi-bin/gbrowse/rice/), 该区间包含了8个候选基因, 其中有7个基因存在引起氨基酸改变的单核苷酸多态(SNP)或InDel (表9)。qRT-PCR检测8个候选基因相对表达量(图6), 在NPB和NIL-中, 基因、和的相对表达量较9311极显著上升; 而基因的相对表达量极显著下降。其他4个基因除的相对表达量在NPB中极显著上升外,、和的相对表达量在NPB 或NIL-中与9311间无显著差异。
标尺为3 cm。Bar: 3 cm.
3 讨论
在QTL分析中, 同一染色体区间存在多个QTL控制多个根系性状, 这可能是一因多效的作用。姜树坤等[23]以笹锦和北陆129杂交衍生的重组自交系群体为材料, 对水稻移栽后的新生根相关性状进行研究, 共检测到10个QTL, 分别控制根平均直径、总根长、平均根表面积、平均根长和根数等性状。并且对第11号染色体的分子标记C477和G320B之间的新主效QTL——同时控制根长、根表面积和根数的QTL、和进行了验证。这些QTL簇体现了QTL的一因多效作用。由于一因多效QTL/基因的存在, 在开展水稻分子育种时, 我们可以考虑选择单个QTL/基因同时改良多个性状。徐晓明等[24]以超级稻协优9308衍生的重组自交系与轮回亲本中恢9308回交多代群体为材料, 采用琼脂无土栽培技术开展了水稻根长主效QTL的精细定位, 最终将其定位在水稻第4号染色体分子标记RM5687与InDel149之间约624.6 kb的范围内。我们定位的是控制最长根长的新QTL, 其位置与报道的在4号染色体上的位置并不相同, 这可能是所用亲本和群体不同所致。幼苗期根的生长为作物获取水和营养物质提供了保障[25]。近等系NIL-在发芽后根的生长受到抑制, 根的总吸收面积和活跃吸收面积相对9311也显著减小(图4和表8)。Raffaele等[26-27]在拟南芥中研究发现, 具有较大根分生组织的突变体往往根较长, 而根分生组织的大小受生长素和细胞分裂素的共同调节。水稻中已克隆了一些与根发育相关的基因, 例如基因调控不定根的数目[15],基因调控不定根的数目与长短[18],基因调控侧根和不定根的数量[28],通过根系与乙烯的特异性反应来影响根的生长发育[29]。调控水稻根长的基因也有报道,基因影响水稻主根长短[30];基因通过外源性蔗糖、葡萄糖和果糖的调控影响水稻根系的长短[13];基因通过影响根细胞的长短与数目影响水稻根系的长短[14];基因影响水稻苗期根长与直径的作用可能与ABA的感知或信号传导有关[20]。我们的研究最终将最长根长QTL精细定位到约68.23 kb的区间内, 依据基因注释发现该区间包含8个候选基因, 其中有7个基因存在引起氨基酸改变的SNP或InDel, 包括编码水解酶、受体蛋白激酶和染色质结构调节因子的基因。根部转录水平的表达分析显示, 4个候选基因的相对表达量在NIL-或NPB与9311之间存在显著差异。因此, 这4个基因作为候选基因的可能性更大。当然, 候选基因的最终确定还有待进一步基因克隆和功能验证。
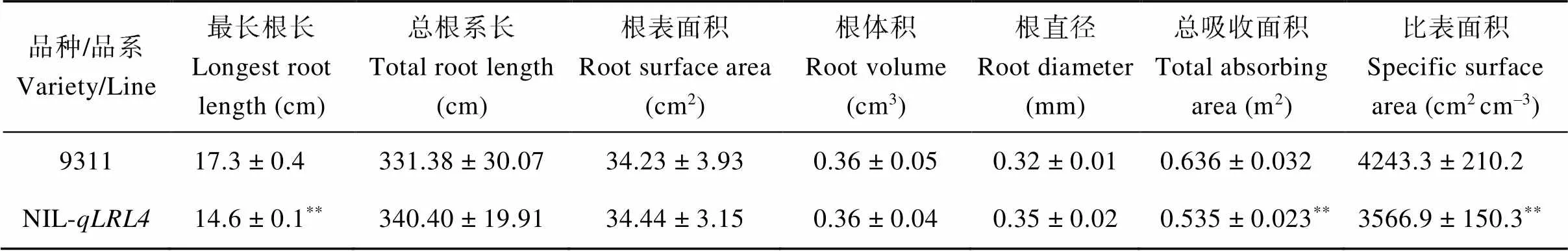
表8 9311和NIL-qLRL4的根系性状和活性
**表示达0.01显著水平差异。
**indicates significant difference at the 0.01 probability level.
被缩小到标记IND4-1和IND4-3之间的68.23 kb区间内。图例的黑色条块代表9311基因型, 白色条块代表NPB基因型。NPB: 日本晴。最长根长表示为均值±标准差。*和**分别表示0.05和0.01显著水平差异。LRL: 最长根长。
was narrowed down to a 68.23 kb interval defined by markers IND4-2 and IND4-3. Black box represents 9311 genotype, and white box represents NPB genotype. NPB: Nipponbare. Values represent means ± SD.*and**indicate significant difference at the 0.05 and 0.01 probability levels compared with NIL-, respectively. LRL: the longest root length.
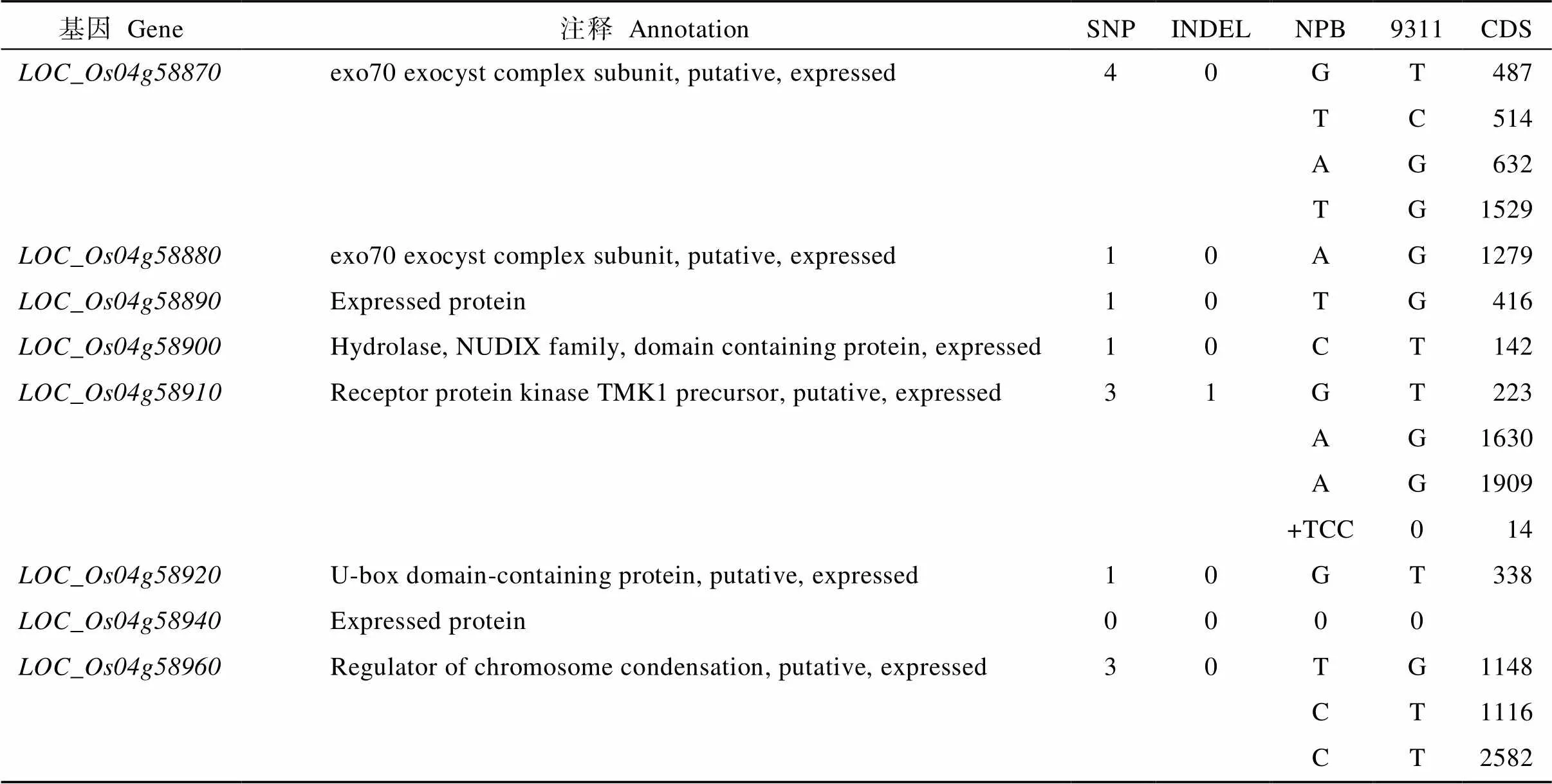
表9 qLRL4精细定位区间的候选基因和候选基因中引起氨基酸改变的序列变化
SNP和InDel为引起氨基酸改变的SNP数目和InDel数目。字母为改变的碱基, 数字为CDS上的位点。
SNP and InDel represent the number of SNPs and InDels that cause amino acid changes.The letters are changed bases and the numbers are positions on the CDS.
相对表达量数据为平均数±标准差,= 3。**表示与9311比较达到0.01的差异显著水平。
Data represent means ± SD of three biological replicates.**indicates significant difference at the 0.01 probability level compared with 9311.
4 结论
利用籼稻9311和粳稻日本晴为亲本的RIL群体对水稻苗期根系相关性状开展QTL分析, 共检测到26个控制最长根长、总根系长、根表面积、根体积和根直径的QTL, 发现水稻第2、4、7和10号染色体上的4个QTL簇, 包括第4号染色体上控制最长根长的QTL。精细定位将定位于InDel标记IND4-1和IND4-3之间约68.23 kb的区间, 包含了8个候选基因。其中7个基因在亲本间有引起氨基酸改变的SNP或InDel, 4个基因在近等基因系NIL-和9311之间的转录水平相对表达量上存在显著差异。此最长根长QTL的精细定位为水稻根系发育相关基因的克隆奠定了基础。
[1] 范楚玉. 西周农事诗中反映的粮食作物选种及其发展. 自然科学史研究, 1982, 3: 267–272.
Fan C Y. Selection and development of grain crops reflected in the poetry of agriculture in the Western Zhou Dynasty, 1982, 3: 267–272 (in Chinese with English abstract).
[2] 陶荣荣, 蔡晗, 朱庆权,周益雷, 王康平, 余超, 侯丹平, 刘海浪, 张耗. 水稻高产高效的根-冠互作机制研究进展. 中国农学通报, 2018, 34(5): 1–4.
Tao R R, Cai H, Zhu Q Q, Zhou Y L, Wang K P, Yu C, Hou D P, Zhang H. Research progress on root-crown interaction mechanism of high-yield and high-efficiency rice., 2018, 34(5): 1–4 (in Chinese with English abstract).
[3] 丁仕林, 刘朝雷, 钱前. 水稻根系遗传研究进展. 中国稻米, 2019, 25(5): 24–29.
Ding S L, Liu C L, Qian Q. Advances in rice root genetics., 2019, 25(5): 24–29 (in Chinese with English abstract).
[4] 徐吉臣, 李晶昭, 郑先武, 邹亮星, 朱立煌. 苗期水稻根部性状的QTL定位. 遗传学报, 2001, 28: 433–438.
Xu J C, Li J Z, Zheng X W, Zou L X, Zhu L H. QTL mapping of rice root traits at seedling stage., 2001, 28: 433–438 (in Chinese with English abstract).
[5] 滕胜, 曾大力, 钱前, 国广泰史, 藤本宽, 黄大年, 朱立煌. 水稻根系活力的遗传分析. 中国水稻科学, 2002, 16: 119–123.
Teng S, Zeng D L, Qian Q, Kunihiro Y, Fujimoto K, Huang D N, Zhu L H. Genetic analysis of root vigor in rice., 2002, 16: 119–123 (in Chinese with English abstract).
[6] 胡兴明, 郭龙彪, 曾大力, 高振宇, 滕胜, 李浩戈, 朱立煌, 钱前. 水稻苗期发根力的QTL和上位性分析. 中国水稻科学, 2004, 18: 396–400.
Hu X M, Guo L B, Zeng D L, Gao Z Y, Teng S, Li H G, Zhu L H, Qian Q. QTL mapping and epistasis analysis of rice root growth ability at seedling stage., 2004, 18: 396–400 (in Chinese with English abstract).
[7] Mitsuhiro O, Wataru T, Takeshi E. Fine-mapping of, a major QTL for root length of rice seedlings grown under a wide range of NH4+concentrations in hydroponic conditions., 2010, 121: 535–547.
[8] 王汝慈. 两个生育时期水稻耐低磷胁迫相关性状的QTL定位. 中国农业科学院硕士学位论文, 北京, 2009.
Wang R C. QTL Mapping of Low Phosphorus Stress-related Traits in Rice during Two Growth Periods. MS Thesis of Chinese Academy of Agricultural Sciences, Beijing, China, 2015 (in Chinese with English abstract).
[9] Obara M, Fukuta Y, Yanagihara S. Genetic variation and QTLs related to root development in upland new rice for(NERICA) varieties., 2019, 69: 94–103.
[10] Kitomi Y, Nakao E, Sawako K. Fine mapping of quick rooting 1 and 2, quantitative trait loci increasing root length in rice., 2018, 8: 727–735.
[11] 章怡兰, 林雪, 吴仪, 李梦佳, 张晟婕, 路梅, 饶玉春, 王跃星. 水稻根系遗传育种研究进展. 植物学报, 2020, 55: 382–393.
Zhang Y L, Lin X, Wu Y, Li M J, Zhang S J, Lu M, Rao Y C, Wang Y X. Research progress on rice root genetics and breeding., 2020, 55: 382–393 (in Chinese with English abstract).
[12] 梁永书, 周军杰, 南文斌, 段东东, 张汉马. 水稻根系研究进展. 植物学报, 2016, 51: 98–106.
Liang Y S, Zhou J J, Nan W B, Duan D D, Zhang H M. Research progress of rice root system., 2016, 51: 98–106 (in Chinese with English abstract).
[13] Yao S G, Mushika J, Taketa S, Ichii M. The short root mutationdefines a sugar-mediated root growth in rice (L.)., 2004, 167: 49–54.
[14] Jia L, Zhang B, Mao C. OsCYT-INV1 for alkaline/neutral invertase is involved in root cell development and reproductivity in rice (L.)., 2008, 228: 51–59.
[15] Inukai Y, Sakamoto T, Ueguchitanka M. Crown rootless1, which is essential for crown root formation in rice, is a target of an auxin response factor in auxin signaling., 2005, 17: 1387–1396.
[16] Liu H, Wang S, Yu X, Yu J, He X, Zhang S, Shou H, Wu P. ARL1, a LOB-domain protein required for adventitious root formation in rice., 2005, 43: 47–56.
[17] Zhao Y, Hu Y F, Dai M G, Huang L M, Zhou D Y. The WUSCHEL-related homeobox geneis required to activate shoot-borne crown root development in rice., 2009, 21: 736–748.
[18] Yang S Q, Li W Q, Miao H, Gan P F, Qiao L, Chang Y L, Shi C H, Chen K M., a gene encoding an unknown function protein which contains DUF630 and DUF632 domains controls leaf rolling in rice., 2016, 9: 1–14.
[19] Dai X Y, Wang Y Y, Zhang W H. OsWRKY74, a WRKY transcription factor, modulates tolerance to phosphate starvation in rice., 2016, 67: 947–960.
[20] Ao S G, Shin T, Masahiko I. Isolation and characterization of an abscisic acid-insensitive mutation that affects specifically primary root elongation in rice (L.)., 2003, 164: 971–978.
[21] Jing H W, Yang X L, Zhang J, Liu X H, Zheng H K, Dong G J, Nian J Q, Feng J, Xia B, Qian Q, Li J Y, Zuo J R. Peptidyl-prolyl isomerization targets rice Aux/IAAs for proteasomal degradation during auxin signaling., 2015, 6: 7395.
[22] McCouch S, Cho Y, Yano M, Paul E, Blinstrub M, Morishima H, Kinoshita T. Report on QTL nomenclature., 1997, 14: 11–131.
[23] 姜树坤, 张凤鸣, 白良明, 孙世臣, 王彤彤, 丁国华, 姜辉, 张喜娟. 水稻移栽后新生根系相关性状的QTL分析. 中国水稻科学, 2014, 6: 598–604.
Jiang S K, Zhang F M, Bai L M, Sun S C, Wang T T, Ding G H, Jiang H, Zhang X J. QTL analysis of the related traits of new roots after transplanting rice., 2014, 6: 598–604 (in Chinese with English abstract).
[24] 徐晓明, 张迎信, 王会民, 任翠, 王汝慈, 沈希宏, 占小登, 吴玮勋, 程式华, 曹立勇. 一个水稻根长QTL的分离鉴定. 中国水稻科学, 2016, 30: 363–370.
Xu X M, Zhang Y X, Wang H M, Ren C, Wang R C, Shen X H, Zhan X D, Wu W X, Cheng S H, Cao L Y. Isolation and identification of a QTLof rice root length., 2016, 30: 363–370 (in Chinese with English abstract).
[25] Rogers E D, Benfey P N. Regulation of plant root system architecture: implications for crop advancement., 2015, 32: 93–98.
[26] Raffaele D I, Francisco S L, Emanuele S. Cytokinins determineroot-meristem size by controlling cell differentiation., 2007, 17: 678–682.
[27] Raffaele D I, Kinu N, Laila M. A genetic framework for the control of cell division and differentiation in the root meristem., 2008, 322: 1380–1384.
[28] Liu W, Xu Z H, Luo D. Roles of OsCKI1, a rice casein kinase I, in root development and plant hormone sensitivity., 2003, 36: 189–202.
[29] Chen H, Ma B, Zhou Y, He S J, Tang S Y, Lu X, Xie Q, Chen S Y, Zhang J S. E3 ubiquitin ligase SOR1 regulates ethylene response in rice root by modulating stability of Aux/IAA protein., 2018, 115: 4513–4518.
[30] Zhang H G, Zhang L J, Si H, Ge Y S, Liang G H, Gu M H, Tang S Z. Rf5 is able to partially restore fertility to Honglian-type cytoplasmic male sterilerice () lines., 2016, 36: 1–10.
Genetic analysis of seedling root traits and fine mapping of the QTLfor the longest root length in rice
TIAN Biao, DING Shi-Lin, LIU Chao-Lei, RUAN Ban-Pu, JIANG Hong-Zhen, GUO Rui, DONG Guo-Jun, HU Guang-Lian, GUO Long-Biao, QIAN Qian, and GAO Zhen-Yu*
China National Rice Research Institute, Hangzhou 310006, Zhejiang, China
In order to analyze the genetic basis of root traits at seedling stage, we performed QTL analysis of root morphology with 148 recombinant inbred lines derived fromvariety 9311 andvariety Nipponbare (NPB). In two repetitions, a total of 26 QTLs were detected for the longest root length, total root length, root surface area, root volume, and root diameter, distributed on chromosomes 1, 2, 4, 7, 9, 10, and 11 in rice. Four QTL clusters on chromosomes 2, 4, 7, and 10 were found, including a major QTLcontrolling the longest root length. To fine mapping of the major QTL, we constructed a near isogenic line NIL-with a segment from NPB between markers IND4-1 and IND4-4 with 9311 background. With a F2population derived from the NIL-and 9311, we fine mapped thewithin ~68.23 kb region between markers HIND4-1 and IND4-3, where eight candidate genes located. Fine mapping of this QTL for root length will help explore genetic mechanism of root elongation and morphogenesis in rice.
rice; root trait; the longest root length;; fine mapping
10.3724/SP.J.1006.2021.02088
本研究由国家自然科学基金项目(32061143039, 31671761)资助。
This study was supported by the National Natural Science Foundation of China (32061143039, 31671761).
高振宇, E-mail: gaozhenyu@caas.cn, Tel: 0571-63370211
E-mail: 82101186057@caas.cn, Tel: 0571-63370483
2020-12-13;
2021-03-19;
2021-04-13.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210413.1521.006.html
